深度学习在脑胶质瘤应用中的研究进展
2021-03-05伍文丽李杰文徐胜生
伍文丽,李杰文,徐胜生*
(1.重庆医科大学附属第一医院放射科,重庆 400016;2.重庆大学光电技术与系统教育部重点实验室,重庆 400044)
0 引言
脑胶质瘤起源于神经胶质细胞,是最常见的原发性中枢神经系统肿瘤,约占成人原发性恶性脑肿瘤的75%。2016 年世界卫生组织(WHO)将脑胶质瘤分为低级别胶质瘤(lowgrade glioma, LGG,包 括WHO Ⅰ- Ⅱ级) 及 高 级 别 胶 质瘤(high-grade glioma, HGG,包 括WHO Ⅲ- Ⅳ级)[1]。MRI是一种非侵入性的医学成像技术,因其具有良好软组织对比度,能为脑胶质瘤的诊断和治疗提供丰富的信息,是脑胶质瘤的评估的首选成像技术[2]。然而,随着医学影像技术及成像设备的发展,逐渐增多的影像图像给放射科医生的工作带来了巨大的挑战。且随着精准医疗的提出,传统的人工阅片等主观的医学图像分析方法已无法满足需求。深度学习(deep learning, DL)是机器学习的一个重要分支,可从大量的原始数据中自动提取图像中抽象、复杂的深度特征,而非手工制作的浅层特征,具有快速高效,准确率高的优势[3],使其迅速成为医学图像分析领域研究的热点。近年来,DL 广泛应用于脑胶质瘤的MRI 图像分析处理中,其在肿瘤分割、分类、基因预测及生存期预测等方面发挥着重要作用。本文就DL 在脑胶质瘤中的应用中的进展予以综述。
1 深度学习概述
DL 是传统的人工神经网络技术的扩展,是通过多层非线性网络结构从原始数据中自动提取并组合低级特征,再转换成更高级、更抽象特征,以完成复杂任务学习的一种深度网络[3,4]。DL 方法包含多种深度模型,其中卷积神经网络(convolutional neural network, CNN)模型是应用最广泛的深度学习模型,在识别、处理和分析图像特征过程与人类神经系统处理视觉信息过程类似[3,4]。CNN 一般由输入层、卷积层、池化层、全连接层及输出层构成,如图1 所示。各层之间相互联系,共同构成复杂而精密的网络。卷积层自动提取输入信息的特征,且通过权值共享减低模型复杂度;池化层紧跟在卷积层之后,其通过降低特征面的分辨率获得具有空间不变性的特征,起到二次提取特征的作用;全连接层中每个神经元与前一层所有神经元进行全连接,通过整合前期所提取的各种局部特征,达到训练分类器及学习目标全局特征的目的。因此CNN 具有局部连接、权值共享、池化操作及多层结构等特点,使其减少训练参数的数量,有效降低了网络计算复杂度,同时使得网络对输入的局部变换具有一定不变性,如平移不变形,从而提升了网络泛化能力[3]。
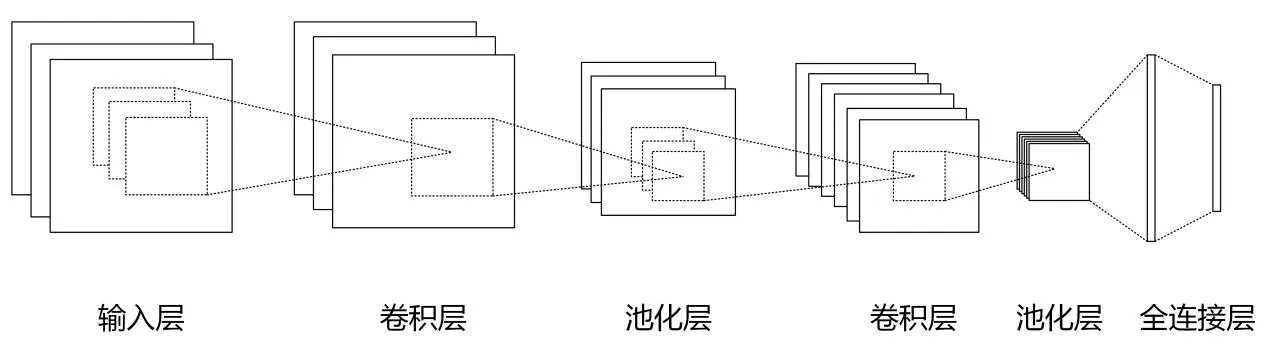
图1 一个基本的卷积神经网络结构
2 DL 在脑胶质瘤分割中的应用
脑胶质瘤的准确分割对疾病诊断、预后和患者治疗计划具有重要作用[5]。目前,基于DL 的分割算法已实现了从多模态MRI 数据中将脑胶质瘤自动分割为坏死区、水肿区、肿瘤非增强区及肿瘤增强区等不同亚区。Pereira 等[6]提出一种基于3×3 卷积核的二维卷积神经网络(2D CNN)自动分割算法,该方法使用的小卷积核可通过减少参数量而防止过拟合的发生。霍智勇等[7]提出了一种基于双通道三维密集连接网络的脑胶质瘤MRI 数据自动分割算法,该算法利用三维密集连接网络加强特征传播,减少了低级特征向高级特征传递过程中的损失,然后采用双通道特征提取网络,选取不同尺度卷积核,获取了多尺度特征,解决了肿瘤区域大小不一致的问题。Cui 等[8]结合两个子网络构建了一种级联深度卷积神经网络,结果表明该方法分割肿瘤整体、肿瘤核心及肿瘤增强区的骰子相似系数(dice similarity coefficient, DSC)分别为0.89,0.77 及0.80,且平均在1.54 秒内快速完成了分割任务。为进一步提高脑胶质瘤分割精度,Prasanna 等[5]采用基于影像组学的CNN 分割技术,即先识别出分割肿瘤不同亚区的影像组学纹理特征,然后将这些特征输入到三维卷积神经网络(3D CNN)模型中,用于脑胶质瘤不同亚区的分割,结果表明相比3D CNN 分割模型,提高了肿瘤增强区和肿瘤整体的分割精度。Mlynarski 等[9]采用2D-3D CNN 分割方法对脑胶质瘤多模态MRI 数据进行分割,该方法结合了2D 和3D 卷积神经网络的优点,捕获了空间上下文特征,结果显示分割肿瘤整体、肿瘤核心及肿瘤增强区的DSC 分别为0.918,0.883 及0.854,优于以上分割算法的精度。
此外,AlBadawy 等[10]研究了CNN 对来自两个机构的胶质母细胞瘤MRI 数据的分割性能,结果显示使用不同机构的数据的分割精度(DSC 为0.68±0.19)显著低于使用同一机构数据的分割精度(DSC 为0.72±0.17)。由此可见,来自不同的机构的数据在成像设备、图像采集参数和对比剂使用方面有所不同,这可能会影响图像质量,从而影响分割精度。
3 DL 在脑胶质瘤分类中的应用
脑肿瘤的病理类型及脑胶质瘤分级很大程度上决定了治疗方案及整体预后[11,12],因此准确进行脑肿瘤分类及分级具有重要临床意义。近年来,一些研究者基于CNN 方法充分挖掘对比增强T1WI (contrast enhanced T1-weighted imaging, CE-T1WI)数据集的深度特征实现了脑胶质瘤、脑膜瘤及垂体瘤分类诊断,如Abiwinanda 等[13]和Paul 等[14]的分类准确率分别为84.19%,90.26%。随后Swati 等[15]使用预训练卷积神经网络模型,提出了一种基于迁移学习的分块微调策略进行分类,准确率高达94.82%。Deepak 等[11]使用迁移学习训练深度卷积神经网络模型,分类准确率为92.3%,当进一步联合CNN 及支持向量机(support vector machine, SVM),准确率提高到97.8%。
除了脑肿瘤定性诊断,DL 还实现了脑胶质瘤的分级诊断。Yang 等[16]使 用GoogLeNet 和AlexNet 模 型 进 行LGG和HGG 分类,两种模型的分类准确率分别为94.5%、93.8%,表明GoogLeNet 具有更佳分类性能。Zhang 等[17]基于CNN模型,从扩散张量成像(diffusion tensor imaging, DTI)数据集中提取深度特征,用于脑胶质瘤分级,结果显示区分LGG 与HGG 准确率为94%;区分WHO Ⅲ级与WHO Ⅳ级脑胶质瘤准确率高达98%,表明在脑DTI 图像上提取的深度特征有助于区分不同级别的脑胶质瘤。为广泛挖掘不同级别胶质瘤间的差异性,Ge 等[18]将多模态MRI 纳入多流2D CNN 模型中,实现了LGG 和HGG 的分类,其中CE-T1WI 具有最高效能,其准确率为83.87%,融合多模态特征分类性能提高了约7%。Mzoughi 等[12]提出一种多尺度3D CNN 架构,基于多模态MRI 数据集进行LGG 及HGG 的分类,并采用数据增强技术对图像进行预处理,其分类准确率从82.5%提高到96.4%,优于2D CNN 模型。上述研究结果均表明DL 有助于脑胶质瘤鉴别及分级,然而目前主要进行的是单中心研究,存在样本量较小的问题,有待进一步进行多中心,大样本的研究以验证其可行性。
4 DL 在脑胶质瘤基因预测中的应用
了解脑胶质瘤异柠檬酸脱氢酶(isocitrate dehydrogenase, IDH),O6-甲基鸟嘌呤-DNA 甲基转移(O6-methylguanine-DNA methyltrans-ferase,MGMT)和1p19q 等肿瘤基因状态有益于治疗方案的制定和预后的评估[19]。近年来,越来越多的研究探讨了DL 预测脑胶质瘤基因状态的价值。Li 等[20]通过增加网络深度和全连接层中的神经元数量构建CNN 模型,用于预测LGG 患者IDH1 突变状态,其工作曲线下面积(the area under the operating characteristic curve, AUC) 为0.92。Korfiatis 等[21]采用三种残差深度神经网络(residual deep neural network, ResNet)模型用于预测MGMT 启动子甲基化状态,结果显示ResNet50 模型优于ResNet34 和ResNet18 模型,其准确率分别为94.90%,80.72%和75.75%。Akkus 等[22]等基于多尺度CNN 模型实现了LGG 患者1p19q 共缺失状态的预测,准确率为87.70%。此外,Chang 等[23]训练的CNN 模型实现了脑胶质瘤的多基因预测,包括IDH1 突变、MGMT 甲基化和1p/19q 共缺失状态,准确率分别为94%,83%及92%,提示利用CNN 可以同时预测多个基因的状态。
为了证明基于不同MRI 模态的预测性能,有研究利用多模态MRI 数据集构建CNN 模型用于预测脑胶质瘤IDH 状态,如Chang 等[24]基于残差卷积神经网络构建预测模型,结果显示FALIR、T2WI、T1WI 及CE-T1WI 数据集的AUC 值分别为0.69,0.73,0.86 及0.92,融合多模态MRI 后AUC 为0.94。另一项研究[25]构建的3D-DenseNet 模型中T2WI 预测能力最好,AUC 为0.82,而基于CE-T1WI 的AUC 为0.75,融合多模态MRI 的AUC 达0.86。这些数据表明融合多模态MRI 数据集可以提供预测脑胶质瘤基因状态的补充信息,其预测性能显著优于单模态模型。然而,Yogananda 等[26]采用3D-Dense-UNets 方法分别构建基于T2WI 及多模态MRI 数据集预测脑胶质瘤IDH 状态模型,准确率分别为97.14%±0.04、97.12%±0.09,AUC 分别为0.98±0.01、0.99±0.01,这表明仅使用T2WI 模态的信息就可以提供较高的预测性能。可见,不同的研究基于不同MRI 模态预测基因状态,其结果并不一致,仍需在更大样本中进一步研究。
5 DL 在脑胶质瘤生存期预测中的应用
既往研究将患者年龄、性别、肿瘤部位、大小等作为预后因素[27],然而这些指标无法反映肿瘤内异质性,存在一定局限。DL 能挖掘肉眼无法识别整个肿瘤的的深层、抽象的特征,更有助于预测患者生存期。Liu 等[28]采用预训练的卷积神经网络对图像深度特征进行筛选,再将筛选后的特征纳入随机森林算法分类模型,其预测生存期准确率达到95.45%。Nie的团队[29]采用深度学习,从多模态、多通道的MRI 数据集中自动提取深度特征,用于预测HGG 患者的总生存期。随后将这些深度特征以及临床特征(如年龄、肿瘤大小和组织学类型)输入SVM 以预测总生存期,其准确率高达90.66%,并进行了一系列比较分析,结果表明融合多模态模型较单一模态模型具有更高性能,在单模态分类性能中静息状态功能磁共振成像具有最佳性能,且使用3D CNN 构建的预测模型较2D CNN 具有更高性能,准确率提高了约10%;另外他们将CNN全连接层中提取的深度特征输入SVM,从而构建CNN+SVM的联合模型,发现其性能较单独的CNN 模型提高了2.5%,且单独的CNN 模型的性能优于SVM 模型。
尽管上述研究均表明DL 方法预测脑胶质瘤生存期方面具有重要价值,但大多数研究并未考虑使用与预后相关的肿瘤基因型,如IDH、MGMT 及1p19q 等。Tang 等[30]提出了一种多任务卷积神经网络模型共同完成肿瘤基因型(IDH,MGMT,1p19q)和总生存期预测任务,具体来说从基因型预测任务中获得的深度特征,用于指导总生存期的预测,反之从总生存期预测任务中学习到的特征也用于肿瘤基因型的预测。结果显示与肿瘤基因型相关特征显著提高了预测总生存期的准确性,反之与总生存期相关的特征也改善了预测基因型的准确性,表明深度特征、基因型和患者生存期之间具有密切关系。
6 总结与展望
目前,基于DL 已采用多模态、多方法对脑胶质瘤MRI 图像进行分割、分类、基因预测及生存期预测的横向及纵向研究,并获得初步进展。相信随着人工智能蓬勃发展的趋势,借助医学影像大数据的发展,不断改进的深度学习方法将有助于更精确地了解脑胶质瘤的特征,在缓解放射科医生的压力同时提高了诊断的速率及准确率,无疑能为精准医疗的实现提供更多的解决方案。
