16S与gyrB基因联合建树快速鉴定海水中的一株地衣芽胞杆菌
2020-05-13林俊杰甘美裕卢源钦肖玉娟
傅 奇, 林俊杰, 甘美裕, 卢源钦, 肖玉娟
16S与基因联合建树快速鉴定海水中的一株地衣芽胞杆菌
傅 奇, 林俊杰, 甘美裕, 卢源钦, 肖玉娟
(厦门华厦学院 环境与公共健康学院, 福建 厦门 361024)
在保守序列高度相似的细菌鉴定中, 单独使用16S rDNA/RNA序列进行比对和构建进化树通常无法准确鉴定到种, 需要增加测序基因数并对多基因进行分析。为实现快速鉴定, 课题组对16S与基因联合建树的方法进行了研究, 将海洋来源的一株杆菌, 分别用通用引物扩增16S和基因并测序, 在GeneBank进行序列比对后, 选择各菌种保藏中心16S和基因均相似的菌株, 取16S和基因序列, 采用Paup* 4.0构建进化树。使用16S与拼接序列构建的进化树中属于同一种的菌株均很好的聚合在一枝, 种间分枝自展值均高于98, 分类结构准确, 筛选得到的杆菌与地衣芽胞杆菌()聚合在一枝, 自展值为100, 鉴定为地衣芽胞杆菌。经生理生化试验验证, 该菌株与地衣芽胞杆菌特征完全一致, 使用16S和基因联合建树得到的鉴定结果准确且快速简便。
16S;; 进化树; 地衣芽胞杆菌 ()
芽胞杆菌()是一个重要的微生物类群, 包括枯草芽胞杆菌()、地衣芽胞杆菌()、凝结芽胞杆菌()、炭疽芽胞杆菌()、蜡样芽胞杆菌()等与发酵工业和生活密切相关的多个菌种[1]。其中地衣芽胞杆菌由于生物安全性好、产生多种功能性代谢产物, 在药品[2]、功能食品[3]、饲料[4]和发酵工业[5-7]得到广泛应用, 其中海洋来源的地衣芽胞杆菌在海水污染处理、新型生物活性物质开发方面具有重要潜力[8, 9]。但在地衣芽胞杆菌的筛选过程中, 由于芽胞杆菌类群保守序列的高度相似性, 无法单独依靠常用的16S rDNA/RNA测序来准确定位到种, 通常需要结合生理生化鉴定[10, 11]或者其他序列, 如5'端16S-23S ITS核苷酸序列[12]、基因[13]、A[14]甚至全基因序列[15, 16]进行分析。基因编码DNA促旋酶的B亚单位, 在细菌中广泛存在, 序列相对保守, 但进化速度快于16S, 目前在细菌鉴定中的使用逐渐增加[17], 积累了较为丰富的比对数据库, 但单独使用时部分芽胞杆菌也难以准确定位, 而16S与序列分别比对建树的方法容易出现进化树结构冲突, 干扰定位的情况。利用16S与基因联合建树, 以较少的序列数和进化树数实现对筛选菌株快速鉴定的方法, 可提高数据的利用率, 减少数据冲突, 为芽胞杆菌种群的分子生物学鉴定方法开发提供参考。
1 材料与方法
1.1 样本
分离菌株样本为海泥, 采集于厦门市集美区红树林区。
1.2 材料与试剂
2216E(液体)、2216E琼脂、营养琼脂、V-P试剂盒、卵黄琼脂培养基、色氨酸肉汤、克氏柠檬酸盐培养基、苯丙氨酸脱羧酶培养基、明胶生化管、硝酸盐还原试剂盒、Kovacs氏靛基质试剂盒等培养基与鉴定试剂均由青岛海博生物科技有限公司生产; 细菌基因组DNA提取试剂盒、胶回收试剂盒、PCR试剂由生工生物工程(上海)股份有限公司生产; 16S通用引物27F/1492R、扩增引物UP-1/UP-2r、测序引物UP-1S/UP-2Sr[18]由生工生物工程(上海)股份有限公司合成。
1.3 仪器与设备
PCR仪: 新加坡ESCO公司; 核酸电泳仪: 美国GE公司; 摇床: 上海苏坤公司; 生化培养箱: 上海一恒公司。
1.4 方法
1.4.1 样品处理方法
取海泥25 g加入225 mL 2216E液体培养基均质后稀释涂布2216E琼脂平板, 30℃培养48 h, 取单菌落进行纯化。
1.4.2 16S与序列提取
用细菌基因组DNA抽提试剂盒, 提取菌株的总DNA; 分别用16S与扩增引物扩增, 所得PCR产物电泳纯化后, 送由生工生物工程(上海)股份有限公司测序。
16S扩增条件: 94℃预变性4 min; 94℃变性30 s, 65℃退火40 s, 72℃延伸90 s, 共35个循环; 72℃延伸10 min。
扩增条件[18]: 94℃预变性4 min; 94℃变性1 min, 60℃退火1 min, 72℃延伸2 min, 共35个循环; 72℃延伸10 min。
1.4.3 16S与序列比对与进化分析
将测序结果在Genbank进行比对。为保障数据的准确性, 取相似度较高的1 000个序列中的各菌种保藏中心菌株进行比对, 包括ATCC、NCTC、BCRC、DSM 4个菌种保藏中心菌株。下载相似菌株的16S与序列, 分别用BioEdit[19]和Clustal X[20]进行比对和编辑, 取有效区间, 分别使用16S、和16S与线形拼接序列建树[21], 采用最大简约算法, 用Paup* 4.0构建进化树[22, 23], 选择大肠杆菌ATCC 25922[24]作为外群。
1.4.4 生理生化验证
参考《伯杰细菌鉴定手册》[1]和行业标准《饲料微生物添加剂地衣芽孢杆菌》[25]进行验证。
2 结果与分析
2.1 菌株分离纯化
从海泥中筛选得到一株革兰氏阳性杆菌, 产芽胞, 菌落湿润, 呈白色, 编号为HX2019004。
2.2 16S与gyrB基因测序
测序所得16S与序列拼接后有效区域分别为1 430 bp和1 115 bp。
2.3 序列比对与进化树构建
16S序列比对结果中, 高相似度菌株过多, 且多数菌株无法获得更多的遗传标记序列; 而序列比对结果中, 多数菌株同时可获取16S序列, 因此, 以相似菌株作为建树群, 下载16S与基因序列(表1), 比较分别使用16S、和16S与线形拼接序列建树结果。
16S比对后去除头尾不整齐片段, 保留中心区域1 440 bp(含gap);比对后去除头尾不整齐片段,保留中心区域1 056 bp(含gap)。构建进化树如图1所示, A树基于16S序列构建, B树基于序列构建, C树基于16S与拼接序列构建, 计算次数为1 000, 参数如表2所示。

表1 GenBank中拥有相似16S与gyrB序列的菌株

续表

图1 基于16S与gyrB序列的进化树(A基于16S序列, B基于gyrB序列, C基于16S与gyrB拼接序列)
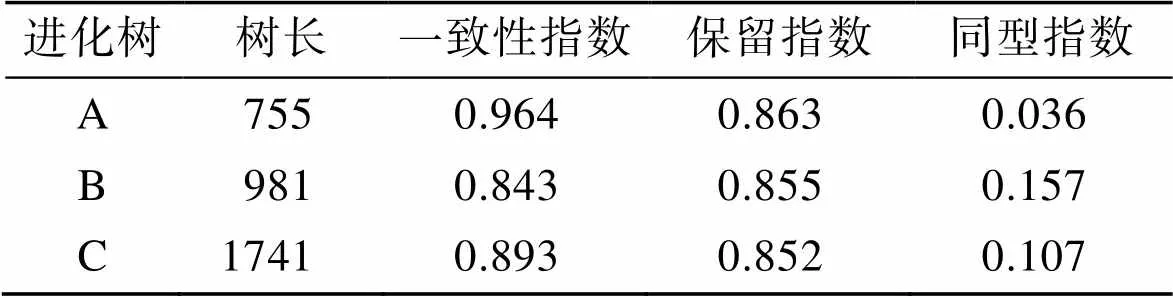
表2 基于16S与gyrB序列的进化树参数表
从图1中可以看出, 仅使用16S序列构建的进化树A分枝自展值低, 无法准确定位HX2019004菌株与、、的进化关系; 仅使用序列构建的进化树B整体分枝自展值较高, 但一致性指数(CI)较低, 且HX2019004菌株与的分类关系存在疑义, 与A树存在较大的结构冲突; 使用16S与拼接序列构建的进化树C中, 同一类菌种均很好的聚合在一枝, 种间分枝自展值均高于98, 表明分类结构准确, HX2019004菌株与地衣芽胞杆菌聚合在一枝, 自展值为100, 表明该菌株为地衣芽胞杆菌可能性高。
2.4 生理生化验证
菌株HX2019004可液化明胶、水解淀粉、还原硝酸盐, 可利用柠檬酸盐做为碳源, 可利用葡萄糖、阿拉伯糖、木糖、甘露醇产酸, 可分解酪素, 可使牛奶胨化, 可在pH 5.5~8.7范围内、0.001%溶菌酶、7%NaCl、厌氧琼脂中生长, V-P反应、接触酶反应阳性; 不能使苯丙氨酸脱氨, 不能使牛奶凝固, 不能分解酪氨酸, 卵黄反应和吲哚反应呈阴性。试验结果与地衣芽胞杆菌生理生化特征吻合[25], 与进化分析结果一致, 命名为HX2019004。
3 结论
利用16S与基因联合建树的方法, 课题组快速将一株从海水中筛选得到的芽胞杆菌HX2019004准确鉴定为地衣芽胞杆菌, 表明该方法在保守序列高度相似菌种间的分类鉴定中具有深入研究的价值。与常用的增加测序基因数的方法相比, 该方法测序量小, 时间短, 数据利用率高; 与多基因分别建树的方法相比, 可避免进化树结构之间的冲突和数据紊乱, 提高了进化树准确性。课题组下一步将继续研究该方法在其他菌群鉴别中的有效性。
[1] Claus D. The genus[M]. New York: Springer, 1986, 2: 1105-1139.
[2] 国家药典委员会. 中华人民共和国药典[M]. 北京: 中国医药科技出版社, 2015: 46-47. Chinese Pharmacopoeia Commission. Pharmacopoeia of the People’s Republic of China[M]. Beijing: China Medical Science and Technology Press, 2015: 46-47.
[3] 方明霞, 齐蕾, 王长泰, 等. 早期肠内营养联合微生态制剂对慢加急性肝功能衰竭病人营养状态和预后的影响[J]. 肠外与肠内营养, 2016, 5: 279-282. Fang Mingxia, Qi Lei, Wang Changtai, et al. Efficacy of early application of enteral nutrition combined with microecologics in treatment of acute-on-chronic hepatitis B liver failure[J]. Parenteral & Enteral Nutrition, 2016, 5: 279-282.
[4] 张红艳, 岳淑宁, 张强, 等. 地衣芽胞杆菌在饲料工业中的应用及研究[J]. 畜牧与饲料科学, 2017, 38(6): 26-28.Zhang Hongyan, Yue Shuning, Zhang Qiang, et al. Application and research ofin feed industry[J]. Animal Husbandry and Feed Science, 2017, 38(6): 26-28.
[5] Saito N. A thermophilic extracellular α-amylase from[J]. Archives of Biochemistry and Biophysics, 1973, 155(2): 290-298.
[6] 王晓丹, 雷安亮, 王婧, 等. 产四甲基吡嗪地衣芽孢杆菌的应用[J]. 中国酿造, 2017, 36(2): 35-38. Wang Xiaodan, Lei Anliang, Wang Jing, et al. Application ofwith tetramethyl pyrazine yield[J]. China Brewing, 2017, 36(2): 35-38.
[7] 乐超银, 邵伟, 刘海军, 等. 一株地衣芽孢杆菌的分离及在大豆肽发酵中的应用[J]. 中国酿造, 2008, 12: 41-44. Yue Chaoyin, Shao Wei, Liu Haijun, et al. Isolation and classification of astrain and its application in soybean peptide fermentation[J]. China Brewing, 2008, 12: 41-44.
[8] 杨波, 司晓光, 郝建安, 等. 海洋地衣芽孢杆菌的絮凝活性研究及机理探讨[J]. 化学与生物工程, 2017, 10: 45-48. Yang Bo, Si Xiaoguang, Hao Jianan, et al. Flocculation activity and mechanism of marine[J]. Chemistry & Bioengineering, 2017, 10: 45-48.
[9] 彭亚林. 印度洋深海热液区盐单胞菌和地衣芽胞杆菌的筛选、鉴定及生理生化特性研究[D]. 青岛: 中国海洋大学, 2008. Peng Yalin. Isolation, identification and biochemical characterization ofandfrom deep-sea hydrothermal vent in Indian Ocean[D]. Qingdao: Ocean University of China, 2008.
[10] Logan N A, Berkeley R C W. Identification ofstrains using the API system[J]. Microbiology, 1984, 130(7): 1871-1882.
[11] O’donnell A G, Norris J R, Berkeley R C W, et al. Characterization of,,, andby pyrolysis gas-liquid chromatography, deoxyribonucleic acid-deoxyribonucleic acid hybridization, biochemical tests, and API systems[J]. International Journal of Systematic and Evolutionary Microbiology, 1980, 30(2): 448-459.
[12] Xu D, Cote J C. Phylogenetic relationships betweenspecies and related genera inferred from comparison of 3′ end 16S rDNA and 5′ end 16S–23S ITS nucleotide sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(3): 695-704.
[13] Wang L T, Lee F L, Tai C J, et al. Comparison ofgene sequences, 16S rRNA gene sequences and DNA- DNA hybridization in thegroup[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57: 1846-1850.
[14] 喻国辉, 牛春艳, 陈远凤, 等. 利用16S rDNA结合gyrA和基因对生防芽孢杆菌R31的快速鉴定[J]. 中国生物防治学报, 2010, 26(2): 160-166. Yu Guohui, Niu Chunyan, Chen Yuanfeng, et al. Use of 16S rDNA,andgene sequence to identify a biocontrolsp. strain R31[J]. Chinese Journal of Biological Control, 2010, 26(2): 160-166.
[15] Lapidus A, Galleron N, Andersen J T, et al. Co-linear scaffold of theandgenomes and its use to compare their competence genes[J]. FEMS Microbiology Letters, 2002, 209(1): 23-30.
[16] Rey M W, Ramaiya P, Nelson B A, et al. Complete genome sequence of the industrial bacteriumand comparisons with closely relatedspecies[J]. Genome Biology, 2004, 5(10): 77.
[17] Watanabe K, Nelson J, Harayama S, et al. ICB database: thedatabase for identification and classification of bacteria[J]. Nucleic Acids Research, 2001, 29(1): 344-345.
[18] Yamamoto S, Harayama S. PCR amplification and direct sequencing ofgenes with universal primers and their application to the detection and taxonomic analysis ofstrains[J]. Applied and Environmental Microbiology, 1995, 61(3): 1104-1109.
[19] Hall T A. Bioedit: a user—friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95-98.
[20] Thomson J D, Gibson T J, Plewniak F, et al. The clustal_X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997; 25: 4876-4882.
[21] Zhao C L, Cui B K, Dai Y C. New species and phylogeny ofbased on morphological and molecular characters[J]. Fungal Diversity, 2013; 58(1): 47-60.
[22] Jiang X Z, Yu H Y, Xiang M C, et al.variabile, a new genus and species offrom soil nematodes[J]. Fungal Diversity, 2011, 46: 43-51.
[23] 肖玉娟, 傅奇, 沈金海, 等. 一株引起厦门地区不同作物感染的分离与鉴定[J]. 科学技术与工程, 2018, 18(14): 20-23. Xiao Yujuan, Fu Qi, Shen Jinhai, et al. Isolation and identification of astrain which infected different crops in Xiamen area[J]. Science Technology and Engineering, 2018, 18(14): 20-23.
[24] Minogue T D, Daligault H A, Davenport K W, et al. Complete genome assembly ofATCC 25922, a serotype O6 reference strain[J]. Genome Announcements, 2014, 2(5): e00969-14.
[25] 全国饲料工业标准化技术委员会. NY/T 1461-2007 饲料微生物添加剂地衣芽孢杆菌[S]. 北京: 中国标准出版社, 2007: 2-3.National Feed Industry Standardization Technical Committee. NY/T 1461-2007. Microorganism feed additives[S]. Beijing: Standards Press of China, 2007: 2-3.
[26] Satomi M, La Duc M T, Venkateswaran K. Bacillus safensis sp. nov., isolated from spacecraft and assembly-facility surfaces[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(8): 1735-1740.
[27] Hsieh F C, Li M C, Lin T C, et al. Rapid detection and characterization of surfactin-producing Bacillus subtilis and closely related species based on PCR[J]. Current Microbiology, 2004, 49(3): 186-191.
[28] Rachinger M, Volland S, Meinhardt F, et al. First insights into the completely annotated genome sequence of Bacillus licheniformis strain 9945A[J]. Genome Announcements, 2013, 1(4): e00525-13.
Identification of astrain from seawater using a phylogenetic tree based on 16S andgene sequences
FU Qi, LIN Jun-jie, GAN Mei-yu, LU Yuan-qin, XIAO Yu-juan
(College of Environment and Public Health, Xiamen Huaxia University, Xiamen 361024, China)
For identifying bacteria with highly similar conserved sequences, 16S rDNA does not provide adequate resolution for comparative sequence analysis; therefore, more DNA sequences and analysis models are required for classification. To develop a rapid identification method, phylogenetic trees based on 16S andgene sequences were investigated. A strain of bacillus was isolated from seawater, and the 16S andgenes of the strain were amplified with universal primers and sequenced. Using BLAST in GenBank, other strains with similar 16S andgenes were selected. Phylogenetic trees based on 16S andgenes were constructed using PAUP* 4.0. In the tree constructed using sequences of both 16S andgenes, strains belonging to the same species were clustered together, the self-expanding values of the branches were higher than 98, and the classification structure was accurate. The isolated strain polymerized within the phylogenetic tree, and the self-expanding value was 100. Physiological and biochemical tests verified that the strain belongs to the species. Thus, the identification of this strain ofobtained by construction of a phylogenetic tree based on the combination of 16S andgenes was accurate, rapid, and simple.
16S;; phylogenetic tree;
Oct. 18, 2019
[Fujian Education and Scientific Research Project for Young and Middle-aged Teachers, No. JAT170821; The Undergraduate Innovation and Entrepreneurship Training Program of Fujian Province, No. 201812709001; Teaching Reform Research Project of Fujian Province, No. FBJG20180124]
Q939
A
1000-3096(2020)04-0090-06
10.11759/hykx20191018002
2019-10-18;
2019-11-08
福建省中青年教师教育科研项目(科技类)(JAT170821); 福建省大学生创新创业训练计划项目(201812709001); 福建省本科高校教育教学改革研究项目(FBJG20180124)
傅奇(1986-), 男, 湖南湘潭人, 讲师, 硕士, 主要从事微生物资源开发, 电话: 0592-6276262, E-mail: fq@hxxy.edu.cn; 肖玉娟,
, 电话: 0592-6276260, E-mail: xyj@hxxy.edu.cn
(本文编辑: 谭雪静)
