基于Gap-LCR反应可视化检测A类大豆皂苷缺失
2020-04-28谈美霞李文帅武国瑞王敏杜维俊岳爱琴赵晋忠
谈美霞,李文帅,武国瑞,王敏,杜维俊,岳爱琴*,赵晋忠*
(1.山西农业大学 文理学院,山西 太谷 030801;2.山西农业大学 农学院,山西 太谷 030801)
大豆皂苷是大豆种子中一类重要的次生代谢产物[1]。大豆皂苷主要分为A类、DDMP类、B类和E类4类[2, 3]。A类大豆皂苷是以大豆皂醇A为配基,而DDMP类、B类和E类是以大豆皂醇B为配基[4, 5]。研究表明A类皂苷末端糖基乙酰化导致大豆及其制品具有苦味和涩味[6],而DDMP类、B类和E类皂苷具有降低胆固醇、抑制结肠癌细胞的增殖、抗血脂氧化、抗炎以及抗HIV病毒等多种对人体有益的生理功能[7~9]。因此,A类皂苷的缺失可以改进大豆及其豆制品的品质。Takada等[10~12]通过对日本种质资源进行筛选,筛选出一个缺失A类大豆皂苷的突变体材料,进一步研究表明由隐性等位基因sg-5控制。Yano等研究者图位克隆GmSg-5基因,该基因编码一种细胞色素P450酶CYP72A69,CYP72A69可以将大豆皂醇B的21位羟基化形成大豆皂醇A[13]。Rehman等[14, 15]通过比对GmSg-5基因和突变体中Gmsg-5基因序列共发现4个SNP位点引起氨基酸变化,其中第1 127位点G→A变异导致CYP72A69第376位氨基酸由精氨酸突变为赖氨酸,使其丧失将大豆皂醇B的21位羟基化形成大豆皂醇A的功能,从而使大豆材料不能合成A类大豆皂苷。
传统检测单核苷酸多态性(single-nucleotide polymorphis,SNP)的方法有直接测序法、限制性片段长度多态性、扩增产物限制性内切酶酶切多态性、等位基因特异PCR技术等[16]。直接测序法直观、准确性高,但周期长、费用高;后3种方法特异性低,并不适用于所有单核苷酸多态性的检测,而且均需要琼脂糖凝胶电泳法,操作繁琐,容易发生交叉污染,因此,亟需建立一套简便、快速、准确、经济的SNP检测方法,以满足大豆分子标记辅助选择育种的需求。唐卓课题组开发了基于DNAzyme的缺口-连接酶链反应(gap ligase chain reaction,Gap-LCR)可视化SNP检测技术,并应用于镰刀形贫血基因突变位点的检测。该技术利用Gap-LCR反应扩增目标DNA,将DNAzyme序列引入连接产物中,然后利用 Lambda Exonuclease降解没有参与连接反应的磷酸化探针,及Exonuclease III释放DNAzyme,释放的DNAzyme和卟啉铁(hemin)结合后可以形成具有过氧化物酶活性的G-四链体结构,可以使显色底物ABTS氧化为绿色的ABTS+,因此可以直接在室温下通过颜色变化来检测单核苷酸多态性[17~22]。Gap-LCR相较于传统SNP检测技术,具有较高的特异性和灵敏度、检测时间短、不需要依赖精密的仪器、不需要通过电泳来检测等优点。
本文基于DNAzyme的Gap-LCR反应检测SNP的方法,分别设计了GmSg-5/Gmsg-5基因的特异探针,建立鉴定A类大豆皂苷缺失材料特异性的可视化快速检测方法,为今后快速筛选A类皂苷缺失大豆材料奠定基础。
1 材料和方法
1.1 试验试剂和仪器
1.1.1 试验试剂
9°N DNA Ligase、Klenow Fragment、Lambda Exonuclease及Exonuclease III均购买于NEB(New England Biolabs)。ATP购买于美国Thermo公司。dTTP、ABTS、hemin均购买于北京索莱宝科技有限公司。dNTP购买于北京全式金生物科技有限公司。
1.1.2 试验仪器
PCR仪(Mastercycler nexus GX2,德国Eppendorf);荧光定量PCR仪(CFX96 Touch,美国Bio-RAD);电泳仪(powerpac Basic,美国Bio-RAD);全自动凝胶成像系统(GeneGenius,英国Syngene);核酸蛋白检测仪(BioSpectrometer kinetic,德国Eppendorf);数码单反相机(EOS 70D,中国Canon);电子分析天平(MSA3.6P-OCE-DM,德国Sartorius)。
1.2 试验方法
1.2.1 探针设计
参照NCBI公布的GmSg-5/Gmsg-5基因序列差异,设计了鉴定Gmsg-5/GmSg-5基因第1 127位点G/A变异的Gap-LCR反应特异探针,如表1。目标DNA序列及探针均由上海生工生物工程有限公司合成。
1.2.2 Gap-LCR反应体系条件及优化
Gap-LCR反应总体积50 μL,其中5×LCR buffer10 μL[100 mmol·L-1Tris-HCl pH 8.4、100 mmol·L-1KCl、50 mmol·L-1(NH4)2SO4、10 mmol·L-1MgSO4],L、R、Ln、Rn探针各6 μL(10 μmol·L-1),1 μL dTTP(1 mmol·L-1),0.3 μL ATP(100 mmol·L-1),0.02 μL 9°N DNA ligase(40 000 U), 0.02 μL Klenow Fragment(5 000 U),1 μLGmSg-5或Gmsg-5(10 μmol·L-1),剩余用ddH2O补足。Gap-LCR反应程序:94 ℃ 3 min,94 ℃ 30 s,65 ℃ 4 min,35个循环,10 ℃保存。
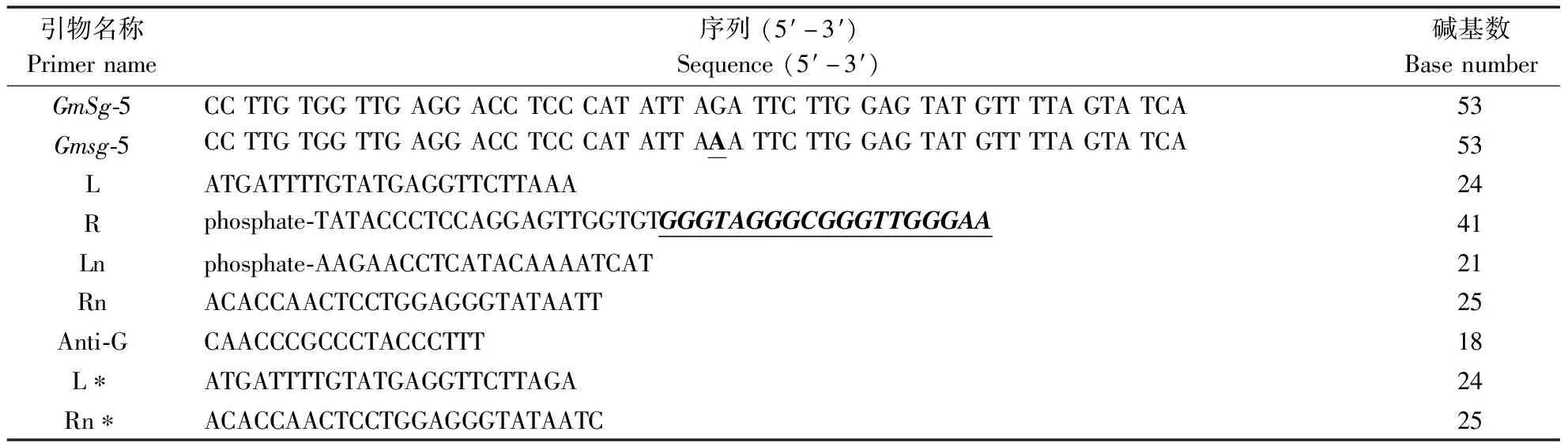
表1 GmSg-5/Gmsg-5基因Gap-LCR反应所涉及的探针和目标序列
注:与GmSg-5相比,Gmsg-5中的下划线和加粗碱基A是突变位点。探针L、R、Ln和Rn与Gmsg-5相同或互补,仅具有单个碱基空位。探针L*、R、Ln和Rn*与GmSg-5相同或互补,仅具有单个碱基空位。探针R的下划线和斜体碱基指DNAzyme序列
模板浓度优化:在其他条件不变的情况下,通过改变50 μL Gap-LCR反应体系中加入的模板的量考察了4个模板浓度对反应结果的影响,分别是0、0.1、0.2、0.3 μmol·L-1。反应结束后经琼脂糖凝胶电泳分离,选择最优的模板浓度。
缓冲溶液pH值优化:考察了7个缓冲溶液pH值(7.8、8.0、8.2、8.4、8.6、8.8、9.0)。反应结束后经琼脂糖凝胶电泳分离和比色检测记录,选择最优的pH值。
探针浓度优化:在其他条件不变的情况下,通过改变50 μL Gap-LCR反应体系中加入的探针的量考察了4个探针浓度对反应结果的影响,分别是0.8、1.0、1.2、1.4 μmol·L-1。反应结束后经琼脂糖凝胶电泳分离,选择最优的探针浓度。
1.2.3 Gap-LCR可视化体系
首先取10 μL PCR产物、10 μL ddH2O、2.5 μL 5×LCR buffer、1 μL Anti-G(10 μmol·L-1)和0.8 μL NaCl(3 mol·L-1),混匀,94 ℃加热1 min,冷至室温;然后向反应体系中加入1 μL Lambda Exonuclease(5 000 U),37 ℃孵育60 min,85 ℃加热15 min使 Lambda Exonuclease失活,冷至室温;然后在混合体系中加0.5 μL Exonuclease Ⅲ(100 000 U),37 ℃孵育5 min,70 ℃加热20 min,冷至室温。经过以上处理后的混合溶液中加2.4 μL hemin(50 μmol·L-1)、1.27 μL ABTS(60 μmol·L-1)和0.6 μL H2O2(50 mmol·L-1),10 min左右观察颜色变化或在核酸蛋白检测仪上测定420 nm处的吸光值。
2 结果与分析
2.1 GmSg-5/Gmsg-5基因Gap-LCR特异探针的设计
下载NCBI网站中的GmSg-5(MF624839.1)和Gmsg-5(LC143441.1)基因序列并进行比对,比对结果(图1),发现第1 127位点存在G/A变异,该SNP位点导致CYP72A69第376位氨基酸由精氨酸突变为赖氨酸,从而使大豆材料中不能合成A类大豆皂苷[14, 15],依此SNP位点的序列差异,根据Gap-LCR反应原理设计了Gap-LCR方法检测Gmsg-5基因的一套特异探针L、R、Ln、Rn(图2)。为了增加试验的准确性,避免假阳性,同步设计检测GmSg-5基因的一套互补特异探针L*、R、Ln、Rn*,以获得与前者相反的结果,两套探针相互验证。本试验的反应原理如图2所示。

图1 GmSg-5/Gmsg-5基因序列比对图
2.2 Gap-LCR检测Gmsg-5基因反应体系的优化
2.2.1 Gap-LCR反应模板浓度的优化
考察模板浓度对Gap-LCR反应体系的影响,从图3可以看出,第2、3、4、5泳道均有L+R/Ln+Rn、R/Rn和L/Ln 3条带,第4泳道的目标条带L+R/Ln+Rn亮度最高,因此选择0.2 μmol·L-1模板浓度作为Gap-LCR反应最佳条件。
2.2.2 Gap-LCR反应缓冲溶液pH值的优化
考察缓冲溶液pH值对Gap-LCR反应体系的影响,从图4A可以看出第1~7泳道均出现L+R/Ln+Rn、R/Rn和L/Ln 3条带,但第4泳道pH为8.4时目标带L+R/Ln+Rn亮度最高;比色检测结果如图4B也可以看出第4管pH为8.4颜色最深,因此选择pH8.4为Gap-LCR反应缓冲液最佳pH值。
2.2.3 Gap-LCR反应探针浓度的优化
考察探针浓度对Gap-LCR反应体系的影响,从图5可以看出第4泳道探针浓度为1.2 μmol·L-1时目标带L+R/Ln+Rn亮度最高,且副产物R/Rn和L/Ln少,因此选择探针浓度1.2 μmol·L-1为Gap-LCR反应最佳反应条件。
2.2.4GmSg-5/Gmsg-5基因互补探针Gap-LCR反应
为了避免检测结果的假阳性,分别根据GmSg-5和Gmsg-5序列差异设计2套互补探针,即基于Gmsg-5序列的一套探针L、R、Ln、Rn和基于GmSg-5序列的一套探针L*、R、Ln、Rn*。利用以上Gap-LCR优化反应体系分别加入两套探针进行Gap-LCR反应,结果如图6。由图6可以看出,第2、3泳道Gap-LCR反应体系中加入的目标片段均为Gmsg-5,分别加入两套探针L*、R、Ln、Rn*(第2泳道)和L、R、Ln、Rn(第3泳道),第2泳道出现目标条带L*+R/Ln+Rn*,而第3泳道没有目标带;第5、6泳道Gap-LCR反应体系中加入目标片段均为Gmsg-5,分别加入2套探针L*、R、Ln、Rn*(第5泳道)和L、R、Ln、Rn(第6泳道),得到相反的结果,第5泳道没有目标带,而第6泳道出现目标条带L+R/Ln+Rn。本试验结果表明在Gap-LCR优化体系条件下,基于GmSg-5/Gmsg-5序列的2套探针L、R、Ln、Rn和L*、R、Ln、Rn*能够特异性的分别识别Gmsg-5和GmSg-5序列。

图2 基于DNAzyme的Gap-LCR反应检测Gmsg-5基因的原理

M:50 bp DNA Marker;1:加L、R引物的普通PCR扩增Gmsg-5基因片段;2~5:模板浓度分别为0、0.1、0.2、0.3 μmol·L-1

M:50 bp DNA Marker;1~7:缓冲溶液pH分别为7.8、8.0、8.2、8.4、8.6、8.8、9.0
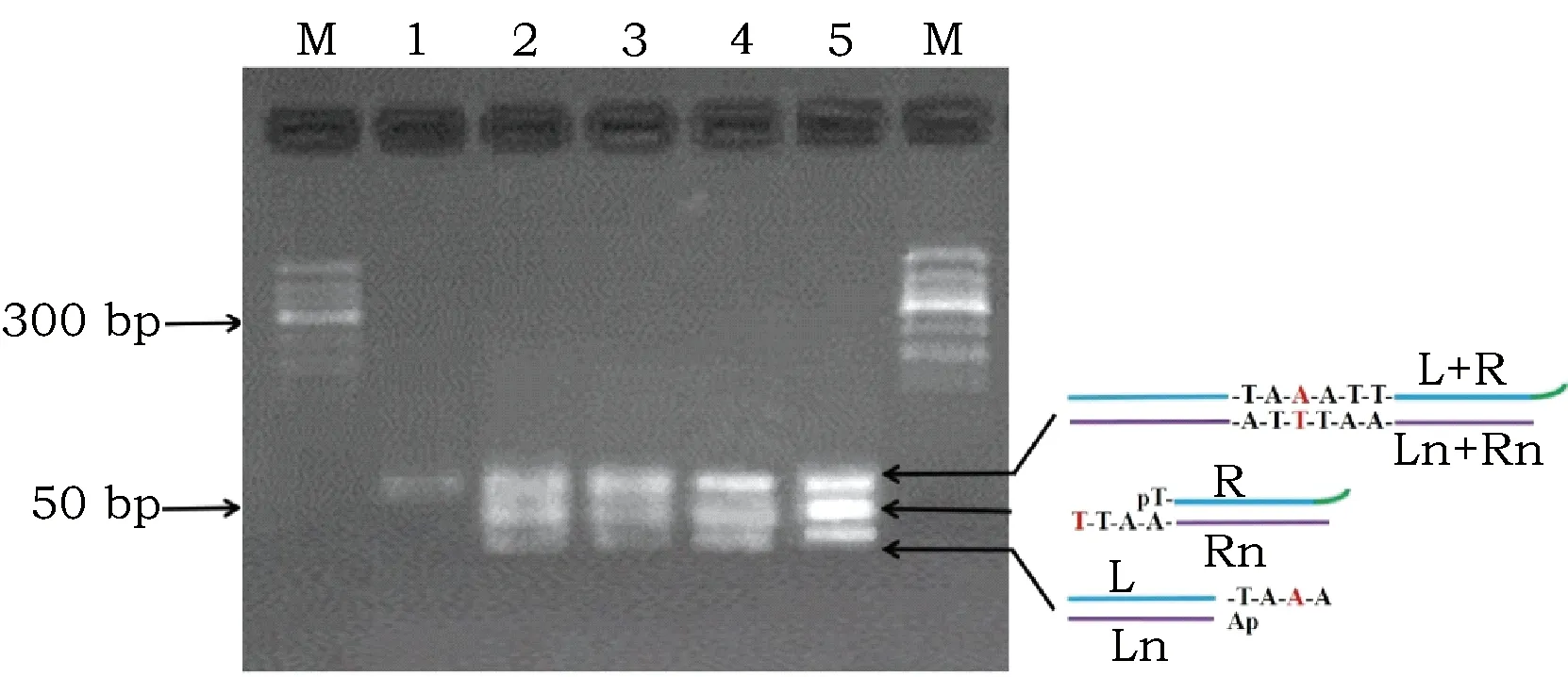
M:50 bp DNA Marker;1:加L、R引物的普通 PCR扩增Gmsg-5基因片段;2~5:探针浓度分别为0.8、1.0、1.2、1.4 μmol·L-1
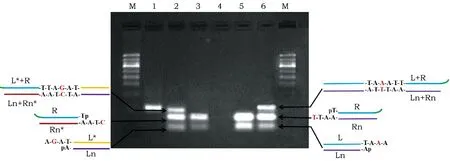
M:50 bp DNA Marker;1:用L、R引物PCR扩增Gmsg-5基因片段;2:模板GmSg-5和探针L*、R、Ln、Rn*;3:模板GmSg-5和探针L、R、Ln、Rn;4:空白对照;5:模板Gmsg-5和探针L*、R、Ln、Rn*;6:模板Gmsg-5和探针L、R、Ln、Rn
2.3 基于DNAzyme的Gap-LCR反应鉴定GmSg-5/Gmsg-5基因的显色反应
由于Gap-LCR反应时设计的探针R序列中引入DNAzyme序列,因此反应结果的检测可以利用DNAzyme的特性来进行SNP 的可视化检测,而不需琼脂糖凝胶电泳。
显色反应前需要在Gap-LCR反应产物中加入Lambda Exonuclease将未参加反应的探针R/Rn和L/Ln或R/Rn*和L*/Ln消去,加入Exonuclease Ⅲ消化互补链Ln+Rn或Ln+Rn*将Gap-LCR产物的DNAzyme序列释放出来。结果如图7 A所示,Gap-LCR反应产物经Lambda Exonuclease和Exonuclease Ⅲ消化后,仅第2、6泳道有一条带L*+R(第2泳道)或L+R(第6泳道),说明经过Lambda Exonuclease和ExonucleaseⅢ消化后,能够完全释放含有DNAzyme目标片段序列释放出来,并且将多余探针形成R/Rn和L/Ln或R/Rn*和L*/Ln全部消化掉,为下一步显色反应做好准备。
将酶切反应后的体系中加入hemin、ABTS和H2O2进行显色反应,结果如图7B所示,添加模板GmSg-5和探针L*、R、Ln、Rn*(管2)、模板Gmsg-5和探针L、R、Ln、Rn(管6)的反应管显绿色,而添加模板GmSg-5和探针L、R、Ln、Rn(管3)、模板Gmsg-5和探针L*、R、Ln、Rn*(管5)、空白对照(管4)均不显色。实验结果表明基于DNAzyme的Gap-LCR反应的可视化检测体系可以简便快捷、准确应用于等位基因Gmsg-5/GmSg-5的SNP的检测,从基因水平上快速准确鉴定出A类皂苷缺失的大豆材料。

M:50 bp DNA Marker;1:用L、R引物PCR扩增Gmsg-5基因片段;2:模板GmSg-5和探针L*、R、Ln、Rn*;3:模板GmSg-5和探针L、R、Ln、Rn;4:空白对照;5:模板Gmsg-5和探针L*、R、Ln、Rn*;6:模板Gmsg-5和探针L、R、Ln、Rn
另外对显色反应的时间进行了研究,从图8可以看出,0~10 min显色液吸光度值不断增加,10 min吸光度值达到最大,显色反应10 min后吸光度值又不断减小,说明显色反应10 min为最佳可视化检测观察时间。

图8 显色反应体系吸光度随反应时间增加的变化情况
3 讨论与结论
现有直接分析检测大豆A类皂苷类型的方法主要包括:薄层色谱法(Thin-Layer Chromatography,TLC)[23]、高效液相色谱法(High Performance Liquid Chromatography,HPLC)[24, 25]、高效液相色谱-质谱联用技术(High Performance Liquid Chromatography-Electrospray Ionization-tandern Mass Spectrometry,HPLC-ESI-MS/MS)[26]。TLC法可以对大豆皂苷组成进行快速筛选,但是不够精确;HPLC和HPLC-ESI-MS/MS克服了薄层色谱法缺点,但使用仪器价格昂贵、运行成本高、费时,很难用于大量大豆种质资源的筛选。因此,有必要建立一种快速、准确、经济检测A类大豆皂苷缺失的方法。
近年来,大豆皂苷代谢途径中的相关基因不断被克隆,尤其是A类皂苷代谢的关键酶基因GmSg-5/Gmsg-5被克隆,为进一步从基因水平检测A类皂苷奠定了基础[10~15]。本研究通过比对大豆A类皂苷关键酶基因GmSg-5/Gmsg-5基因序列,找出SNP位点,根据GmSg-5/Gmsg-5基因SNP位点分别设计了GmSg-5/Gmsg-5基因的2套特异性互补探针L*、R、Ln、Rn*和L、R、Ln、Rn。同时优化了Gap-LCR反应体系,该体系在0.2 μmol·L-1模板浓度、缓冲液pH值8.4、探针浓度1.2 μmol·L-1条件效果最好,显色反应10 min即可分别实现对GmSg-5/Gmsg-5基因的检测。Gap-LCR反应体系中加入Gmsg-5基因的一套特异探针L、R、Ln、Rn,显色反应显绿色;而加入GmSg-5基因的一套特异探针L*、R、Ln、Rn*显色反应不显色说明该大豆材料不合成A类大豆皂苷,为A类皂苷缺失材料。
本研究建立的基于DNAzyme的Gap-LCR反应检测A类大豆皂苷缺失的可视化检测方法,与直接测序法、限制性片段长度多态性、扩增产物限制性内切酶酶切多态性、等位基因特异PCR技术等从基因水平SNP检测手段相比[16],上述几种方法均需要首先进行普通PCR,而本研究所用的是Gap-LCR,由于Gap-LCR只添加几个碱基,所以程序简单省时,只需要变性和退火两步。另外上述几种方法扩增结果的观察均需采用琼脂糖凝胶电泳法,操作繁琐,容易发生交叉污染,特异性低,本研究在Gap-LCR扩增体系的一个探针R的3′端引入了DNAzyme序列,可以直接在室温下通过颜色变化来检测单核苷酸多态性。Abravava等[27]比较了Gap-LCR与等位基因特异性PCR(ASPCR)在检测HIV逆转录酶基因突变体时的差异,证明Gap-LCR是一种灵敏而特异的扩增技术,可以准确检测单碱基突变。不足之处是本研究建立的可视化检测方法只能定性分析A类大豆皂苷的有无,不能进行定量分析,要进行定量分析还有待进一步研究。
通过本研究建立了一种从基因水平上鉴定A类大豆皂苷缺失的可视化检测方法,该方法快速、简单、实用、经济。
