利玛原甲藻PL11共附生菌多样性研究
2020-03-26李月月田晓清韩清华樊成奇马丽艳陆亚男
李月月,田晓清,韩清华,樊成奇,马丽艳,陆亚男
(1.上海海洋大学,上海 201306;2.中国水产科学研究院东海水产研究所,上海 200090;3.农业农村部东海渔业资源开发利用重点实验室,上海 200090)
原甲藻(Prorocentrum)是导致赤潮的重要生物之一,其代表产物大田软海绵酸(okadaic acid,OA)及其结构类似物鳍藻毒素-1(dinophysistoxin-1,DTX-1)是腹泻性贝类毒素(diarrheic shellfish poisoning,DSP)的主要组分。腹泻性贝类毒素引起的中毒事件具有全球性,自20世纪60年代以来在欧洲、亚洲、北美洲、南非等地都曾有报道[1],DSP不仅会危害海洋生态平衡,还会通过海洋食物链的富集威胁人类健康[2],同时也是蛋白磷酸酶2A、1和2B的有效抑制剂,可以导致人体消化系统中癌症的发生[3]。而利玛原甲藻(P.lima)是原甲藻属甲藻中主要的产毒藻株,可以作为腹泻性贝毒的稳定性来源,因此利玛原甲藻在很长一段时间内一直是人们研究的热点。然而直到目前,对于利玛原甲藻产毒机制方面的研究仍较少。事实上,利玛原甲藻中生物毒素的表达水平受营养和环境因素的影响很大[4],但是包括环境微生物在内的各种因素对其产毒的具体影响仍未有定论。虽然目前认为利玛原甲藻相关细菌并不能独立产生毒素[5],但是很多学者认为其对藻毒素的产生有着直接或间接的影响[6]。有研究表明,细菌可能对利玛原甲藻细胞产生毒素有一定的协同作用,在利玛原甲藻的衰老阶段,产毒水平增加,这些细菌可能开始降解利玛原甲藻细胞,会加速微藻的衰老,裂解作用可能会增加溶解有机碳的数量,这可能为增加毒素的产生提供更多的资源,从而促进毒素的生成[1]。目前,关于细菌对利玛原甲藻产毒的影响只进行了初步的探索,它们之间的相互作用机制仍然需要更深入的研究。
本文以利玛原甲藻藻株为研究材料,通过高通量测序及微生物纯培养技术,对利玛原甲藻PL11可能的共附生菌群的多样性进行分析,包括其群落的组成以及相对丰度。并通过纯培养的方法得到9株可培养细菌,以期为后期继续研究细菌在微藻产毒过程中的作用提供必要的生物材料及基础。
1 材料与方法
1.1 材料与仪器
生物材料:利玛原甲藻PL11。
试剂材料:Ezup柱式基因组DNA抽提试剂盒(上海生工生物有限公司);16s引物(上海生工生物有限公司);PCR扩增体系(康为世纪);PBS缓冲液(上海生工生物有限公司)。
培养基:f/2培养基,2216E培养基。
实验仪器:高压灭菌锅:股海申安医疗器械厂,LDZX-75KBS型;大型恒温培养箱:上海化学仪器有限公司,ATB-032型;超净工作台:北京东联哈尔仪器有限公司,DL-CJ-INDII型;台式高速离心机:湖南湘仪离心机仪器有限公司,H-1560型;超声仪:上海汉克科学仪器有限公司,SK250LH型;PCR仪:杭州朗基科学仪器有限公司,AG-22331型;凝胶成像系统:上海天能科技有限公司,GIS-1600型。超纯水仪:上海智岩科学仪器有限公司,Milli-QAdvantage型。
1.2 实验方法
1.2.1 藻培养
利玛原甲藻采用f/2培养基进行培养。f/2培养基由4种储备液配制而成,分别为75.0 g·L-1NaNO3溶液、5.0 g·L-1NaH2PO4·H2O溶液、L1 Trace Metal Solution和Vitamin Solution。其中 NaNO3溶液、NaH2PO4·H2O、L1 Trace Metal Solution以超纯水配制后,高温灭菌放入4℃冰箱内储存备用;Vitamin Solution以高温灭菌后的超纯水配制备用。L1 Trace Metal Solution和Vitamin Solution为混合溶液,具体配方见表1。
f/2培养基配制过程如下:将超纯水用盐卤将其盐度调至23,分别加入1 mL NaNO3溶液、NaH2PO4·H2O溶液以及L1 Trace Metal Solution,高压灭菌锅高温灭菌30 min(120℃),冷却至20℃加入0.5 mL Vitamin Solution。利玛原甲藻的培养条件:光照强度为4 500~5 000 lx,光照周期为12L/12D,温度为24℃。

表1 f/2培养基配方Tab.1 Medium formula of f/2
1.2.2 高通量测序及数据分析
实验室培养藻的密度达到1×106个·L-1后,取50 mL藻液于无菌离心管中,离心(转速6 000 r·min-1,时间 5min),收集藻细胞,以 30 mL pH 7.2的PBS缓冲液洗涤藻细胞,重复4次放置备用。藻共附生菌总基因组DNA以Stool DNA Kit试剂盒(美国OMEGA)提取,取1mL以扩增引物338F和806R进行扩增。上游引物为338F:5′-ACTCCTACGGGAGGCAGCAG-3′,下游引物 为 806R:5′-GGACTACHVGGGTWTCTAAT-3′[7]。PCR扩增反应体系如下:模板 DNA:10 ng,BSA:0.2μL,DNA多聚酶:0.4μL,5×FastPfu缓冲液:4μL,2.5 mmol·L-1dNTPs:2μL,上游引物(5μmol·L-1):0.8μL,下游引物(5μmol·L-1):0.8μL,加入灭菌后的超纯水使总体积达到50μL。PCR反应条件:预变性时间5 min(95℃),进入循环,变性时间40 s(95℃),退火时间40 s(55℃),延伸时间60 s(72℃),循环30次,最后延伸15 min(72℃)。通过电泳来检验PCR完成的产物是否合格,如条带清晰,无降解则符合后续测序要求。如条带不清晰或未出现,则重新进行PCR扩增,若再次扩增后仍没出现清晰的条带则要重新提取。回收PCR产物样品进行Illumina Miseq高通量测序(上海美吉生物公司)。
数据质控:根据paired-end reads之间的互相重叠进行序列拼接,对reads的质量和拼接的效果进行质控分析[8],具体操作如下:以 FLASH对每个样品的读长进行拼接,得到原始连接数据(raw tags),对原始连接数据进行进一步的过滤以得到高质量的连接数据(clean tags);后续进行tags截取,截取质量阈值为≤19,碱基数默认长度为3,得到tags数据集,过滤掉连续高质量碱基长度小于tags长度75% 的 tags。最后进行去除嵌合体序列的处理,通过与数据库的比对,移除掉Chimera序列,得到有效数据(effective tags)[9]。
数据分析:按照序列间的距离进行聚类分析,从中选取与代表序列相似度大于97%的序列生成操作分类单元(OTU)。选取Silva作为细菌16SrRNA数据库、RDP、Greengene为功能基因数据库,使用Qiime平台与 RDP Classifier分别作为计算软件及算法,设定0.7为置信度阈值[10]。菌落丰富度(community richness)指数分别为Chao和ACE;菌落多样性(community diversity)指数为Shannon以及 Simpson。稀释曲线(rarefaction curve)采用对序列进行随机抽样的方法,以抽到的序列数与它们对应的物种(如OTU)数目或多样性指数来构建。
1.2.3 可培养共附生菌株的分离及鉴定
取1.5 mL藻液(对数生长期)于2 mL的EP管中,离心(转速 6 000 r·min-1,5 min)。弃去上清液,加入1.5 mL无菌超纯水轻轻震荡混匀,重复离心一次,弃上清液加入1.5 mL无菌超纯水震荡混匀,冰浴下超声波破碎藻细胞处理10 min,然后用灭菌超纯水将其稀释10倍,分别取200μL破碎藻细胞原液与稀释后的藻液均匀的涂布在2216E平板培养基上,28℃培养48 h,期间不断进行观察,待培养基上生长出单菌落后,在2216E平板培养基上再次进行划线纯化,彻底纯化后的单菌落接种到固体斜面培养基上,28℃培养48 h后接种到2216E液体培养基中并放入摇床中培养48 h(23℃)[10-11]。
16S rRNA测序及比对:使用细菌基因组DNA提取试剂盒(日本 TaKaRa)提取基因组DNA,按照试剂盒的说明书进行具体实验步骤:取1 mL菌液至1.5 mL无菌离心管中,离心(转速8 000 r·min-1,时间 1 min),弃上清收集菌体。加入180μL溶菌酶溶液重悬菌液,37℃水浴30~60 min,再加入400μL Buffer Digestion,震荡混匀,65℃水浴1 h。加入200μL Buffer PB,充分混匀,-20℃放置5 min后离心(转速10 000 r·min-1,时间5 min),将上清液转移至新的离心管中。加入1 mL异丙醇,充分混匀后离心(转速10 000 r·min-1,时间 5 min),弃上清。加入 1 mL 75%乙醇,离心(转速10 000 r·min-1,时间2 min),弃上清。打开离心管的盖子倒置5~10 min使乙醇完全挥发,得到所需DNA。加入50 μL TE Buffer溶解DNA,以16S rRNA扩增通用引物27F及1492R进行扩增,其序列分别为上游引物 27F:5′-AGAGTTTGATCCTGGCTCAG-3′;下游引物 1492R:5′-GGTTACCTTGTTACGACTT-3′。PCR扩增体系包括:23μL的 2×TaqPCR MasterMix:,1μL上游引物,1μL下游引物,5μL模板DNA,20μL去离子水[10]。PCR扩增程序如下:预变性时间4 min(95℃),进入循环,变性时间40 s(95℃),退火时间40 s(57℃),延伸时间2 min(72℃),循环 40次,最后延伸 10 min(72℃)。PCR扩增结束后进行电泳。回收样品进行基因测序(上海美吉生物公司),对获得的16SrRNA基因序列进行分析,通过EzBioCloud进行菌株序列同源性对比,建立菌株的16S rRNA基因系统发育进化树(MEGA软件)[7]。
2 结果与分析
2.1 样品信息统计
利玛原甲藻PL11共附生微生物的测序结果如表2所示,通过高通量测序一共获得42 065条序列。稀释曲线(rarefaction curve)可以用来比较测序样本中物种的丰富度、均一性或多样性,也可以用来说明样本的测序数据量是否合理[12]。以Shannon为多样性指数构建稀释曲线(图1),样品的曲线逐渐趋向水平,说明测序数据量已经足够大,可以反映样品中的物种多样性,样本的测序数量是合理的。

表2 利玛原甲藻PL11共附生菌群高通量测序样本信息表Tab.2 Summary of the high-throughput sequencing sample from P.lima PL11
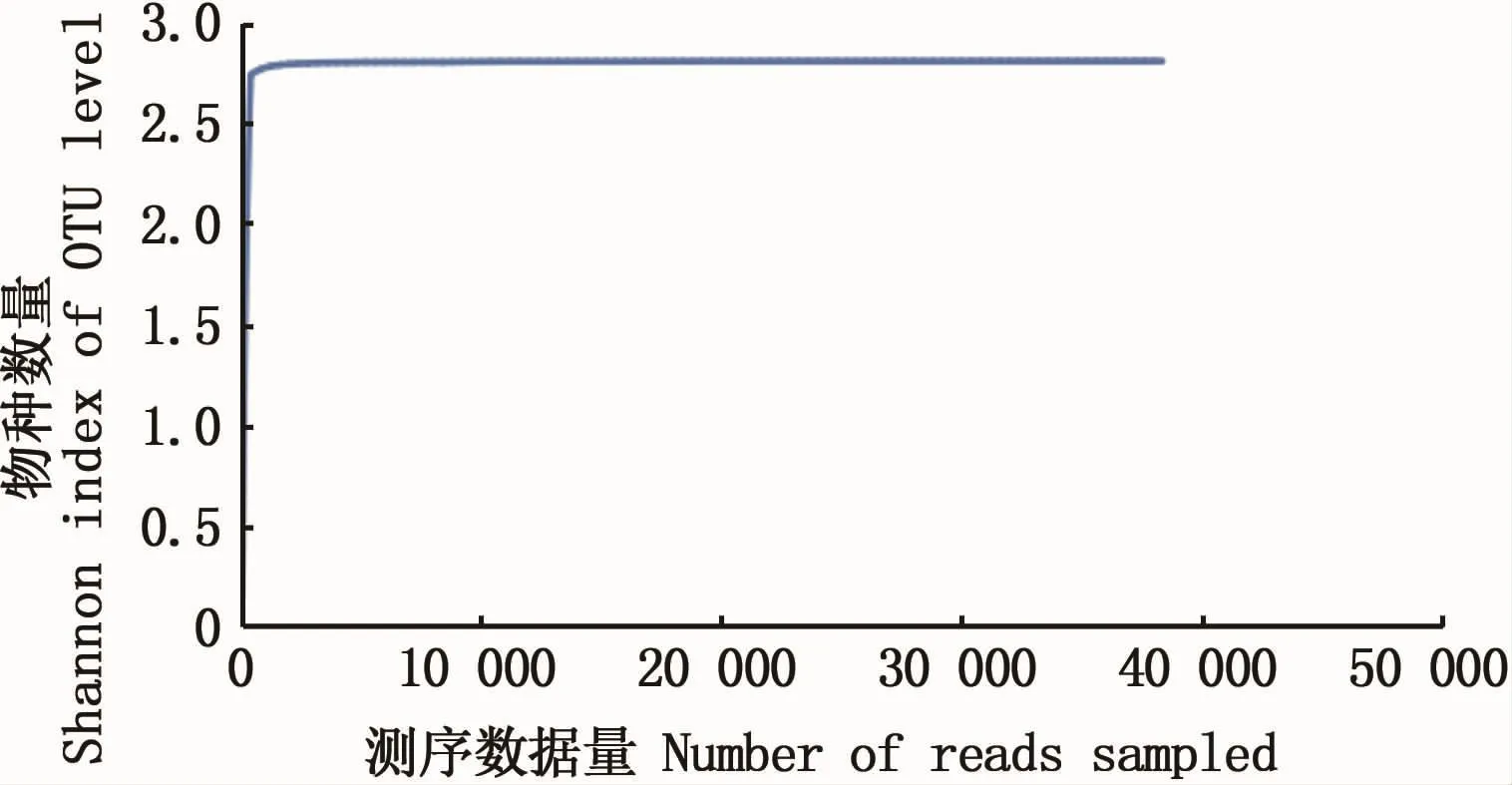
图1 利玛原甲藻PL11共附生菌群高通量测序样品Shannon-Wiener曲线Fig.1 Shannon-Wiener curve of the sequencing sample from P.lima PL11
2.2 利玛原甲藻的共附生菌多样性分析
MiSeq高通量测序结果显示,利玛原甲藻PL11共附生菌群共68个OTU,包括5门,14纲,26目,38科及54属。PL11共附生菌群属水平相对丰度分布如图2所示。其中优势门(>5%)3个,包括变形菌门(Proteobacteria,75.5%)、拟杆菌门(Bacteriodetes,11.5%)以及浮霉菌门(Planctomycetex,8.5%)。优势纲(>5%)4个,包括 α-变形菌纲(Alphaproteobacteria,51.7%)、δ-变形菌纲(Deltaproteobacteria,31.8%)、鞘脂杆菌纲(Sphingobacteria,9.9%)以及 OM190纲(8.9%);优势属(>5%)6个,包括侏囊菌科下未 知 属 (norank Nannocystaceae,21.8%)、Pyruvatibacter属(11.6%)、Phaeodactylibacter属(9.4%)、生丝单胞菌科下未知属(norank Hyphomonadaceae,8.5%)、OM190纲下未知属(8.1%)以及Roseovarius属(7.9%)。用MEGA6建立的利玛原甲藻PL11共附生菌群系统发育进化树见图3。
2.3 利玛原甲藻可培养共附生菌的系统发育进化分析
从利玛原甲藻PL11培养物中分离出了9株细菌,用MEGA6建立的系统发育进化树见图4,通过序列同源性比对(表3)以及系统发育进化树的结果显示,这些菌株分属于8个属,如表3所示,分 别 为Ochrobactrumsp.(2株)及Microbacteriumsp.、Algoriphagussp.、Ponticoccussp.、Hoefleasp.、Labrenziasp.、Erythrobactersp.、Sphingopyxissp.各1株。其中,菌株PL11-1与已知菌株的16S rRNA基因同源性最高值为97.55%,说明其为Ponticoccus属潜在新种,其多相分类学鉴定分析目前正在进行中。
3 讨论

图3 利玛原甲藻PL11共附生菌群系统发育进化树Fig.3 Phylogenetic tree of bacterial community associated with P.lima PL11
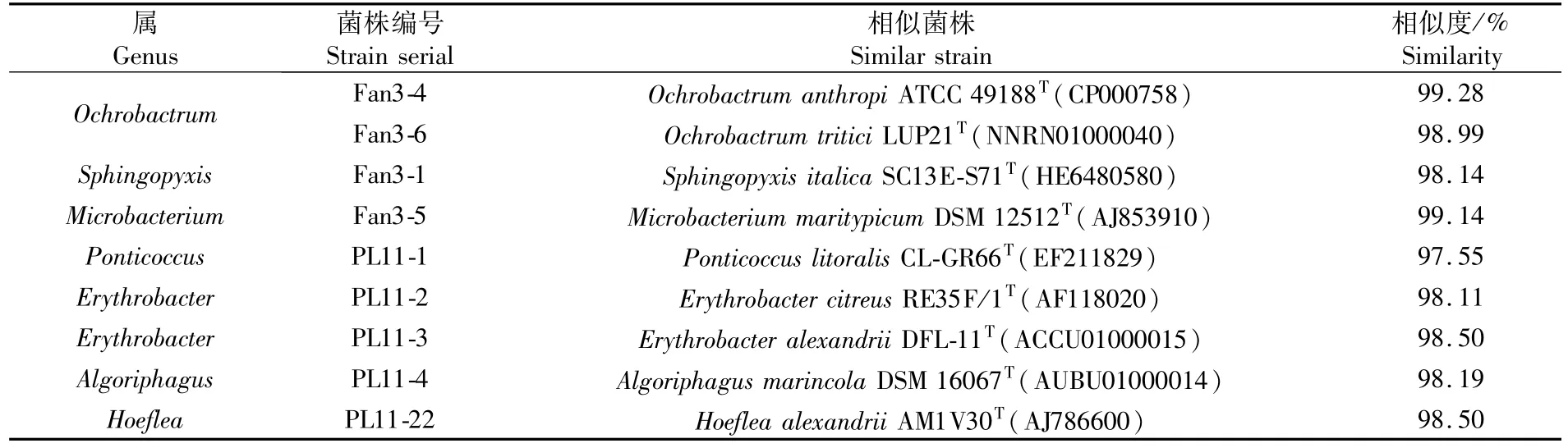
表3 菌株种属的分类结果Tab.3 Classification results of strain species

图4 基于16Sr RNA序列构建的可培养共附生菌的系统发育进化树Fig.4 Phylogenetic tree based on neighbor-joining analysis of 16S r RNA sequences
细菌和浮游植物之间的相互作用越来越多地被认为是毒素产生和赤潮发生的重要因素[13],了解毒素的产生机制有利用解决其对环境以及人类健康产生的危害,藻菌之间的相互作用关系是了解毒素产生的关键环节。高通量测序技术不经过分离培养微生物就可以分析微生物群落的多样性,从而为优化利玛原甲藻共附生微生物的分离并增加其可培养性带来有价值的参考。本文利用MiSeq高通量测序首次对利玛原甲藻PL11共附生菌群多样性进行了分析。从获得的68个OTU中归类出54个属,其优势属6个。先前电子显微镜观察研究表明,在无菌培养的利玛原甲藻细胞中没有检测到细菌。通过高通量测序可以清楚的看到利玛原甲藻PL11具有丰富且多样的微生物群落。
此外,基于微生物纯培养技术从利玛原甲藻PL11中分离获得9株细菌,包括一株潜在新株PL11-1。PL11-1属于玫瑰杆菌簇,红杆菌科,Ponticoccus属;Poncticoccussp.与藻类赤潮爆发关系密切,TAN等[14]等发现Ponticoccus属下一株菌在发生赤潮时能与藻类相互作用,表现出一定的主导作用。Ponticoccussp.PD-2是首次发现的有群体感应的菌株,它对多种赤潮藻的生长都能产生抑制作用[15]。从利玛原甲藻PL11中得到的PL11-1可能与利玛原甲藻生长和产毒都有着密切的关系。fan3-1属于鞘氨醇单胞菌(Sphingopyxissp.),鞘氨醇单胞菌可以降解微囊藻毒素,通常情况下同一株菌只能降解一种藻毒素,Sphingopyxis属下一株菌却能够同时降解 MCYR、MC-LR和 MC-RR 3种毒素[16]。进一步研究Ponticoccus属下PL11-1与Sphingopyxis属下fan3-1和利玛原甲藻之间的相互作用关系,可以更好地了解利玛原甲藻产毒或毒素降解的机理。关于利玛原甲藻与其共附生微生物的相互作用,本团队其他成员正在利用获得的菌株开展试验研究。
通过高通量测序技术首次解析了利玛原甲藻PL11共附生微生物种类、丰度及多样性信息,还通过分离纯化获得了纯培养的PL11共附生细菌9株,对比发现纯培养获得的菌株种属与高通量测序中相对丰度较高的种属一致性不高,可能是菌株分离时的培养基、温度等因素以及微生物自身的可培养性造成的影响。此结果对于深化对利玛原甲藻共附生微生物多样性的认识,指导其可培养共附生微生物的分离具有重要意义。
