CHAP抗生酶数据库的构建及其信息分析
2019-09-02唐立成吴宏宇黄青山
唐立成 吴宏宇,2 黄青山,*
(1 复旦大学生命科学院遗传学研究所遗传工程国家重点实验室,上海 200433;2 上海高科联合生物技术研发有限公司,上海 201206)
近年来,抗生素滥用导致多重耐药菌出现频率大幅提高,已成为全球关注的公共卫生问题,因此迫切需要寻找抗生素的合适替代品[1]。抗生酶是一类可以使致病微生物死亡的酶,它最早被定义为通过降解细菌细胞壁从而具有抗菌能力的噬菌体裂解酶[2],现在一般也将细菌素、自溶素和溶菌酶[3]包含在内。CHAP抗生酶是一类含有CHAP结构域的抗生酶。CHAP结构域(CHAP domain)是属于抗生酶催化域中的一种,与其他抗生酶催化域不同的是,它是一类半胱氨酸、组氨酸依赖性的酰胺水解酶或肽酶(cysteine,histidine-dependent amidohydrolases/peptidases,CHAP),一般由110~140个氨基酸组成,在亲核攻击机制中利用催化半胱氨酸残基发挥催化活性作用[4]。CHAP结构域由于普遍拥有优异的抑菌活性[5],被应用于新型嵌合体抗菌酶的设计和改造,并且取得了众多研究成果[6-7]。目前已有一些含CHAP结构域抗生酶的报道,并且对其结构和功能也有了不少研究,例如LysGH15的CHAP结构域首次被确证为是一个EF-hand-like钙结合蛋白[8];通过为PlyTW的CHAP结构域添加SH3b结合域可以增加CHAP结构域活性[9]等。
相对于已有大量数据库研究的抗菌肽来说,目前抗生酶数据库仅有EnzyBase[10],phiBIOTICS[11]和GMEnzy[12]3个数据库。它们均未对抗生酶中的CHAP抗生酶进行系统分析,所收集的CHAP抗生酶数据较少甚至没有。为此,本课题从权威数据库和已发表的科学文献中收集了1572条含有CHAP结构域的抗生酶,对这些抗生酶进行了详细注释和分类,包括蛋白质名称、来源、抗菌活性数据、CHAP结构域长度、结构和抗菌活性等信息,并建立了基于Web的数据库,可以直接从浏览器访问和检索所有数据。这些信息不仅可以用作已报道的含有CHAP结构域抗生酶之间的比较,也可以对新型CHAP结构域进行预测分析。
1 材料及方法
1.1 数据收集
首先通过关键词("CHAP","enzybiotic","lysin","lysozyme")检索PubMed中包含CHAP抗生酶实验的文献,然后从文献中抽取CHAP抗生酶信息,同时对UniProtKB蛋白数据库检索CHAP结构域蛋白,得到初步的CHAP抗生酶数据,共计6416条,留作数据整理。
1.2 数据整理与数据库设计
对手动收集的文献与数据库中的CHAP抗生酶信息进行数据整合,删除冗余与不确定信息,参考对应的文献与数据库,最终本研究获得了1572条含有CHAP结构域的抗生酶。对这些CHAP抗生酶进行信息关联和信息注释,信息来源于UniProtKB、InterPro、PDB和GO等公共数据库。同时根据收集到的CHAP抗生酶蛋白序列,运用BioPerl程序包计算CHAP抗生酶的物化性质参数,包括序列长度、分子量、电荷量、pI值等信息。整理后的CHAP抗生酶信息主要包含基本信息、活性信息、序列信息、结构信息和文献信息。据此进行对应的关系表设计,并在MySQL数据库中将其实现。目前数据库中数据收集的截止日期为2018年5月30日。
1.3 Web接口与功能开发
CHAP抗生酶数据库构建在一个运行WAMP架构的64位的Windows (2008 R2)虚拟服务器上。服务器整合了Apache HTTP Server (V2.4.23) Web服务器和PHP (V5.4.45)以及MySQL Server (V5.5.20)数据库服务器。所有数据保存在MySQL数据库中。服务器端程序主要使用PHP语言编写,部分功能使用Perl语言编写,通过PHP进行功能调用。前端使用jQuery JavaScript Library (V1.6.2)和Highchart jQuery插件以及Cascading Style Sheets (CSS)技术实现。Apache、MySQL、PHP、Perl和jQuery都是开源软件且都是平台独立的技术,非常适合用于学术研究。
2 实验结果
2.1 CHAP抗生酶数据收集与整理结果
从数据库和文献信息中收集了1572条CHAP抗生酶,包含1394条来自251种细菌的CHAP抗生酶和178个来自12种噬菌体的抗生酶。数据库对CHAP抗生酶的基本信息、活性信息、序列信息、结构信息和文献信息进行了详细描述。基本信息主要包含来源、功能、物化性质、数据库链接等,结构信息包含结构域信息和三维结构信息。
2.2 数据库设计结果
当前CHAP抗生酶数据库主要由5个MySql关系表组成,分别是Chap、Sequence、Activity、Reference、Domain和Dom2Go表,这些表之间的关系如图1所示。Chap表主要保存CHAP抗生酶的基本信息,其中Reference表保存Chap抗生酶相关的文献信息,Sequence表和Activity表分别保存CHAP抗生酶的蛋白序列信息和抗菌活性信息,此外,Domain表存储CHAP抗生酶的结构域信息,而衍生出的Dom2Go表则是保存结构域对应的GO注释信息。从关系表模式图(图1)可以清晰了解整个数据库的构架。

图1 CHAP抗生酶数据库关系表模式图Fig.1 Schema of the CHAP enzybiotic database
2.3 数据库Web接口
数据库Web接口如图2所示。接口主要包含浏览(Browse)、搜索(Search)、统计信息(Statistical Info) 和指南(Guide)。通过浏览接口用户可以浏览数据库的所有CHAP抗生酶数据,并且可以在此页面下载FASTA格式的所有CHAP抗生酶数据,方便用户进行独立的数据分析和设计研究。搜索接口主要包含快速搜索和高级搜索。用户可以使用关键词进行快速搜索,也可以利用高级搜索功能针对蛋白名称、来源和靶向目标等进行单一或者组合搜索。数据库提供了数据查询功能,浏览和搜索结果显示的条目可以对应数据库中的每个CHAP抗生酶,每个CHAP抗生酶都有唯一的数据库ID,点击ID可以链接到这一CHAP抗生酶的详情页面(图2),在详细信息中记录了该抗生酶的基本信息、结构域信息、GO注释信息、3D结构信息、序列信息、活性信息和文献信息。同时还提供了评论接口供用户对其进行注释和交流。

图2 CHAP数据库Web接口截图Fig.2 Web interfaces of CHAP enzybiotic database
2.4 数据库中信息分析
2.4.1 抗生酶来源统计
CHAP抗生酶数据库收集了1572条CHAP抗生酶,其中1394条CHAP抗生酶来自251种细菌。细菌来源统计如图3所示,排名前5的细菌来源占数据库数据的36.3%(570/1572)。其中大部分来自于革兰阳性菌链球菌属(Streptococcus)、葡萄球菌属(Staphylococcus)和乳球菌属(Lactococcus),占数据库数据的27.3%(429/1572)。来自于革兰阴性菌的CHAP抗生酶主要来自于双歧杆菌属(Bifidobacterium)和埃希菌属(Escherichia),占数据库数据的9.0%(141/1572)。
对178个来自12种噬菌体的CHAP抗生酶产生的噬菌体种属的统计(图4)发现,CHAP抗生酶主要来自于葡萄球菌噬菌体(Staphylococcusphage),占数据库数据的8.8%(139/1572),其次是链球菌噬菌体(Streptococcusphage),占数据库数据的1.5%(24/1572)。
2.4.2 活性统计
对活性数据的分析表明,CHAP抗生酶主要对革兰阳性菌有活性,包括葡萄球菌属、链球菌属、乳球菌属等。对革兰阴性菌也有活性,包括军团菌属、铜绿假单胞菌属和沙门菌属等。

图3 CHAP抗生酶的细菌来源Fig.3 Bacterial source of CHAP enzybiotics
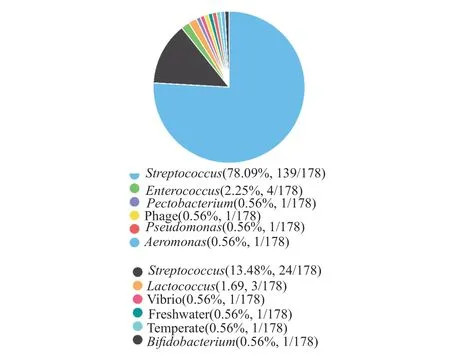
图4 CHAP抗生酶的噬菌体来源Fig.4 Bacteriophage source of CHAP enzybiotics
2.4.3 物化性质统计
本数据库中的CHAP抗生酶的序列在43~1819个氨基酸长度,最短的CHAP抗生酶SLE-1仅仅有43个氨基酸,仅有1个CHAP结构域构成,而最长的CHAP抗生酶Antireceptor有1819个氨基酸,来自于链球菌噬菌体,由2个催化域组成。大部分(1330/1572,84.6%)的CHAP抗生酶分子量介于20~80kDa之间(图5),与抗菌肽相比,分子量相对还是较大的。对CHAP抗生酶等电点的统计分析(图6)显示,数据库中绝大多数(1344/1572,85.5%)CHAP抗生酶等电点位于4~11之间,同时也有2个CHAP抗生酶的等电点特别低(<1),4个特别高(>13),是否具有特别的意义,值得深入研究。
3 结论和讨论

图5 CHAP抗生酶的分子量分布Fig.5 Distribution of molecular weight for the CHAP enzybiotics
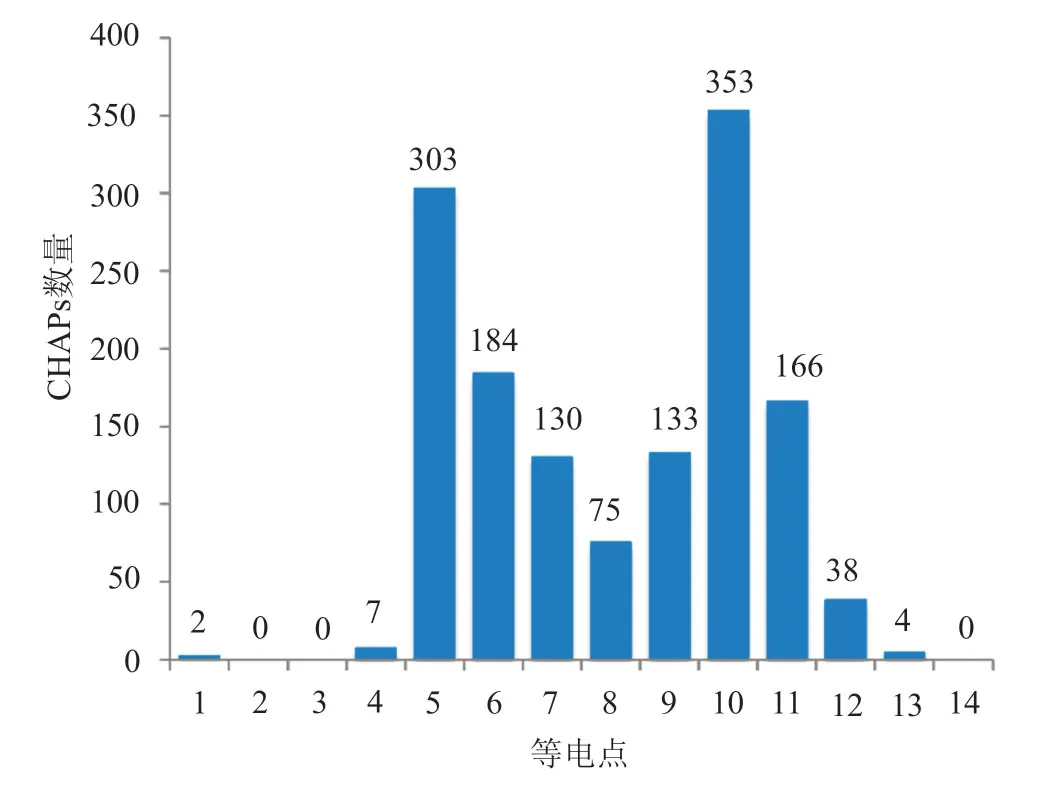
图6 CHAP抗生酶的等电点分布Fig.6 Distribution of isoelectric points for the CHAP enzybiotics
随着细菌耐药问题的日益严峻,寻找具有全新作用机制的抗菌物质显得十分重要。抗生酶和抗菌肽就是其中比较重要的两种。当前国内外已经有多种抗生酶和抗菌肽在进行治疗耐药菌感染相关的临床试验[13-14]。国内上海高科联合生物技术研发有限公司开发的用于治疗烧伤创面金黄色葡萄球菌感染的溶葡萄球菌酶涂剂已经完成III期临床试验[15]。建立相关的抗生酶数据库有助于推动此领域的研究,开发更多的抗生酶用于耐药菌感染的治疗。
目前,针对抗菌肽已建立了非常多的数据库,例如APD[16-18]、ANTIMIC[19]、AMPer[20]、CAMP[21-22]、DAMPD[23]、YADAMP[24]、LAMP[25]、BaAMPS[26]和DRAMP[27]。它们含有许多来自不同来源或特定家族的抗菌肽序列,对抗菌肽的研究提供了巨大的帮助。但对抗生酶进行数据库分类则相对较少,仅有2012年的EnzyBase[10]、2013年的phiBIOTICS[11]和2014年的GMEnzy数据库[12]。其中phiBIOTICS是一个仅针对治疗性抗生酶的数据库,只有29个抗生酶及其69项相关研究信息,数据相对较少;EnzyBase最后更新于2012年,收录了216种来源的1144种抗生酶。GMEnzy收集了157种遗传修饰的抗生酶。与这些数据库相比,本研究构建的数据库重点研究含有CHAP结构域的抗生酶,针对含有这一类独有结构的抗生酶进行系统分析整理。其它抗生酶数据库均未对CHAP结构域这一广泛存在于抗生酶中的活性结构域进行描述和分析,本数据库收录了目前已有报道的含有CHAP结构域的抗生酶信息。
CHAP抗生酶数据库可以帮助科研人员探索CHAP抗生酶的使用和设计新的抗生酶。通过数据库,可以对所有含有CHAP结构域的抗生酶进行进化分析,了解不同抗生酶的进化关系,对CHAP结构域的差异性和共同性以及它们的抗菌活性的关系研究有着重要的意义。建立数据库可以将目前零散的含有CHAP结构域的抗生酶的研究系统化,用户可以迅速地查找相关的理化性质并进行比较,可以快速找到针对一类或多类特定病原体的CHAP抗生酶,并对它们的活性和作用条件进行筛选。并且本文的数据库也可以为目前热门的嵌合体抗生酶[28]的研究提供极大的帮助。通过比较和筛选针对特定病原菌的CHAP结构域,可以为研究人员构建嵌合体抗生酶提供新的思路。CHAP抗生酶数据库的构建将增加研究这些生物活性蛋白的效率和便利性。
对于潜在药物来说,活性与毒性信息同样重要。目前本文的CHAP抗生酶数据库,活性数据并不全面,并且没有包含细胞毒性的相关数据。在未来的工作中,本文将专注于收集完善CHAP抗生酶的活性数据以及细胞毒性信息,将数据库进一步整合更新,以便更准确地对CHAP抗生酶进行评估。另外,由于目前关于CHAP抗生酶的结构研究较少,结构信息非常缺乏,我们计划将一些结构预测和分析工具整合入数据库中,以便研究者更好地利用CHAP抗生酶数据库进行CHAP抗生酶设计和结构功能领域的探索。
致谢:上海高科联合生物技术研发有限公司对本研究工作的支持。
