高效液相色谱法提高红曲细菌分离与分析的效果
2019-07-30邱丹腾张海玲刘树滔林士森陈玲琳饶平凡
邱丹腾 张海玲 刘树滔* 林士森 陈玲琳 饶平凡
(1 福州大学生物工程研究所 福州350108 2 中国科学院上海生命科学研究院-浙江工商大学食品营养科学联合研究中心 杭州310035)
传统的微生物分离、 分析方法是建立在纯培养技术上发展起来的,如平板涂布法、平板划线分离法以及稀释混合平板法。 然而这些传统方法不但分离周期长、操作繁琐,而且难以得到绝对的纯种,并且对于难培养和不可培养的微生物而言,这些方法并不适用[1-2]。 随着分子生物学技术的快速发展,克服了传统培养方法的限制,可以从基因的水平定量分离、分析微生物,主要包括免疫磁性颗粒分离技术[3-4]、场流分离技术[5]、实时荧光定量PCR 技术[6-7]以及PCR-DGGE 技术[8-10]等,这些技术具有高效、快速、操作简单等优点,能够大大提高微生物分离、 分析的效率, 缩短研究周期[11]。PCR-DGGE 技术是近年来国内外应用较为广泛的分子生物学技术之一, 它不仅可以通过对电泳图谱的观察来判定微生态系统的多样性和相似性,而且还可以通过对差异条带进行胶回收、克隆和测序完成定性分析。 然而DGGE 技术存在一定的局限性,它只能对单条带进行测序分析,无法完全将有序列差异的DNA 片段分开,从而出现不同序列DNA 的共迁移现象,给条带回收和测序带来困难[11-13]。 条带回收及其测序过程不仅试验周期长,还费力耗材。分子生物学技术普遍存在干扰成分多, 检测灵敏度和准确性受限以及无法回收菌体细胞,无法分析菌体表面特性,检测样品制备繁琐等问题。
高效液相色谱技术是微生物分离、 分析和制备方法的一种创新性选择。细菌作为带电颗粒,具有不同的表面带电特性, 其细胞表面的羧基、氨基、 磷酸等基团在不同的pH 条件下会发生电离,细菌可以像生物大分子一样, 利用离子交换色谱技术进行分离[14-15]。高效液相色谱技术作为一种快速、灵敏的崭新手段已应用于青枯菌、金黄色葡萄球菌、 致泻性大肠杆菌以及动物肠道菌群等的初步分析,该方法不仅能以细菌细胞的种类、鞭毛长短、致病力的强弱而分离成不同的色谱组分,还能将不同生长阶段的细菌细胞分离[16-20]。
本文选取红曲作为模式体系, 通过红曲细菌样品制备条件优化, 应用HPLC 技术分离红曲生产菌株, 回收菌体细胞并结合PCR-DGGE 技术,利用细菌16S rDNA 对色谱分离得到的菌株进行鉴定。 探讨HPLC 技术在红曲细菌分离、分析中的应用潜力,为复杂微生物体系的分离、分析提供新思路, 为发酵食品生产菌株的筛选和制备提供新方法。
1 材料与方法
1.1 仪器与材料
高效液相色谱仪(DL2000),日本Hitachi 公司;高速离心机(CF15RXⅡ),日本Hitachi 公司;PCR 仪 (S1000TMThermalCycler), 美国BIO-RAD公司;变性梯度凝胶电泳仪(BIO-RAD DCodeTM),美国BIO-RAD 公司;pH 计(FE20),梅特勒-托利多(上海)有限公司;凝胶成像分析仪(JS-380A),上海培清科技有限公司; 光学显微镜(BM1000),南京江南永新光学仪器有限公司。
SuperQ-650C 强阴离子交换树脂(TSKgel SuperQ-TOYOPEARL 650C),日本TOSOH 公司。
平衡缓冲液 (A 液):20 mmol/L 哌嗪-盐酸缓冲液(pH 8.0);洗脱缓冲液(B 液):20 mmol/L 哌嗪-盐酸缓冲液(pH 8.0)+1 mol/L NaCl。
1.2 试验方法
1.2.1 红曲细菌样品的制备 称取10.0 g 红曲粉末于90 mL A 液中,搅拌30 min 后用灭菌的两层纱布过滤, 去除大颗粒物质。 滤液400×g 离心2 min 去除中等大小杂质, 上清经5 μm 滤膜抽滤后,将滤液12 000×g 离心10 min,收集沉淀,缓冲液洗涤3 次后重悬于10 mL 样品处理液中, 即为红曲细菌样品。
1.2.2 红曲细菌的HPLC 分析 色谱系统: 采用日本Tosch 公司的强阴离子交换树脂TSKgel SuperQ-TOYOPEARL 650C 色谱柱 (200 mm×4.6 mm i.d);流速1 mL/min;泵压范围0.5~1.5 MPa;温度范围23~28 ℃。
洗脱方法:红曲细菌样品进样后,0~10 min 用A 液平衡,使样品中的细菌充分吸附到色谱柱上;10~20 min 进行0~25%的B 液线性梯度洗脱;20~40 min 进行25%~50%的B 液线性梯度洗脱;40~80 min 进行50%~100%的B 液线性梯度洗脱;80~100 min 完全用B 液洗脱,将残留在树脂上的细菌全部洗脱下来。
检测条件: 采用HPLC 紫外检测池对通过色谱柱的细菌进行在线检测,检测波长260 nm。
1.2.3 洗脱组分的镜检 采用灭菌的试管重复多次收集洗脱峰, 分别于4 ℃下12 000×g 离心5 min,取少许沉淀涂片镜检,采用结晶紫染色法,在油镜(100X)下观察。
1.2.4 洗脱组分的DGGE 分析 洗脱组分基因组DNA 的提取: 使用Bioteck 公司土壤基因组DNA快速提取试剂盒的操作步骤对细菌DNA 进行提取, 并采用1.0%琼脂糖凝胶电泳对基因组DNA的提取效果进行验证。
洗脱组分16S rDNA 基因V3 区的巢式PCR扩增:首先,以提取的基因组DNA 为模板,采用通用引物27F (5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-GGCTACCTTGTTACGACTT-3’)对细菌16S rDNA 进行扩增。 PCR 反应体系(50 μL):PCR Mix 25 μL,模板1 μL,引物各1 μL,去离子水22 μL。PCR 反应程序:94 ℃预变性4 min;接着30 个循环:94 ℃变性30 s,55 ℃退火40 s,72 ℃延伸1 min 30 s;最后72 ℃终延伸10 min。
接着,以扩增的16S rDNA 为模板,采用通用引物341F-GC (5’-CGC CCG CCG CGC GCG CGG CGG GCG GGG CGG GGG CAC GGG GGG CCT ACG GGA GGC AGC AG-3’)和518R (5’-ATT ACC GCG GCT GCT GG-3’)对V3 可变区进行扩增。 PCR 反应体系 (25 μL):PCR Mix 12.5 μL,模板0.5 μL,引物各0.5 μL,去离子水11 μL。PCR 反应程序:95 ℃预变性5 min;接着30 个循环:94 ℃变性30 s,61 ℃退火30 s,72 ℃延伸30 s;最后72 ℃终延伸10 min。 以上产物均用1.0%的琼脂糖凝胶电泳检测。
洗脱组分的DGGE 电泳: 采用BIO-RAD 公司的DCodeTM 基因突变检测系统对V3 区PCR产物进行检测: 使用梯度制胶器制备变形梯度30%~60%的8%的聚丙烯酰胺凝胶。 待胶完全凝固后, 将胶板安装进60 ℃缓冲液的电泳槽内,分别加入8 μL 的PCR 产物。电泳条件为200 V、6.5 h、60 ℃。 电泳完毕后,用EB 染色,清水漂洗,置于凝胶成像系统拍照分析。
1.2.5 洗脱组分的16S rDNA 测序及系统发育学分析 将DGGE 图谱单一条带对应的洗脱峰细菌16S rDNA 送上海生工生物技术有限公司测序,测得的序列与GeneBank 数据库做相似性分析,并采用MEGA5.1 软件中的Neighbor-Joining 方法构建进化树。
2 结果与讨论
2.1 红曲细菌的色谱分析
本研究采用HPLC 方法对红曲细菌进行分离、分析。 试验结果如图1 所示,紫外检测图谱上共出现5 个明显的洗脱峰, 分别为P1、P2、P3、P4、P5。 根据洗脱时间的不同,对各洗脱峰进行重复多次收集。 260 nm 紫外检测波长下,除了核酸有吸收外,其余组分也有响应值,例如细菌表面脱落蛋白、表面蛋白以及表面多糖等。 因此,为了确认洗脱峰是否为细菌组分, 需要进一步做相关的细菌鉴定分析。
2.2 洗脱组分的确认
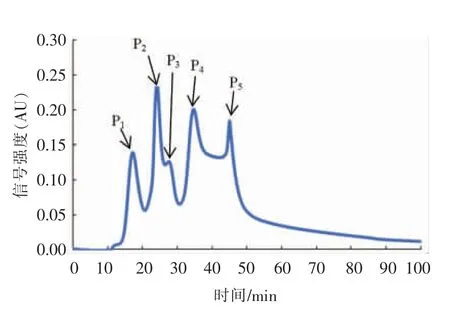
图1 红曲细菌的HPLC 色谱图Fig.1 Chromatograms of the bacteria obtained from Red Kojic Rice
为了确认收集到的洗脱组分是否为细菌,对5 个洗脱峰进行显微镜检查,结果如图2 所示,表明收集到的各洗脱峰组分为细菌, 包括杆菌和球菌。
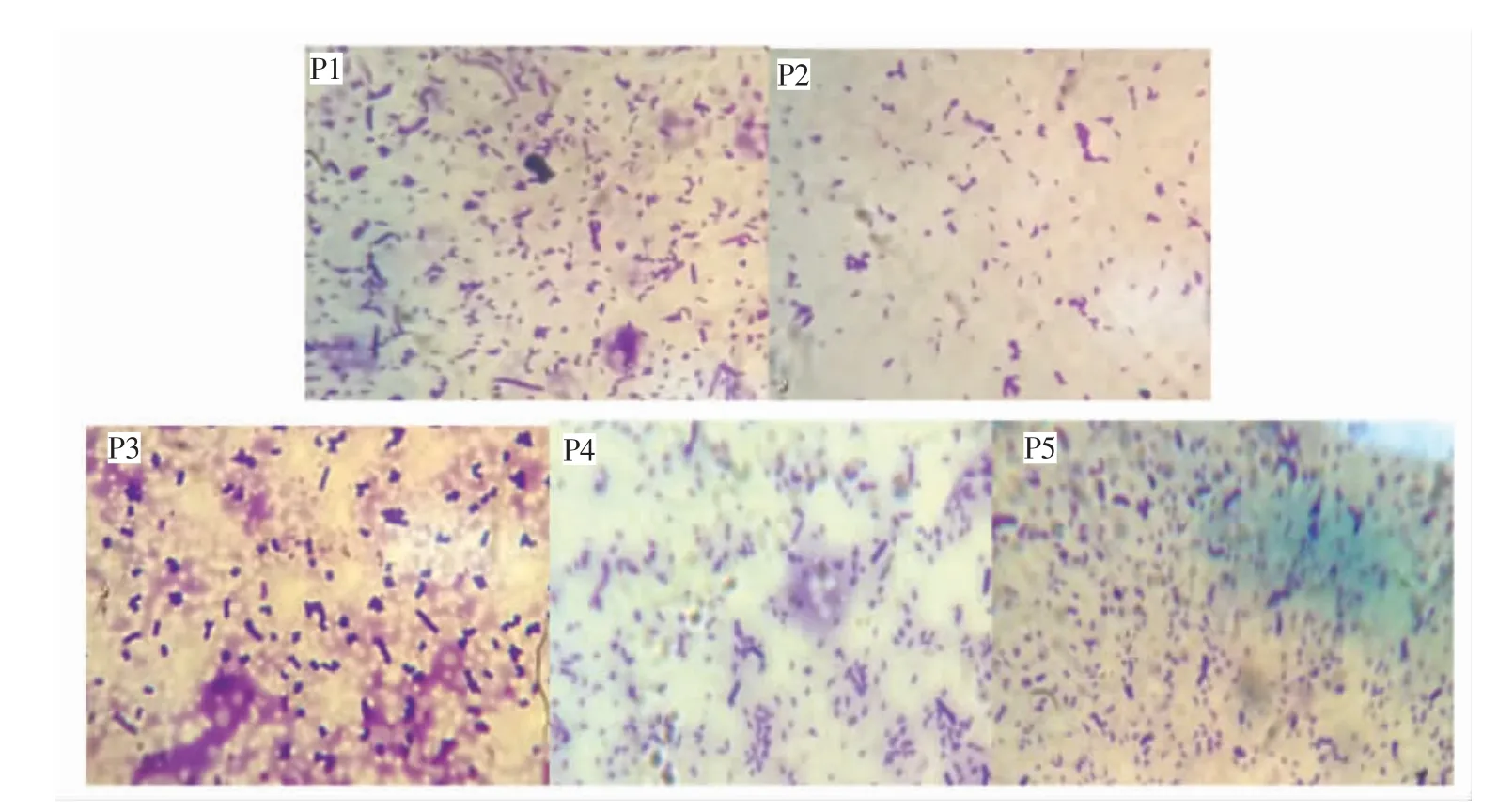
图2 洗脱组分镜检图Fig.2 Microscope graph of the eluted fractions
2.3 洗脱组分的DGGE 分析
2.3.1 基因组DNA 的提取 提取的各洗脱峰的细菌基因组DNA, 在1.0%琼脂糖凝胶电泳上进行检测,电泳结果如图3,表明提取的基因组DNA 纯度较好,浓度较高,可以作为PCR 扩增模板。
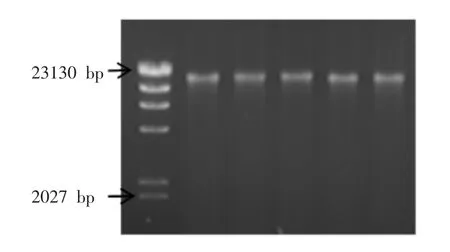
图3 细菌峰基因组DNA 电泳图Fig.3 Agarose gel electrophoresis (1.0%)of genome DNA of eluted fractions
2.3.2 16S rDNA 可变区V3 的巢式PCR 扩增 分别采用细菌通用引物27F 和1492R 对各洗脱峰进行细菌16S rDNA 的扩增,341F-GC 和518R 对其中V3 可变区进行扩增。电泳结果见图4、图5,可以看出,在1 500 bp 位置附近有明亮的特异性条带,符合16S rDNA 基因片段大小,表明16S rDNA 扩增效果较好;以该16S rDNA 为模板,扩增的V3可变区产物,其电泳条带在250 bp 附近,符合目标片段大小且不存在非特异性条带, 可作为DGGE 分析的样品。
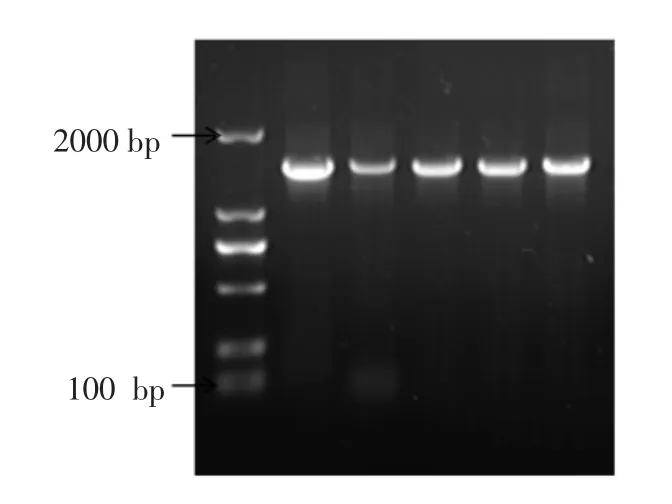
图4 细菌峰16S rDNA PCR 产物电泳图Fig.4 Agarose gel electrophoresis (1.0%)of 16S rDNA PCR products
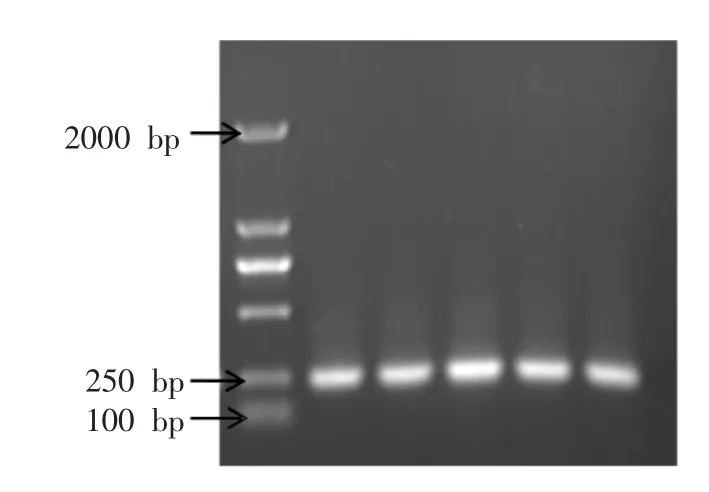
图5 洗脱峰V3 可变区PCR 产物电泳图Fig.5 Agarose gel electrophoresis (1.0%)of V3 region PCR products
2.3.3 DGGE 电泳分析 理论上认为,DGGE 技术最低可检测到只有1 个碱基差异的DNA 片段[10],DGGE 图谱上的每个条带就很可能代表一种微生物,基于该原因,可以通过DGGE 图谱检验高效液相色谱对红曲细菌的分离效果。
对各洗脱峰16S rDNA V3 可变区进行DGGE 分析,结果如图6 所示。根据DGGE 原理,5个洗脱峰中有3 个可能为单菌,分别是:P1、P2、P5,说明HPLC 技术能够实现复杂细菌样品的纯菌分离。
2.4 洗脱组分的测序及系统发育学分析
将DGGE 图谱中单条带对应的3 个洗脱峰的16S rDNA 送往上海生工生物技术有限公司测序,均测序成功。测序得到的3 个16S rDNA 序列输入NCBI 数据库中,利用Blastn 进行比对分析,结果见表1。鉴定结果显示,P1的相似菌株为脂肪酸芽孢杆菌(Alicyclobacillus)和人参土芽孢杆菌(Bacillus ginsengihumi),P2的相似菌株为假单胞菌属 (Pseudomonas sp),P5的相似菌株为环状芽孢杆菌(Bacillus circulans)。
为了显示各菌株的系统发育关系和分类学地位, 选取其中的相似菌株脂肪酸芽孢杆菌(KC893647.1)、人参土芽孢杆菌(NR_041378.1)、假单胞菌 (KT380612.1)、 环状芽孢杆菌(KT720033.1)以及细菌峰菌株, 用MEGA5.1 的Neighbor-Joining 法构建进化树,结果如图7 所示。
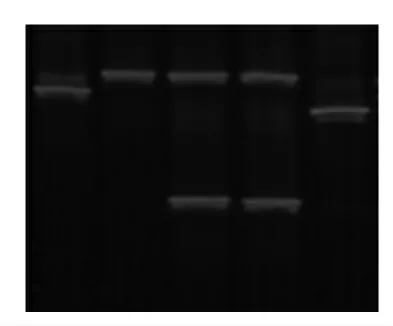
图6 细菌峰V3 区DGGE 电泳图Fig.6 DGGE pattern of PCR products of the V3 region obtained from eluted fractions
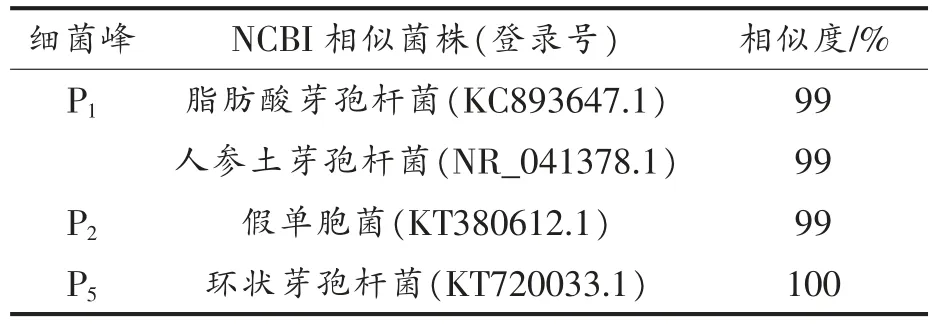
表1 红曲细菌高效液相色谱峰的鉴定结果表Table1 Phylogenetic identification results of bacteria peaks
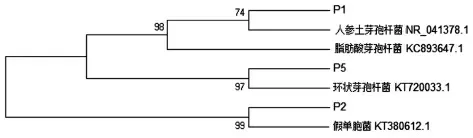
图7 基于16S rDNA 序列构建进化树Fig.7 Phylogenetic tree based on the complete 16S rDNA sequences of 3 bacterial strains obtained and 4 related strains
从系统进化树可以看出, 得到的细菌峰菌株与数据库相似菌株可分为3 个分支:P1与脂肪酸芽孢杆菌和人参土芽孢杆菌聚为一支,P2归类于假单胞菌,P5归类于环状芽孢杆菌。
3 结论
采用PCR-DGGE 技术对高效液相色谱的5个细菌峰的16S rDNA V3 可变区进行分析,其中3 个洗脱峰为单条带。DGGE 单一条带对应样品的16S rDNA 序列经NCBI 序列同源性分析和进化树构建分析,结果表明,高效液相色谱分离得到的3 个洗脱峰与数据库中序列同源性均达到99%及以上, 分析可知得到的细菌峰菌株属于芽孢杆菌属和假单胞菌属, 包括: 脂肪酸芽孢杆菌(Alicyclobacillus)、人参土芽孢杆菌(Bacillus ginsengihumi)、环状芽孢杆菌(Bacillus circulans)和假单胞菌(Pseudomonas sp),说明了HPLC 技术能够作为一种快速、 简便的方法从复杂细菌体系中分离出单菌。
HPLC 技术在红曲细菌分离、分析方面显示出很大的创新性和优越性。 通过HPLC 与PCRDGGE 技术相结合的分析方法, 能够实现色谱分离细菌16S rDNA 全长序列的直接测序, 无需进行胶回收。 与传统PCR-DGGE 技术相比,具有鉴定序列较长、准确性高、试验周期短、样品可回收等优点; 克服了现有分子生物学技术在对微生物进行分离、分析时存在准确性受限、菌体细胞无法回收、样品制备困难以及试验周期长等缺点,为复杂微生物体系的分离、分析提供新的思路。
下一代测序技术的不断进展以及价格不断下降,对深入分析复杂微生态体系具有重要的意义。但无论是现有的分子生物学技术还是下一代测序技术都是对分析样品中的DNA 分子进行分析,其分析结果直接受制于微生物样本DNA 提取和PCR 效果,具备理想的纯度、浓度和完整性样品的总DNA 的提取是研究的前提条件和关键。 所以在下一步研究中, 本课题组将继续探讨液相色谱技术在高质量DNA 提取,从而辅助高通量测序技术分离、 分析肠道菌群等复杂微生态系统的应用潜力。
