糖尿病肾病芯片数据挖掘及生物信息学分析
2019-01-14闫瑞娟臧雯雯
闫瑞娟 臧雯雯
(1.泰山医学院,山东 泰安 271016; 2.山东省立第三医院,山东 济南 250000;3.泰山医学院附属聊城医院,山东 聊城 252000)
糖尿病肾病是糖尿病的主要并发症之一,最终会导致终末期肾病[1]。目前临床上尚缺乏灵敏度高的诊断糖尿病肾病的早期标志物。因此,通过筛选糖尿病肾病患者肾脏组织的差异表达基因来挖掘灵敏度高的标志物,对于糖尿病肾病的诊断和治疗具有重要意义。
随着基因芯片技术的发展和应用,产生了许多基因表达谱数据,如何挖掘有价值的数据信息,已成为生物信息学的研究热点[2-3]。生物信息学的发展也为糖尿病肾病的研究提供了新的思路,本研究对来自GEO数据库的糖尿病肾病组织的基因芯片进行分析,筛选出核心基因和重要的信号通路,构建差异基因的互作网络,这些核心基因和信号通路可为糖尿病肾病的研究提供新思路。
1 材料与方法
1.1 芯片数据的选择及数据标准化处理
在GEO数据库中以“diabetic nephropathy”为检索词,可获得多组与糖尿病肾病相关的表达谱数据,本研究将GEO数据库中编号为GSE30529的芯片表达谱数据纳入研究对象。该芯片是含有22个样本的人肾小管基因表达的数据集,包含正常肾小管组织12个样本和糖尿病肾病肾小管组织10个样本。在线GEO2R工具用于分析和查找差异基因,以|logFC|>2和P<0.05对数据进行标准化处理后筛选差异基因。
1.2 差异基因的GO富集分析和KEGG通路分析
生物学信息注释数据库(DAVID)是一个在线生物信息学资源网站,可以为大规模基因或蛋白质进行系统综合的生物功能注释信息,找出最显著富集的生物学注释。本研究将筛选出的差异基因上传至DAVID在线软件后,确定对应的种属,选择DAVID软件中的基因功能注释(functional annotation),即可得到差异基因分别参与的生物过程、分子功能,细胞组成和生物学信号通路分析(KEGG)。
1.3 差异蛋白互作网络分析
蛋白质互作关系数据库(STRING)包含2031个组织的9643763种蛋白质,是最大的蛋白质数据库之一,通过数据库我们可以直接或间接的预测蛋白质间的相互关系。将筛选出的差异基因导入STRING在线软件后,导出蛋白互作网络的数据信息,cytoscape软件可将导入的数据信息转换为直观的蛋白互作网络图,分析网络内每个差异蛋白的连通度,连通度高的即为核心基因。同时,以degree cutoff = 2, node score cutoff = 0.2, k-core = 2, and max. depth = 100为标准,利用cytoscape内的MCODE软件对互作网络进行评分,可得出评分表较高的蛋白互作网络,找出在关键点的蛋白质。
2 结 果
2.1 差异表达基因及构建互作网络
GSE30529数据集中总的差异表达基因为4280个,其中下调2620个,上调1660个。对数据进行标准化处理后筛选出差异比较大的基因98个,其中包含12个下调的基因和86个上调的基因,和9个核心基因(表1),其中包括CD44、PTPRC、EGF、FN1、SPARC、VIM、KNG1、TIMP1、CXCR4。
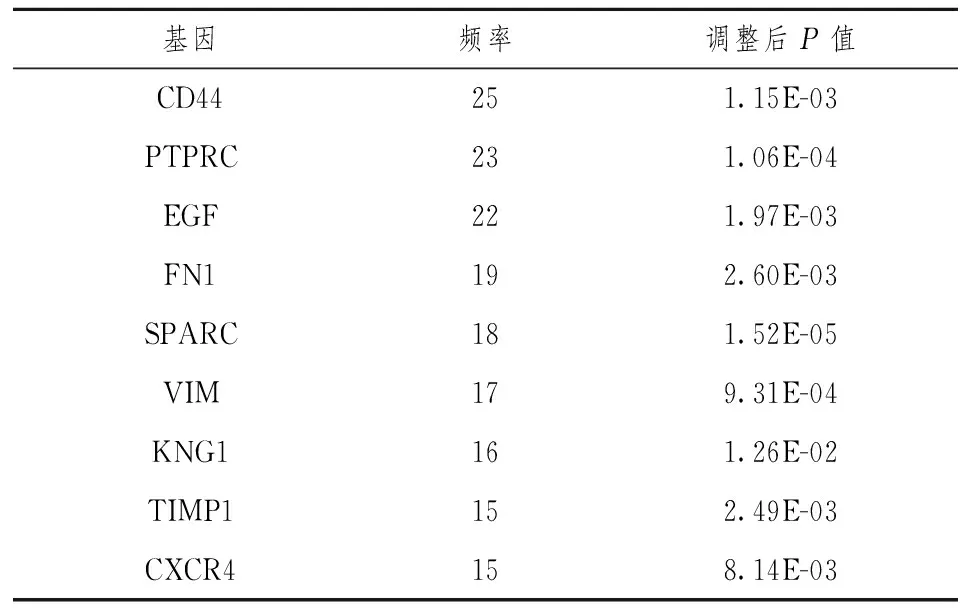
表1 连接度比较高的9个核心基因
2.2 差异基因的GO富集分析和KEGG信号通路分析
用DAVID软件对差异蛋白进行GO富集分析和KEGG信号通路富集分析,GO富集分析显示这些差异表达基因主要位于细胞外基质区域和细胞质膜的构成,主要的分子功能是蛋白结合、胶原蛋白结合和受体结合等,主要参与炎症反应、细胞黏附反应等生物过程(表2)。KEGG通路分析显示这些差异基因主要参与PI3K/Akt信号通路、ECM受体相互作用、细胞黏附和趋化因子信号转导通路等(表3)。

表2 差异基因的GO富集分析列表
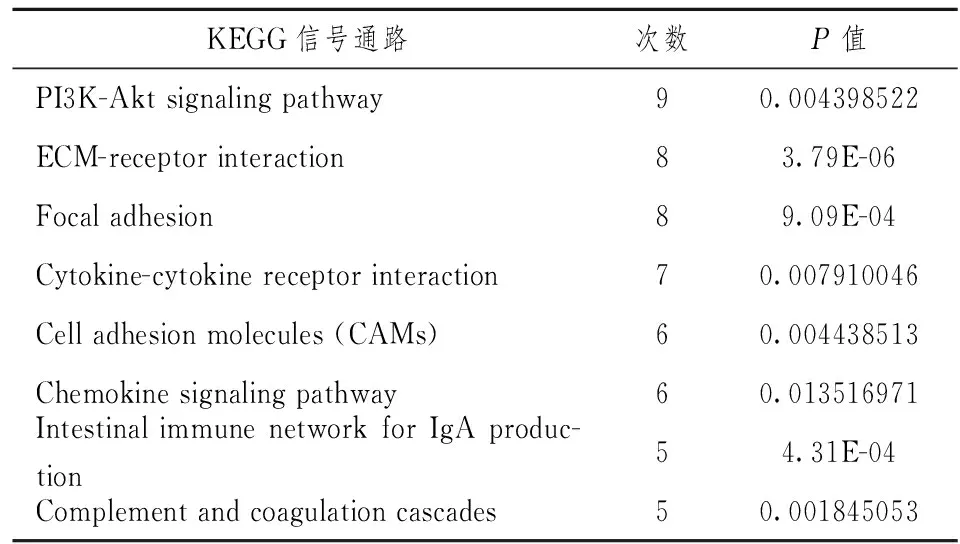
表3 差异基因的KEGG通路分析列表
2.3 差异基因编码蛋白的互作网络分析
通过STRING在线工具和cytoscape软件对98个差异基因进行蛋白质相互作用网络分析(被STRING识别的基因有94个)(图1),在总的蛋白网络中以degree cutoff = 2, node score cutoff = 0.2, k-core = 2, and max. depth = 100为标准,利用cytoscape内的MCODE软件对互作网络进行评分,找出蛋白质相互作用网络中的主要节点,其中核心基因PTPRC、EGF、FN1、SPARC、VIM、KNG1、TIMP1、CXCR4是这些节点的重要组成部分。
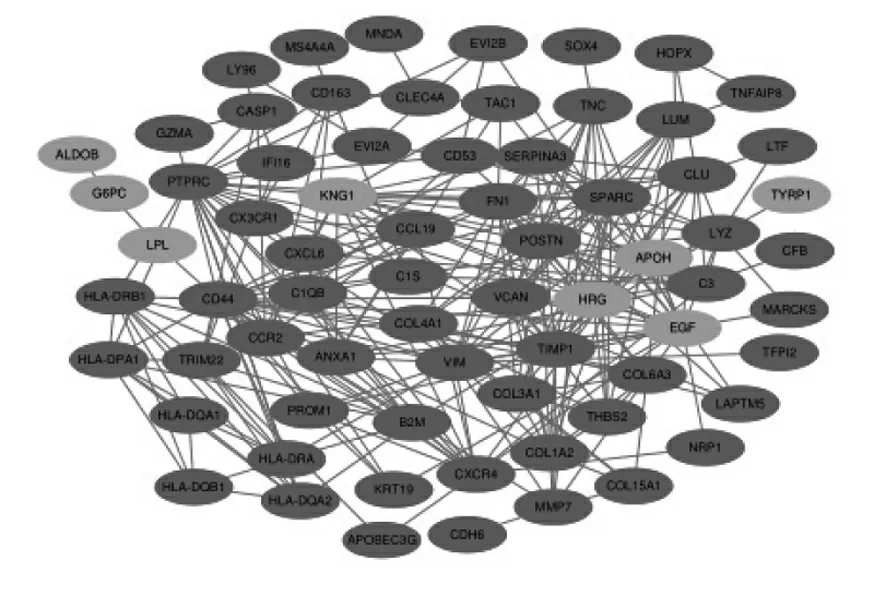
蓝色:表达量下降的基因,红色:表达量上升的基因
图1 用STRING在线分析工具和cytoscape软件对
98个差异基因进行蛋白质相互作用网络分析
3 讨 论
糖尿病肾病是糖尿病最常见的微血管并发症,并且是全球终末期肾病的主要原因之一[4],糖尿病肾病的分子机制仍不清楚,临床上亦缺乏有效的治疗靶点。糖尿病肾病是多基因相互作用的结果,基因表达谱的大量应用,为本研究提供了生物信息学方面研究糖尿病肾病发病机制的可能性,对基因芯片数据进行进一步的生物学分析,是对基因芯片数据挖掘的一个重要方向。本研究利用生物信息学软件对芯片GSE30529进行分析,共发现98个差异表达基因,并对这些差异基因进行GO富集分析、KEGG信号通路分析和蛋白质互作网络分析,筛选出了9个与糖尿病肾病相关的蛋白,其中表达量下调的蛋白有EGF、KNG1,表达量上调的蛋白有PTPRC、FN1、SPARC、VIM、TIMP1、CXCR4。核心蛋白中PTPRC、EGF、FN1、SPARC、VIM、KNG1、TIMP1、CXCR4同时处于蛋白互作网络的核心位点,这些蛋白可能成为早期诊断糖尿病肾病的指标。
对这些核心基因深入研究,表皮生长因子(epidermal growth factor,EGF)通过与表皮生长因子受体结合参与多种生物过程的调控,包括上皮细胞增殖、代谢、分化、存活和组织损伤与修复。越来越多证据表明,EGF参与多种肾损伤,肾衰竭患者中EGF明显降低[5],肾脏慢性肾损伤时,表皮生长因子受体信号通路的激活可以诱导TGF-β信号通路,诱导上皮细胞细胞周期阻滞于G2/M期,同时产生促炎症因子和趋化因子[6]。激肽原1(KNG1,kininogen 1)在激肽原酶的作用下释放激肽,激活激肽系统,改善肾脏微循环,芯片结果显示糖尿病肾病组中KNG1的表达明显下降。尚未有文献直接指出KNG1在糖尿病肾病中的作用机制,但是Nathalie Vionnet等人[7]指出KNG1参与血管并发症的病理生理过程,亦可作为糖尿病肾病的潜在靶点。受体型酪氨酸蛋白磷酸酶C(receptor-type tyrosine-protein phosphatase C,PTPRC)参与自然杀伤细胞介导的细胞毒性的反应、趋化因子信号通路,白细胞迁移,T细胞受体信号转导和细胞因子间受体相互作用等炎症反应[8]。SPARC是一种广泛表达的促纤维化蛋白,通过调节系膜细胞转化生长因子(TGF-β1)和一型胶原的产生,抑制系膜细胞增殖和影响细胞外基质的产生。Taneda S等[9]实验证明,糖尿病肾病小鼠模型中,SPARC在高糖状态下通过促进TGF-β1表达来导致肾小球硬化和肾小管间质损伤。fibronectin 1 (FN1)和VIM是上皮间质转化的主要蛋白,具有间质细胞的属性,是肾小管上皮细胞向间质转化的代表性蛋白[10]。CXCR4是一种G蛋白偶联受体,可以与趋化因子SDF-1结合,CXCR4/CXCR7/SDF-1信号通路参与调解肾脏生长和肾脏固有细胞对外界刺激的病理反应[11]。
筛选出来的9个核心蛋白质主要是以PI3K-Akt信号通路为主线,通过调节细胞间黏附因子及趋化因子等因子的释放和上皮间质转化等过程参与糖尿病肾病的发展。通过筛选出的蛋白功能可以看出,细胞黏附导致的微血管病变和细胞外基质的沉积为糖尿病肾病的主要机制。
