用全基因组关联作图和共表达网络分析鉴定油菜种子硫苷含量的候选基因
2018-05-14魏大勇崔艺馨汤青林梅家琴李加纳
魏大勇 崔艺馨 熊 清 汤青林 梅家琴 李加纳 钱 伟,*
用全基因组关联作图和共表达网络分析鉴定油菜种子硫苷含量的候选基因
魏大勇1,2,3崔艺馨3熊 清4汤青林1,2梅家琴3李加纳3钱 伟3,*
1西南大学园艺园林学院, 重庆 400715;2南方山地园艺学教育部重点实验室, 重庆 400715;3西南大学农学与生物科技学院, 重庆 400715;4西南大学计算机与信息科学学院, 重庆 400715
油菜籽饼粕是畜禽养殖中重要的蛋白原料, 但饼粕中的硫苷是一种抗营养物质, 食用过多会对禽畜产生毒害, 因此挖掘油菜籽粒硫苷含量的候选基因对油菜种子低硫苷育种具有重要现实意义。本研究连续4年种植1个含157份材料的油菜自然群体, 结合重测序数据对种子硫苷含量进行全基因组关联分析(GWAS), 并对15份低硫苷和15份高硫苷材料进行种子发育早期的转录组测序, 通过权重基因共表达网络分析(WGCNA)鉴定种子硫苷含量的候选基因。用GWAS共检测到45个与种子硫苷含量显著相关的SNP, 单个位点解释的表型变异为13.5%~23.3%, 主要分布在A09、C02和C09染色体的3个区间中, 覆盖5个已知的硫苷代谢基因。用WGCNA分析发现高、低硫苷材料之间的2275个差异表达基因, 可分为12个基因模块, 其中1个模块的基因显著富集在已知的硫苷生物合成途径, 对该模块内163个基因的权重分析得到13个候选基因。经检测, GWAS和WGCNA共得到的18个候选基因中, 有14个候选基因的表达量与种子硫苷含量显著相关(= 0.376~0.638,<0.05)。用两种方法鉴定到1个共同的候选基因(基因名), 与该基因连锁的5个SNP构成5种单体型, 等位基因效应分析发现, 自然群体中63%的材料(99/157)为Hap 5, 平均硫苷含量为50.79 μmol g–1, 与另外4种单体型(95.04~110.28 μmol g–1)存在极显著差异(<0.01)。本研究结合GWAS和WGCNA两种方法鉴定了油菜种子硫苷含量的候选基因, 可为复杂性状候选基因的筛选提供参考。
甘蓝型油菜; 全基因组关联分析; 权重基因共表达网络分析; 重测序; 转录组测序; 种子硫苷含量
油菜(L., 基因组为AACC)由白菜(L., 基因组为AA)和甘蓝(L., 基因组为CC)种间自然杂交并染色体加倍形成[1-2], 是我国乃至世界主要的油料作物。油菜籽粒榨油后的饼粕含丰富的蛋白质, 是良好的天然动物饲料[3]。然而硫代葡萄糖苷(glucosinolate, 简称硫苷)及其降解产物是油菜饼粕中主要的抗营养因子, 严重影响其作为动物饲料的食用安全和适口性, 限制了菜籽饼粕蛋白资源的开发利用[4], 因此降低油菜籽粒中硫苷的含量是油菜育种的重要目标之一。
硫苷是一类含氮和硫的次生代谢产物, 广泛存在于十字花科植物中[5]。硫苷的种类根据侧链R基团的来源不同, 分为脂肪族、芳香族和吲哚族三大类[6]。硫苷的生物合成途径大致可以分为前体氨基酸侧链延长、核心结构合成和次级修饰3个主要阶段, 主要基因在模式植物拟南芥中均已被证实[6-10]。随着油菜及其亲本种(白菜和甘蓝)参考基因组的释放, 大量的硫苷合成和降解基因已被鉴定, 其中在白菜[11]、甘蓝[12]和油菜[1]中分别鉴定出123、127和181个硫苷代谢相关基因。目前对油菜种子硫苷含量的研究主要是通过QTL定位[13-15]、全基因组关联分析(GWAS)[16-17]和关联转录组分析(associative transcriptomic)[18-19]挖掘候选基因和功能位点。
随着测序技术的不断更新和测序费用的不断降低, 海量的测序数据需要分析, 由此一种新的方法应运而生,即权重基因共表达网络分析(WGCNA)[20]。其首先假定基因网络服从无尺度分布, 根据功能相似的基因往往具有类似的表达变化, 构建基因模块。然后对模块进行深入挖掘, 比如筛选模块的枢纽基因、关联模块与性状、模块富集分析和建立基因表达网络等[20-21]。该技术已被广泛应用于人类的疾病研究, 比如Farber[22]结合GWAS和WGCNA分析策略, 找到调控人类骨密度的关键枢纽基因。
本研究将结合GWAS和WGCNA研究方法, 从全基因组和转录组水平挖掘油菜种子硫苷总量候选基因, 为油菜种子低硫苷分子育种和农作物复杂性状候选基因鉴定提供参考。
1 材料与方法
1.1 供试材料和表型测定与分析
157份具有广泛变异的甘蓝型油菜(L.)自交种由重庆市油菜工程技术研究中心提供, 于2013¾2016连续4年种植于西南大学北碚实验基地, 3行区播种, 每行10株, 每年2次田间重复。采用当地常规田间管理方式。用福斯(FOSS)近红外光谱分析仪(NIRSystem, TR-3750)测定157份油菜自然群体的自交种籽粒硫苷总量, 取每份材料3株自交种, 每袋样品重复测定2次。以SAS软件(版本9.13)[23]对表型数据进行方差分析和相关性分析。根据4年数据从上述自然群体中选择稳定遗传的15份极端低硫苷和15份极端高硫苷材料继续种植, 于次年始花期统一剥蕾自交, 并于自交后第15天取角果提取RNA。
1.2 油菜全基因组重测序
于八叶期选择鲜嫩叶片迅速冷冻在液氮中, 由北京百迈客生物科技公司完成总DNA的提取及全基因组重测序。质检合格的文库用Illumina HiSeq 4000平台的双末端150 bp (paired-end, PE150)模式测序, 共得到34.5亿对reads, 平均每个材料的测序深度大约为5倍, 每个材料的覆盖率达到85%。用BWA (版本0.6.1-r104)软件[24]将过滤后得到的干净有效的reads (clean reads)与油菜参考基因组v4.1 (http://www.genoscope.cns.fr/brassicanapus/data/)进行比对; 用GATK (版本2.4-7-g5e89f01)软件[25]检测单核苷酸多态性(single nucleotide polymorphisms, SNP)和插入缺失(insertions and deletions, Indel)。
1.3 全基因组关联分析
采用R软件的GenABEL包[26]进行GWAS分析。通过Merk等[27]方法对4年油菜种子的硫苷含量数据进行最佳线性无偏预测(best linear unbiased prediction, BLUP), 估计种子硫苷含量的BLUP值作为表型数据。采用PCA+K (控制主成分和亲缘关系)的混合线性模型对BLUP和SNP标记进行关联位点的检测, 设定阈值为<8.56×10–8(0.05/所使用的标记, –lg () = 7)。将与显著SNP处于同一单体型块(2>0.5)的区间, 定义为候选关联区间, 在此区间按以下标准预测候选基因: (1)在甘蓝型油菜或拟南芥参考基因组上与性状相关的已知功能的基因; (2)SNP直接落在基因内部; (3)参考已报道QTL定位的结果。
1.4 油菜种子RNA提取及转录组测序
采用植物总RNA提取试剂盒DP432 (天根生化科技有限公司, 北京)提取30份极端材料发育15 d角果的总RNA, 然后将样品送北京百迈客生物科技有限公司构建文库, 并以HiSeq 2000基因分析系统(Illumina公司, USA)进行RNA-Seq PE100测序分析。
1.5 差异表达基因鉴定
采用Tophat-Cufflinks-Cuffmerge-Cuffdiff分析流程鉴定DEGs, 利用TopHat 2.0.0调用bowtie 2将reads比对到甘蓝型油菜参考基因组, 以Cufflinks 2.0.0对测序reads进行组装[28], 分析30个样品中所有转录本的表达量, 以每1百万个map上的reads中map到外显子的每千个碱基上的reads数(reads per kilobase of exon per million mapped reads, RPKM)表示。采用Cuffdiff进行DEGs的筛选鉴定和统计检验, 筛选标准如下: (1)过滤掉在30份材料中基因表达量最大值小于1的低丰度基因; (2)差异表达倍数大于2, 即log2绝对值大于1; (3)错误发现率(false discovery rate,) q-value小于0.05。
1.6 权重基因共表达网络分析
通过基于R语言的WGCNA包[20]对筛选后得到的2275个DEGs构建基因模块, 经过计算设加权参数软阈值为9, 按照基因间两两不相似性聚类得到基因的系统聚类树(附图1-a), 然后根据动态混合剪切法对聚类树切割修剪, 得到17个分支模块(附图1-b), 计算每个模块的模块特征值(module eigengene, ME), 并对ME聚类, 通过相似度高的ME合并分支模块, 最终得到12个整合的基因模块(附图1-c)。每个模块含有50个到534个基因不等, 有200个基因未能分配到任何1个基因模块中。随后计算ME与样本种子硫苷含量的相关性和显著性, 确定模块在样本中的表达情况, 正(负)相关越高(低), 说明在这个模块内的基因表达量越高(低)。通过基因的连通性确定每个基因的权重值, 连通性高的基因在该模块中可能起枢纽作用。因此, 我们将显著模块内单个基因的平均连通性排名前10%的基因作为枢纽基因(hub gene)。
1.7 GO富集和KEGG代谢通路分析
以BLASTP将每个模块的油菜蛋白序列与拟南芥蛋白参考序列(TAIR10)比对, 阈值为1×10–10。根据比对结果, 分别采用在线软件agriGO (http://bioinfo. cau.edu.cn/agriGO/)和在线工具DAVID (https://david. ncifcrf.gov/), 以及共同采用Fisher精确检测(<0.05)和Benjamini Hochberg多重比对检验(FDR<0.05), 以同源性最高的拟南芥蛋白为油菜相对应的蛋白进行GO富集和KEGG代谢途径分析。
2 结果与分析
2.1 表型数据分析
157份油菜自然群体的种子硫苷含量存在广泛的遗传变异, 介于27.38~169.20, 平均为(70.13± 36.50) μmol g–1(表1和附表1)。该性状4年均表现频率为连续近正态分布, 表明油菜种子硫苷含量是1个数量性状, 受多基因控制。

表1 关联分析群体中油菜种子硫苷含量的表型变异
方差分析结果表明, 基因型、环境以及基因型与环境互作间都存在显著差异(<0.01), 但年度内的重复间不存在显著差异(附表2)。4年种子硫苷含量相关性在0.92~0.96之间。
2.2 以GWAS预测控制种子硫苷含量的候选基因
以全基因组重测序共检测到5 294 158个SNP和1 307 151个InDel。过滤最小等位基因频率小于5%和杂合率大于25%的位点, 剩下690 953个特异的SNP位点用于GWAS分析, GWAS共检测到45个与油菜种子硫苷含量显著相关的SNP, 主要分布在A09 (10个, 物理位置位于2 372 597~3 118 196 bp)、C02 (5个, 物理位置位于44 926 609~44 991 771 bp)和C09 (29个, 物理位置位于2 375 598~3 198 893 bp)染色体的3个区间(图1), 单个位点解释的表型变异介于13.45%~23.34%。这个结果与以前报道的QTL结果一致[14,16,18-19], 表明上述3个区间存在控制油菜种子硫苷含量的主效位点。
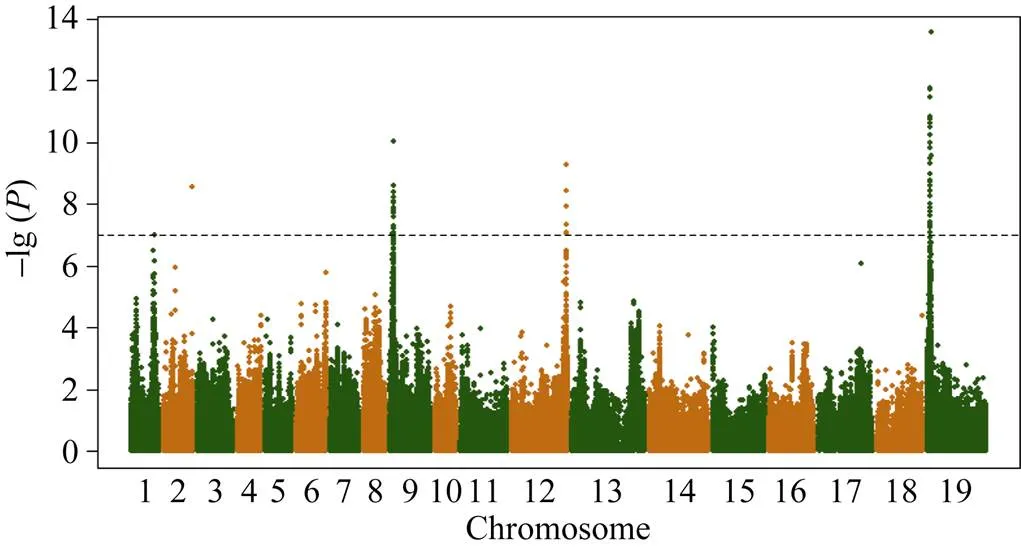
图1 全基因组关联分析曼哈顿图
虚线代表矫正后的阈值–lg () = 7。
The dashed lines represent the bonferroni-adjusted significance threshold –lg () = 7.
与油菜参考基因组的基因注释比对, 在上述3个区间共发现285个基因, 其中5个为已知的硫苷代谢基因。候选基因位于A09染色体2.699 Mb处(显著SNP区间内); 候选基因和分别位于C09染色体2.927 Mb和3.100 Mb处(显著SNP区间内); 候选基因和分别位于C02染色体44.600 Mb和44.703 Mb处(显著SNP上游大约0.2~0.3 Mb处)。与模式植物拟南芥的参考基因组比对后发现, 候选基因与拟南芥基因0 ()同源, 候选基因与()同源, 其他3个候选基因均与()同源, 反映出油菜在进化过程中的基因组复制事件。
2.3 以WGCNA鉴定控制种子硫苷含量的枢纽基因
WGCNA分析发现ME与种子硫苷含量的相关性在–0.76~0.76之间, 其中显著正和负相关的模块分别有4个(Brown、Blue、Purple和Yellow)和2个(Greenyellow和Magenta)(<0.001)。进一步对显著相关模块内的基因进行GO富集和KEGG分析, 只有1个模块(Brown)内的基因显著富集在硫苷生物合成进程(GO: 0019761)和硫苷生物合成代谢通路(KEGG: ko00966)(表2), 说明该模块内存在控制油菜种子硫苷含量的关键基因。
进一步对Brown模块内163个基因进行权重分析, 确定枢纽基因。一共检测到6100条节点线, 每条线的节点连接着1个基因。单个基因的权重值在0.02~0.28之间, 平均值为0.10。删除位于随机染色体的基因, 将剩下的126个基因蛋白序列与拟南芥蛋白参考序列进行同源比对和功能注释分析, 共检测到33个(占26.2%)已知的硫苷代谢基因, 包括13对同源基因, 每对同源基因分别来自于油菜的A和C亚基因组, 说明这些同源基因共表达可能参与种子硫苷的代谢。权重分析中, 连通性排名前10% (权重值>0.20)的基因有13个, 功能注释显示, 其中9个为已知的硫苷代谢基因, 且3对为同源基因, 这些基因在油菜种子硫苷代谢中可能起枢纽作用(表3)。

表2 Brown模块GO富集和KEGG通路分析

表3 Brown模块枢纽基因分析
2.4 候选基因与种子硫苷含量的相关性及效应分析
对GWAS鉴定到的5个已知硫苷代谢基因和利用WGCNA发现的13个枢纽基因进行表达分析发现, 其中14个基因的表达量与种子硫苷含量的积累显著正相关(= 0.376~0.638,<0.05), 1个基因显著负相关(= –0.489,<0.01)(附图2)。此外, 两种方法鉴定到1个共同的基因(), 该基因与C02染色体上通过GWAS得到的5个显著SNP构成1个单体型块(2>0.5)(图2-a), 等位基因效应分析发现, 删除自然群体中11份单体型数目小于3的材料, 剩下146份材料被分为五种单体型, 其中99份材料(占群体总数的63%)均为Hap 5, 其平均硫苷含量为50.79 μmol g–1, 与另外四种单体型材料的种子硫苷含量(95.04~110.28 μmol g–1)存在极显著差异(<0.01)(图2-b)。上述结果表明, 两种方法鉴定到的候选基因与表型性状具有较高的相关性, 并且结合两种方法可能鉴定到比较重要的关键基因。
3 讨论
3.1 GWAS得到的候选基因比较分析
基于全基因组重测序相对高的图谱密度, 本研究通过GWAS共检测到5个已知的硫苷代谢基因。其中, 调控吲哚族硫苷代谢的候选基因的3个同源拷贝分别分布在染色体A09 ()、C02 ()和C09 ()。另外, 控制脂肪族硫苷生物合成的基因仅仅1个拷贝在染色体C09 ()被检测到, 该候选基因的低表达与种子低硫苷显著相关, 这个结果与以前报道的结果一致。研究发现在A09和C02染色体上的2个拷贝在低硫苷油菜中已经丢失[18], 而本研究重测序数据比对的甘蓝型油菜参考基因组是1个法国双低冬性油菜品种“Darmor-”, 所以通过GWAS在A09和C02染色体上未检测到的SNP变异。事实上, 基因组的插入和缺失在作物上是很普遍的, 并且是性状变异的重要机制。在玉米上, 基因启动子上117 bp的插入和内含子35 bp的缺失, 导致了生育酚含量显著的提升[29]。

图2 连锁不平衡和单体型效应分析
a: 候选基因与C02染色体5个显著SNP构成的单体型连锁不平衡分析, 箭头指示核心基因和显著SNP的物理位置。b: C02染色体5个显著SNP构成的单体型效应分析; **表示在0.01水平显著。
a: the LD analysis between the candidate geneand five significant SNPs from GWAS, the arrows shows physical location of the candidate gene and five significant SNPs on chromosome C02. b: the haplotype effect analysis for five significant SNPs in chromosome C02; ** indicates significant difference at<0.01.
3.2 GWAS与WGCNA重复鉴定到关键候选基因MAM1
WGCNA对差异表达基因进行模块化分类, 可以有效分离出真实与性状相关的基因, 提高功能富集的检测功效, 对复杂性状的候选基因挖掘提供新的途径[22]。本研究通过WGCNA共检测到13个枢纽基因, 其中9个已被证实参与硫苷的代谢途径[1], 但只有1个基因()在GWAS中被重复检测到, 该基因在拟南芥中已被证实编码甲硫烷基化苹果酸合酶(methylthioalkylmalate synthase), 是控制甲硫氨酸起源的脂肪族硫苷侧链延长的1个主要基因[30]。转录组表达分析发现, 该基因的表达与种子硫苷含量的积累显著正相关(=0.376,<0.05)。比较五种单体型发现, Hap 5与另外4种单体型的差别主要是C02染色体上SNP位点44 943 847等位基因由C变为A, 使得种子硫苷含量降低52%。根据该位点开发基于PCR的功能标记, 将使油菜进一步降低种子硫苷含量成为可能。
3.3 两种方法检测效率分析及其应用
两种方法只检测到1个重复的基因, 原因可能是: (1) WGCNA中的其他12个枢纽基因在本研究油菜自然群体的DNA水平上没有差异, 导致通过GWAS检测不到显著的位点; (2) WGCNA中单一枢纽基因对油菜种子硫苷的贡献度太小, 它们的显著性淹没在背景噪音中, 通过GWAS难以准确挖掘出来, 对微效多基因控制的位点检测能力不足, 正是GWAS面临的主要问题。因此, 在植物复杂农艺性状候选基因挖掘中, 结合GWAS和WGCNA分析是一种新的思路, 可以提高对微效位点的检测能力和构建关键基因的调控网络。另外, 硫苷总量是由各分量构成, 控制硫苷各组分的相关基因的变异(包括转录水平的差异)均能影响硫苷总量的表现型。本研究通过GWAS和WGCNA方法试图挖掘影响硫苷总量的基因, 但这些基因可能参与硫苷分量的代谢, 需进一步研究确定。
另外, 用WGCNA可以深入挖掘显著模块内枢纽基因, 并对功能未知的基因进行功能注释预测。因为, 在权重网络中, 节点线连接的2个基因表达模式是相似的, 且有潜在的相似功能, 若节点线一端基因功能已知的话, 就可以预测另一端基因的功能, 为以后验证功能未知基因提供参考。
4 结论
通过GWAS和WGCNA共鉴定到18个控制油菜种子硫苷含量的候选基因, 其中多数为已知的硫苷基因, 且其表达量与种子硫苷含量显著相关。1个控制脂肪族硫苷侧链延长的关键基因()被两种方法共同检测出, 与该基因连锁的5个SNP构成5种单体型, 其中Hap 5覆盖了63%的材料, 其种子硫苷含量显著低于含其他4种单体型的材料。因此, 结合GWAS和WGCNA是一种鉴定复杂性状候选基因的新策略, 可兼顾微效位点的检出率以及对关键基因的确定。
[1] Chalhoub B, Denoeud F, Liu S, Parkin I A, Tang H, Wang X, Chiquet J, Belcram H, Tong C, Samans B, Correa M, Da Silva C, Just J, Falentin C, Koh C S, Le Clainche I, Bernard M, Bento P, Noel B, Labadie K, Alberti A, Charles M, Arnaud D, Guo H, Daviaud C, Alamery S, Jabbari K, Zhao M, Edger P P, Chelaifa H, Tack D, Lassalle G, Mestiri I, Schnel N, Le Paslier M C, Fan G, Renault V, Bayer P E, Golicz A A, Manoli S, Lee T H, Thi V H, Chalabi S, Hu Q, Fan C, Tollenaere R, Lu Y, Battail C, Shen J, Sidebottom C H, Wang X, Canaguier A, Chauveau A, Berard A, Deniot G, Guan M, Liu Z, Sun F, Lim Y P, Lyons E, Town C D, Bancroft I, Wang X, Meng J, Ma J, Pires J C, King G J, Brunel D, Delourme R, Renard M, Aury J M, Adams K L, Batley J, Snowdon R J, Tost J, Edwards D, Zhou Y, Hua W, Sharpe A G, Paterson A H, Guan C, Wincker P. Early allopolyploid evolution in the post-Neolithicoilseed genome., 2014, 345: 950–953
[2] Nagaharu U. Genomic analysis inwith special reference to the experimental formation ofand peculiar bode of fertilization., 1935, 7: 389–452
[3] 刘后利. 油菜遗传育种学. 北京: 中国农业大学出版社, 2000. pp 146–154 Liu H L. Genetics and Breeding in Rrapeseed. Beijing: Chinese Agricultural Universitatis Press, 2000. pp 146–154 (in Chinese)
[4] Mithen R. Glucosinolates-biochemistry, genetics and biological activity., 2001, 34: 91–103
[5] Fahey J W, Zalcmann A T, Talalay P. The chemical diversity and distribution of glucosinolates and isothiocyanates among plants., 2001, 56: 5–51
[6] Halkier B A, Gershenzon J. Biology and biochemistry of glucosinolates., 2006, 57: 303–333
[7] Bak S, Feyereisen R. The involvement of two p450 enzymes, CYP83B1 and CYP83A1, in auxin homeostasis and glucosinolate biosynthesis., 2001, 127: 108–118
[8] Grubb C D, Abel S. Glucosinolate metabolism and its control., 2006, 11: 89–100
[9] Mikkelsen M D, Naur P, Halkier B A.mutants in the C-S lyase of glucosinolate biosynthesis establish a critical role for indole-3-acetaldoxime in auxin homeostasis., 2004, 37: 770–777
[10] Wittstock U, Halkier B A. Cytochrome P450 CYP79A2 fromL. Catalyzes the conversion of L-phenylalanine to phenylacetaldoxime in the biosynthesis of benzylglucosinolate., 2000, 275: 14659–14666
[11] Wang X, Wang H, Wang J, Sun R, Wu J, Liu S, Bai Y, Mun J H, Bancroft I, Cheng F, Huang S, Li X, Hua W, Wang J, Wang X, Freeling M, Pires J C, Paterson A H, Chalhoub B, Wang B, Hayward A, Sharpe A G, Park B S, Weisshaar B, Liu B, Li B, Liu B, Tong C, Song C, Duran C, Peng C, Geng C, Koh C, Lin C, Edwards D, Mu D, Shen D, Soumpourou E, Li F, Fraser F, Conant G, Lassalle G, King G J, Bonnema G, Tang H, Wang H, Belcram H, Zhou H, Hirakawa H, Abe H, Guo H, Wang H, Jin H, Parkin I A, Batley J, Kim J S, Just J, Li J, Xu J, Deng J, Kim J A, Li J, Yu J, Meng J, Wang J, Min J, Poulain J, Wang J, Hatakeyama K, Wu K, Wang L, Fang L, Trick M, Links M G, Zhao M, Jin M, Ramchiary N, Drou N, Berkman P J, Cai Q, Huang Q, Li R, Tabata S, Cheng S, Zhang S, Zhang S, Huang S, Sato S, Sun S, Kwon S J, Choi S R, Lee T H, Fan W, Zhao X, Tan X, Xu X, Wang Y, Qiu Y, Yin Y, Li Y, Du Y, Liao Y, Lim Y, Narusaka Y, Wang Y, Wang Z, Li Z, Wang Z, Xiong Z, Zhang Z. The genome of the mesopolyploid crop species., 2011, 43: 1035–1039
[12] Liu S Y, Liu Y M, Yang X H, Tong C B, Edwards D, Parkin I A P, Zhao M X, Ma J X, Yu J Y, Huang S M, Wang X Y, Wang J Y, Lu K, Fang Z Y, Bancroft I, Yang T J, Hu Q, Wang X F, Yue Z, Li H J, Yang L F, Wu J, Zhou Q, Wang W X, King G J, Pires J C, Lu C X, Wu Z Y, Sampath P, Wang Z, Guo H, Pan S K, Yang L M, Min J M, Zhang D, Jin D C, Li W S, Belcram H, Tu J X, Guan M, Qi C K, Du D Z, Li J N, Jiang L C, Batley J, Sharpe A G, Park B S, Ruperao P, Cheng F, Waminal N E, Huang Y, Dong C H, Wang L, Li J P, Hu Z Y, Zhuang M, Huang Y, Huang J Y, Shi J Q, Mei D S, Liu J, Lee T H, Wang J P, Jin H Z, Li Z Y, Li X, Zhang J F, Xiao L, Zhou Y M, Liu Z S, Liu X Q, Qin R, Tang X, Liu W B, Wang Y P, Zhang Y Y, Lee J, Kim H H, Denoeud F, Xu X, Liang X M, Hua W, Wang X W, Wang J, Chalhoub B, Paterson A H. Thegenome reveals the asymmetrical evolution of polyploid genomes., 2014, 5: 3930
[13] Fu Y, Lu K, Qian L W, Mei J Q, Wei D Y, Peng X H, Xu X F, Li J N, Frauen M, Dreyer F, Snowdon R J, Qian W. Development of genic cleavage markers in association with seed glucosinolate content in canola., 2015, 128: 1029–1037
[14] Howell P M, Sharpe A G, Lydiate D J. Homoeologous loci control the accumulation of seed glucosinolates in oilseed rape ()., 2003, 46: 454–460
[15] Zhao J, Meng J. Detection of loci controlling seed glucosinolate content and their association with Sclerotinia resistance in., 2003, 122: 19–23
[16] Li F, Chen B Y, Xu K, Wu J F, Song W L, Bancroft I, Harper A L, Trick M, Liu S Y, Gao G Z, Wang N, Yan G X, Qiao J W, Li J, Li H, Xiao X, Zhang T Y, Wu X M. Genome-wide association sudy dissects the genetic architecture of seed weight and seed quality in rapeseed (L.)., 2014, 21: 355–367
[17] Qu C M, Li S M, Duan X J, Fan J H, Jia L D, Zhao H Y, Lu K, Li J N, Xu X F, Wang R. Identification of candidate genes for seed glucosinolate content using association mapping inL., 2015, 6: 1215–1229
[18] Harper A L, Trick M, Higgins J, Fraser F, Clissold L, Wells R, Hattori C, Werner P, Bancroft I. Associative transcriptomics of traits in the polyploid crop species., 2012, 30: 798–802
[19] Lu G, Harper A L, Trick M, Morgan C, Fraser F, O'Neill C, Bancroft I. Associative transcriptomics study dissects the genetic architecture of seed glucosinolate content in., 2014, 21: 613–625
[20] Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis., 2008, 9: 559
[21] 宋长新, 雷萍, 王婷. 基于WGCNA算法的基因共表达网络构建理论及其R软件实现. 基因组学与应用生物学, 2013, 32: 135–141 Song C X, Lei P, Wang T. Gene co-expression network analysis ased onWGCNA algorithm-theory and implementation in R Software., 2013, 32: 135–141 (in Chinese with English abstract)
[22] Farber C R. Systems-level analysis of genome-wide association data., 2013, 3: 119–129
[23] SAS V9.13 software. SAS Institute, Cary, NC, USA, 2005
[24] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform., 2009, 25: 1754–1760
[25] McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo M A. The genome analysis toolkit: a mapreduce framework for analyzing next-generation DNA sequencing data., 2010, 20: 1297–1303
[26] Aulchenko Y S, Ripke S, Isaacs A, Van Duijn C M. GenABEL: an R library for genome-wide association analysis., 2007, 23: 1294–1296
[27] Merk H L, Yarnes S C, Van Deynze A. Trait diversity and potential for selection indices based on variation among regionally adapted processing tomato germplasm., 2012, 137: 427–437
[28] Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley D R, Pimentel H, Salzberg S L, Rinn J L, Pachter L. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks., 2012, 7: 562–578
[29] Li Q, Yang X H, Xu S T, Cai Y, Zhang D L, Han Y J, Li L, Zhang Z X, Gao S B, Li J S, Yan J B. Genome-wide association studies identified three independent polymorphisms associated with alpha-tocopherol content in maize kernels., 2012, 7: e36807
[30] Kroymann J, Textor S, Tokuhisa J G, Falk K L, Bartram S, Gershenzon J, Mitchell-Olds T. A gene controlling variation in Arabidopsis glucosinolate composition is part of the methionine chain elongation pathway., 2001, 127: 1077–1088
附表1 材料来源
Supplementary table 1 Source of the accessions in this study
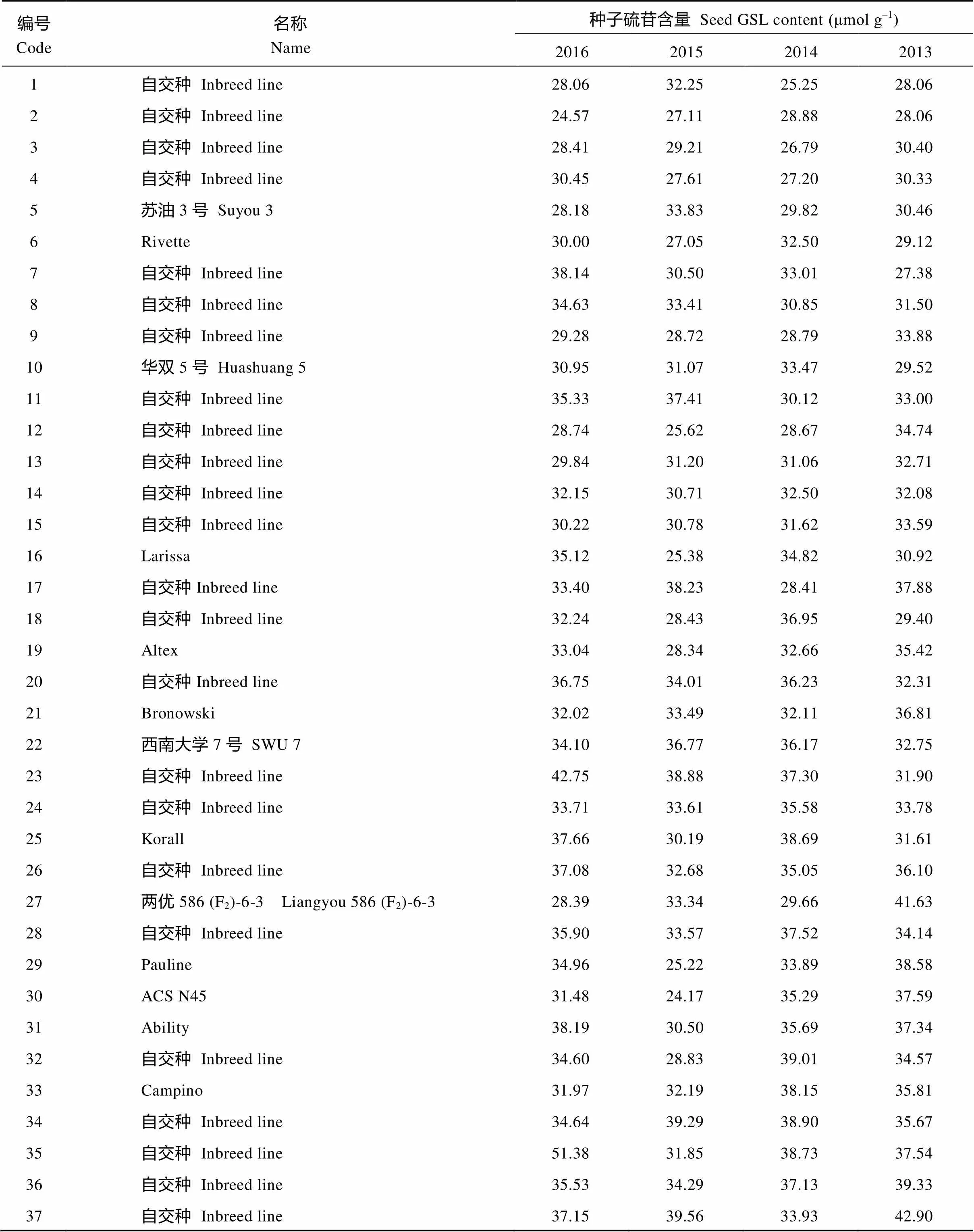
编号Code名称Name种子硫苷含量 Seed GSL content (μmol g–1) 2016201520142013 1自交种 Inbreed line28.0632.2525.2528.06 2自交种 Inbreed line24.5727.1128.8828.06 3自交种 Inbreed line28.4129.2126.7930.40 4自交种 Inbreed line30.4527.6127.2030.33 5苏油3号 Suyou 328.1833.8329.8230.46 6Rivette30.0027.0532.5029.12 7自交种 Inbreed line38.1430.5033.0127.38 8自交种 Inbreed line34.6333.4130.8531.50 9自交种 Inbreed line29.2828.7228.7933.88 10华双5号 Huashuang 530.9531.0733.4729.52 11自交种 Inbreed line35.3337.4130.1233.00 12自交种 Inbreed line28.7425.6228.6734.74 13自交种 Inbreed line29.8431.2031.0632.71 14自交种 Inbreed line32.1530.7132.5032.08 15自交种 Inbreed line30.2230.7831.6233.59 16Larissa35.1225.3834.8230.92 17自交种Inbreed line33.4038.2328.4137.88 18自交种 Inbreed line32.2428.4336.9529.40 19Altex33.0428.3432.6635.42 20自交种Inbreed line36.7534.0136.2332.31 21Bronowski32.0233.4932.1136.81 22西南大学7号 SWU 734.1036.7736.1732.75 23自交种 Inbreed line42.7538.8837.3031.90 24自交种 Inbreed line33.7133.6135.5833.78 25Korall37.6630.1938.6931.61 26自交种 Inbreed line37.0832.6835.0536.10 27两优586 (F2)-6-3 Liangyou 586 (F2)-6-328.3933.3429.6641.63 28自交种 Inbreed line35.9033.5737.5234.14 29Pauline34.9625.2233.8938.58 30ACS N4531.4824.1735.2937.59 31Ability38.1930.5035.6937.34 32自交种 Inbreed line34.6028.8339.0134.57 33Campino31.9732.1938.1535.81 34自交种 Inbreed line34.6439.2938.9035.67 35自交种 Inbreed line51.3831.8538.7337.54 36自交种 Inbreed line35.5334.2937.1339.33 37自交种 Inbreed line37.1539.5633.9342.90
(续附表1)

编号Code名称Name种子硫苷含量 Seed GSL content (μmol g–1)2016201520142013 38自交种 Inbreed line41.5338.4435.1341.86 39自交种 Inbreed line42.8773.0037.9439.10 40Omega104.2391.6138.3838.71 41自交种 Inbreed line37.4738.8242.3635.69 42Q290.6456.7336.8244.85 43自交种 Inbreed line31.3130.9344.4834.73 44自交种 Inbreed line31.2933.7751.0329.67 45SW Sinatra39.6126.6730.6351.22 46Andor36.1725.7840.1542.52 47Tenor37.5938.3345.8937.66 48Aurora36.0434.3447.4836.77 49Clipper39.7845.4140.6443.75 50自交种 Inbreed line41.8045.2844.5240.35 51自交种 Inbreed line41.5936.3245.4839.77 52自交种 Inbreed line41.3041.8444.0942.22 53Granit38.6634.0639.9244.87 54自交种 Inbreed line33.5934.0442.2344.32 55自交种 Inbreed line38.7348.8843.7643.72 56Montego42.9334.9444.6743.51 57Allure49.9141.9946.3942.28 58自交种 Inbreed line40.0841.4146.0442.97 59自交种 Inbreed line38.5445.5044.6244.85 60NK Passion48.4946.2046.1343.47 61Campari46.7352.8548.1242.50 62Lord61.0981.4745.9545.95 63自交种 Inbreed line43.3641.1553.4139.47 64DRAKKAR33.9730.3048.1445.05 65中双5号 Zhongshuang 553.7147.4548.1445.61 66Lilian48.6642.9450.8743.28 67Rapid50.2749.0049.9244.69 68Aragon54.0443.7955.1539.59 69Lisandra48.3731.8554.8740.51 70821选×品93-498 F8 821 Xuan × Pin 93-498 F857.5246.8243.1753.81 71油研2号× 84-24016 Youyan 2 × 84-2401676.7079.3041.6756.24 72Pivot31.0531.3558.4241.61 73SLM 051256.1730.5853.7248.36 74Musette61.0559.7353.4149.47 75Lion92.53103.6555.5848.03 76Baros52.4338.7359.5445.92 77Korinth58.7750.0047.2057.10 78Odin68.9760.0060.2050.25 79Beluga59.4252.5954.8058.19 80Express 61783.7846.9760.9052.10 81SWGospel65.6452.6662.8652.86 82Fortis77.7245.9765.6653.94 83Remy68.0353.2965.3654.30 84BRISTOL76.1964.9471.3649.38 85Jantar69.9147.8161.9860.50 86Binera86.6881.6062.2460.40 87Boston74.5552.6064.1359.12 88Licapo60.2344.5466.0857.42 89Alesi73.3250.7566.7859.90 90Nugget73.9852.7668.2758.44 91Jessica71.9273.4769.8057.61 92HANNA76.3576.0262.7867.88 93Duplo74.1876.9374.0557.65 94Wesroona71.1979.2867.3764.48 95Laser76.8158.8775.3357.31 96Recital70.9452.2572.3662.24 97Escort82.0465.8075.9565.02 98Darmor96.64141.0078.3863.00 99Ww 128676.3997.9773.0870.24 100(D57×Oro)×油研2号-F6 (D57×Oro)×Youyan 2-F672.9256.8167.0776.86 101Falcon72.2173.1176.7571.03 102华油6号 Huayou 684.0491.2873.5078.47 103贵农78-6-112 Guinong 78-6-11278.6389.8671.6185.40 104Chuosenshu86.26108.8488.6488.43 105Lirabon109.95104.4998.2886.98 106Pera91.5298.4776.04111.30 107Manitoba87.8994.70114.9973.71 108Nugget105.0194.40104.7187.41 109Regina II89.1895.8393.3399.70 110Oro74.4088.1585.84108.59 111Galant94.2273.9287.67106.82 112V8108.7089.26109.7785.73 113Nakate Chousen126.30124.3691.44104.60 114CRESOR92.3684.7389.55106.61 115Hankkija’s Lauri99.37128.2896.18100.79 116TANTAL103.22115.3495.86104.73 117云油14 Youyou 1496.84102.85106.2196.13 118K26-96114.75102.00103.92100.34 119Spaeths Zollerngold104.30106.79100.98108.09 120Baltia112.0193.61106.74102.92 121Mali101.14105.85102.20114.77 122RESYN-H048112.25106.00104.38113.97 123Daichousen (fuku)114.21106.34114.41106.62 124Orpal95.2496.20110.46111.72 125Orriba110.66112.91112.52111.31 126EMERALD110.66112.91109.30114.72 127湘油16 Xiangyou 16100.0594.64110.22116.66 128Daichousen (mizuyasu)102.05100.97113.01114.26 129Zephyr114.07105.58117.34112.79 130Wesreo114.03101.87118.95113.23 131Miyauchi Na106.39138.65119.01110.30 132Olivia115.30115.82119.21113.47 133Taisetsu107.13116.00111.10122.19 134JANETZKIS133.00129.88115.00119.73 135JetNeuf112.14132.64120.52114.68 136Gulliver110.21102.68113.37119.94 137Leonessa123.22112.63114.65122.08 138Ceska Krajova107.40105.93123.48115.89 139Skrzeszowicki143.05131.53119.89119.54 140CANARD74.88122.32114.91125.84 141Mansholt102.00107.84122.01121.82 142Palu140.92128.05126.79118.74 143Parapluie131.00142.78124.71122.51 144Kruglik119.41114.38118.23129.02 145Czyzowska108.55122.55116.10131.66 146Edita147.21115.40131.10119.89 147Sonnengold128.77144.22133.00125.70 148Conny139.65122.66134.30128.54 149湘农油-1 Xiangnongou-1120.54101.40135.57123.31 150西农长角×((D57×Oro)×85-64)F7Xinongchangjiao×((D57×Oro)×85-64)F7106.86128.13122.10141.81 151MOANA139.74145.62138.69125.34 152Mlochowski118.99120.69133.42131.32 153Samo169.19152.80137.73132.82 154Suigenshu124.93144.33147.49126.56 155Nunsdale134.00151.62144.68131.49 156Gisora150.75148.00131.50159.43 157Dippes149.04153.73149.56143.58
附表2 关联分析种子硫苷含量表型方差分析
Supplementary table 2 ANOVA of phenotype for seed GSL in association population

变异来源Source自由度df均方MS概率值P-value 基因型 Genotype1569238.54<0.001 环境 Environment3406.700.0002 基因型×环境 Genotype×Environment459165.83<0.001 重复 Repeat10.180.9561
附图1 基因聚类树及模块切割
Supplementary fig. 1 Gene cluster dendrogram and module splitting
a: 基因聚类树, 纵坐标表示各基因间的聚类距离; b: 动态混合切割得到的分支模块; c: 合并相似度高的模块。
a: gene cluster dendrogram tree,-coordinate indicates the cluster distance between genes; b: modules assignment cut using dynamic tree; c: merging of modules whose expression profiles are very similar.

附图2 核心基因在30份极端材料中表达水平与种子硫苷含量的相关性分析
Supplementary fig. 2 Correlation analysis between core gene expression levels and seed GSL contents in 30 extreme accessions
*和**分别表示在0.05和0.01水平上相关显著。
*and**indicate significant correlation at<0.05 and 0.01, respectively.
Identification of Candidate Genes for Seed Glucosinolate Content of Rapeseed by Using Genome-wide Association Mapping and Co-expression Networks Analysis
WEI Da-Yong1,2,3, CUI Yi-Xin3, XIONG Qing4, TANG Qing-Lin1,2, MEI Jia-Qin3, LI Jia-Na3, and QIAN Wei3,*
1College of Horticulture and Landscape Architecture, Southwest University, Chongqing 400715, China;2Key Laboratory of Horticulture Science for Southern Mountainous Regions, Ministry of Education, Chongqing 400715, China;3College of Agronomy and Biotechnology, Southwest University, Chongqing 400715, China;4School of Computer and Information Science, Chongqing 400715, China
Seed meal of rapeseed (L.) is a valuable protein source for livestock raising. However, high seed glucosinolates (GSL) content is harmful and toxic to livestock. Therefore, identifying candidate genes of seed GSL content is important in rapeseed breeding for low seed GSL. In this study, a genome-wide association study (GWAS) for seed GSL content was conducted using 157 rapeseed lines grown in four consecutive years. Meanwhile, a weighted gene co-expression network analysis (WGCNA) was carried out in early seed development stage of 15 low and 15 high seed GSL content lines for detecting candidate genes. In total, 45 SNPs found by GWAS significantly associated with seed GSL contents, explaining 13.5%–23.3% of the phenotypic variance per SNP. These SNPs were mainly detected from three intervals on chromosomes A09, C02, and C09, covering five known GSL metabolism genes. A total of 2275 differentially expressed genes (DEGs) were identified by RNA-Seq between rapeseed lines with low and high seed GSL contents. These DEGs were clustered into 12 modules by WGCNA, of which one module (contains 163 DEGs) was mainly enriched in the GSL biosynthetic process. By using a weighted analysis for this module, 13 hub-genes were detected including nine known GSL metabolic genes. Among the 18 candidate genes identified by GWAS and WGCNA, 14 genes showed significant correlation between their expressions and the seed GSL contents (= 0.376–0.638,< 0.05). Furthermore, one gene,(), was detected by both GWAS and WGCNA. Five haplotypes were formed by five SNPs that significantly linked with, and 63% of the rapeseed population (99/157) were found to carry Hap 5 with significant lower seed GSL contents (an average of 50.79 μmol g–1) compared with those carrying the other four haplotypes (95.04–110.28 μmol g–1). By GWAS and WGCNA, our study not only identified the candidate genes for seed GSL content of rapeseed, but also provided a guidance for digging candidate genes for other complex traits.
;GWAS; WGCNA; Re-sequencing; RNA-seq; Seed GSL content
2017-12-10;
2018-03-15;
2018-03-16.
10.3724/SP.J.1006.2018.00629
本研究由国家自然科学基金项目(31601333), 国家重点基础研究发展计划(973计划)项目(2015CB150201)和中央高校基本科研业务专项(XDJK2017B036)资助。
This study was supported by National Natural Science Foundation of China (31601333), the National Basic Research Program of China (973 Program) (2015CB150201), and the Fundamental Research Funds for the Central Universities (XDJK2017B036).
钱伟, E-mail: qianwei666@hotmail.com, Tel: 023-68250701
E-mail: swuwdy@swu.edu.cn
http://kns.cnki.net/kcms/detail/11.1809.S.20180316.0853.002.html
