甘蔗品种的AFLP和SSR标记鉴定及其应用
2018-05-14汪洲涛高世武王春风马晶晶阙友雄许莉萍
汪洲涛 游 倩 高世武 王春风 李 竹 马晶晶 阙友雄 许莉萍 罗 俊
甘蔗品种的AFLP和SSR标记鉴定及其应用
汪洲涛 游 倩 高世武 王春风 李 竹 马晶晶 阙友雄 许莉萍 罗 俊*
福建农林大学农业部福建甘蔗生物学与遗传育种重点实验室, 福建福州 350002
品种遗传多样性和指纹图谱是育种、品种权保护和新品种推广等工作的重要参考和依据。本研究选用38个来自国家甘蔗品种区域试验的甘蔗新品种(系), 以9对AFLP标记扩增出348个位点, 多态性位点248个, 多态性比率为72.26%; 15对SSR标记扩增出180个位点, 多态性位点176个, 多态性比率为97.78%。38个新品种(系)的遗传相似系数分布在0.668~0.847之间, 其箱线图分布特征显示, 其中的FN、MT、YZ、YG、GT等系列品种(系)遗传基础相似。通过UPGMA聚类表明, 可在遗传相似系数为0.732处将38个甘蔗新品种(系)划分为2个群体, 其中福农09-2201和桂糖06-1492作为一个子群体最先被划分出来, 它们在群体中的异质性较强; 另外, 在遗传相似性系数为0.770处划分出一个子群体a, 其中包含参照品种ROC22、福农07-3206、福农40、海蔗22、桂糖09-12、柳城07-150等品种(系)。ROC22具有广适应性和高产高糖等优良特性, 子群体a中的另外几个品种(系)则更有可能拥有这些特性, 具有更高的推广潜力。本研究选择60个SSR位点构建了38个甘蔗新品种(系)的指纹图谱, 对品种鉴定及品种权的保护具有重要作用, 可望直接应用于指导甘蔗种质资源的遗传多样性评价和分子指纹图谱鉴定, 并将为这些品种(系)推广布局或作为杂交亲本利用提供参考和借鉴。
甘蔗; 分子标记; 种质鉴定; 群体划分; 推广布局
受人口增长、物质消费水平提高、气候变化、城市化侵占耕地和老龄化导致劳动力短缺等因素促使, 粮食安全与能源安全早已成为人类面临的严峻挑战[1-4]。甘蔗是最主要的糖料作物和重要的能源作物, 其需求量不断增长[5-6], 在耕地面积限制的情况下, 需要不断育成新的优良品种来提高甘蔗的产量。从水稻、玉米等粮食作物的生产发展过程可以看出, 粮食产量的每次重大提升都得益于关键种质资源的发掘与利用, 种质资源及其高效利用十分重要[7-8]。甘蔗种质资源十分丰富, 在我国云南国家甘蔗种质资源圃中, 保育近2200份, 美国和印度的世界甘蔗种质收集中心保育的分别为近6000和4000多份。如此庞大的种质资源群体蕴含着巨大的基因潜质, 对其鉴定和评价是育种工作的重要基础。形态标记、细胞标记、生化标记和分子标记等是近些年被广泛使用的植物种质资源鉴定方法, 在甘蔗种质鉴定工作中也发挥着重要作用[9-12]。如Praveen等[13]在2015年报道了SGDB (Sugarcane Germplasm Database), 该数据库便是根据形态特征、农艺性状、品质性状、抗病虫性等信息鉴定划分甘蔗种质资源的, 这些信息很大程度上提高了育种亲本的选择效率。在我国, 自“九五”以来, 育种工作者也主要应用形态学、细胞学或生理生化等方法完成了大量甘蔗种质资源鉴定评价工作, 获得一批优良特异的甘蔗种质[14]。这些鉴定工作对育种工作具有重要的实用价值, 不过这些方法也均存在较多的局限性, 如易受环境和植物生育时期影响、标记的位点有限等, 其鉴定结果的准确性和稳定性较差。DNA分子标记技术从基因水平寻找生物个体间的遗传差异, 不受环境等因素的干扰, 并且标记位点丰富[15]。目前, DNA分子标记技术已经成为鉴定和评价农作物种质资源最主要的方法, 得到广泛应用[16-18]。
在甘蔗种质资源的分子标记技术鉴定和评价等工作中, 目前主要涉及的标记技术有AFLP (Amplified Fragment Length Polymorphism)[19]、SSR (Simple Sequence Repeat)[20]、SNP (Single Nucleotide Polymorphisms)[21]和ISSR (Inter-simple Sequence Repeat)[22]等。AFLP和SSR标记相对于其他DNA分子标记, 具备检测成本低、多态性位点丰富、在植物基因组中分布广泛且均匀等优点, 已被众多学者深入讨论[23-26]。利用AFLP标记和SSR标记开展甘蔗种质资源遗传多样性的研究已有诸多报道, 如刘新龙等[27]运用AFLP标记技术研究了41份甘蔗近缘属植物滇蔗茅()无性系, 发现滇蔗茅的遗传多样性与地理分布有着明显的关系; Lima等[28]将基于AFLP标记获取的79个甘蔗品种的遗传相似系数对比甘蔗品种的系谱关系时发现, 前者能比后者提供更多的甘蔗品种间血缘的信息。这与先前Barret等[29]比较AFLP标记法和系谱法评价小麦遗传多样性效用的研究结论一致; Singh等[30]对印度亚热带地区割手密和商业种的SSR标记鉴定显示, 割手密的变异水平远高于商业种, 而这些高变异的割手密材料并未有效应用于甘蔗育种中, 该结果为目前甘蔗育种亲本的开拓提供了方向; 齐永文等[31]运用SSR标记技术对中美甘蔗品种的遗传距离评估表明, 中美种质间遗传基础差异较明显, 我们不仅要注重美国种质的引进, 也要重视中国种质的发掘利用, 协同拓宽中国甘蔗品种的遗传基础。另外, 利用分子标记技术构建甘蔗指纹身份证或指纹图谱也有诸多报道, 如刘新龙等[32]通过筛选多态性丰富、品种聚类区分率高、易统计的引物组成核心引物, 构建了云南自育的27个品种与10个蔗区主栽品种的分子身份证, 为品种间的区分提供了精准快捷的依据; 姚春雪等[33]利用SSR标记研究崖城89-9和昆明蔗茅及其67份杂交后代(F1、BC1和BC2)材料, 构建各份材料的DNA指纹图谱, 为追踪蔗茅野生种血缘在不同世代中的传递情况及其在甘蔗育种中的进一步利用提供科学依据。
农作物品种区域试验是新育成品系正式成为推广品种的关键环节[34]。目前, 新品种推广主要依据AMMI模型或GGE-Biplot分析生成的新品种丰产性、稳定性和适应性等指标[35-36], 还未见从分子水平为甘蔗新品种推广布局提供建议的报道。本研究主要应用9个AFLP和15个SSR标记分析来自三轮国家甘蔗品种区域试验(以下简称“区试”)的参试品种的遗传多样性并绘制SSR标记指纹图谱, 同时从分子水平的分析结果出发讨论甘蔗新品种的推广布局策略。本研究可以让我们了解38个新育成甘蔗栽培品种(系)的共祖度(遗传背景相似度), 从而间接评估目前甘蔗育种所用亲本的遗传基础; 区试品种(系)也是种质资源的重要部分, 今后可作为育种亲本的选用对象, 研究结果对今后育种的亲本选择有一定的参考价值; 同时, 本研究尝试从实际的品种推广工作出发, 从分子标记鉴定结果的角度, 讨论新品种(系)可能的推广策略, 为区试品种的推广工作提供参考; 通过品种(系)指纹图谱的绘制, 为品种身份鉴别及品种权保护等工作提供可靠依据。
1 材料与方法
1.1 实验材料及其DNA提取
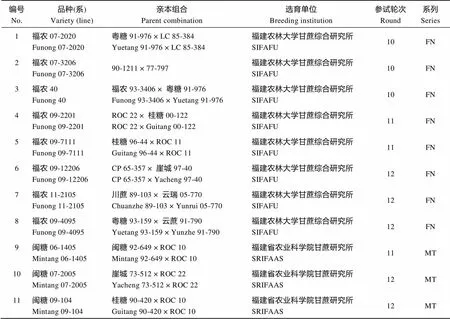
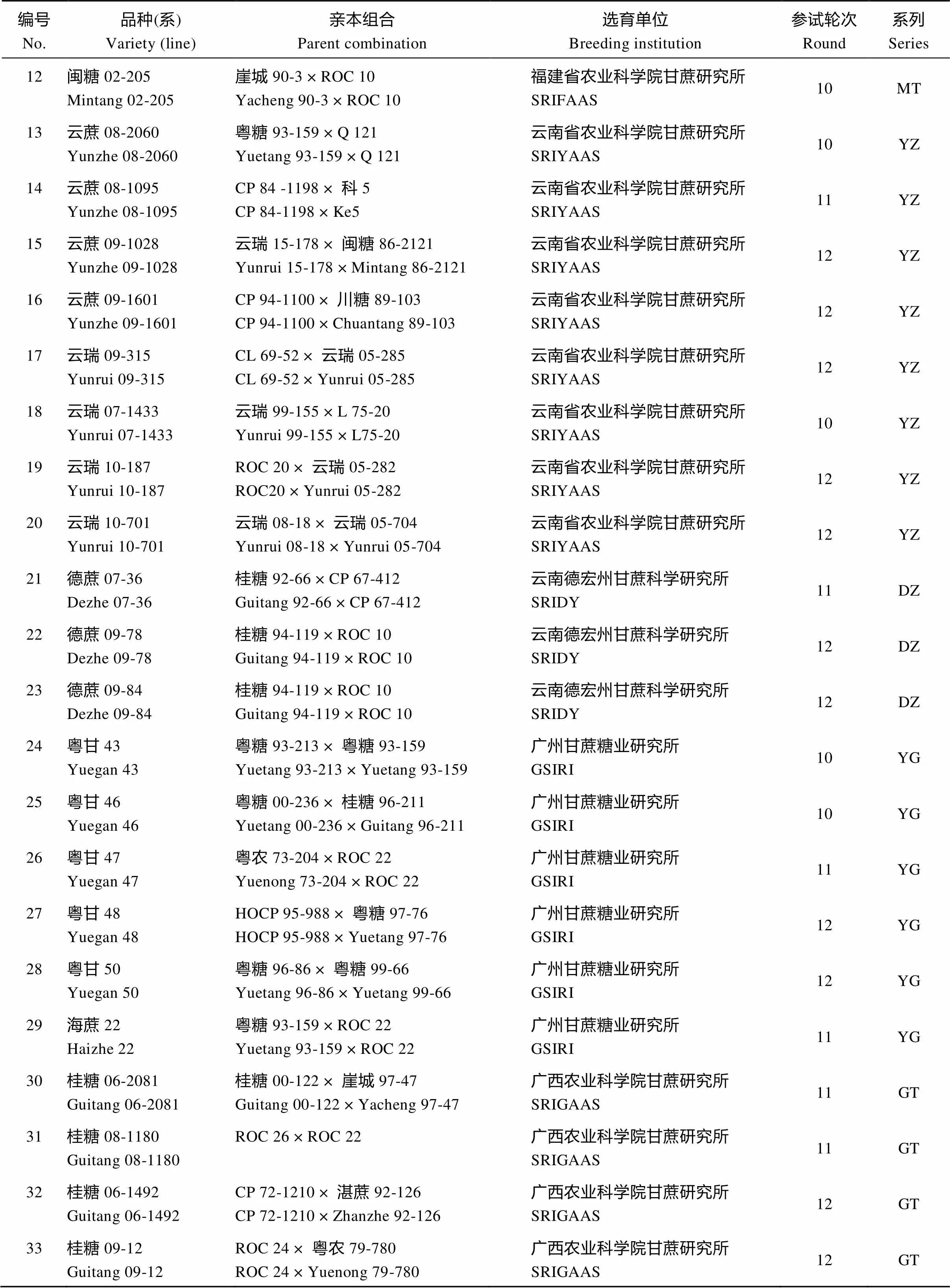
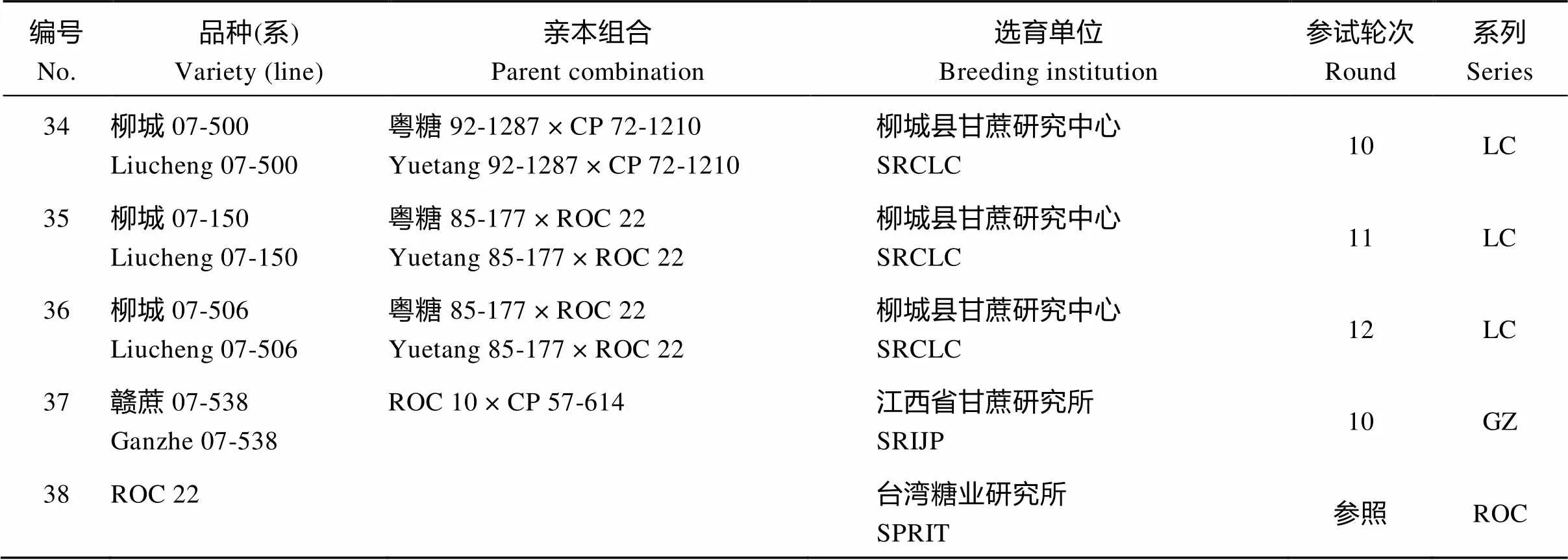
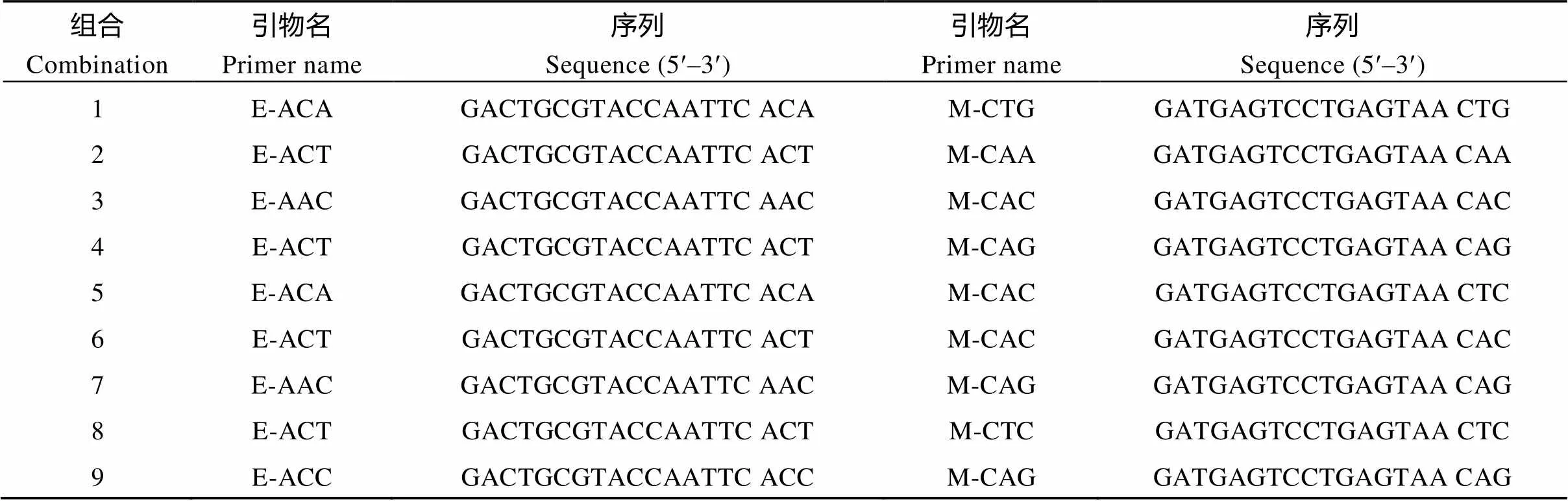
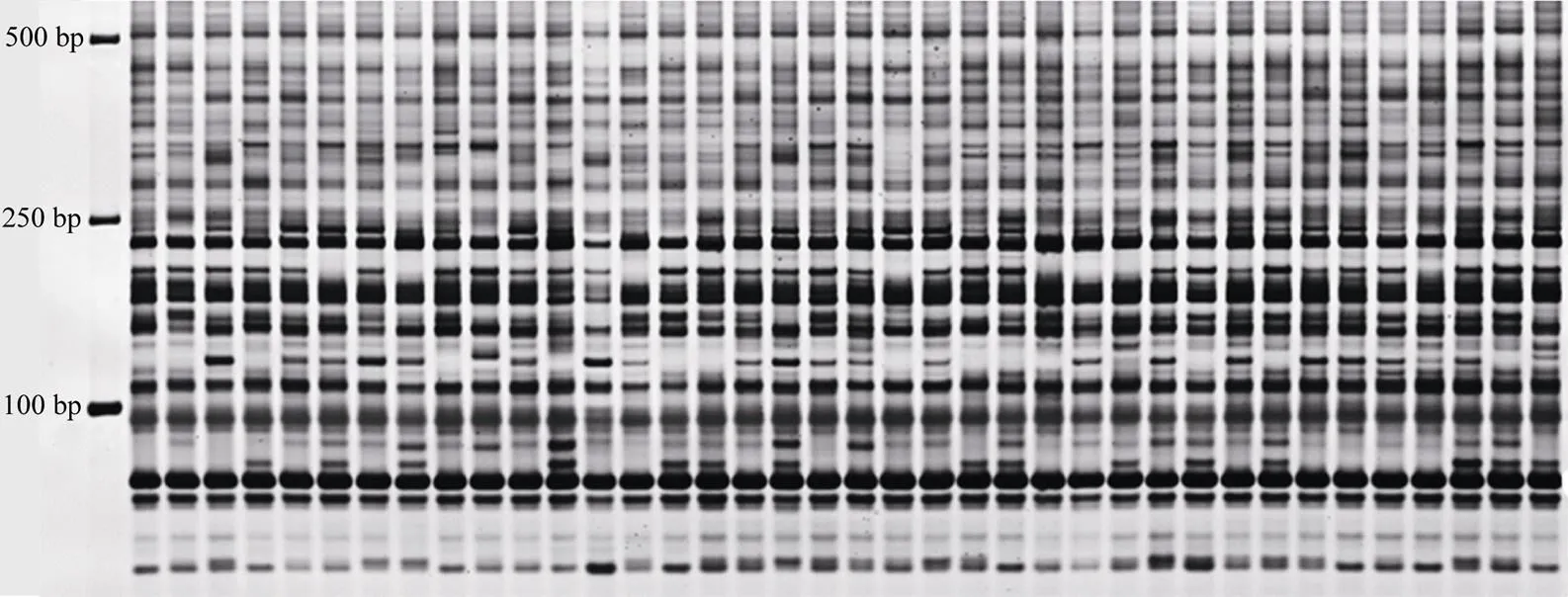
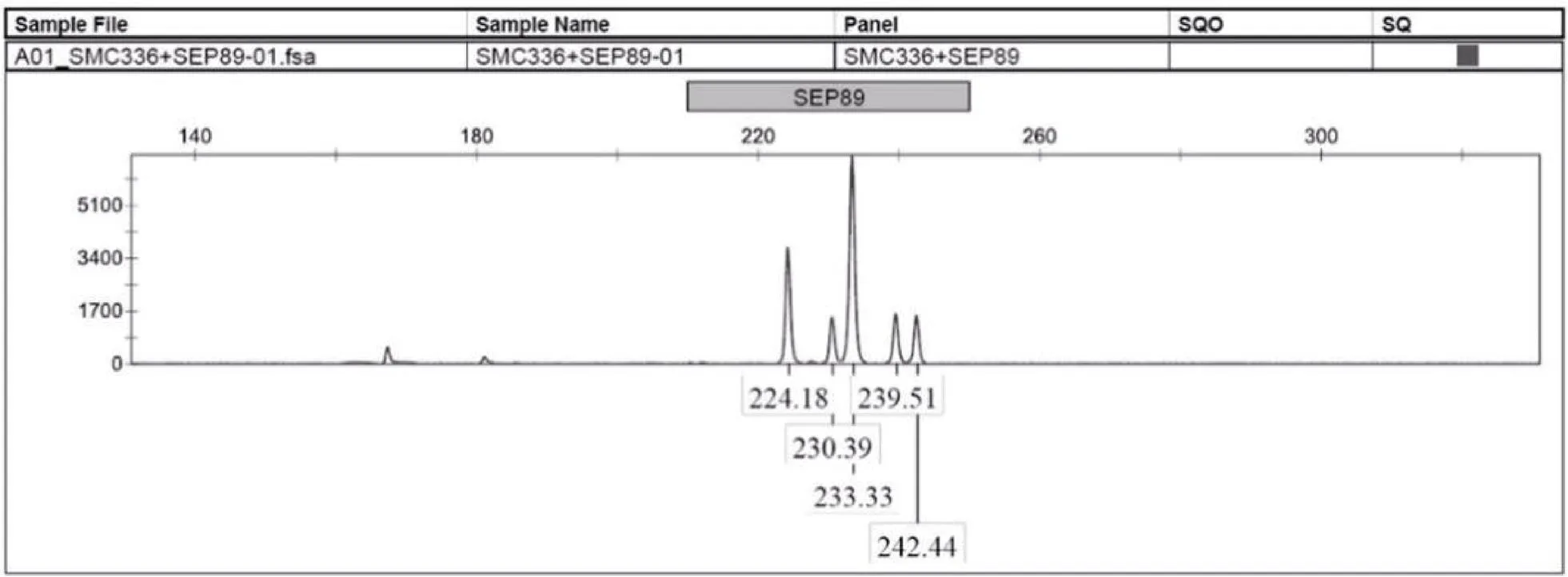
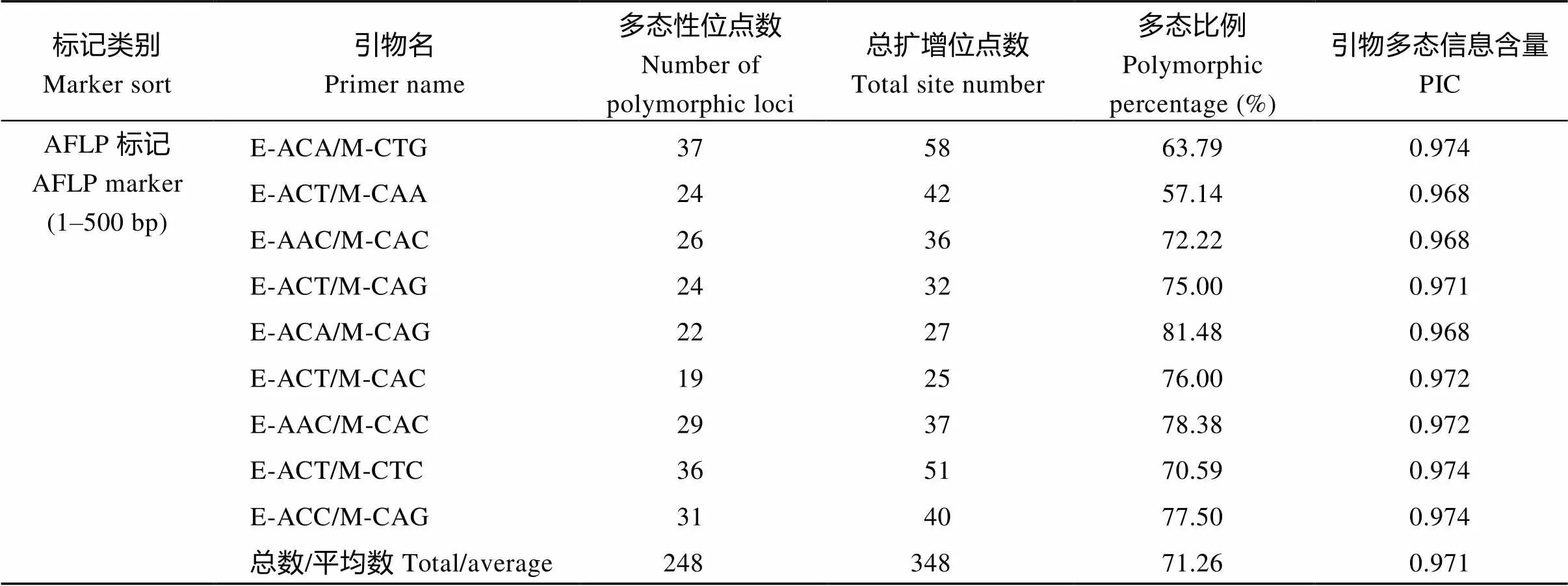
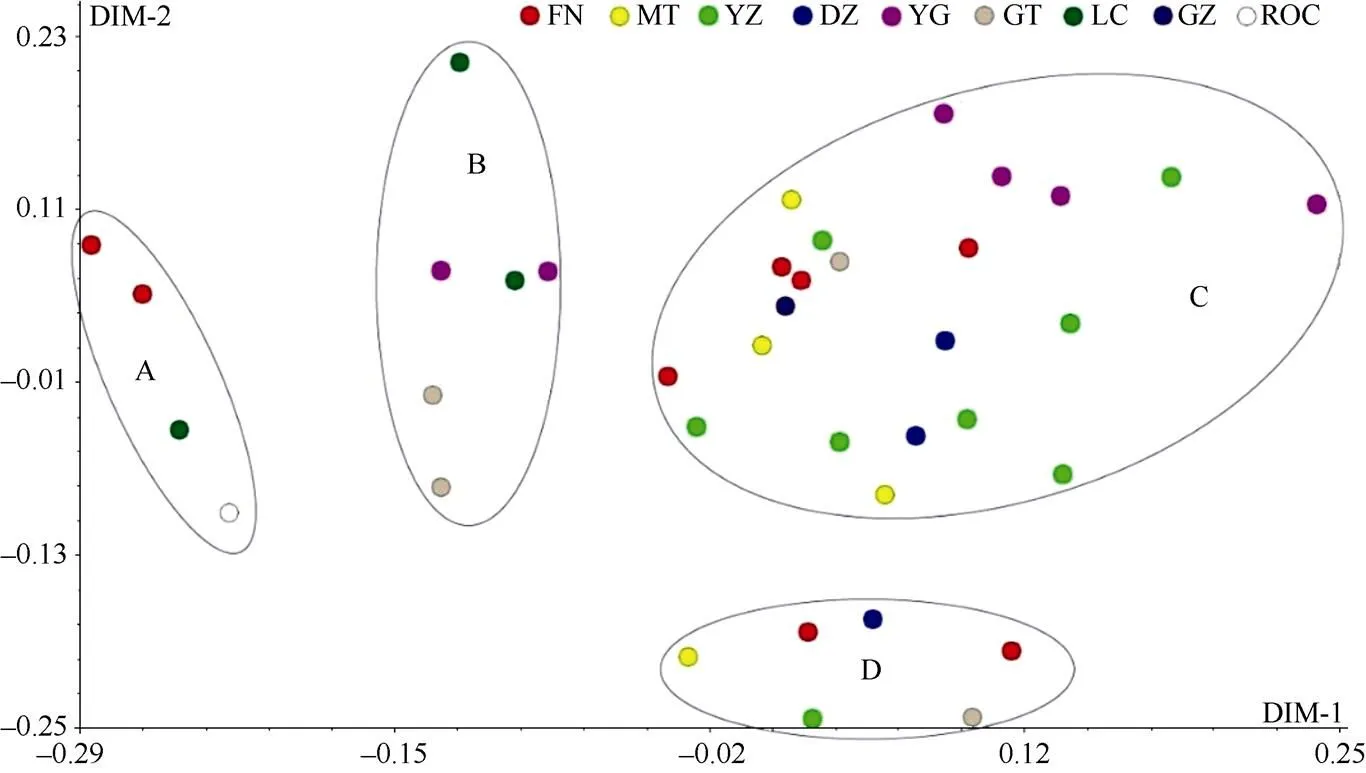
38个供试甘蔗新品种(系)(附表1)由福建农林大学农业部福建甘蔗生物学与遗传育种重点实验室提供。其中10个来自第10轮区试, 10个来自第11轮区试, 17个来自第12轮区试, 以及参照品种ROC22 (全国推广面积最大的品种), 均属于栽培种。在福建农林大学大学城试验基地种植所有材料, 在甘蔗分蘖期选取每个品种(系) 3片无病虫健康完全展开叶片, 用保鲜袋包装后带回实验室, 液氮速冻后置–80℃低温冰箱保存, 用于提取DNA样品。使用天根DNA提取试剂盒提取甘蔗基因组DNA, Synergy HT多功能酶标仪检测DNA质量, 选择1.8 对Vos等[19]建立的AFLP标记方法进行部分调整优化, 以9对多态性良好、扩增条带清晰的AFLP引物组合(表1)扫描38个甘蔗新品种(系)基因组。甘蔗基因组DNA经内切酶I和R I在37℃水浴酶切10 h; 酶切产物在37℃水浴连接特定的I和R I接头, 反应时间为2 h, 将反应产物稀释10倍后作为预扩增模板; 使用含有1个选择性核苷酸的I和R I的引物在反应条件为94℃ 30 s, 56℃ 60 s, 72℃ 60 s, 20个循环下预扩增, 将预扩增产物稀释20倍作为选择性扩增的模板; 以含3个选择性核苷酸的I和R I引物进行选择性扩增, PCR条件分为3个阶段, 第1阶段94℃ 30 s, 65℃ 30 s, 72℃ 60 s, 1个循环; 第2阶段94℃ 30 s, 65℃ 30 s, 72℃ 60 s, 12个循环, 从第2个循环开始退火温度每循环降低0.7℃; 第3阶段以94℃ 30 s, 56℃ 30 s, 72℃ 60 s, 23个循环结束。 表1 9对AFLP标记引物组合 表2中, 15对SSR引物由两个主要途径获取, 一是根据潘永保等[37-39]从国际微卫星标记联盟设计的221对引物筛选出的21对应用于甘蔗研究的引物中, 选择7对(编号1~7)多态性好的引物; 二是根据闫学兵等[40]从National Center for Biotechnology Information网站下载的EST序列设计筛选的26对EST-SSR引物中选择8对(编号8~15)多态性好的引物。将15对SSR引物和38个甘蔗DNA样品送到北京阅微基因有限公司进行毛细管电泳分型。 使用Microsoft Excel 2016软件将变性聚丙烯酰胺电泳结果和毛细管电泳结果都统计成0/1矩阵格式。SSR标记的电泳结果以吸收峰的形式表示, 参考Cordeiro等[41]的指纹数据统计方式, 在某位点出现吸收峰记“1”, 未出现记“0”, 统计范围为100~350 bp; 对AFLP标记使用“参考线法”记带, 即在电泳图上从下到上逐步移动参考线, 当遇到条带立即停止, 所停止位置记为一个位点, 在该位点参考线上出现条带的品种即在其相应的统计单元格记“1”, 未出现记“0”, 统计范围为1~500 bp。 表2 15对SSR标记引物 在数据分析与可视化上, 使用NTSYS 2.10e和PowerMarker V3.25软件计算各品种间的遗传相似性系数[42]、多态性位点数以及引物多态信息含量(Polymorphism Information Content, PIC)[43], 并使用基于Nei氏遗传距离的非加权类平均法(UPGMA)绘制聚类图。 式中,M表示2个品种共有的条带数,M+M表示两个品种一共被扩增出来的条带数;P和P分别为第个和第个等位基因频率,为等位基因数。使用R3.3.3绘制遗传相似性系数分布箱线图和甘蔗品种(系)的指纹图谱。 从图1可以清晰地辨识1~500 bp (统计区)内出现的变异位点。用AFLP标记的9对引物组合共扩增出348个位点, 多态性位点有248个, 多态性比率为71.26%。 图2中每个吸收峰代表1个扩增片段, 共5个扩增片段, 吸收峰所在X轴位置显示了扩增片段的长度, 具体数值标明在吸收峰的底部; Y轴代表扩增片段的浓度, 由吸收峰的高度决定。用SSR标记的15对引物共扩增出180个位点, 多态性位点有176个, 多态性比率达到92.78%。 由表3可知, SSR标记中每对引物的多态性条带的比率都处于很高水平, 12对引物达到100%, 其中多态性条带的比率最低的引物SMC334BS也达到83.33%, 表明15对SSR的引物的多态性良好, 鉴别品种间遗传差异的能力较高。另外, 各引物的平均PIC值达到0.920, 引物SEP84的PIC值最低, 为0.695。组合2的扩增片段多态性较差, 低于60%。PIC值平均水平达到0.971, 且各个引物间的PIC值相差很小。在总体统计基础上, 可看出SSR标记和AFLP标记的效用都达到较高水平, 彼此相当。 从图3可以看出, 着色部分为箱体, 箱体的上边框称为第三四分位数(Third Quartile), 下边框称为第一四分位数(First Quartile), 箱体内部靠近中部的线为中位数线(Median), 整个箱体部分表示数据的主要集中趋势。图中总体(ALL) 38个甘蔗新品种(系)和各系列(FN、MT、YZ、YG和GT)甘蔗品种(系)内部的遗传相似性系数都向0.725~0.770集中。虚线轴的最顶部为上限值(upper extreme), 最底部为下限值(lower extreme), 上下限值之间的范围就代表整个相似性系数的分布范围[44-45], 6组数据的分布范围都在0.680~0.830之间。FN系列品种(系)相似度水平的集中趋势和YZ系列接近, 并且与38个甘蔗品种总体的相似水平集中趋势接近; 另外, GT系列品种(系)的相似系数集中区域低于其他几个系列的品种, YG系列品种(系)的相似系数集中区域高于其他几个系列的品种。 图1 AFLP标记聚丙烯酰胺电泳图 图2 SSR标记毛细管电泳图 表3 24对引物的高效性评价 (续表3) ALL系列和FN系列品种(系)的箱线图上下边缘外存在异常值(黑色实心小圆点, Outlier), 异常值代表该组品种内部存在遗传距离很近(异常值处于上限值上部)或很远(异常值处于下限值下部)的极端情况(相对于本组数据而言), 这是表现相似系数分布特征的重要方面。在总体品种中(ALL), 因为品种数量多, 出现高相似组合或低相似组合的可能性更大, 也因此比FN系列出现了更多异常值。箱线图在比较不同群体的遗传相似系数的分布范围时具有较强的简明性, 同时各个群体的遗传相似系数的分布特征也能得到很好的体现(图3)。 图3 遗传相似性系数的分布特征 缩写同附表1。The abbreviations are the same as those given in Supplementary table 1. 从图4可以看出, 38个甘蔗新品种(系)遗传基础较为狭窄, 彼此间遗传距离接近。在遗传相似性系数为0.732处可将38个甘蔗品种(系)分为A、B两个群体。群体A包含4 (福农09-2201)和32 (桂糖06-1492)两个品种(系), 这2个品种(系)最先被划分出来, 说明它们在整个群体中异质性较强; 群体B包含剩下的36个品种(系)。在相似性系数为0.770处分割群体B, 可获得一个小的子群体a, 该子群体含有参照品种38 (ROC22)、2 (福农07-3206)、3 (福农40)、29 (海蔗22)、33 (桂糖09-12)和35 (柳城07-150), 说明这些品种(系)与参照品种ROC22同质性较高。从附表1可知, 29 (海蔗22)和35 (柳城07-150)的亲本中包含品种ROC22, 但2 (福农07-3206)、3 (福农40)和33 (桂糖09-12)的亲本不包括品种ROC22。以不同的遗传相似系数水平划分38个甘蔗品种的群体可以得到不同的子群体或不同的划分类型, 在不同群体间或类型间选择亲本搭配的组合, 其血缘关系的同质性相对于同一群体内或同一划分类型内的品种(系)组合会相对减小, 因此更有可能选育出优良品种。 图4 38份甘蔗品种(系)的聚类分析 从图5可以看出, MT系列、YZ系列和DZ系列甘蔗品种集中水平较高, 它们主要分布在主成分C中, 说明福建省农业科学院甘蔗研究所、云南省农业科学院甘蔗研究所、云南德宏州甘蔗科学研究所选用的甘蔗育种亲本具有较高的同质性, 也可能是育种者选择亲本时具有一定的偏好性。FN系列品种(系)分布在主成分A、C和D中, YG系列分布在B和C中, GT系列分布在B、C和D中, 它们的分散范围较广, 说明福建农林大学甘蔗综合研究所、广州甘蔗糖业研究所和广西农业科学院甘蔗研究所选用亲本的遗传范围较广, 育种者亲本的选择偏好性不强。总体来看, 各系列品种(系)的主成分区域都彼此靠近, 有些具有部分重合甚至完全重合, 可推测各个育种单位所选用的育种亲本的遗传背景接近, 不存在明显的差异性或者选择的偏向性。我们也可以依据图5进行亲本的选择, 依据分布距离与血缘同质性的关系, 图中距离较远(或分布于不同主成分中)的品种(系)都将是较为理想的亲本组合。 图5 38个甘蔗新品种(系)二维主成分分析 缩写同附表1。The abbreviations are the same as those given in Supplementary table 1. 从图6可以看出, 该图谱的指纹分布均匀, 并且品种(系)间都具有较为明显的差异, 说明构建图谱的位点选择恰当, 图谱的区分力度强。 利用分子标记技术鉴定种质资源遗传多样性, 首先需要考虑标记方法和引物的选择问题。Creste等[46]对AFLP、SSR和TRAP鉴定的82个甘蔗品种遗传系数分布结果比较发现, 3种标记鉴定的遗传系数分布特征不同, 它们各有自己的正态分布区域, SSR标记对整体品种亲缘关系的鉴定效率最高, 而AFLP标记更擅长于亲缘关系相近甘蔗品种的鉴别, 这告诉我们分子标记的选择需要从研究目的考虑, 每种标记方法有其自身的特点和优势。Mohamed等[47]通过对比SSR、SRAP和CAPS-SNP标记对柑橘品种多样性的鉴定结果, 也指出不同的标记各有优缺点, 并提出不同标记的结合使用对柑橘品种多样性的研究更为客观。位点信息量是影响分子标记技术鉴定结果稳定性最主要的因素, 其次是引物数量。对引物位点信息量和数量的评价可以预估分析结果的准确性[48], 所用引物的多态信息含量高、数量足, 得到的品种指纹信息更加丰富, 由此深入探讨的结果才更加可信。本研究的38个甘蔗新品种(系)的遗传背景接近, 将AFLP标记和SSR标记结合使用, 在保证足够的引物数量的同时, 也可以结合两种标记优势, 减少单分子标记鉴定的误差。另外, 15对SSR和9对AFLP引物组合的位点信息量高, 由此得到的甘蔗品种DNA指纹数据为后续的分析提供了可靠基础。 本研究首次使用箱线图分析各个品种(系)间的遗传相似性系数, 根据其分布特征判断和比较38个甘蔗新品种(系)以及各个系列甘蔗品种(系)的遗传基础。另外, 我们所拥有的种质资源群体越大, 越有可能找到极高或极低遗传差异的不同品种(系)组合, 这一点体现在箱线图的箱体宽度与异常值数量的关系上。38个甘蔗新品种(系)间相似度集中在0.725~0.780的水平, 说明第10、第11和第12轮甘蔗区试品种(系)遗传背景相似度都较高, 亲本的共祖度较高, 这也一定程度解释了为什么近些年来难有特别优异的新品种出现。在甘蔗生产中品种的单一化问题日渐严重, ROC系列品种在中国的种植面积超过80%, 种植时间已有20余年, 种质退化和减产等问题日益突出, 急需新的优良品种更新换代[49]。 亲本选择是育种的主要步骤, 亲本高效鉴定是杂交育种的基础工作, 对亲本的选择具有重要的指导作用[50-51]。育种中选择具有一定的遗传差异的种质资源作亲本, 可以提高杂种一代基因型的杂合性。对作物种质资源的遗传距离评估有利于鉴定和组合最佳亲本, 以产生遗传变异最大的子代群体和促进不同种质资源的优良基因渗透到新选育品系中,达到获取理想杂种优势的目的[51-52]。前人研究表明, 亲本的遗传距离与F1的杂种优势呈正相关, 据此可以预先评估新育品系的杂种优势[53]。本研究的聚类分析中, 4 (福农09-2201)和32 (桂糖06-1492)最先被划分出来, 说明它们与剩下的36个品种(系)存在较大遗传差异, 在群体中的异质性最强, 育种工作中可作为重点选用材料。 图6 38份甘蔗品种(系)的指纹图谱 Y轴方向的左边代表高特异性的扩增位点名(共60个位点, 位点名的前半部分是引物名, 后半部分是扩增片段长度, 全部选自SSR标记的扩增位点), X轴顶部代表各品种(系); 黑色或深灰色(方便相邻2个品种间的区分)的长方格子代表某品种(系)在该位点处有条带, 白色表示没有。 On the left of the Y-axis direction, 60 names of amplification loci with high specificity were listed (Those loci came from the SSR mark. The first half of the locus name is the primer name, and another half corresponds to the length of the amplified fragment). The serial numbers of the varieties (lines) were enumerated on the top of the X-axis direction. Each black or gray rectangular (distinguishes between adjacent varieties) represents an amplification band, and the white indicates no amplification band. 新品种通过审定(鉴定、认定或登记)后如何推广布局, 丰产性、稳定性和地区适应性是首先考虑的几个因素, 这些因素可由GGE-biplot与AMMI模型分析[5,34,36,49]; 其次, 根据基因丰富度对自然环境变化适应能力的关系原理, 考虑新品种的遗传背景因素。遗传背景过于相似的品种搭配推广, 对丰富群体的遗传多样性贡献有限, 不利于品种的多系布局[54], 而遗传差异大的品种在同一地区推广, 可以增加作物群体的基因范围, 增强其抗病抗逆等潜力。即推广与某地区甘蔗主栽品种遗传差异较大的优良甘蔗新品种, 不仅可以提高该地区甘蔗产量, 也可以在一定程度上增加该地区甘蔗群体的基因丰富度、增强甘蔗抵抗不良环境的能力和控制病虫害流行等[55]。合理分布和推广这些优异新品种, 能使甘蔗生产效益和生产安全性得到显著提高。在评估新品种的推广潜力时, 我们应该考虑新育成品种与现有的广受种植户认可的推广品种的遗传相似度, 这种相似度越大, 新品种在适应性、高产高糖性上越可能与现有优良品种接近, 也越容易受到种植户的接受而增加推广的机会。在本研究中, 2 (福农07-3206)、3 (福农40)、29 (海蔗22)、33 (桂糖09-12)和35 (柳城07-150)的遗传背景跟ROC22接近, 与ROC22聚到非常小的子群中的品种(系), 大多含有ROC22的血缘, 遗传了ROC22的广适应性特性, 产量品质与ROC22相当或优于ROC22, 具有被大面积推广的潜质。关于新品种推广或布局的建议和讨论, 主要是从遗传多样性的角度出发, 在实际的推广中还需要结合区域试验结果、抗病性表现和遗传系谱图等信息综合考虑。在2 (福农07-3206)、3 (福农40)、29 (海蔗22)、33 (桂糖09-12)、35 (柳城07-150)等几个品种(系)中, 2 (福农07-3206)、3 (福农40)和33 (桂糖09-12)的亲本不包括ROC22, 但是它们在亲缘关系上比10 (闽糖07-2005)、26 (粤甘47)、36 (柳城07-506)等亲本组合中包含ROC22的品种(系)更接近ROC22, 其原因可能是甘蔗育种中所使用的亲本之间具有较强的血缘同质性, 具体还要结合系谱图进一步分析。 在品种权保护、种质资源管理、种苗销售和新品种试验等工作中都需要品种(系)真实性的鉴定, 甘蔗品种(系)指纹图谱的绘制在规范品种和种质资源管理、规范甘蔗种苗市场和保护品种权益上都有十分重要的作用[32]。 将SSR和AFLP标记技术结合使用, 在获取丰富的甘蔗品种(系)指纹信息的同时, 互补了两种标记的优点。基于指纹信息利用箱线图研究各品种(系)间遗传相似性系数分布特征, 可清晰地展现出整个研究群体遗传基础的范围, 并可从中判断是否存在亲缘关系很远或很近的品种(系)组合。4 (福农09-2201)和32 (桂糖06-1492)等异质性较强的品种可作为重点利用的育种材料; 根据参照品种ROC22在聚类图中的位置, 可判定与其聚集到越小的群体中的品种(系)有较大可能拥有与其相似的田间优良性状(高产、稳产、高糖、抗病等), 从而更容易受到种植户的认可而具有更大的推广潜力。选择与参照品种异质性较强的品种与ROC22同时推广, 可一定程度上扩大甘蔗种植品种的多样性, 增强实际生产中甘蔗群体抵抗不良环境或病虫害等能力。本文在研究甘蔗遗传多样性的基础上讨论种质资源遗传基础的判定方法、种质资源的划分与利用方法、新品种的推广策略以及品种指纹图谱的构建与应用, 能给甘蔗育种等相关科研人员提供参考和借鉴。 [1] Menhas R, Umer S, Shabbir G. Climate change and its impact on food and nutrition security in Pakistan., 2016, 45: 549–550 [2] Kumar M. Impact of climate change on crop yield and role of model for achieving food security., 2016, 188: 1–14 [3] Tiepolo M. Urbanization and food security in Niamey, Niger., 2002, 26: 29–58 [4] Matsumoto K. Energy structure and energy security under climate mitigation scenarios in China., 2015, 10: e0144884 [5] Luo J, Pan Y B, Xu L P, Grisham M P, Zhang H, Que Y X. Rational regional distribution of sugarcane cultivars in China., 2015, 5: 15721 [6] Que Y X, Pan Y B, Lu Y H, Yang C, Yang Y T, Huang N, Xu L P. Genetic analysis of diversity within a Chinese local sugarcane germplasm based on start codon targeted polymorphism., 2014, (5): 468375 [7] 袁隆平. 杂交水稻超高产育种. 杂交水稻, 1997, 12(6): 1–3 Yuan L P. Hybrid rice breeding for super high yield., 1997, 12(6): 1–3 (in Chinese with English abstract) [8] Tang S X, Wei X H, Javier E L. Introduction and utilization of INGER rice germplasm in China., 2004, 3: 561–567 [9] Erskine W, Muehlbauer F J. Allozyme and morphological variability, outcrossing rate and core collection formation in lentil germplasm., 1991, 83: 119–125 [10] Nayak S, Naik P K, Acharya L, Mukherjee A K, Panda P C, Das P. Assessment of genetic diversity among 16 promising cultivars of ginger using cytological and molecular markers., 2005, 60: 485–492 [11] Wang C L, Singh D, Mitra S K. Biochemical markers: A useful tool for assessing genetic diversity in jackfruit (Lam.)., 2011, 890: 91–101 [12] Li H, Cao S Y, Niu J, Yuan P, Zhao D. The types and application of molecular markers in the study of pomegranate germplasm resources., 2015, 1089: 127–132 [13] Praveen K, Kumar M H, Umamaheshwari A, Reddy D M, Sudhakar P, Sabita N. SGDB: a sugarcane germplasm database., 2015, 17: 150–155 [14] Chen R K, Xu L P, Lin Y Q. Modern Sugarcane Genetic Breeding. Beijing: China Agriculture Press, 2011. pp 2–12 [15] 刘庆昌. 遗传学. 北京: 科学出版社, 2015. pp 286–288 Liu Q C. Genetics. Beijing: Science Press, 2015. pp 286–288 (in Chinese) [16] Ashfaq M, Khan A S. Genetic diversity in basmati rice (L.) germplasm as revealed by microsatellite (SSR) markers., 2012, 48: 53–62 [17] Shoaib A, Arabi M I E. Genetic diversity among syrian cultivated and landraces wheat revealed by AFLP markers., 2006, 53: 901–906 [18] Lenka D, Tripathy S K, Kumar R, Behera M, Ranjan R. Assessment of genetic diversity in quality protein maize (QPM) inbreds using ISSR markers., 2015, 36: 985–992 [19] Vos P, Hogers R, Bleeker M, Reijans M, van de Lee T, Hornes M, Frijters A, Pot J, Peleman J, Kuiper M. AFLP: a new technique for DNA fingerprinting., 1995, 23: 4407–4414 [20] Liu X L, Li X J, Xu C H, Lin X Q, Deng Z H. Genetic diversity of populations of, with different ploidy levels using SSR molecular markers., 2016, 18: 365–372 [21] Lander E S. The new genomics: global views of biology., 1996, 274: 536–539 [22] Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification., 1994, 20: 176–183 [23] Duarte Filho L S C, Silva P P, Santos J M, Barbosa G V S, Ramalho-Neto C E, Soares L, Andrade J C F, Almeida C. Genetic similarity among genotypes of sugarcane estimated by SSR and coefficient of parentage., 2010, 12: 145–149 [24] Russell J R, Fuller J D, Macaulay M, Hatz B G, Jahoor A, Powell W, Waugh R. Direct comparison of levels of genetic variation among barley accessions detected by RFLPs, AFLPs, SSRs and RAPDs., 1997, 95: 714–722 [25] Pejic I, Ajmone-Marsan P, Morgante M, Kozumplick V, Castiglioni P, Taramino G, Motto M. Comparative analysis of genetic similarity among maize inbred lines detected by RFLPs, RAPDs, SSRs, and AFLPs., 1998, 97: 1248–1255 [26] Guichoux E, Lagache L, Wagner S, Chaumeil P, Léger P, Lepais O O, Lepoittevin C, Malausa T, Revardel E, Salin F, Petit R J. Current trends in microsatellite genotyping., 2011, 11: 591–611 [27] 刘新龙, 蔡青, 毕艳, 陆鑫, 马丽, 应雄美. 中国滇蔗茅种质资源遗传多样性的AFLP分析. 作物学报, 2009, 35: 262–269 Liu X L, Cai Q, Bi Y, Lu X, Ma L, Ying X M. Genetic diversity analysis for germplasm ofin China., 2009, 35: 262–269 (in Chinese with English abstract) [28] Lima M L, Garcia A A, Oliveira K M, Matsuoka S, Arizono H, De Souza C L Jr, De Souza A P. Analysis of genetic similarity detected by AFLP and coefficient of parentage among genotypes of sugar (spp.)., 2002, 104: 30–38 [29] Barret B A, Kidwell K K. Comparison of AFLP and pedigree-based genetic diversity assessment methods using wheat cultivars from the Pacific Northwest., 1998, 38: 1271–1278 [30] Singh R K, Singh R B, Singh S P, Mishra N, Rastogi J, Sharma M L, Kumar A. Genetic diversity amongclones and commercial hybrids through SSR markers., 2013, 15: 109–115 [31] 齐永文, 劳方业, 张垂明, 樊丽娜, 何慧怡, 刘少谋, 李奇伟, 邓海华. 中美重要甘蔗种质SSR遗传多样性比较. 热带作物学报, 2011, 32: 99–104 Qi Y W, Lao F Y, Zhang C M, Fan L N, He H Y, Liu S M, Li Q W, Deng H H. Comparative analysis of genetic diversity of Chinese and American sugarcane (spp.) using SSR makers., 2011, 32: 99–104 (in Chinese with English abstract) [32] 刘新龙, 马丽, 陈学宽, 应雄美, 蔡青, 刘家勇, 吴才文. 云南甘蔗自育品种DNA指纹身份证构建. 作物学报, 2010, 36: 202–210Liu X L, Ma L, Chen X K, Ying X M, Cai Q, Liu J Y, Wu C W. Establishment of DNA fingerprint ID in sugarcane cultivars in Yunnan, China., 2010, 36: 202–210 (in Chinese with English abstract) [33] 姚春雪, 王先宏, 何丽莲, 李富生. 甘蔗与蔗茅杂交不同世代的SSR指纹图谱构建. 分子植物育种, 2011, 9: 381–389Yao C X, Wang X H, He L L, Li F S. DNA fingerprint construction of different generations ofspp. ×using SSR marker., 2011, 9: 381–389 (in Chinese with English abstract) [34] 汪洲涛, 苏炜华, 阙友雄, 许莉萍, 张华, 罗俊. 应用AMMI和HA-GGE双标图分析甘蔗品种产量稳定性和试点代表性. 中国生态农业学报, 2016, 24: 790–800Wang Z T, Su W H, Que Y X, Xu L P, Zhang H, Luo J. Analysis of yield stability and test site representativeness of sugarcane trials using combined AMMI and HA-GGE biplot models., 2016, 24: 790–800 (in Chinese with English abstract) [35] 罗俊, 邓祖湖, 阙友雄, 袁照年, 陈如凯. 国家甘蔗第七轮区域试验品种的丰产性及稳定性. 应用与环境生物学报, 2012, 18: 734–739Luo J, Deng Z H, Que Y X, Yuan Z N, Chen R K. Productivity and stability of sugarcane varieties in the 7th round national regional trial of China., 2012, 18: 734–739 (in Chinese with English abstract) [36] Luo J, Pan Y B, Que Y X, Zhang H, Grisham M P, Xu L P. Biplot evaluation of test environments and identification of mega-environment for sugarcane cultivars in China., 2015, 5: 15505 [37] Pan Y B. Application of microsatellite and RAPD fingerprints in the Florida sugarcane variety program., 2003, March/April: 19–28 [38] Pan Y B, Scheffler B E, Richard Jr E P. High-throughput molecular genotyping of commercial sugarcane clones with microsatellite (SSR) markers., 2007, 9: 176–181 [39] Pan Y B. Databasing molecular identities of sugarcane (spp.) clones constructed with microsatellite (SSR) DNA markers., 2010, 1: 87–94 [40] 闫学兵, 阙友雄, 许莉萍, 郭晋隆, 陈如凯, 潘永保. 甘蔗EST序列的SSR信息分析. 热带作物学报, 2010, 31: 1497–1501Yan X B, Que Y X, Xu L P, Guo J L, Chen R K, Pan Y B. Analysis of SSR information in EST resources of sugarcane., 2010, 31: 1497–1501 (in Chinese with English abstract) [41] Cordeiro G M, Pan Y B, Henry R J. Sugarcane microsatellites for the assessment of genetic diversity in sugarcane germplasm., 2003, 165: 181–189 [42] Aitken K S, Jackson P A, McIntyre C L. A combination of AFLP and SSR markers provides extensive map coverage and identification of homo(eo)logous linkage groups in a sugarcane cultivar., 2005, 110: 789–801 [43] Milbourne D, Meyer R, Bradshaw J E, Baird E, Bonar N, Provan J, Powell W, Waugh R. Comparison of PCR-based marker systems for the analysis of genetic relationships in cultivated potato., 1997, 3: 127–136 [44] Streit M, Gehlenborg N. Bar charts and box plots., 2014, 11: 117 [45] Nuzzo R L. The Box plots alternative for visualizing quantitative data., 2016, 8: 268–272 [46] Creste S, Sansoli D M, Tardiani A C S, Silva D N, Goncalves F K, Favero T M, Medeiros C N F, Festucci C S, Carlini-Garcia L A, Landell M G A, Pinto L R. Comparison of AFLP, TRAP and SSRs in the estimation of genetic relationships in sugarcane., 2010, 12: 150–154 [47] Mohamed H A, Manosh K B, Zhang Z W, Guo W W. Exploitation of SSR, SRAP and CAPS-SNP markers for genetic diversity of citrus germplasm collection., 2011, 128: 220–227 [48] 高伟, 王坤波, 刘方, 王春英, 张香娣, 王玉红, 黎绍惠. SSR引物及多态性位点数对陆地棉野生种系聚类结果的影响. 植物遗传资源学报, 2013, 14: 237–242Gao W, Wang K B, Liu F, Wang C Y, Zhang X D, Wang Y H, Li S H. Effection of the quantity of SSR primer and allele on cluster analysis ofLinn races., 2013, 14: 237–242 (in Chinese with English abstract) [49] Luo J, Pan Y B, Xu L P, Zhang H, Yuan Z N, Deng Z H, Chen R K, Que Y X. Cultivar evaluation and essential test locations identification for sugarcane breeding in China., 2014: 302753 [50] Benin G, Matei G, Costa d O A, Silva G O, Hagemann T R, Lemes da Silva C, Pagliosa E S, Beche E. Relationships between four measures of genetic distance and breeding behavior in spring wheat., 2012, 11: 2390–2400 [51] Yao J, Li H, Ye J, Shi L L. Relationship between parental genetic distance and offspring’s heterosis for early growth traits in Liriodendron: implication for parent pair selection in cross breeding., 2016, 47: 163–177 [52] Longin F H, Liu W X, Ranc N, Reif J C. Association of progeny variance and genetic distances among parents and implications for the design of elite maize breeding programs., 2011, 56: 227–231 [53] Wegary D, Vivek B, Labuschagne M. Association of parental genetic distance with heterosis and specific combining ability in quality protein maize., 2013, 191: 205–216 [54] 戎俊, 杨小强, 耿宇鹏, 宋志平, 卢宝荣. 分子生态学(第2版). 北京: 高等教育出版社, 2015. pp 249–250 Rong J, Yang X Q, Geng Y P, Song Z P, Lu B R. Molecular Ecology (2nd edn). Beijing: Higher Education Press, 2015. pp 249–250 (in Chinese) [55] 朱有勇. 农业生物多样性与作物病虫害控制. 北京: 科学出版社, 2013. pp 9–27Zhu Y Y. Agricultural Biodiversity and Control of Crop Diseases and Insect Pests. Beijing: Science Press, 2013. pp 9–27 (in Chinese) 附表1 38份来自3轮国家甘蔗品种区域试验的品种(系) Supplementary table 1 Thirty-eight sugarcane varieties (lines) from three rounds of national variety regional testings 编号No.品种(系)Variety (line)亲本组合Parent combination选育单位Breeding institution参试轮次Round系列Series 1福农07-2020Funong 07-2020粤糖91-976 × LC 85-384Yuetang 91-976 × LC 85-384福建农林大学甘蔗综合研究所SIFAFU10FN 2福农07-3206Funong 07-320690-1211 × 77-797福建农林大学甘蔗综合研究所SIFAFU10FN 3福农40Funong 40福农93-3406 × 粤糖91-976Funong 93-3406 × Yuetang 91-976福建农林大学甘蔗综合研究所SIFAFU10FN 4福农09-2201Funong 09-2201ROC 22 × 桂糖00-122ROC 22 × Guitang 00-122福建农林大学甘蔗综合研究所SIFAFU11FN 5福农09-7111Funong 09-7111桂糖96-44 × ROC 11Guitang 96-44 × ROC 11福建农林大学甘蔗综合研究所SIFAFU11FN 6福农09-12206Funong 09-12206CP 65-357 × 崖城97-40CP 65-357 × Yacheng 97-40福建农林大学甘蔗综合研究所SIFAFU12FN 7福农11-2105Funong 11-2105川蔗89-103 × 云瑞05-770Chuanzhe 89-103 × Yunrui 05-770福建农林大学甘蔗综合研究所SIFAFU12FN 8福农09-4095Funong 09-4095粤糖93-159 × 云蔗91-790Yuetang 93-159 × Yunzhe 91-790福建农林大学甘蔗综合研究所SIFAFU12FN 9闽糖06-1405Mintang 06-1405闽糖92-649 × ROC 10Mintang 92-649 × ROC 10福建省农业科学院甘蔗研究所SRIFAAS11MT 10闽糖07-2005Mintang 07-2005崖城73-512 × ROC 22Yacheng 73-512 × ROC 22福建省农业科学院甘蔗研究所SRIFAAS12MT 11闽糖09-104Mintang 09-104桂糖90-420 × ROC 10Guitang 90-420 × ROC 10福建省农业科学院甘蔗研究所SRIFAAS12MT (续附表1) 编号No.品种(系)Variety (line)亲本组合Parent combination选育单位Breeding institution参试轮次Round系列Series 12闽糖02-205Mintang 02-205崖城90-3 × ROC 10Yacheng 90-3 × ROC 10福建省农业科学院甘蔗研究所SRIFAAS10MT 13云蔗08-2060Yunzhe 08-2060粤糖93-159 × Q 121Yuetang 93-159 × Q 121云南省农业科学院甘蔗研究所SRIYAAS10YZ 14云蔗08-1095Yunzhe 08-1095CP 84 -1198 × 科5CP 84-1198 × Ke5云南省农业科学院甘蔗研究所SRIYAAS11YZ 15云蔗09-1028Yunzhe 09-1028云瑞15-178 × 闽糖86-2121Yunrui 15-178 × Mintang 86-2121云南省农业科学院甘蔗研究所SRIYAAS12YZ 16云蔗09-1601Yunzhe 09-1601CP 94-1100 × 川糖89-103CP 94-1100 × Chuantang 89-103云南省农业科学院甘蔗研究所SRIYAAS12YZ 17云瑞09-315Yunrui 09-315CL 69-52 × 云瑞05-285CL 69-52 × Yunrui 05-285云南省农业科学院甘蔗研究所SRIYAAS12YZ 18云瑞07-1433Yunrui 07-1433云瑞99-155 × L 75-20Yunrui 99-155 × L75-20云南省农业科学院甘蔗研究所SRIYAAS10YZ 19云瑞10-187Yunrui 10-187ROC 20 × 云瑞05-282ROC20 × Yunrui 05-282云南省农业科学院甘蔗研究所SRIYAAS12YZ 20云瑞10-701Yunrui 10-701云瑞08-18 × 云瑞05-704Yunrui 08-18 × Yunrui 05-704云南省农业科学院甘蔗研究所SRIYAAS12YZ 21德蔗07-36Dezhe 07-36桂糖92-66 × CP 67-412Guitang 92-66 × CP 67-412云南德宏州甘蔗科学研究所SRIDY11DZ 22德蔗09-78Dezhe 09-78桂糖94-119 × ROC 10Guitang 94-119 × ROC 10云南德宏州甘蔗科学研究所SRIDY12DZ 23德蔗09-84Dezhe 09-84桂糖94-119 × ROC 10Guitang 94-119 × ROC 10云南德宏州甘蔗科学研究所SRIDY12DZ 24粤甘43Yuegan 43粤糖93-213 × 粤糖93-159Yuetang 93-213 × Yuetang 93-159广州甘蔗糖业研究所GSIRI10YG 25粤甘46Yuegan 46粤糖00-236 × 桂糖96-211Yuetang 00-236 × Guitang 96-211广州甘蔗糖业研究所GSIRI10YG 26粤甘47Yuegan 47粤农73-204 × ROC 22Yuenong 73-204 × ROC 22广州甘蔗糖业研究所GSIRI11YG 27粤甘48Yuegan 48HOCP 95-988 × 粤糖97-76HOCP 95-988 × Yuetang 97-76广州甘蔗糖业研究所GSIRI12YG 28粤甘50Yuegan 50粤糖96-86 × 粤糖99-66Yuetang 96-86 × Yuetang 99-66广州甘蔗糖业研究所GSIRI12YG 29海蔗22Haizhe 22粤糖93-159 × ROC 22Yuetang 93-159 × ROC 22广州甘蔗糖业研究所GSIRI11YG 30桂糖06-2081Guitang 06-2081桂糖00-122 × 崖城97-47 Guitang 00-122 × Yacheng 97-47广西农业科学院甘蔗研究所SRIGAAS11GT 31桂糖08-1180Guitang 08-1180ROC 26 × ROC 22广西农业科学院甘蔗研究所SRIGAAS11GT 32桂糖06-1492Guitang 06-1492CP 72-1210 × 湛蔗92-126CP 72-1210 × Zhanzhe 92-126广西农业科学院甘蔗研究所SRIGAAS12GT 33桂糖09-12Guitang 09-12ROC 24 × 粤农79-780ROC 24 × Yuenong 79-780广西农业科学院甘蔗研究所SRIGAAS12GT (续附表1) 编号No.品种(系)Variety (line)亲本组合Parent combination选育单位Breeding institution参试轮次Round系列Series 34柳城07-500Liucheng 07-500粤糖92-1287 × CP 72-1210Yuetang 92-1287 × CP 72-1210柳城县甘蔗研究中心SRCLC10LC 35柳城07-150Liucheng 07-150粤糖85-177 × ROC 22Yuetang 85-177 × ROC 22柳城县甘蔗研究中心SRCLC11LC 36柳城07-506Liucheng 07-506粤糖85-177 × ROC 22Yuetang 85-177 × ROC 22柳城县甘蔗研究中心SRCLC12LC 37赣蔗07-538Ganzhe 07-538ROC 10 × CP 57-614江西省甘蔗研究所SRIJP10GZ 38ROC 22台湾糖业研究所SPRIT参照ROC FN: 福建农林大学甘蔗综合研究所选育的甘蔗品系; MT: 福建省农业科学院甘蔗研究所选育甘蔗品系; YZ: 云南省农业科学院甘蔗研究所选育的甘蔗品系; DZ: 云南德宏州甘蔗科学研究所选育的甘蔗品系; YG: 广州甘蔗糖业研究所选育的甘蔗品系; GT: 广西农业科学院甘蔗研究所选育的甘蔗品系; LC: 柳城县甘蔗研究中心选育的甘蔗品系; GZ: 江西省甘蔗研究所选育的甘蔗品系; ROC: 台湾糖业研究所选育的甘蔗品种。 SIFAFU: Sugarcane Institute, Fujian Agriculture and Forestry University; SRIFAAS: Sugarcane Research Institute, Fujian Academy of Agricultural Sciences; SRIYAAS: Sugarcane Research Institute, Yunnan Academy of Agricultural Sciences; SRIDY: Sugarcane Research Institute, Dehong, Yunnan Province; GSIRI: Guangzhou Sugarcane Industry Research Institute; SRIGAAS: Sugarcane Research Institute of Guangxi Academy of Agricultural Sciences; SRCLC: Sugarcane Research Center of Liucheng County; SRIJP: Sugarcane Research Institute of Jiangxi Province; SPRITP: Sugar Processing Research Institute of Taiwan Province. FN: all sugarcane lines bred by SIFAFU; MT: all sugarcane lines bred by SRIFAAS; YZ: all sugarcane lines bred by SRIYAAS; DZ: all sugarcane lines bred by SRIDY; YG: all sugarcane lines bred by GSIRI; GT: all sugarcane lines bred by SRIGAAS; LC: all sugarcane lines bred by SRCLC; GZ: all sugarcane lines bred by SRIJP; ROC: sugarcane variety bred by SPRITP. Identification of Sugarcane Varieties by AFLP and SSR Markers and Its Application WANG Zhou-Tao, YOU Qian, GAO Shi-Wu, WANG Chun-Feng, LI Zhu, MA Jing-Jing, QUE You-Xiong, XU Li-Ping, and LUO Jun* Key Laboratory of Sugarcane Biology and Genetic Breeding (Fujian), Ministry of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China Genetic diversity and fingerprint of varieties are crucial reference for breeding, cultivar right protection and new cultivar extension. We used nine pairs of AFLP markers and 15 pairs of SSR markers to scan 38 new sugarcane varieties (lines) from the national variety regional testings, obtaining abundant fingerprint data. Among the total 348 AFLP bands, 248 were polymorphic, with a polymorphism rate of 72.26 %. In addition, among the total 180 SSR bands, 176 were polymorphic, with a polymorphism rate of 97.78 %. The genetic similarity coefficients of these 38 new varieties (lines) were distributed from 0.668 to 0.847. We explored the distribution characteristics of genetic similarity coefficients using the boxplot, observing the genetic basis of five series (FN, MT, YZ, YG, and GT) of the 38 new sugarcane varieties (lines) was approximately similar. The clustering analysis manifested that these 38 new sugarcane varieties (lines) were divided into two groups at the genetic similarity coefficient of 0.732, with a subgroup including FN09-2201 and GT06-1492 that had high heterogeneity. Moreover, there was a small subgroup containing ROC22 at the genetic similarity coefficient of 0.770. Except for ROC22, the subgroup also contained FN 07-3206, FN 40, HZ 22, GT 09-12, and LC 07-150. ROC22 has wide adaptability, high yield, high sugar content and other excellent characteristics, other varieties (lines) in the same subgroup should be more likely to have these excellent characteristics and higher extention potential. Finally, through selection of 60 efficient amplification sites in the identification of SSR markers, we constructed the fingerprints of these 38 new sugarcane varieties (lines), which should play an important role in variety identification and variety protection. This study is expected to be directly applied to guide the genetic diversity assessment and molecular fingerprinting identification of sugarcane germplasm, and also to provide references for the extention and layout of these varieties or their utilization as hybrid parents. sugarcane; molecular marker; germplasm identification; group division; variety extension 2017-10-13; 2018-03-15; 2018-03-16. 10.3724/SP.J.1006.2018.00723 本研究由国家现代农业产业技术体系建设专项(CARS-17)和引进国际先进农业科学技术计划(948计划)项目(2014-S18)资助。 This study was supported by China Agriculture Research System (CARS-17) and the Program of Introducing International Super Agricultural Science and Technology (948 Program) (2014-S18). 罗俊, E-mail: sisluojun@126.com http://kns.cnki.net/kcms/detail/11.1809.S.20180316.0853.004.html1.2 AFLP标记
1.3 SSR标记
1.4 数据统计与分析

2 结果与分析
2.1 AFLP标记与SSR标记电泳结果
2.2 引物的有效性
2.3 遗传相似性系数的分布特征


2.4 聚类分析

2.5 主成分分析
2.6 甘蔗品种(系)指纹图谱绘制
3 讨论

4 结论