食品中转基因成分核酸检测技术研究进展
2018-04-11徐春祥颜春荣吴小芹
方 昕,徐春祥,颜春荣,吴小芹
(1.江苏省食品药品监督检验研究院,江苏 南京 210008;2.南京林业大学 林学院,江苏 南京 210037)
转基因生物是指利用DNA重组技术,将外源基因导入受体基因组,从而使受体获得某种新的性状而得到的生物[1]。以转基因生物为原料加工生产得到的食品为转基因食品。合理运用转基因技术,可以培育具有优良特性的作物新品种,从而提高单位面积产量,改善作物品质,减少作物对农药的依赖,降低生产成本,减少环境污染,提高经济效益[2-4]。
1994年,美国首次批准耐储存的转基因番茄进入市场销售,随后,抗虫棉花、抗除草剂大豆、抗虫玉米和油菜等一系列具有改良性状的转基因作物相继在多个国家开始大规模种植[5]。自1996年起,全世界种植转基因作物面积逐年递增,2016年达1.85亿公顷(图1),覆盖全球26个国家[6]。截至2016年,我国转基因作物面积为280万公顷,占世界种植面积的2%[6]。我国的转基因作物主要有两种来源途径,一类是农业部批准商业化种植和生产的作物,包括抗虫棉花和抗病毒番木瓜;另一类是经过安全评价审批的进口转基因作物,包括玉米、大豆、油菜、甜菜和棉花。
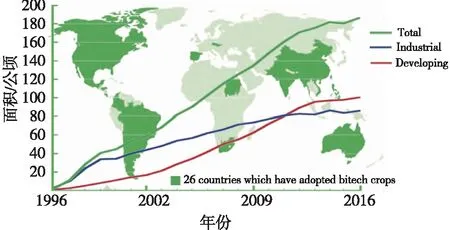
图1 1996—2016年全球转基因作物种植面积[6](百万公顷)Fig.1 Global area of GM crops from 1996 to 2006[6] (million hectares)
转基因技术在带来产量、经济效益大幅提升的同时,其产品安全性却一直备受争议[7]。针对转基因食品的潜在风险,全面且严格的监管措施不可或缺,而精准的检测和科学评估则是转基因食品监管重要的技术基础。因此,对转基因产品的科学评估和准确的检测对转基因食品监管有着重要的意义。
转基因成分检测方法可以从核酸、转录水平、蛋白表达及表型等层面进行探索。基因的修改可以通过转录水平的方法来检测,如合成新的转录子或RNA沉默[8]。然而,基因的转录调控容易受到外在环境条件的影响,并且,在生物不同的生长发育阶段,同一基因的转录水平也不尽相同,这些因素容易干扰检测的稳定性和可重复性。外源基因的表达蛋白,也可以作为转基因检测的目标物。然而,基于蛋白的检测技术有一定的局限性:首先,检测对象中必须含有已知蛋白,抗体制备困难大,制备周期长,不适合新品种转基因食品的检测;其次,检测的准确性受到样品基质的影响,不适合深加工或复杂样品;再次,和核酸检测技术相比,蛋白类方法灵敏度较低,不适合转基因成分较少的样品[9]。转基因作物新表型的出现往往也可以作为判断转基因的依据,但是,这种方法需要对植物进行培育和观察,不仅周期长,同时还需要排除自发突变株干扰,因此,并不适合作为检测转基因成分的技术[10]。
实践中, 基于核酸的转基因检测技术因其较高的灵敏度和良好的特异性而获得广泛认可和应用[11-13]。本文中,笔者主要针对当今国内外转基因成分的核酸分析检测技术的原理、应用及方法评价进行综述,以期为转基因食品的分析检测提供更多思路。
1 检测技术
1.1 目标基因选择策略
依据核酸目标序列的位置和特征,转基因检测技术可以分为外源基因特异性(gene specific)检测、筛选/元件特异性(screeing/element specific)检测、构建特异性(construct specific)检测和转化事件特异性(event specific)检测[10]。外源基因特异性检测是以插入的外源基因为检测靶序列,此方法不能区别导入相同外源基因的不同作物及品系,具有一定的局限性。筛选/元件特异性检测是以CaMV35S启动子、nos终止子等转录元件或者标记作为检测靶序列,此方法可以对转基因成分进行快速筛选。构建特异性检测的目标序列是外源基因和转录元件的链接区域,然而,该方法不能区别包含相同外源基因的不同转基因品系,不能提供具体的转基因品系信息[14]。转化事件特异性检测的目标序列是导入的外源DNA与受体结合位点边界的序列。该方法包含了受体信息和转基因构建信息,不仅确定了外源基因的插入位点,还明确了外源基因的拷贝数,是目前特异性最高的转基因检测技术[15]。
1.2 常规聚合酶链式反应(PCR)
PCR是核酸检测中最常用的方法,目前该方法的研究成熟、应用广泛,常见的PCR方法包括常规PCR、巢式PCR(nested PCR)、多重PCR(multiplex PCR)和实时荧光定量PCR(real-time PCR)等。
常规PCR是在DNA聚合酶作用下,经过变性、退火和延伸3个步骤的循环而达到DNA扩增的技术,是定性判断转基因成分的最简单有效的方法之一。该方法特异性好、灵敏度高、速度快,早在2003年就被我国出入境检验检疫系统列为行业标准方法。
巢式PCR是利用内、外两对引物的PCR反应。第一对引物(外引物)扩增片段与常规PCR相似,第二对引物(内引物)结合在第一次PCR产物内部。巢式PCR的优势主要体现在两个方面:一是错配率低、特异性高,两轮PCR引物不同,降低了非特异性反应被连续放大的可能性;二是灵敏度高,两轮PCR克服了单次扩增平台期效应的限制,使扩增倍数提高,可以实现对DNA被破坏的深加工食品中转基因成分的检测。Ao等[16]采用多重巢式PCR 检测了深加工食品中大米、玉米和大豆转基因成分,灵敏度高达到0.005%。Brod等[17]运用巢式PCR 技术成功检测出大豆制品中的转基因成分。该方法的缺点在于操作步骤多,增加了污染的几率,假阳性可能性较大。
多重PCR 是在同一个反应体系中加入多对引物、同时扩增多个目标序列的PCR反应。该技术的优势在于快速高效,可以同时检测多个基因,适合识别包含受体信息和外源信息的转化事件,曾一度成为转基因检测研究的热点。Cheah等[18]开发出以EPSPS和Cry1Ab为目标基因的大豆及玉米转基因成分的多重PCR检测方法,检测限达0.25%。在欧洲,多重PCR被用来同时筛选多种转基因食品或作物,检测过程高效,结果准确[19-20]。多重PCR技术的难点在于引物的设计,必须保证各对引物的特异性,多对引物之间不形成引物二聚体,多对引物最佳退火温度接近。
实时荧光定量PCR是在常规PCR的基础上,通过添加荧光染料或者使用荧光探针来实时监控扩增反应过程的技术。常用的实时荧光定量PCR主要有两类:一是TaqMan探针法,其原理是利用双荧光标记的TaqMan探针解离产生的荧光来监测PCR产物;二是荧光染料法,常用的染料为SYBR Green,其原理是利用染料分子与双链DNA特异性结合而产生荧光来监测PCR反应。Kaur等[21]运用TaqMan实时荧光定量PCR成功检测出转基因玉米中8个外源基因。Köppel等[22]建立了以大豆和玉米为原料的转基因食品多重实时荧光PCR检测方法。和常规PCR相比,实时荧光定量PCR有两点优势:一是可以实时监测扩增产物,无需产物分析步骤,检测过更加简便快速;二是可以通过构建标准曲线,实现扩增产物的定量分析。目前,荧光定量PCR方法已经成为国内外最常用的转基因检测方法。尽管如此,该方法还是受到仪器设备的限制,不适合快速检测和现场检测。
1.3 数字PCR (Digital PCR)
数字PCR是继常规PCR和实时荧光定量PCR之后,可以绝对定量核酸拷贝数的第三代PCR技术。该技术将样品分散到数万个反应单元中,使得反应单元仅包含模板单分子,并在反应结束后收集各反应单元的荧光信号,最后通过统计学原理计算样品中核酸的含量[23]。目前常见的数字PCR主要有两大类,一是芯片数字PCR,二是微滴数字PCR。前者利用芯片上的数万个微孔将样品模板单分子化,后者是将扩增体系分散为数百万油状液体包裹的小液滴,从而实现样品模板单分子化。
2013年,Morisset等[24]运用数字PCR对食品及饲料中的转基因成分进行了精确的定量分析。Dobnik等[25]基于微滴数字PCR技术,建立了针对欧盟授权的12 种转基因玉米品系转基因成分的多重定量分析方法。Iwobi等[26]运用微滴数字PCR快速分析不同食品的转基因成分含量,检出限可达到0.1%。
和荧光定量PCR相比,数字PCR不依赖扩增曲线和标准曲线就可以实现绝对定量分析。同时,该技术结合了荧光定量PCR和高通量检测,为大量样本筛选提供了技术支持,在转基因检测方面具有较大的应用前景。
1.4 等温扩增技术
等温扩增技术是指在恒定温度下对DNA进行扩增的技术。与常规的PCR相比,等温扩增技术不需要DNA的热变形和温度的循环,在较低的温度下就能实现DNA的扩增。根据单链模板形成的方式不同,等温扩增可以分为以下4类[27]:① 链置换DNA聚合酶介导的反应,如环介导等温扩增(LAMP)、滚环扩增(RCA);② 酶促解旋引物退火反应,如依赖解旋酶的扩增(HDA)、重组酶介导扩增(RPA);③ 基于RNA转录的扩增,如转录介导扩增(TMA);④ 需要单链剪切酶辅助的反应,如链置换扩增(SDA)。
LAMP技术由日本Eiken公司[28]2000年研发,该技术利用特殊设计的4条引物,特行性地识别DNA模板上的6个区域,在聚合酶作用下,外侧引物延伸使得内侧引物形成的子链脱离,从而再生出新的引物结合位点,继而引发新的扩增。LAMP的扩增效率高,可在短时间内产生大量的扩增产物及副产物,从而衍生出了浊度法、钙黄绿素法和SYBR Green荧光法等产物分析方法。其中,比较常见的是浊度法,LAMP扩增中产生大量的副产物焦磷酸根离子能够与反应液中的Mg2+结合生成沉淀,大量累积时就会形成肉眼可见的浑浊。和常规PCR相比,LAMP具有灵敏度高、特异性高、不依赖精密仪器和结果判读快速等优点。该方法简化了扩增产物分析的步骤,适合快速检测和现场检测。闫兴华等[29]以cordapA作为靶基因设计4条引物,运用LAMP技术在50 min内成功检测出转基因玉米LY038。
SDA是利用限制性内切酶活性和具有链置换活性DNA聚合酶实现的扩增反应。SDA在两对引物和两种功能酶的作用下,通过“酶切-延伸-替代”的循环方式使靶序列得以扩增。
NASBA是基于RNA的等温核酸扩增技术。模板RNA逆转录形成RNA-DNA杂交体,RNase H酶降解杂交体的RNA,单链DNA作为模板合成新的双链DNA,后者被T7RNA聚合酶转录出靶RNA,由此进入新一轮扩增循环,不断形成靶核酸分子[30]。Morisset等[31]利用NASBA技术建立了转基因玉米MON863和MON810的多重检测方法,证明了该技术在转基因检测方面的可应用性。
等温扩增技术的出现,降低了检测过程对设备的要求,并使得检测结果可视化。但该方法设计较为复杂,检测成本高,也在一定程度上限制了其应用发展。
1.5 侧翼序列PCR 技术
侧翼序列是指目标基因两侧的DNA序列,其中包含着许多重要的转录和表达调控因子。在转基因作物研究中,外源基因插入位点的侧翼序列对目的基因的正常转录和表达有着重要影响。传统的侧翼序列分析方法包括反向PCR[32]、连接介导PCR[33]、交错式热不对称PCR(Tail-PCR)[34-36]、随机引物PCR[37]和特异连接PCR[38]等。
反向PCR是将基因组酶切破碎,通过环化实现反向引物扩增,最后测序判断产物序列信息的一项技术[9]。反向PCR 可以快速扩增一个已知基因片段的两侧序列。由于侧翼序列位置,因此选择合适的内切酶是该技术的关键。Zimmermann等[32]通过反向PCR获得了转基因玉米bt11基因5′端序列,从而建立了转基因玉米定量竞争PCR技术。Xu等[39]运用反向PCR技术获得转基因大豆的侧翼序列,建立出一套成熟的转基因大豆检测方法。
Tail-PCR是利用3个嵌套的特异性引物,分别和1个低Tm值的简并引物组合,进行连续的PCR循环反应,利用不同的退火温度选择性地扩增靶序列[40]。Tail-PCR技术简单易行,反应高效,灵敏度高,产物的特异性高。邵彦春等[41]运用该技术分离到红曲霉色素突变株T-DNA的侧翼序列,获得了一个类似发育调控子基因的序列。Wang等[36]运用Tail-PCR分析了转基因水稻外源基因插入位点的侧翼序列,依据转化事件PCR原理开发出高特异性的转基因水稻鉴定方法。
近年来,随着第二代高通量测序(NGS)的发展,侧翼序列PCR有了进一步的发展[42],先后出现了long template-rapid amplification of genomic DNA ends (LT-RADE)[43-44]、位点搜索PCR(site finding-PCR)[45]和随机破碎片段PCR (randomly broken fragment PCR,RBF-PCR)[45]等技术。
随机破碎片段PCR的原理是利用超声破碎的方法获得基因小片段,两端平端化处理后在3′端添入碱基A,利用通用接头进行染色体步移[9]。该技术已经被成功用于检测转基因玉米LY038中未知的侧翼序列,该方法不依赖于酶切位点的选择,适用于所有种类的转基因品系,具有很高的应用价值[46]。
LT-RADE是一种基于引物延伸来识别外源基因侧翼序列的方法[43-44]。和传统的RADE相比,LT-RADE可以延伸出800~1 000 bp的长DNA片段。该方法分为单引物延伸、产物纯化、同聚物加尾和两次巢式PCR共5个步骤,运用此法可以检测出样品中微量的转基因成分,但其特异性还有待提高[43]。
1.6 核酸检测试纸条技术
免疫层析试纸条是常用的快速检测方法之一。该方法基于免疫学原理,结合层析法,能够快速、简便、准确地筛选到目标物。常见的免疫层析试纸是以蛋白为抗原抗体进行特异性反应的,但由于核酸相较于蛋白更加稳定,因此,近年来基于免疫学原理的核酸试纸条技术受到了越来越多的关注[47]。
核酸试纸条可以结合多种PCR技术,用异硫氰酸荧光素(FITC)、地高辛(DIG)或生物素(biotin)等标记引物或探针获得的扩增产物作为抗原,胶体金和检测线处分别标记亲和素、抗荧光素等相应抗体,通过抗原抗体特异性结合获得可视化的检测结果[48]。Woo等[49]用双重PCR 结合一次性核酸试纸条检测了含转基因玉米59122 的玉米粉中的外源CaMV35S启动子和内源基因SSIIb,检测限为1%。Kolm等[50]结合恒温核酸扩增技术,用核酸试纸条检测转基因作物中的外源CaMV35S启动子,结果与琼脂糖凝胶电泳检测一致。
然而,核酸试纸条技术尚不成熟,在实际应用中还存在一些缺点和限制[51]。第一,方法的特异性和准确度还有待提高。扩增中引物二聚体和非特异性扩增产物会造成检测结果的假阳性,扩增效率不高则会造成结果的假阴性。第二,该技术依赖于核酸的提取及PCR仪的使用,步骤繁琐耗时,不适用于现场检测。
2 总结与展望
目前,转基因成分检测技术的难度主要体现在两个方面:一方面,随着生物技术的发展,越来越多的新技术(如CRISPR基因组定点突变技术)被应用于转基因产品的研发[52],使得转基因食品日趋多样化。然而,利用新技术生产出的转基因食品的基因编辑信息往往不被公开,未知的导入序列增加了转基因成分的检测难度;另一方面,食品加工技术水平和加工精度的不断提高,使得原料DNA的破坏程度更为严重,低质量、低浓度的模板DNA给基于核酸分析的转基因检测带来了困难。
基于核酸分析的转基因成分检测技术在国内外已得到广泛应用。这类方法不受转录水平限制,能同时分析多个目标基因,并且能获得更多如载体构建、植物来源和植物品系等转基因信息,对转基因产品的特征描述更全面,具有广阔的应用价值。针对转基因食品检测的现状,一方面需要进一步优化复杂样品或深加工样品的核酸提取和纯化技术,另一方面也应研发如高通量测序技术、生物传感器技术等特异性和灵敏度更高、检测更快速、结果更准确的新型核酸检测技术。在实际检测中,可以依据各技术优劣势、食品样品的加工类型及程度,组合使用不同的检测技术,优化检测方法,提高检测效率。未来,高通量、自动化、微型化、低成本、高灵敏度、高特异性、快速简便和准确高效的转基因检测技术及技术组合将成为转基因成分检测技术研究与应用的发展方向。
参考文献:
[1]SUSLOW T,THOMAS B,BRADFORD K J.Biotechnology provides new tools for plant breeding[M].Davis:Seed Biotechnology Center,University of California Davis Publications,2002.
[2]QAIM M.The economics of genetically modified crops[J].Annu Rev Resour Econ,2009,1(1):665-694.
[3]JONES L.Science,medicine,and the future:genetically modified foods[J].British Med J,1999,318:581-584.
[4]QAIM M,ZILBERMAN D.Yield effects of genetically modified crops in developing countries[J].Science,2003,299:900-902.
[5]JAMES C,KRATTIGER A F.Global review of the field testing and commercialization of transgenic plants:1986 to 1995[R].ISAAA Briefs,1996,1:31.
[7]UZOGARA S G.The impact of genetic modification of human foods in the 21st century:a review[J].Biotechnol Adv,2000,18(3):179-206.
[8]FRIZZI A,HUANG S.Tapping RNA silencing pathways for plant biotechnology[J].Plant Biotechnol J,2010,8(6):655-677.
[9]王晨光,许文涛,黄昆仑,等.转基因食品分析检测技术研究进展[J].食品科学,2014,35(21):297-305.
[10]HOLST-JENSEN A,BERTHEAU Y,DE LOOSE M,et al.Detecting un-authorized genetically modified organisms (GMOs) and derived materials[J].Biotechnol Adv,2012,30(6):1318-1335.
[11]MARMIROLI N,MAESTRI E,GULLM,et al.Methods for detection of GMOs in food and feed[J].Anal Bioanal Chem,2008,392(3):369-384.
[13]AHMED F E.Detection of genetically modified organisms in foods[J].Trends Biotechnol,2002,20(5):215-223.
[14]ARULANDHU A J,VAN DIJK J P,DOBNIK D,et al.DNA enrichment approaches to identify unauthorized genetically modified organisms (GMOs)[J].Anal Boanal Cem,2016,408(17):4575-4593.
(4)经分析、判断和查清渗水范围或裂缝分布、走向、宽度等,然后对其凿至一定深度,一般为8-10cm,并在渗水范围以外各延长20 cm,将周围松动的混凝土全部凿除,在凿开的凹槽内用快凝水泥浆埋设压浆咀,其间距通常为1. 0 m左右。在压浆嘴上接上轧头、伸缩竹、软管等装置,用压浆机将防水砂浆或化学浆液(聚氨酯、丙凝、水玻璃等)压人,填满孔隙和裂缝。压浆一般自上而下进行,并记录时间,直到孔隙内浆液已经饱满,才能停比压浆并记下压入浆量。
[15]HOLST-JENSEN A,RØNNING S B,LØVSETH A,et al.PCR technology for screening and quantification of genetically modified organisms (GMOs)[J].Anal Bioanal Chem,2003,375(8):985-993.
[16]AO J,LI Q,GAO X,et al.A multiplex nested PCR assay for the simultaneous detection of genetically modified soybean,maize and rice in highly processed products[J].Food Control,2011,22(10):1617-1623.
[17]BROD F C A,DOS SANTOS FERRARI C,VALENTE L L,et al.Nested PCR detection of genetically modified soybean in soybean flour,infant formula and soymilk[J].LWT-Food Sci Technol,2007,40(4):748-751.
[18]CHEAH Y K,CHONG Y T,KHOO S P,et al.Development of multiplex-PCR for genetically modified organism (GMO) detection targetingEPSPSandCry1Abgenes in soy and maize samples[J].Int Food Res J,2011,18(2):515-522.
[20]HEIDE B R,HEIR E,HOLCK A.Detection of eight GMO maize events by qualitative,multiplex PCR and fluorescence capillary gel electrophoresis[J].Eur Food Res Technol,2008,227(2):527-535.
[21]KAUR J,RADU S,GHAZALI F M,et al.Real-time PCR-based detection and quantification of genetically modified maize in processed feeds commercialised in Malaysia[J].Food Control,2010,21(11):1536-1544.
[22]KÖPPEL R,SENDIC A,WAIBLINGER H U.Two quantitative multiplex real-time PCR systems for the efficient GMO screening of food products[J].Eur Food Res Technol,2014,239(4):653-659.
[23]DUBE S,QIN J,RAMAKRISHNAN R.Mathematical analysis of copy number variation in a DNA sample using digital PCR on a nanofluidic device[J].PloS ONE,2008,3(8):e2876.
[26]IWOBI A,GERDES L,BUSCH U,et al.Droplet digital PCR for routine analysis of genetically modified foods (GMO):A comparison with real-time quantitative PCR[J].Food Control,2016,69:205-213.
[27]NIEMZ A,FERGUSON T M,BOYLE D S.Point-of-care nucleic acid testing for infectious diseases[J].Trends Biotechnol,2011,29(5):240-250.
[28]TOMITA N,MORI Y,KANDA H,et al.Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products[J].Nature Protoc,2008,3(5):877-882.
[29]闫兴华,许文涛,商颖,等.环介导等温扩增技术 (LAMP) 快速检测转基因玉米 LY038[J].农业生物技术学报,2013,21(5):621-626.
[30]KIEVITS T,VAN GEMEN B,VAN STRIJP D,et al.NASBATM isothermal enzymatic in vitro nucleic acid amplification optimized for the diagnosis of HIV-1 infection[J].J Virol Methods,1991,35(3):273-286.
[31]MORISSET D,DOBNIK D,HAMELS S,et al.NAIMA:target amplification strategy allowing quantitative on-chip detection of GMOs[J].Nucleic Acids Res,2008,36(18):e118.
[32]ZIMMERMANN A,LÜTHY J,PAULI U.Event specific transgene detection in Bt11 corn by quantitative PCR at the integration site[J].LWT-Food Sci Technol,2000,33(3):210-216.
[33]SIEBERT P D,CHENCHIK A,KELLOQQ D E,et al.An improved PCR method for walking in uncloned genomic DNA[J].Nucleic Acids Res,1995,23(6):1087-1088.
[34]YANG L,XU S,PAN A,et al.Event specific qualitative and quantitative polymerase chain reaction detection of genetically modified MON863 maize based on the 5'-transgene integration sequence[J].J Agric Food Chem,2005,53(24):9312-9318.
[35]PAN A,YANG L,XU S,et al.Event-specific qualitative and quantitative PCR detection of MON863 maize based upon the 3′-transgene integration sequence[J].J Cereal Sci,2006,43(2):250-257.
[36]WANG W X,ZHU T H,LAI F X,et al.Event-specific qualitative and quantitative detection of transgenic rice Kefeng-6 by characterization of the transgene flanking sequence[J].Eur Food Res Technol,2011,232(2):297-305.
[37]LIU Y G,CHEN Y.High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J].Biotechniques,2007,43(5):649-656.
[38]YAN Y,AN C,LI L,et al.T-linker-specific ligation PCR (T-linker PCR):an advanced PCR technique for chromosome walking or for isolation of tagged DNA ends[J].Nucleic Acids Res,2003,31(12):e68.
[39]XU W T,ZHANG N,LUO Y B,et al.Establishment and evaluation of event-specific qualitative and quantitative PCR method for genetically modified soybean DP-356043-5[J].Eur Food Res Technol,2011,233(4):685-695.
[40]王亚萍,谭玉梅,刘永翔,等.冠突曲霉产孢相关基因的克隆及功能分析[J].基因组学与应用生物学,2017,36(9):3767-3774.
[41]邵彦春,丁月娣,陈福生,等.TAIL-PCR法快速分离红曲霉色素突变株T-DNA插入位点侧翼序列[J].微生物学通报,2007,34(2):323-326.
[42]FRAITURE M A,HERMAN P,PAPAZOVA N,et al.An integrated strategy combining DNA walking and NGS to detect GMOs[J].Food Chem,2017,232:351-358.
[43]SPALINSKAS R,VAN DEN BULCKE M,VAN DEN EEDE G,et al.LT-RADE:an efficient user-friendly genome walking method applied to the molecular characterization of the insertion site of genetically modified maize MON810 and rice LLRICE62[J].Food Anal Methods,2013,6(2):705-713.
[44]SPALINSKAS R,VAN DEN BULCKE M,MILCAMPS A.Efficient retrieval of recombinant sequences of GM plants by cauliflower mosaic virus 35S promoter-based bidirectional LT-RADE[J].Eur Food Res Technol,2013,237(6):1025-1031.
[45]TAN G,GAO Y,SHI M,et al.SiteFinding-PCR:a simple and efficient PCR method for chromosome walking[J].Nucleic Acids Res,2005,33(13):e122.
[46]XU W,SHANG Y,ZHU P,et al.Randomly broken fragment PCR with 5′ end-directed adaptor for genome walking[J].Sci Rep,2013,3:3465.
[47]RICCI F,VOLPE G,MICHELI L,et al.A review on novel developments and applications of immunosensors in food analysis[J].Anal Chim Acta,2007,605(2):111-129.
[48]KAMLE S,ALI S.Genetically modified crops:detection strategies and biosafety issues[J].Gene,2013,522(2):123-132.
[49]WOO H J,CHUNG C M,SHIN K S,et al.Application of polymerase chain reaction with disposable amplicon detection device for identification of regulatory gene introduced into genetically modified maize[J].J Korean Soc Appl Biol Chem,2011,54(6):860-864.
[50]KOLM C,MACH R L,KRSKA R,et al.A rapid DNA lateral flow test for the detection of transgenic maize by isothermal amplification of the 35S promoter[J].Anal Methods,2015,7(1):129-134.
[51]夏启玉,李美英,杨小亮,等.免疫层析试纸条技术及其在转基因检测中的应用[J].中国生物工程杂志,2017,37(2):101-110.
[52]蔡军,李慧,胡梦龙,等.转基因成分分析检测技术研究进展[J].食品安全质量检测学报,2016,7(2):706-714.
