12S rRNA、Cyt b基因标记在几类野生动物检材鉴定中的差异
2018-01-06黄娅琳
黄娅琳
(1.南京森林警察学院,南京 210023;2.国家林业局森林公安司法鉴定中心,南京 210023)
12S rRNA、Cyt b基因标记在几类野生动物检材鉴定中的差异
黄娅琳1,2
(1.南京森林警察学院,南京 210023;2.国家林业局森林公安司法鉴定中心,南京 210023)
【目的】通过比较线粒体DNA上的12S rRNA和Cyt b基因分子标记在几类野生动物物种鉴定中的效果差异,以确定基因标记技术在野生动物物种鉴定中的有效性。【方法】提取各类野生动物样本DNA,PCR扩增线粒体DNA上的12S rRNA基因片段和Cyt b基因片段,对PCR产物进行电泳检测及测序分析,测序结果在GenBank上进行BLAST搜索,同源性分析。并将实验序列和NCBI上下载的与该序列存在同源性的序列用软件MEGA 7.0进行比对并构建进化树。【结果】从各类样本中成功地提取到了基因组总DNA,并成功扩增出了用于动物种属鉴定的12S rRNA基因片段、Cyt b基因片段。测序分析结果表明用12S rRNA基因片段和Cyt b基因片段均可准确鉴定黑熊、藏羚羊、鬣羚、穿山甲样本的动物种属,但12S rRNA基因片段无法准确鉴定北山羊样本,BLAST搜索结果为其近缘物种山羊,而Cyt b基因片段却可准确鉴定北山羊。各样本基于12S rRNA、Cyt b基因序列构建进化树的结果与BLAST搜索结果相符。【结论】单一位点基因标记能够准确鉴别亲缘关系较远的物种。但对某些近缘物种,单一位点基因标记的鉴别能力较差,故野生动物物种鉴定宜选用2个以上的基因标记位点。
野生动物;种属鉴定;12S rRNA;Cyt b;基因标记
随着非法猎杀、贩卖野生动物案件的与日俱增,越来越多的野生动物物种面临灭绝的危险。快速、准确地鉴定其种属将有助于准确量刑,打击犯罪,保护濒危的野生动物资源。案件中常常涉及小块肌肉组织、血痕、毛发等微量检材或炮制加工过的动物药材,针对此类动物物证,目前往往通过线粒体DNA(mtDNA)保守片段测序技术准确确定动物物种。线粒体DNA由于其结构简单、进化速度快、在世代传递中没有基因重组等优点,常常被用作动物种属鉴定的工具。文献报道可用于物种鉴定的基因标记位点有 16S rRNA、12S rRNA、Cyt b、D-loop基因片段等。其中,Cyt b基因片段、12S rRNA基因片段是最常用于动物物种鉴定的基因片段[1]。王义权等报道了用DNA序列分析方法通过Cyt b基因片段序列来鉴定中药材乌梢蛇及其伪品[2]。吴平等通过对中华鳖、山瑞鳖以及一些龟类的12S rRNA基因片段的测序,通过对待检样品12S rRNA基因片段的序列分析,从数据库中判断待检样品是否为正品[3]。迄今,已有大量研究通过测定物种的12S rRNA序列或Cyt b序列,然后向GenBank中搜索类似序列并对序列进行分析以确定物种[4-10]。采用此方法成功鉴定了黑麂、黄麂、黑熊、穿山甲、非洲弯角羚、非洲跳羚、非洲黑斑羚、非洲象、孔雀、孟加拉巨蜥以及各种家畜家禽,为海关进出口监管和重点保护野生动物的执法工作提供了重要帮助[11]。
直接测序法用于物种的鉴定可操作性强,结合了PCR技术可用于毛发、血痕、唾液等微量物证,对于未知物种只需用通用引物扩增后测序就可获得相应的基因片段。但在日常案件的鉴定中,由于时间和经费的限制,通常是将单一基因位点分子标记用于物种鉴定。但单一基因位点分子标记是否适用于各类野生动物以及案件中所涉及到的各种状态的样本,本研究拟通过比较12S rRNA和Cyt b基因位点分子标记在检测几类常见涉案野生动物样本中的效果差异来进行探究。
1 材料和方法
1.1 材料
实验材料均来自国家林业局森林公安司法鉴定中心日常检案,检材物种(已知)及状态如表1所示,检材图片如图1所示。
1.2 方法
1.2.1 样本DNA的提取
针对不同的样本采用不同的取样方法,肌肉组织剪取约20 mg置于1.5 mL EP管中,骨骼、动物角、甲片均用锉子磨取约30 mg置1.5 mL EP管中,毛发剪取约3~5 cm、血痕剪取约0.5 cm2置1.5 mL EP管中。血痕样本DNA采用TIANamp Genomic DNA Kit血液/细胞/组织基因组DNA提取试剂盒(天根公司)提取,其他样本的基因组总DNA均用DNA提取试剂盒(Takara公司)提取,提取方法参照试剂盒说明书,骨骼、动物角、甲片、毛发的组织消化时间延长为25~30 h。用分光光度法检测样本DNA的纯度和浓度。将样品总DNA置于-20℃保存,待后续试验用。

表1 样本物种及状态Table 1 Species and status of samples

图1 实验样本Figure 1 Experimental samples
1.2.2 PCR扩增
扩增线粒体DNA上12S rRNA基因片段采用通用引物:L1091和H1478[12],扩增线粒体DNA上Cyt b基因片段采用自设计引物,利用Primer Premier 5.0软件进行引物设计。引物序列如下:
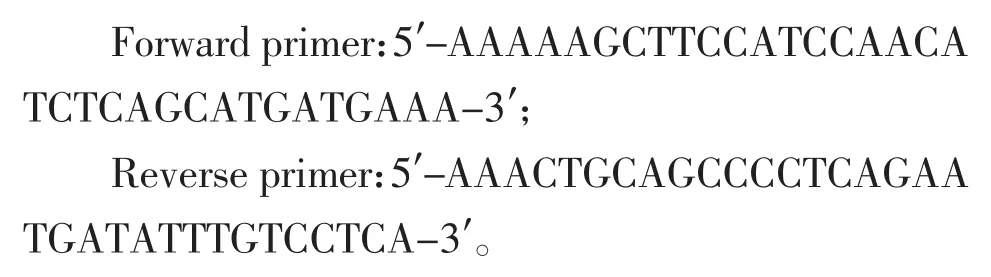
扩增反应在Veriti PCR仪(AB公司)上进行。12S rRNA基因片段和Cyt b基因片段均采用相同反应体系:总反应体积为25 μL,其中含2.5 pmol/μL引物各 2 μL,2.5 mM dNTPs 2 μL,1.25 U Ex Taq 酶0.25 μL,10×buffer 2.5 μL,25 mM MgCl21.5 μL,10~20 ng/μL 模板 1~2 μL,其他为灭菌蒸馏水。PCR 反应所需试剂及引物均购自Takara公司。PCR反应程序:94℃预变性 3 min;94℃变性 30 s,56℃退火30 s,72℃延伸 1 min,38个循环;72℃延伸 7 min。扩增产物经1%琼脂糖凝胶电泳,在凝胶成像系统(GENEGENUS)上检测、成像。
1.2.3 PCR扩增片段测序
PCR产物用PCR产物(小量)纯化试剂盒(Takara公司)纯化。以纯化产物为模板DNA,用ABI公司测序试剂盒进行测序PCR扩增(操作按试剂盒说明),扩增产物经甲酰胺变性后,ABI公司3130xl测序仪测序。
1.2.4 BLAST搜索及序列对比
将测序结果在GenBank中进行BLAST搜索,并进行同源性分析比对,以确定动物种属,用DNAMAN生物学软件进行同源性分析。
1.2.5 系统进化树的构建
在GenBank下载与本研究涉及的5个物种存在同源性的序列,保证每个物种至少有两个来自不同实验室的序列,同时尽可能下载完所有的近源物种序列。1号样本近缘物种12S rRNA基因片段序列信息如表2所示,其他样本近缘物种序列略。通过软件MEGA 7.0进行序列比对和进化树构建。去除与样本序列相对应以外的区段后,将序列比对文件保存为CLUSTER格式。其后,将该CLUSTER文件导入MEGA 7.0软件,将其转化为Meg文件后进行系统进化树构建。统计分析采用邻接(Neighbor Joining,NJ)方法,进化距离的计算采用Kimura 2-Parameter模型,步差值(bootstrap)设置为 1 000。

表2 GenBank来源序列信息Table 2 Sequence information downloaded from GenBank
2 结果
2.1 电泳检测结果及分析
分光光度法测得1号至6号样本基因组总DNA 的 OD260/OD280 分 别 为 1.67、1.23、1.82、1.75、1.65、1.25,表明各样本DNA纯度较高。样本总DNA 浓度为 15 ng/μL、2 ng/μL、18 ng/μL、15 ng/μL、17 ng/μL、3 ng/μL。
图2所示为1号至6号样本基因组总DNA电泳结果,由图可见,1号、3号、4号、5号样本DNA裂解液中检测到基因组总DNA(箭头所示),表明该样品中提取到的总DNA质量较高。但电泳检测未检测到2号、6号样本DNA裂解液中的基因组总DNA,推测这是由于2号、6号样本DNA含量过低的原因。
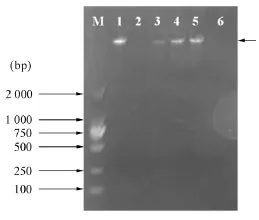
图2 样本基因组总DNA琼脂糖电泳图Figure 2 Electropherogram of genome total DNA
以所检动物样本基因组总DNA为模板,扩增线粒体DNA上12S rRNA和Cyt b基因片段,结果如图3所示。由图可见,检材DNA通过PCR扩增,1~6号样本在 12S rRNA(约 430 bp)和 Cyt b(约 360 bp)目的基因片段处均得到了大量的PCR产物(箭头所示),其中2号毛发样本和6号炮制过的穿山甲片样本虽然基因组总DNA含量较低,但通过PCR扩增均获得了大量的扩增产物。
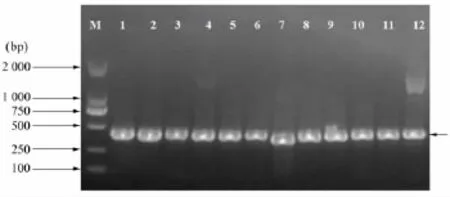
图3 12S rRNA和Cyt b基因片段PCR扩增产物电泳图谱Figure 3 Electropherogram of PCR products of 12S rRNA gene and Cyt b gene
2.2 基因序列比对
将PCR产物纯化后经3130xl测序仪进行测序,各样本测序所得序列经BLAST搜索,同源性比对分析,发现所检测的1~6号样本的Cyt b基因片段序列分别与黑熊、藏羚羊、北山羊、鬣羚、穿山甲线粒体DNA Cyt b基因序列的部分片段的同源性均高达99%至100%,均能准确鉴定样本所属物种。而1~6号样本的12S rRNA基因片段序列与黑熊、藏羚羊、山羊、鬣羚、穿山甲线粒体DNA 12S rRNA基因序列的部分片段的同源性均高达99%至100%,表明本研究所用12S rRNA基因片段能准确鉴定1号、2号、4号、5号、6号样本的所属物种,但不能准确鉴定3号样本北山羊肌肉组织的种属。
结果表明,12S rRNA基因序列和Cyt b基因序列在黑熊、藏羚羊、鬣羚、穿山甲的鉴定中都可得出相同的结果,能准确确定其所属物种。而在北山羊的鉴定中,本研究所扩增的Cyt b基因片段,与NCBI数据库中的北山羊的Cyt b基因序列同源性达99%,通过DNAMAN生物学软件分析,发现该序列与已有的山羊的Cyt b基因序列同源性达95%,表明本研究扩增的Cyt b基因片段能区分出山羊和北山羊;而本研究所扩增的12SrRNA基因片段与NCBI数据库中山羊的同源性最高,达99%,DNAMAN生物学软件分析结果与之相符,表明本研究所扩增的12S rRNA基因片段不能区分山羊和北山羊这两个近缘物种。
2.3 采用系统进化树验证基因序列比对结果
基于12S rRNA序列构建的1号样本及熊科近源物种的系统进化树如图4所示,结果表明1号样本(Sample 1)在熊科Ursidae的5个属、8个种中与亚洲黑熊Ursus thibetanus来源个体距离最近,被聚为一支。基于K2P模型计算进化距离,1号样本的12S rRNA序列与亚洲黑熊的分支长度最小,介于0~0.005之间;与大熊猫 Ailuropoda melanoleuca进化距离最远,分支长度达0.076 6。因此,1号样本被判断为来自亚洲黑熊,这与BLAST搜索结果相符。采用相同的方法构建2号~6号样本的系统进化树,聚类结果均与BLAST搜索结果一致。
3 讨论
在样本的选择上,本研究选用了骨骼、毛发、肌肉组织、血痕、角制品、已炮制过的穿山甲甲片等野生动物案件中常常遇到的,具有代表性的检材样本。不同检材由于DNA含量的差异,其DNA提取的难易程度不同。血痕、肌肉组织是野生动物案件中最常见的检材,由于其DNA含量大,片段长度较长,DNA提取及PCR扩增的难度较小,鉴定成功率较高。而本研究所选用的骨骼、毛发、角制品、已炮制过的穿山甲甲片均为较为陈旧的检材,由于角质化程度较高或经过人为加工,其DNA含量较低或已大量降解,其中毛发和炮制过的甲片DNA含量极低,其DNA提取液的浓度未达到琼脂糖电泳检测的要求,电泳检测未见清晰条带(见图2)。
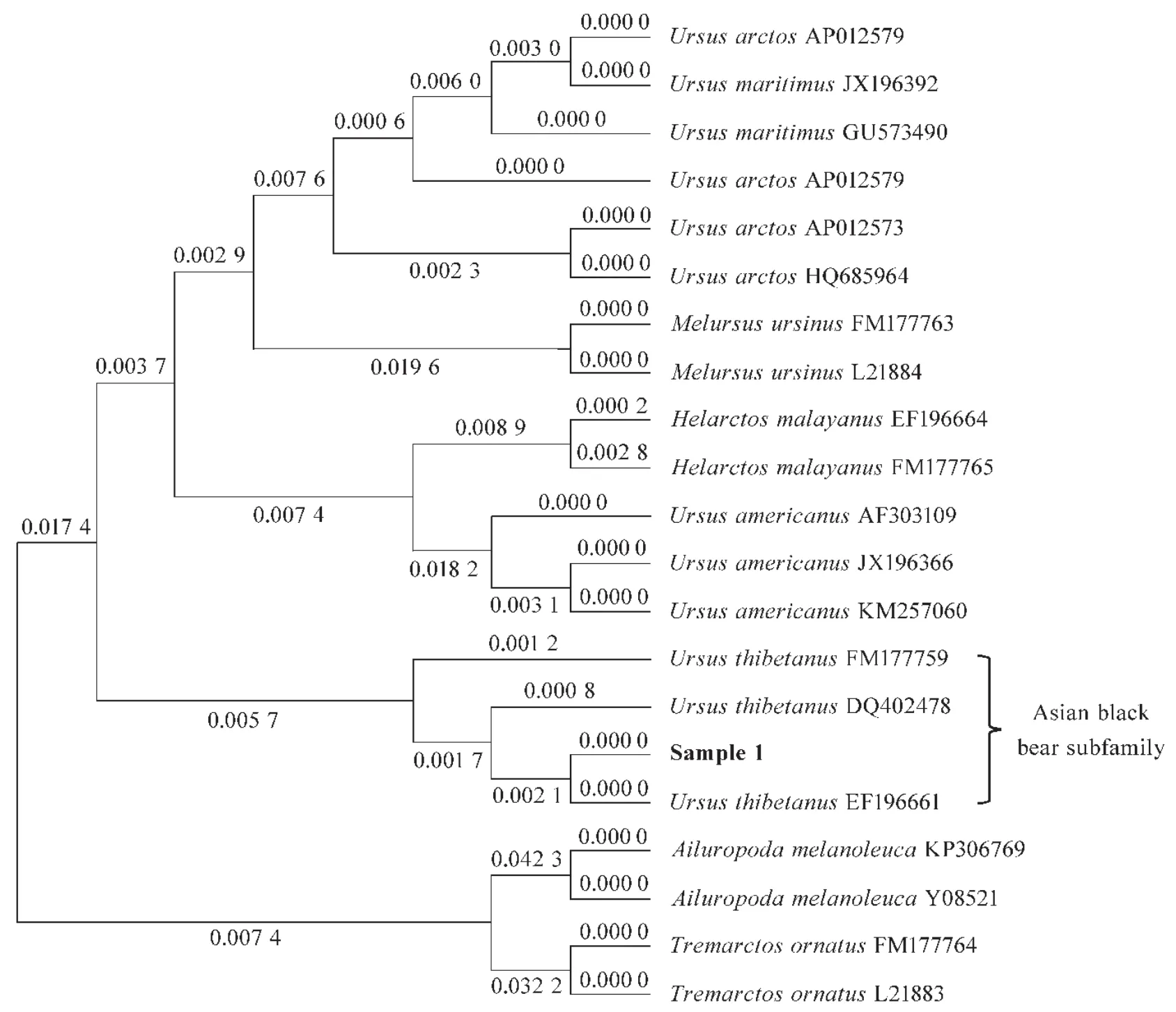
图4 基于12S rRNA序列构建的1号样本(Sample 1)及熊科近源物种的系统进化树Figure 4 Phylogenetic tree based on 12S rRNA sequences from sample 1 and its homologous species in bear family
野生动物案件涉及的检材常常是降解、陈旧、高度角质化、炮制加工过的动物检材,通过细胞核DNA是无法提取到符合鉴定需求的长度的DNA片段,通常高于200 bp以上的片段就无法扩增出来,而线粒体DNA具有环状双链的稳定结构、在细胞中拷贝数多等优点,非常适合降解、陈旧检材的鉴定。本研究表明降解、陈旧、高度角质化、炮制加工过的动物检材中仍能提取到符合较长片段(如400~500 bp)PCR扩增需求的模板DNA。
在基因位点的选择上,本研究选用了线粒体DNA上的12S rRNA和Cyt b基因位点,由于线粒体12S rRNA基因的进化速率较快,不同物种间序列差异大,有利于设计针对目标物种的高特异性引物,且扩增该基因的稳定性和可重复性好。而Cyt b基因是动物线粒体上一个编码蛋白质的基因,有一定的保守性,根据蛇类药材Cyt b基因片段序列的分析[2],表明12S rRNA和Cyt b基因这类在种内个体间的序列差异很小,而种间的序列差异却较大的DNA片段,正是物种鉴别的理想标记。此外,NCBI的GenBank中有大量的12S rRNA基因、Cyt b基因片段比对序列资源,为案件中未知物种的鉴定提供了极大的方便。近年来,12S rRNA基因片段和Cyt b基因片段已被广泛用于野生动物刑事物证鉴定和动物类药材的鉴定研究。虽然大量研究表明单一的12S rRNA基因位点或Cyt b基因位点在大多数动物上均能得到准确的鉴定结果,但本研究表明单一基因位点分子标记对于一些亲缘关系很近的动物物种鉴别能力较差,如12S rRNA基因位点用于山羊和国家一级重点保护动物北山羊的鉴定,这也可能是由于本研究是选用的12S rRNA基因的通用引物,鉴定的特异性稍差的缘故,在后续的研究中将设计特异性引物进一步研究。因此,在野生动物未知物种的鉴定中宜同时选用2个以上的基因标记位点,以提高鉴定的准确性。此外,COI也是目前广泛应用的线粒体分子标记,常用于昆虫及野生动物物种的种属鉴定,尤其是在动物类中药材基原动物的种属鉴定及其混伪品的鉴定中应用极为广泛[13-19]。根据2009年以“生命条形码与人类未来”为主题的中国科协第31期新观点新学说学术沙龙的报道,COI条形码具有很高的物种鉴定可靠性,但同时也有部分研究证实COI并不适用于所有的动物类群,还需要一个辅助性的条形码序列。本课题组曾经在45种野生动物样本上做过比较,发现COI与本文所用的12S rRNA、Cyt b在鉴定的准确性方面差别不大。在所检测的大多数动物上,这3个基因位点均能准确鉴定物种,但在个别的物种上它们存在差异,此外,对于不同的检材COI与12S rRNA、Cyt b扩增成功的能力存在一定差异,文献中COI基因的通用引物扩增片段是580 bp以上的片段,作者研究发现对于某些陈旧、DNA有所降解的检材,其PCR扩增的成功率有所下降,相对来说,本研究扩增的12S rRNA、Cyt b基因片段均为400 bp左右的片段,陈旧的或高温处理过的检材均能成功扩增出目的片段[20]。
大量研究表明,核基因组中存在大量的线粒体假基因[21-22]。由于本文设计的引物是根据高度保守的区域设计引物,所以很可能会导致线粒体DNA正常基因和核假基因共扩增。但假基因有一个重要来源是以mRNA为中介的正常基因的间接复制品:即在真核生物细胞中的mRNA先经反向转录形成DNA,再整合到染色体上而形成的。因此,核基因组假基因中没有正常基因中存在的内含子;3’末端有Poly A结构;有些假基因与相应的正常基因密码子偏好不同。Cai等使用通用引物对来自总DNA的COI扩增产物测序发现,具有核DNA污染的个体,测序峰图具有很多双峰现象;而只来自线粒体DNA的样品没有出现这种现象[23]。鉴于线粒体DNA正常基因和核假基因存在的以上序列特征差异,本研究通过对实际扩增产物峰图和序列的分析,排除了本研究扩增所得序列是受核基因组假基因干扰的可能性,进一步增强了本文研究结果的可靠性。
通过利用所获得的序列进行BLAST在NCBI上进行同源搜索鉴定物种的方法已被广泛应用。但这种方法的科学性有待商榷。这是因为NCBI上的序列的真实性、可靠性都有待验证;测序得到的序列头尾两端准确性不高,网上BLAST比对前没有去除。一个替代方法可以是通过将NCBI上所有的与该序列存在同源性的序列都下载下来,尽量保证每个物种至少有两个来自不同实验室的序列,同时尽可能下载完所有的近源物种序列。利用CLUSTER对比后,裁剪去除与样品核心序列相对应以外的区段,然后构建进化树,根据进化树来判断待检样品属于哪个物种。本研究的结果表明,这两种方法所得结果具有高度的一致性。相对来讲,BLAST法简单快速;而进化树分析方法科学性更强,因此两者可以相互验证补充。
[1]黄璐琦,唐仕欢,李军德,等.动物药材分子鉴定研究策略[J].中国中药杂志,2011,36(3):234-236.
[2]王义权,周开亚,徐珞珊,等.中药材乌梢蛇及其混淆品的DNA 序列分析鉴别[J].药学学报,1999,34(1):67-71.
[3]吴平,周开亚,杨群.亚洲淡水和陆生龟鳖类12S rRNA基因片段的序列分析和系统发生研究[J].动物学报,1999,45(3):260-267.
[4]刘中权,王义权,周开亚,等.中药材龟甲及原动物的高特异性PCR 鉴定研究[J].药学学报,1999,34(12):941-945.
[5]黄娅琳.mtDNA 12S rRNA基因序列测定在腐烂动物肌肉样本鉴定中的应用[J].刑事技术,2005(4):10-11.
[6]黄娅琳.12S rRNA和Cyt b基因序列测定在獐乳制品鉴定中的应用[J].四川动物,2008,27(6):1111-1114.
[7]LEE J C I,TSAI L C,HUANG M T,et al.A novel strategy for avian species identification by cytochrome b gene [J].Electrophoresis,2008,29(11):2413-2418.
[8]NATONEK-WISNIEWSKA M,SLOTA E,KALISZ B.Use of cytochrome b polymorphism for species identification of biological material derived from cattle,sheep,goats,roe deer and red deer[J].Folia Biologica-krakow,2010,58(1):47-50.
[9]NATONEK-WISNIEWSKA M.Species identification of feline DNA based on analysis of cytochrome b [J].Animal Science,2009,9(4):379-383.
[10]TOBE SS,LINACRE A.DNA typing in wildlife crime:recent developments in species identification [J].Forensic Science,Medicine,and Pathology,2010(6):195-206.
[11]杨光,蔡垚,刘海.mtDNA标记在几种海关进出口动物产品鉴定中的应用[J].动物学杂志,2004,39(5):40-43.
[12]KOCHER T D,THOMAS W K,MEYER A,et al.Dynamics of mitochondrial DNA evolution in animals:amplification and sequencing with conserved primers[J].Proceedings of the National Academy of Sciences USA,1989(86):6196-6200.
[13]蔡继峰,刘敏,应斌武,等.mtDNA中COI分子标记在常见食尸性蝇类鉴定中的应用[J].昆虫学报,2005,48(3):380-385.
[14]HAJIBABAEI M,JANZEN D H,BURNS J M,et al.DNA barcodes distinguish species of tropical Lepidoptera[J].Proceedings of the National Academy of Sciences USA,2006(103):968-971.
[15]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biologicalidentifications through DNA barcodes[J].Proceedings of the Royal Society of London.Series B:Biological Sciences,2003(270):313-321.
[16]HEBERT P D N,RATNASINGHAM S,DE WAARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society of London B,2003,270(s1):96-99.
[17]DALTON D L,KOTZE A.DNA barcoding as a tool for species identification in three forensic wildlife cases in South Africa[J].Forensic Science International,2011(207):51-54.
[18]陈士林,姚辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则[J].中国中药杂志,2013,38(2):141-148.
[19]张辉,姚辉,崔丽娜,等.基于COI条形码序列的《中国药典》动物药材鉴定研究[J].世界科学技术·中医药现代化,2013,15(3):371-380.
[20]黄娅琳.高温烹饪对动物肌肉组织DNA降解的影响[J].四川动物,2012,31(2):222-225.
[21]DU BUY H G,RILEY F L.Hybridization between the nuclear and kinetoplast DNA's of Leishmania enriettii and between nuclear and mitochondrial DNA's of mouse liver[J].Proceedings of the National Academy of Sciences USA,1967,57(3):790-797.
[22]DEXING Z,GODFREY H.Nuclear integrations:challenges for mitochondrial DNA marke[rJ].Tree,1996,11(6):247-251.
[23]CAI Y,CHENG X Y,DUAN D,et al.Mitochondrial COI gene transfers to the nuclear genome of Dendroctonus valens and its implications[J].Journal of Applied Entomology,2010,135(4):302-310.
The Differences of 12S rRNA and Cyt b Gene Marker in Several Wildlife Species Identification
HUANG Ya-lin1,2
(1.Nanjing Forest Police College,Nanjing 210023,China;2.Wildlife Material Evidence Appraisal Center of the State Forestry Administration,Nanjing 210023,China)
【Objective】To study the effectiveness of gene markers for wildlife species identification,we compared the differences between mtDNA 12S rRNA and Cyt b gene markers in wildlife species identification.【Method】The isolation of DNA from wildlife samples was carried out.Partial mitochondrion DNA 12S rRNA and Cyt b genes were amplified and sequenced.The original animal identifications of different wildlife samples were determined by BLAST search of the sequences in GenBank.Then,genetic distance and phylogenetic tree were analyzed by MEGA 7.0.【Result】The genome DNAs were successfully extracted from wildlife samples and partial mitochondrion DNA 12S rRNA and Cyt b genes were amplified.For the identification of Ursus thibetanus,Pantholops hodgsonii,Capricornis milneedwardsii,and Manis javanica sample species,both the 12S rRNA gene marker and Cyt b gene marker can identify the wild species successfully.However,in terms of identification of Capra ibex,Cyt b gene marker could distinguish Capra ibex from Capra hircus gene sequences,while 12S rRNA gene marker not.Phylogenetic tree constructed based on 12S rRNA and Cyt b sequences was consistent with BLAST analysis.【Conclusion】Most of wild animals can be identified by single locus marker.When comes to some closely relative species,the identification ability of single locus marker is poor,thus two or more gene marker loci are recommended to identify wild animal species.
wild animal;species identification;12S rRNA;Cyt b;gene marker
Q959 文献标志码:A 文章编号:1000-2650(2017)02-0273-07
10.16036/j.issn.1000-2650.2017.02.021
2016-12-02
中央高校基本科研业务费专项资金项目(LGYB201518)资助。
黄娅琳,博士,副教授,主要从事野生动物DNA鉴定及相关遗传学研究,E-mail:huangyalin228@163.com。
(本文审稿:徐怀亮;责任编辑:秦碧雯;英文审稿:刘益平)
