滇重楼GAox基因的克隆与生物信息学分析△
2017-12-26张梦幻廖登群孙鹏李先恩祁建军
张梦幻,廖登群,孙鹏,李先恩,祁建军
(中国医学科学院 北京协和医学院 药用植物研究所,北京 100193)
·基础研究·
滇重楼GAox基因的克隆与生物信息学分析△
张梦幻,廖登群,孙鹏,李先恩,祁建军*
(中国医学科学院 北京协和医学院 药用植物研究所,北京 100193)
目的克隆编码滇重楼赤霉素合成代谢途径中关键酶GA氧化酶(Gibberellic oxidase,GAox)的全长cDNA序列,并进行生物信息学分析。方法根据滇重楼转录组测序序列,设计特异性PCR引物,利用RT-PCR 技术扩增目标基因编码区的全长序列连接至pMD18-T载体上,进行序列测定及序列同源性比较分子系统进化树构建、结构域搜索及蛋白质三维结构预测等生物信息学分析。结果成功克隆了滇重楼的的6条GAox基因,大小在1000 bp左右,分别命名为PpGA2ox1、PpGA2ox2、PpGA2ox3、PpGA2ox4、PpGA20ox1 和PpGA3ox1。6条基因分别属于GA氧化酶三个不同的亚家族。滇重楼GAox蛋白氨基酸数量均在300~400,理论等电点在4~7左右,带正电荷。PpGA2ox1、PpGA2ox2和PpGA2ox4为不稳定蛋白,PpGA2ox3、PpGA20ox1和PpGA3ox1为稳定蛋白,均为没有跨膜结构域和信号肽的亲水蛋白。除了PpGA2ox1亚细胞定位在叶绿体,其他蛋白均定位在细胞质。滇重楼GAox蛋白与不同物种同源序列的序列相似性较高,保守性比较强。结论首次从滇重楼中克隆了6条GAox基因并对其进行了初步的生物信息学分析。
滇重楼;GA氧化酶;基因克隆;生物信息学分析
滇重楼ParispolyphyllaSmith var.yunnanensis种子是重要的繁殖方式之一,其繁殖系数较高[1],但存在形态休眠和生理休眠[2],大量种子在漫长的休眠期失去活力[3],出苗率极低。赤霉素(GA)又是一种促进生长的植物激素,可以调节种子发育,促进种子萌发[4-7],与植物种子休眠具有重要关系。GA的生物合成途径中包括GA20ox、GA3ox和GA2ox 3种重要的氧化酶[8],这些基因在拟南芥、水稻等植物中进行了克隆和功能研究[9-16],为研究GAs在滇重楼种子休眠与萌发中的作用提供了重要的参考。
前期对滇重楼不同器官及不同发育时期的种子进行的转录组测序发现了GA氧化酶的候选基因,其中4条GA2ox基因、1条GA20ox基因和1条GA3ox基因为全长基因。本文利用RT-PCR技术对这6条全长GAox基因进行物理克隆和测序,并进行了生物信息学分析,为下一步利用RT-qPCR分析GA基因的时空表达特性和功能分析奠定了基础。
1 材料与方法
1.1 材料
滇重楼种子于2015年11月采自云南临沧道地药材种植科技有限公司,由中国医学科学院药用植物研究所祁建军研究员鉴定为滇重楼ParispolyphyllaSmith var.yunnanensis的种子。
1.2 方法
1.2.1 种子处理 成熟的滇重楼种子去除杂物后轻轻搓去种皮,室内阴干1~2 d,蒸馏水中浸泡12 h,20 ℃蛭石层积,取不同萌发阶段的种子,自来水冲洗干净后,蒸馏水冲洗,75%乙醇冲洗,吸水纸擦干,锡箔纸包裹,置液氮中备用。
1.2.2 植物总RNA的提取及cDNA的合成 取不同发育阶段的滇重楼种子按照快速通用植物RNA提取试剂盒3.0(北京华越洋生物科技有限公司)的操作步骤提取样品的总RNA,并用NanoDrop 2000检测总RNA的浓度与质量,用1.2%的琼脂糖电泳检测其完整性。按照PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis Kit试剂盒(大连宝生物公司)的操作步骤反转录合成第一链cDNA。
1.2.3GAox基因的扩增、克隆及测序 通过分析转录组数据,其中14条Unigene序列注释为GA2ox基因,8条Unigene序列注释为GA20ox基因,1条Unigene序列注释为GA3ox基因,经分析其中4条GA2ox基因,1条GA20ox基因,1条GA3ox基因为全长基因。通过NCBI的ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)确定序列开放阅读框( open reading frame,ORF),使用primer premer 5设计扩增ORF的引物(见表1),引物由生工生物工程(上海)股份有限公司北京分部合成。

表1 RT-PCR 引物
RT-PCR反应体系为:5×PrimeSTAR Buffer (Mg2+plus) 10 μL,PrimeSTAR HS DNA Polymerase 0.5 μL,dNTP Mixture 4 μL,模板cDNA 2 μL,引物各1 μL,灭菌蒸馏水31.5 μL。PCR反应程序:94 ℃预变性5 min,98 ℃变性10 s,60 ℃退火5 s,72 ℃延伸60 s,40个循环,循环结束后72 ℃延伸5 min,4 ℃保温。
1.2%琼脂糖凝胶电泳检测PCR产物,对扩增所得目的片段按照DNA纯化回收试剂盒(天根生物公司)操作说明进行切胶回收。
将回收产物连接到pMD18-T载体上,转化大肠杆菌(DH5α菌株),在含有氨苄的LB培养基上进行培养,约12 h后观察菌落情况,挑取阳性克隆放入含有氨苄的LB培养液中培养约16 h后进行菌落PCR扩增,用质粒PCR鉴定阳性重组子,电泳检测后送上海生工生物公司测序。
1.2.4 序列分析 运用SeqMan软件对两头测序的序列进行拼接,拼接结果运用DNAMAN软件与转录组测序结果进行比对。利用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)获得滇重楼GAox基因的蛋白序列。
1.2.5 滇重楼GAox基因系统发育分析 构建利用滇重楼GAox基因进化树选用了基因功能研究比较广泛的4个物种(单子叶植物水稻、玉米,双子叶植物拟南芥、番茄)的GAox基因家,这4个物种的GAox基因蛋白序列由Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)数据库中下载[13]。首先用Clustalw对滇重楼GAox蛋白序列与4个物种的蛋白质序列进行多序列比对,然后运用MEGA6.0 构建 Neighbor-joining(NJ)系统进化树。根据系统发育分析,选取与滇重楼GAox蛋白亲缘关系较近的同源序列,运用DNAMAN进行蛋白质序列比对。
1.2.6 滇重楼GAox 蛋白的物理特性预测 利用ExPASy Protparam(http://www.expasy.ch/tools/protparam.html)分析相对分子质量、等电点等[17];利用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜区分析;WOLF PSORT(http://wolfpsort.org/)进行亚细胞定位分析;利用SignalP软件(http://www.cbs.dtu.dk/services/SignalP/)分析信号肽[18]。
1.2.7 滇重楼GAox基因蛋白质的结构域和同源建模分析 利用 InterProScan (http://www.ebi.ac.uk/interpro/search)、NCBI Conserved Domains(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和ExPASy Prosite(http://prosite.expasy.org/)进行蛋白质结构域和功能位点分析。采用SWISS-MODEL(http://swissmodel.expasy.org/)同源建模方法进行3D结构预测[19]。
2 结果
2.1 总RNA提取及检测结果
NanoDrop 2000检测滇重楼提取的总RNA,A260/280≈2.0~2.2,A260/230>1.8,质量浓度在200 ~ 400 ng·μL-1。取5 μL RNA进行琼脂糖凝胶电泳检测,28 S和18 S条带清晰(见图1),表明提取的总RNA完整性较好,可进行下步实验。
2.2 cDNA第一链的合成结果
经NanoDrop 2000检测,cDNA的质量浓度均大于1000 ng· μL-1,A260/280≈1.8,A260/230>2,均能扩增出参照基因,说明反转录成功。
2.3 GAox基因的扩增、克隆及测序
用反转录得到的cDNA 为模板,基于RT-PCR技术用PrimeSTAR© HS DNA Polymerase进行PCR扩增,凝胶电泳跑出6条条带(见图2),大小与预期理论值一致。挑取阳性克隆进行测序,测序结果与预期序列比对,序列相似性均在99%以上,表明克隆成功。
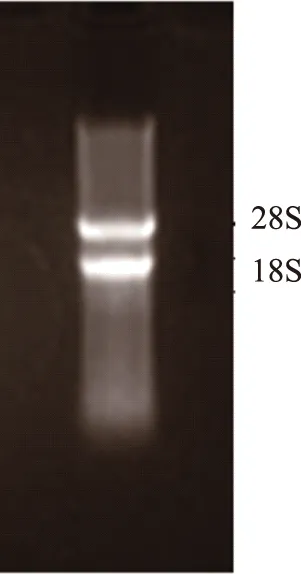
图1 滇重楼总RNA琼脂糖凝胶电泳图
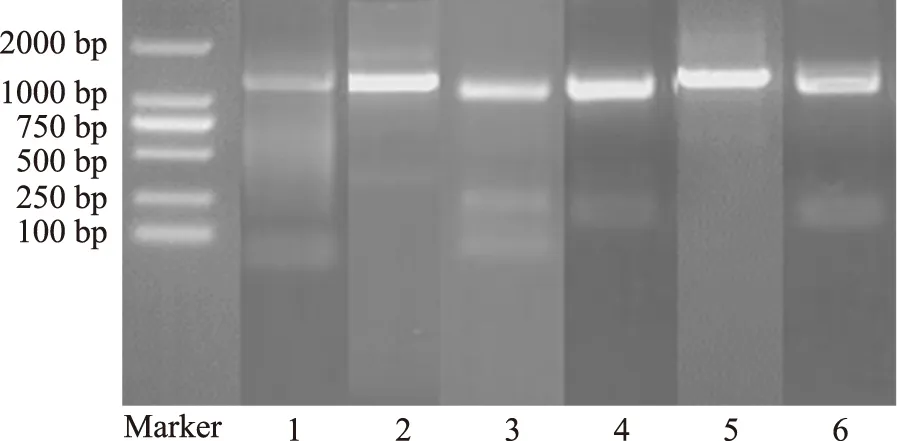
注:1~6泳道对应的基因分别为PpGA2ox1、PpGA2ox2、PpGA2ox3、PpGA2ox4、PpGA20ox1和PpGA3ox1。图2 GAox基因电泳图
2.4 序列分析
第一次成功克隆到6条滇重楼GAox基因全长编码区,其中,PpGA2ox1全长为1092 bp,编码363个氨基酸,NCBI登录号为KY921589;PpGA2ox2全长为1032 bp,编码343个氨基酸,NCBI登录号为KY921590;PpGA2ox3全长为978 bp,编码325个氨基酸,NCBI登录号为KY921591;PpGA2ox4全长为969 bp,编码322个氨基酸,NCBI登录号为KY921592;PpGA20ox1全长为1122 bp,编码373个氨基酸,NCBI登录号为KY921593;PpGA3ox1全长为1050 bp,编码349个氨基酸,NCBI登录号为KY921594。
2.5 滇重楼GAox基因系统发育分析
与水稻等4个物种构建的GAox系统进化树显示,GAox氨基酸聚类为8个亚枝(见图3),分别代表了C19-GA2ox、C20-GA2ox、GA20ox、GA3ox、GAox-A、GAox-B、GAox-C和GAox-D亚家族[19]。PpGA2ox1和PpGA2ox2属于C19-GA2ox亚家族成员,PpGA2ox3和PpGA2ox4属于C20-GA2ox亚家族成员,PpGA20ox1属于GA20ox亚家族成员,PpGA3ox1属于GA3ox亚家族成员。此外由图3还可以发现,PpGA2ox1与双子叶植物拟南芥和番茄的GA2ox序列聚为一个亚枝,PpGA2ox2与单子叶植物水稻和玉米的GA2ox序列聚为一个亚枝,PpGA2ox3与单子叶植物水稻和玉米的GA2ox序列聚为一个亚枝,而与PpGA2ox4亲缘关系较近的物种中既有单子叶植物也有双子叶植物,PpGA20ox1与单子叶植物水稻和玉米的GA20ox序列聚为一个亚枝,PpGA3ox1与双子叶植物拟南芥和番茄的GA3ox序列聚为一个亚枝。滇重楼不同的基因没有被截然聚在单子叶或者双子叶植物类群中,这可能与重楼属植物在被子植物系统发育中的特殊地位有关,重楼属植物子叶为一枚属于单子叶植物,但其叶脉网状、心皮多数、花基数在4以上等特征与双子叶植物有共同之处,所以在被子植物系统发育的研究中具有重要的意义[20]。
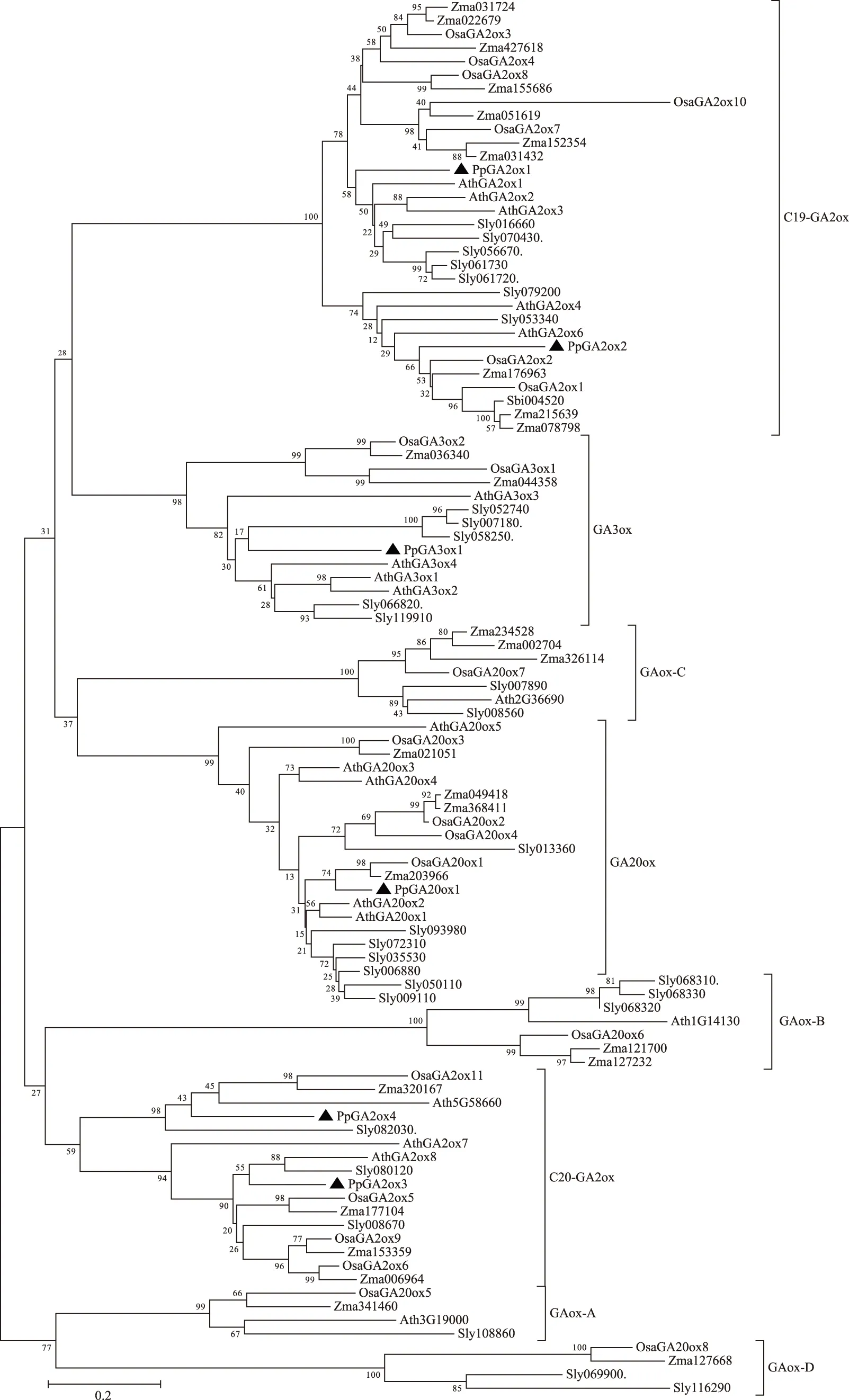
注:▲标记基因为滇重楼基因。图3 GAox基因的系统进化树
2.6 滇重楼GAox蛋白的物理特性预测
滇重楼GAox蛋白的物理特性见表2。滇重楼GAox蛋白的氨基酸数量均在300~400,理论等电点在4~7左右,带正电荷。PpGA2ox1、PpGA2ox2和PpGA2ox4的不稳定系数>40%,为不稳定蛋白;PpGA2ox3、PpGA20ox1和PpGA3ox1的不稳定系数<40%,为稳定蛋白。总平均亲水系数均<0,为亲水蛋白。均没有跨膜结构域和信号肽。除了PpGA2ox1亚细胞定位在叶绿体,其他蛋白均定位在细胞质。
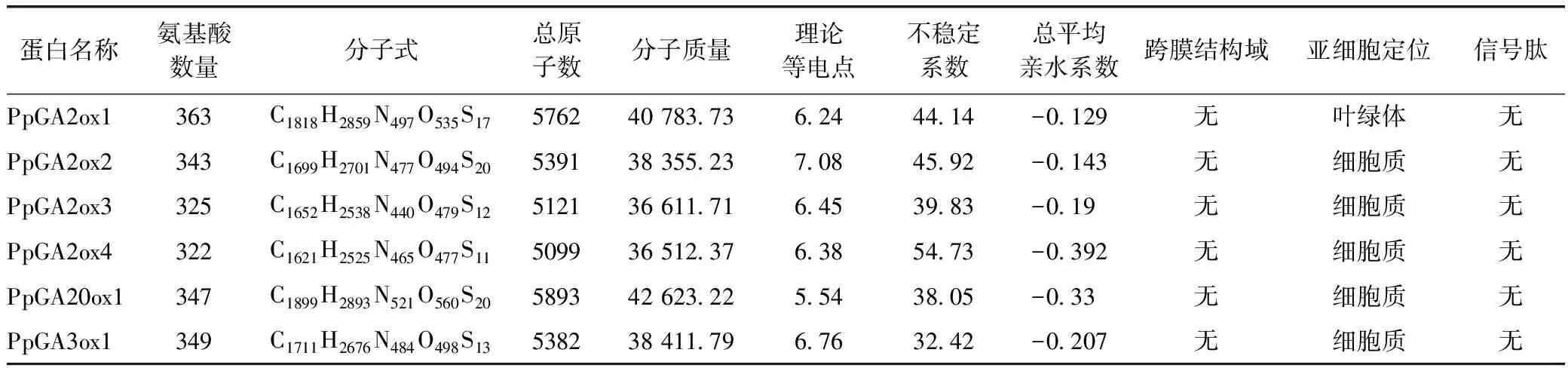
表2 滇重楼GAox蛋白的物理特性预测
2.7 滇重楼GAox蛋白结构域分析
根据系统发育分析的结果选取与PpGAox基因亲缘关系较近的3个蛋白质序列进行多序列比对(见图4~9)。PpGA2ox1与同源GA2ox蛋白的序列相似性为61.73%,PpGA2ox2与同源GA2ox蛋白的序列相似性为61.51%,PpGA2ox3与近缘物种GA2ox蛋白的序列相似性为56.42%,PpGA2ox4与同源GA2ox蛋白的序列相似性为50.75%,PpGA20ox1与同源GA20ox蛋白序列的序列相似性为67.59%,PpGA3ox1与同源GA3ox蛋白的序列相似性为65.23%。由此可见滇重楼GAox蛋白与不同物种同源序列的相似性比较高,保守性比较强。
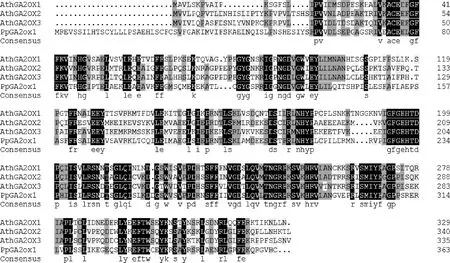
图4 滇重楼PpGA2ox1与同源GA2ox蛋白序列的多序列比对
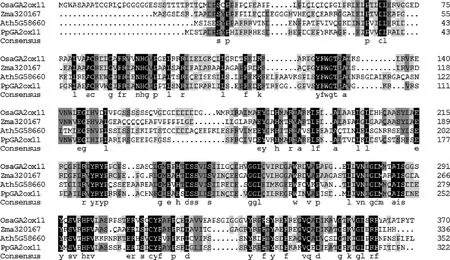
图7 滇重楼PpGA2ox4与同源GA2ox蛋白序列的多序列比对

图8 滇重楼PpGA20ox1与同源GA20ox蛋白序列的多序列比对
InterProScan和conserved domains预测结构域表明,GAox蛋白序列均有3个保守结构域,分别是异青霉素N合成酶(IPNS)结构域、非血红素加氧酶的N-末端(DIOX_N)结构域和依赖于2-酮戊二酸和Fe2+的双加氧酶超家族(2OG-FeII_Oxy)结构域。PpGAox蛋白的上述结构域在氨基酸残基之间的位置见表3。异青霉素N合成酶(IPNS)是一种非血红素Fe2+依赖性酶。它属于一类含有非血红素Fe2+酶,其包括2-酮戊二酸依赖双加氧酶、2-酮戊二酸依赖羟基酶和参与乙烯和花色素苷生物合成的酶。非血红素加氧酶的N-末端(DIOX_N)结构域是具有2-酮戊二酸/Fe(II)-依赖性双加氧酶活性的蛋白质的高度保守的N-末端区域。Fe(II)2OG双加氧酶域酶催化植物激素的形成,如乙烯、赤霉素、花青素等。利用Prosite分析GAox蛋白的功能位点,显示保守功能域均为依赖于2-酮戊二酸和Fe2+的双加氧酶结构域,Fe2+结合于组氨酸(His)残基、天冬氨酸(Asp)残基、组氨酸(His)残基、2-酮戊二酸结合于精氨酸(Arg)残基(见图10)。

图9 滇重楼PpGA3ox1与同源GA3ox蛋白序列的多序列比对
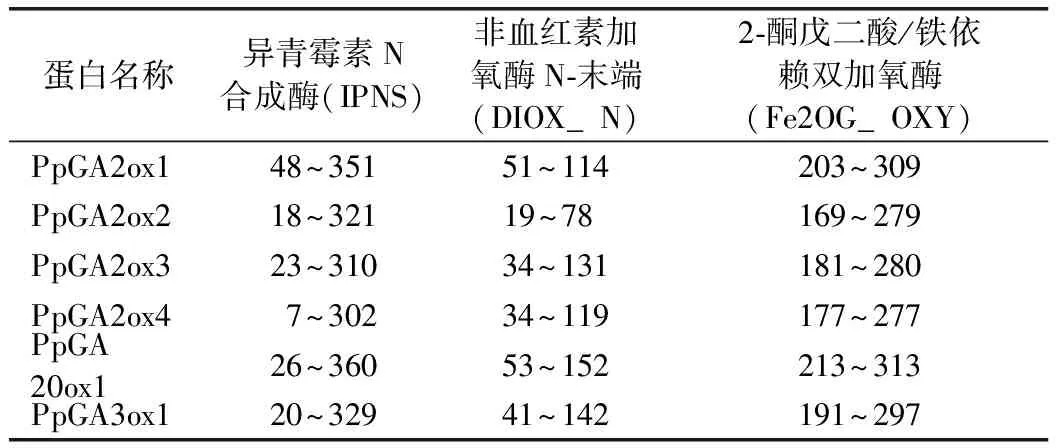
蛋白名称异青霉素N合成酶(IPNS)非血红素加氧酶N⁃末端(DIOX_N)2⁃酮戊二酸/铁依赖双加氧酶(Fe2OG_OXY)PpGA2ox148~35151~114203~309PpGA2ox218~32119~78 169~279PpGA2ox323~31034~131181~280PpGA2ox47~30234~119177~277PpGA20ox126~36053~152213~313PpGA3ox120~32941~142191~297
2.8 滇重楼PpGA2ox1及PpGA2ox1蛋白三维结构建模
基于较高可靠性的PDB模板(4xae.2.A)利用SWISS-MODEL 进行同源建模,GAox蛋白三维结构模型见图11。根据三维蛋白模型可以发现GAox的特征结构域位于GAox蛋白的缝隙内,特征结构域多由无规卷曲和β折叠片组成,在GA生物合成中起重要作用。

图10 GAox蛋白的功能位点
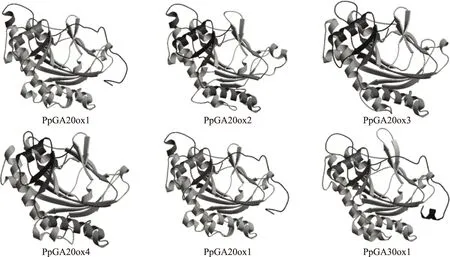
图11 GAox蛋白三维结构预测
3 讨论
近年来,在GA生物合成与代谢的关键酶基因的研究在拟南芥、水稻等植物中取得了比较突出的结果。如水稻的GA2ox基因(OsGA2ox5),大麦和小麦种GAox基因(TaGA20ox3、TaGA3ox3和TaGA2ox7)[22]等。药用植物GAox基因的研究也取得了一定的成果,如丹参GAox基因(SmGA2ox、SmGA20ox和SmGA3ox)[23]、西洋参的GA2ox基因(PqGA2ox)[24]、GA20ox基因(PqGA20ox)[25]、铁皮石斛的GA3ox基因(DoGA3ox)[26]等。GAox家族属于2OGDs超家族[20]。Huang等根据GAox基因的基因结构和特征结构域,GAox基因家族被分为C19-GA2ox、C20-GA2ox、GA20ox、GA3ox、GAox-A、GAox-B、GAox-C和GAox-D8个亚家族[20]。基因结构和保守结构域的研究不仅能够深入了解GAox基因的分类,而且可以对其进化历史和功能多样化进行分析。
此研究基于滇重楼转录组测序数据,应用Primer Premier 5.0软件设计了特异性PCR引物,基于RT-PCR技术第一次成功地克隆了滇重楼的6条GAox基因的全长编码框区,大小在1000 bp左右,分别命名为PpGA2ox1、PpGA2ox2、PpGA2ox3、PpGA2ox4、PpGA20ox1和PpGA3ox1。其中PpGA2ox1和PpGA2ox2属于C19-GA2ox亚家族成员,PpGA2ox3和PpGA2ox4属于C20-GA2ox亚家族成员,PpGA20ox1属于GA20ox亚家族成员,PpGA3ox1属于GA3ox亚家族成员。此外通过系统发育学分析,从分子角度验证了滇重楼在植物系统发育学上的特殊地位:子叶一个属于单子叶植物,但也有属于双子叶植物的一些特征,在研究植物系统发育上具有重要意义。滇重楼GAox蛋白氨基酸数量均在300~400,理论等电点在4~7左右,带正电荷。PpGA2ox1、PpGA2ox2和PpGA2ox4的不稳定系数>40%,为不稳定蛋白;PpGA2ox3、PpGA20ox1和PpGA3ox1的不稳定系数<40%,为稳定蛋白。总平均亲水系数均<0,为亲水蛋白。均没有跨膜结构域和信号肽。除了PpGA2ox1亚细胞定位在叶绿体,其他蛋白均定位在细胞质。滇重楼GAox蛋白与不同物种同源基因的序列相似性在50%~70%,序列相似性高,保守性比较强。结构域预测表明,PpGAox蛋白序列均含有3个保守结构域:异青霉素N合成酶(IPNS)结构域、非血红素加氧酶的N-末端(DIOX_N)结构域和依赖于2-酮戊二酸和Fe2+的双加氧酶超家族(2OG-FeII_Oxy)结构域。利用Prosite分析GAox蛋白的功能位点,显示保守功能域均为为依赖于2-酮戊二酸和Fe2+的双加氧酶结构域,Fe2+结合于组氨酸(His)残基、天冬氨酸(Asp)残基和组氨酸(His)残基,2-酮戊二酸结合于精氨酸(Arg)残基。基于较高可靠性的PDB模板(4xae.2.A)利用SWISS-MODEL 进行同源建立了蛋白的三维模型,根据三维蛋白模型可以发现GAox的特征结构域位于GAox蛋白的缝隙内,特征结构域多由无规卷曲和β折叠片组成,在GA生物合成中起重要作用。研究结果为下一步基于实时定量PCR技术分析滇重楼GAox基因时空表达特性及其调控赤霉素生物合成代谢机制奠定了一定基础。
[1] 李绍平,杨丽英,杨斌,等.滇重楼高效繁育和高产栽培研究[J].西南农业学报,2008,21(4):956-959.
[2] Ye Y,Chen S,Wang L,et al.Effects of stratification at different temperatures on the embryo differentiation and development ofParispolyphyllavar.yunnanensis seeds[J].Pak J Bot,2011,43(3):1507-1512.
[3] 李运昌.重楼属植物引种栽培的研究——Ⅰ.滇重楼的有性繁殖试验初报[J].云南植物研究,1982(4):429-431.
[4] Finkelstein R,Reeves W,Ariizumi T,et al.Molecular aspects of seed dormancy[J].Annu Rev Plant Biol,2008,59:387-415.
[5] Bewley J D,Black M.Seeds:physiology of development and germination[M].New York:Plenum Press,1994:117-145.
[6] Weitbrecht K,Müller K,Leubner-Metzger G.First off the mark:early seed germination[J].Journal of experimental botany,2011,62(10):3289-3309.
[7] Matakiadis T,Alboresi A,Jikumaru Y,et al.The Arabidopsis abscisic acid catabolic gene CYP707A2 plays a key role in nitrate control of seed dormancy[J].Plant Physiology,2009,149(2):949-960.
[8] Hedden P,Phillips A L.Gibberellin metabolism:new insights revealed by the genes[J].Trends in plant science,2000,5(12):523-530.
[9] Hedden P,Thomas S G.Gibberellin biosynthesis and its regulation[J].Biochemical Journal,2012,444(1):11-25.
[10] Kang H G,Jun S H,Kim J,et al.Cloning and molecular analyses of a gibberellin 20-oxidase gene expressed specifically in developing seeds of watermelon[J].Plant physiology,1999,121(2):373-382.
[11] Vidal A M,Gisbert C,Talón M,et al.The ectopic overexpression of a citrus gibberellin 20-oxidase enhances the non-13-hydroxylation pathway of gibberellin biosynthesis and induces an extremely elongated phenotype in tobacco[J].Physiologia Plantarum,2001,112(2):251-260.
[12] Zhao H,Dong J,Wang T.Function and expression analysis of gibberellin oxidases in apple[J].Plant Molecular Biology Reporter,2010,28(2):231-238.
[13] Biemelt S,Tschiersch H,Sonnewald U.Impact of altered gibberellin metabolism on biomass accumulation,lignin biosynthesis,and photosynthesis in transgenic tobacco plants[J].Plant Physiology,2004,135(1):254-265.
[14] Croker S J,García-Lepe R,Lewis M J,et al.Modification of gibberellin production and plant development in Arabidopsis by sense and antisense expression of gibberellin 20-oxidase genes[J].The Plant Journal,1999,17(5):547-556.
[15] Cai P,Long H,Deng G B,et al.Molecular cloning,characterization,and expression analysis of genes encoding gibberellin 20-oxidase in Dasypyrum villosum dwarf mutant[J].Plant Molecular Biology Reporter,2012,30(5):1110-1116.
[16] Walker J M.The proteomics protocols handbook[M].Totowa,NJ:Humana Press,2005.
[17] Petersen T N,Brunak S,von Heijne G,et al.4.0:discriminating signal peptides from transmembrane regions[J].Nat Meth,2011,8 (10):785-786.
[18] Biasini M,Bienert S,Waterhouse A,et al.SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J].Nucleic acids research,2014:42:W252-W258.
[19] 吴建明,陈荣发,黄杏,等.高等植物赤霉素生物合成关键组分GA20-oxidase氧化酶基因的研究进展[J].生物技术通报,2016(7):1-12.
[20] Huang Y,Wang X,Ge S,et al.Divergence and adaptive evolution of the gibberellin oxidase genes in plants[J].BMC evolutionary biology,2015,15(1):207.
[21] 叶晓霞.广西重楼属植物分类学研究及资源现状[D].桂林:广西师范大学,2010.
[22] Stephen P,Huttly A K,Prosser I M,et al.Heterologous expression and transcript analysis of gibberellin biosynthetic genes of grasses reveals novel functionality in theGA3oxfamily[J].BMC Plant Biology,2015,15(1):1-19.
[23] 杜清.丹参赤霉素合成与代谢相关基因家族的鉴定、分子克隆及表达分析[D].北京:北京协和医学院,2015.
[24] 孙同玉,祝娟,孙鹏,等.西洋参PqGA2ox基因的克隆与序列分析[J].中草药,2014,45(24):3599-3606.
[25] 孙同玉,祝娟,孙鹏,等.西洋参赤霉素20-氧化酶基因的克隆与序列分析[J].中草药,2015,46(11):1656-1660.
[26] 刘思思,张岗,陈晓梅,等.铁皮石斛赤霉素3-氧化酶基因的克隆及表达分析[J].中草药,2016,47(6):990-996.
CloningandBioinformaticAnalysisofGAoxGenesfromParispolyphyllavar.yunnanensis
ZHANG Menghuan,LIAO Dengqun,SUN Peng,LI Xian’en,QI Jianjun*
(InstituteofMedicinalPlantDevelopment,ChineseAcademyofMedicalSciences&PekingUnionMedicalCollege,Beijing100193,China)
Objective:To clone the full-length cDNA sequences of gibberellic oxidase genes inParispolyphyllavar.yunnanensiswhich encode the key enzymes involved in GA biosynthetic pathway and analyze their bioinformatic properties.MethodsThe sequences of candidate genes in our previous transcriptomic database ofParispolyphyllavar.yunnanensis were used to design the gene-specific PCR primers for RT-PCR cloning of their full-length coding regions.The amplified cDNA fragments were transformed into pMD18-T vector andE.coliDH5α competent cells for sequencing.A series of bioinformatic analysis were carried out,including sequence comparison of homologies,reconstruction of GAox phylogenetic tree,domain search,and 3D structure prediction.ResultsSequencing results showed that the full-length CDS of sixPpGAoxgenes were about 1000 bp.Six genes,named asPpGA2ox1,PpGA2ox2,PpGA2ox3,PpGA2ox4,PpGA20ox1 andPpGA3ox1,belonged to three GAox subfamilies,encoding 300-400 amino acids.Their theoretical pI values were about 4-7 and in a positive charge.The proteins ofPpGA2ox1,PpGA2ox2 andPpGA2ox4 were unstable andPpGA2ox3,PpGA20ox1 andPpGA3ox1 were stable.However,all of them were hydrophilic,signal peptide and had no transmembrane domain.PpGA2ox1 was predicted to be located in the chloroplast,and the rest d in the cytoplasm.PpaGAox proteins had higher homologous sequences with those of other species w and strong domain conservatism.ConclusionThe full-length cDNA sequences of sixGAoxwere cloned for the first time fromP.polyphyllavar.yunnanensis and the bioinformatic analysis were carried out.
Parispolyphyllavar.yunnanensis;GA oxidase;gene cloning;bioinformatic analysis
10.13313/j.issn.1673-4890.2017.9.002
国家自然科学基金(31471575)
*
祁建军,博士,研究员,硕士生导师,研究方向:药用植物生物技术;E-mail:jjqi@implad.ac.cn
2016-04-11)
