大肠杆菌利用合成生物学策略生产聚羟基脂肪酸酯的研究进展
2017-12-06庄倩倩
庄倩倩
(齐鲁工业大学 生物工程学院 山东省微生物工程重点实验室,山东 济南 250353)
大肠杆菌利用合成生物学策略生产聚羟基脂肪酸酯的研究进展
庄倩倩
(齐鲁工业大学 生物工程学院 山东省微生物工程重点实验室,山东 济南 250353)
合成生物学作为近年来发展迅速的一门交叉学科,为微生物的生物合成提供了强有力的平台工具。微生物细胞工厂可以合成一系列不同种类的聚羟基脂肪酸酯(PHA),而大肠杆菌作为最常用的底盘,正不断运用合成生物学的策略发掘PHA的多样性并降低成本、提高产量。本文中,笔者综述了大肠杆菌利用合成生物学策略生产生物基材料PHA的研究进展,并对其开发与应用前景进行了展望。
大肠杆菌;合成生物学;聚羟基脂肪酸酯
聚羟基脂肪酸酯(PHA)是生物体内一类碳源和能源的天然贮藏材料,它们在碳源过剩、其他大量元素(N、O、P、S)被耗尽的情况下,可以由不同种类的微生物合成[1-3]。PHA主要是由含有不同碳链长度的单体即羟基脂肪酸(hydroxyalkanoate,HA)聚合而成的,组成PHA的羟基脂肪酸主要是3-羟基脂肪酸,也有4-羟基脂肪酸和5-羟基脂肪酸等,其分子量一般为5.0×104~10.0×105[4]。PHA分子量的大小与底盘微生物的种类、底物组成、接种状态、培养条件以及下游加工技术等相关[5]。尽管目前有超过300种微生物可以用来积累PHA聚合物,但只有极少数可以合成工业应用规模的PHA,它们常常具有相对较慢的生长速度和较低的最适生长温度,且体内同时拥有PHA的降解途径,这些对PHA的生产都是极为不利的[6]。大肠杆菌作为一种模式生物,因其生长速度快、培养条件简单、遗传背景清楚、操作简单而被广泛用来生产各种为人类所用的高附加值产品,如生物燃料、生物材料和大宗化学品等。虽然在自然条件下,大肠杆菌无法合成PHA,但是重组大肠杆菌仍然被认为是PHA合成的理想宿主。
合成生物学是21世纪初兴起的一门交叉学科,是在工程学思想的指导下对现有的生物系统进行再设计和改造或从头设计并构建新的生物元件、装置和生物系统,目的是产生天然生物系统所不具备的功效[7]。利用合成生物学的策略,不仅可以设计构建基因线路,还能通过重构细胞代谢网络实现生物基化学品、生物材料、生物能源和生物医药产品等的高效合成。伴随着合成生物学技术和手段的不断革新,大肠杆菌高效生产PHA的研究也在不断突破,许多不同的途径及相关酶被整合,用以合成不同类型的PHA,为生产满足工业应用规模、适用于不同领域的PHA奠定了基础。本文中,笔者综述了近年来大肠杆菌利用合成生物学策略生产PHA的研究进展,以期为低成本高附加值的PHA合成提供理论依据和研发动力。
1 PHA简介
PHA的一般结构式如图1所示。从图1中可以看出,PHA的结构具有多样性。首先,侧链的R1、R2基团是可变基团,它们可以是具有不同碳原子数的饱和或不饱和的烷基基团。主链单体X可以是从1到4变化的,n代表的聚合度(100~30 000),通常直接决定PHA的分子量大小。
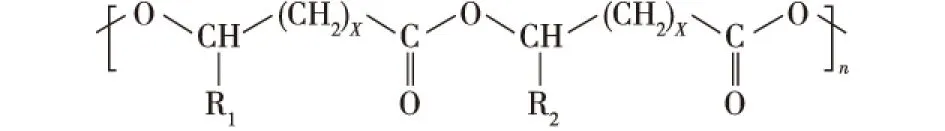
图1 PHA 的结构通式(R1/R2=烷基基团,C1~C13 的饱和或不饱和脂肪酸基团)Fig.1 General structure of PHA (R1/R2=acyl group,C1-C13 unsaturated or saturated fatty acid group)
随着目前追求“绿色聚合物”潮流的日益兴盛,PHA作为一类生物可降解、具有生物兼容性的环境友好型生物基材料,已经被用来作为石油基塑料的替代品[8]。由于原油价格不断攀升,加之石油资源的面临耗竭问题和由传统塑料造成的环境污染日益严重等状况频发,PHA的生产与合成越来越受到研究者们的关注。到目前为止,已经有超过150种的PHA单体组成被报道[9-10]。
PHA的分类依据主要有2个:单体中所含的碳原子数目不同以及所含单体种类的不同。
根据单体中所含碳原子组成数目的不同,PHA可以被分成3种主要的类型:①短链PHA,即scl-PHA,含有3~5个碳原子,例如聚3-羟基丁酸酯(P(3HB))、聚3-羟基戊酸酯(P(3HV))、聚3-羟基丁酸-3-羟基戊酸酯(P(3HB-co-3HV))和聚3-羟基丁酸-4-羟基丁酸酯(P(3HB-co-4HB))等,通常可以由广泛产碱杆菌或罗尔斯通氏菌等产生[11];②中长链PHA,即mcl-PHA,含有6~14个碳原子,例如聚3-羟基己酸酯(PHHx)、聚3-羟基辛酸酯(PHO)和聚3-羟基癸酸酯(PHD)[3,12-13]等,mcl-PHA常由气单胞菌和恶臭假单胞菌等微生物产生[14];③短链-中长链共聚的PHA,即scl-mcl PHA,含有3~14个碳原子,例如聚3-羟基丁酸-3-羟基己酸酯(P(3HB-co-3HHx))、聚3-羟基丁酸-3-羟基己酸-3-羟基辛酸酯(P(3HB-co-3HHx-co-3HO))等[11]。其中,含有中链单体PHA 共聚物的组成主要依赖于培养条件、碳源和PHA合成途径的代谢工程改造[15]。
根据所含单体种类的不同,又可以将PHA 分成均聚物(又称为同聚物)和共聚物两大类。均聚物(同聚物)指的是只含有一种单体的PHA 聚合物;而共聚物是指由两种或两种以上的PHA 单体聚合而成的聚合物。因为共聚物含有的单体种类不同,又有了二元聚合物和三元聚合物之说。二元聚合物有P(3HB-co-3HHx),三元聚合物有P(3HB-co-3HV-co-3HHx)等[16-17]。另外,共聚物还可分为随机共聚物和嵌段共聚物等,其中嵌段共聚物有P3HB-b-P4HB等[18]。因为共聚物是由不同种类的单体聚合而成,因而具有了同种单体聚合而成的均聚物所不具备的某些特性,能够很好地改善均聚物理化性质上的某些不足,从而在一定程度上拓展了PHA的应用范围和适用性[19]。
上述PHA的多样性主要是由3种PHA生物合成途径和PHA聚合酶的特异性及生物加工过程产生的。第一种PHA生物合成途径是乙酰乙酰辅酶A途径,主要用于合成scl-PHA(C3~C5)[20-21];第二种途径是脂肪酸β-氧化途径,主要是利用脂肪酸为碳源合成mcl-PHA[22];第三种途径是脂肪酸从头合成途径,同第二种途径一样,主要是以迭代循环的方式来发挥作用[23-24]。Mcl-PHA的主要前体物是3-酮酰基辅酶A,(R)-3-羟酰基载体蛋白(ACP),(S)-3-羟酰基辅酶A和烯酰基辅酶A,这些都是脂肪酸代谢的中间产物。在过去的20年里,通过引入mcl-PHA合成酶基因,同时过表达烯酰基辅酶A水合酶或3-酮酰基-ACP还原酶,大肠杆菌mcl-PHA的生物合成途径已经被成功构建[25]。
应用基因组编辑技术和代谢工程手段,可以通过控制PHA聚合物中单体组成来调节和改善PHA的生物可降解性、生物兼容性及热力学性能、力学性能等[26]。近年来,利用合成生物学的方法,全球的研究者们试图构建满足不同类型PHA高效生产的平台。
2 DNA克隆和组装的新技术及其在大肠杆菌PHA生物合成中的应用
合成生物学在研究内容上可以分为3个层次:生物元件、模块和底盘。生物元件是合成生物学研究中的最小单位,而保证生物元件在不同的环境中都能正常发挥其生物学功能是合成生物学研究的基石。生物元件主要分为表达特定酶的功能元件和起调控作用的调控元件。模块是由生物元件组装而成的。底盘是承载模块发挥功能的细胞系统。
生物元件和模块的构建优化需要利用DNA克隆和组装技术来实现。传统的克隆方法是通过利用DNA限制性内切酶和连接酶进行酶切、连接的方式实现的。该方法虽然被广泛应用,但无法解决大片段基因簇的克隆难题,不能有效地进行多个片段的一次性组装[27]。近年来,代谢途径改造手段发展迅速,理性的代谢工程可通过使用多样的DNA重组技术和理性的目标鉴定来修饰细胞代谢,从而提高底盘细胞中功能性生物元件的活力,以高效合成目标产物[28]。最新的克隆方法包括BioBrick组装法[29]、Gibson组装法[30]、Golden Gate组装法[31-32]、序列和连接酶不依赖的克隆方法(sequence and ligation-independent cloning,SLIC)[33]等,但这些方法仍存在着大片段基因簇处理成本高、效率低的问题。为了解决这些难题,国内外的研究者们探索了多种方法。
2.1 SSOA法
陈国强教授的实验室开发了一种基于体外单链互补突出末端退火黏连的基因片段提取方法(SSOA)[34]。对于已知基因组序列的大肠杆菌,在基因组的目的基因簇上下游找到特定的限制性内切酶酶切位点,利用得到的酶切位点切割含有目的基因簇的基因组,并经过核酸外切酶消化得到单链DNA互补末端,同时用核酸外切酶处理带有与基因簇末端同源序列的环形或线性载体,经过处理的基因簇和环形或线性载体骨架分别通过特异性的退火黏连及共价结合,进一步形成包含目的基因簇的环形或线形载体。该技术结合KEGG数据库的遗传信息,能够以较高的效率克隆大肠杆菌基因组上的任意一段小于28 kb的基因组片段[34]。但上述方法同时也存在一个问题,即所克隆的基因组片段上必须带有抗性筛选标记。基于此,另一种无需使用抗性基因,而是依赖于体内同源重组的大片段DNA 克隆技术得以开发。该方法主要是通过同源重组将载体插入到基因组上目的基因簇的上游或下游;提取该基因组后利用在基因簇中不存在而在其上下游存在的酶进行酶切消化,并将消化的基因组在体外自连,从而得到带有目的基因簇和克隆载体的新质粒。该方法也被应用到了PHB的生产中,大肠杆菌过表达氢酶-3基因簇和(或)乙酰辅酶A,能够在厌氧条件下将产氢与PHB的合成相偶联,无论外源是否添加乙酸为碳源,都能提高H2和PHB的合成,使得PHB产量提高9倍,H2含量提高10倍以上[35]。
2.2 多片段组装(NE-LIC)法
2013年,Wang等[36]开发了基于切刻酶系统的多片段组装技术(NE-LIC),该方法是基于不依赖于连接的DNA克隆LIC技术实现的。首先切刻酶系统在双链DNA末端同源臂上产生互补单链的3′-,5′-突出末端,并通过互补单链突出末端体外拼接不同长度的多个片段,而后利用特异性重组位点将体外形成的线性多基因片段在体内重组成环,该方法效率高、成本低,可以实现多个基因片段的一次性组装。同年,该种组装方法成功应用于PHB生物合成途径的组装中,将合成所需的6个DNA片段在大肠杆菌中一次性组装,经过发酵发现,重组菌共积累了占细胞干质量50%的PHB。
2.3 基于cyclization after transformation(CAT)策略的DNA组装
研究表明,构建启动子、核糖体结合位点以及终止子等的标准化元件库,可以提高模块的表达水平,同时减少外源功能元件给菌体造成的负担,以此来提高合成模块的利用效率,从而实现目标产物的高效产出[37-39]。2014年,Wu等[40]利用串联启动子策略,将细菌血红蛋白启动子Pvgb在微好氧条件下串联组装在PHB合成基因前,该组装方法与CAT相结合,能够显著提高多基因片段组装的构建效率。经实验表明,当8个串联重复序列作为启动子时,大肠杆菌S17-1的PHB合成量最高,达到5.37 g/L。
2.4 寡聚接头介导的组装(OLMA)法
2016年,Li等[41]在大肠杆菌中利用理性设计的核糖体结合位点库,构建高、中、低拷贝数的质粒,用以过表达PHB合成基因,构建过程应用了寡聚接头介导的组装方法(OLMA),结果表明,菌株在经过了平板筛选和高通量筛选之后,能够积累占细胞干质量0~92%的PHB。该项研究所应用的建库设计、组装和筛选方法为今后其他种类PHA的多酶途径优化提供了有力借鉴。
3 大肠杆菌构建新合成途径合成非天然PHA的研究
应用合成生物学的策略和方法,可以在大肠杆菌中构建全新的代谢途径来实现非天然PHA的合成。2012年,Meng等[12]在大肠杆菌中构建了一个新的合成途径用以合成P(3HP-co-4HB),其中包括5个外源基因,即来自克氏梭菌的编码4-羟基丁酰辅酶A转移酶的orfZ基因、来自橙色绿屈挠菌的编码丙酰辅酶A连接酶ACS区域的pcs’基因、来自恶臭假单胞菌KT2442的编码脱水酶和乙醛脱氢酶的dhaT和aldD基因以及编码PHA合成酶的phaC1基因。该新构建的途径最终合成了单体比例可调控的新型P(3HP-co-4HB),丰富了PHA的多样性[10]。
聚3-羟基丙酸酯(P3HP)以较大的拉伸强度著称,而聚4-羟基丁酸酯(P4HB)是一种具有较高弹性的聚合物。重组大肠杆菌可以合成P3HP和P4HB,从而构建嵌段式共聚物。当外源添加1,4-丁二醇作为4HB前体时,可以合成P4HB,随后添加1,3-丙二醇作为3HP的前体时,就能够共聚成嵌段物P3HP-b-P4HB。该研究合成的非天然PHA即P3HP-b-29%P4HB和P3HP-b-37%P4HB比P(3HP-co-4HB)具有更优越的性能[42]。
传统的mcl-PHA生产主要是通过脂肪酸β-氧化和脂肪酸从头合成途径实现的。这两个途径分别有自己的合成缺陷,脂肪酸β-氧化途径是以脂肪酸为碳源来合成PHA,而脂肪酸是一种较昂贵的碳源且对微生物细胞有毒害作用,因此利用该途径合成PHA具有很大的限制。脂肪酸从头合成途径虽然是利用碳水化合物为碳源生产PHA,但是细胞内积累的PHA含量较低。基于上述原因,迫切需要构建一个利用廉价、不相关碳源高效生产PHA的平台,从而提供充足的(R)-3-羟酰基辅酶A底物。2014年,Zhuang等[43]着眼于解决上述问题,在重组大肠杆菌中构建了逆向脂肪酸β-氧化循环中所需酶的过表达质粒,试图直接利用葡萄糖为碳源,通过构建的逆向脂肪酸β-氧化循环,合成含有不同单体的mcl-PHA共聚物。当在单敲除硫酯酶基因菌株的发酵培养基中添加30 g/L葡萄糖时,敲除tesB基因的菌株合成的mcl-PHA含量最高,约占细胞干质量的4.01%;敲除yciA基因的菌株次之,约占细胞干质量的2.04%;敲除tesA基因的菌株合成的mcl-PHA含量最低。为了进一步提高产量,在重组大肠杆菌中同时敲除了2个硫酯酶基因,得到双敲除菌株,其中敲除tesB和yciA基因的菌株合成的mcl-PHA的含量最高,可达6.62%(质量分数)。为了合成scl-mcl PHA,笔者用来自施氏假单胞菌的PHA聚合酶-PhaC2Ps替换合成mcl-PHA的来自铜绿假单胞菌的PHA聚合酶-PhaC2Pa,构建新的合成质粒,转入大肠杆菌进行发酵。在添加 30 g/L葡萄糖的情况下,能够合成占细胞干质量约12.10%的scl-mcl PHA,其中3HB占21.18%(摩尔百分数),中链单体占78.82%(摩尔百分数)。该生产途径可以在经过优化之后用于工业化的大规模生产。
4 大肠杆菌调控合成途径产生非天然PHA的研究
合成生物学的策略不仅可以构建新的PHA合成途径,也可以用来调控PHA生物合成的代谢流。例如CRISPR(clustered regularly interspaced short palindromic repeats)系统作为gRNA介导的转录调控平台高效的合成生物学工具,能够抑制功能元件的表达量,从而实现对代谢途径的调控[44]。该系统也可以应用到PHA的合成代谢流调控和单体组成改造上。Lv等[45]在重组大肠杆菌中,以葡萄糖为碳源,利用CRISPRi技术合成P3HB4HB。其中,通过设计不同的sgRNA,不同程度地抑制了编码琥珀酸半醛脱氢酶的sad基因,从而调控了流向4HB合成途径的代谢流强度,最终合成了含有不同摩尔分数(1%~9%)4HB的P3HB4HB;继续抑制琥珀酰辅酶A合成酶基因和琥珀酸脱氢酶基因(sucC、sucD、sdhA和sdhB),可进一步增强4HB合成途径的代谢流,使得P3HB4HB中4HB的组成比例扩大到了18.4%(摩尔分数)。本研究结果表明,CRISPRi是一种能够在大肠杆菌中同时调控多个基因表达,从而调控细胞代谢流的方法。
5 展望
当前,随着合成生物学策略和方法的不断发展与日趋完善,越来越多的技术手段被应用于PHA的生物合成之中。但是,由于传统PHA的单体结构和组成难以调控、生产成本较高,而且已有的mcl-PHA和scl-mcl PHA种类单一、应用范围有限,因此,适用于生物材料、医药化工等领域的新型PHA亟待开发合成。
此外,大肠杆菌作为目前合成PHA最常用的底盘之一,可以通过进一步对其进行理性的设计优化和代谢改造,阻断竞争性的代谢旁支路径,使之进行基本生命活动所需的能量更小,利于代谢流量更多地进入PHA的合成途径,从而提高PHA的合成量。同时应用更为廉价的碳源,开发更为简便易行的下游生物加工过程,都为实现PHA的可持续高产和大规模商业化的“绿色制造”提供了新的研究方向。
[1] RAI R,KESHAVARZ T,ROETHER J A,et al.Medium chain length polyhydroxyalkanoates,promising new biomedical materials for the future[J].Mater Sci Eng R,2011,72(3):29-47.
[2] LI R,CHEN Q,WANG P G,et al.A novel-designedEscherichiacolifor the production of various polyhydroxyalkanoates from inexpensive substrate mixture[J].Appl Microbiol Biotechnol,2007,75(5):1103-1109.
[3] SATOH Y,MURAKAMI F,TAJIMA K,et al.Enzymatic synthesis of poly(3-hydroxybutyrate-co-4-hydroxybutyrate) with CoA recycling using polyhydroxy-alkanoate synthase and acyl-CoA synthetase[J].J Biosci Bioeng,2005,99(5):508-511.
[4] REHM B.Polyester synthases:natural catalysts for plastics[J].Biochem J,2003,376:15-33.
[5] LEE S Y.Plastic bacteria:progress and prospects for polyhydroxyalkanoate production in bacteria[J].Trends Biotechnol,1996,14(11):431-438.
[6] 庄倩倩.利用逆向脂肪酸β-氧化合成中长链聚羟基脂肪酸酯[D].济南:山东大学,2015.
[7] ENDY D.Foundations for engineering biology[J].Nature,2005,438:449-453.
[8] PARK S J,AHN W S,GREEN P R,et al.Production of poly(3-hydroxybutyrate-co-3-hydroxyhexanoate) by metabolically engineeredEscherichiacolistrains[J].Biomacromolecules,2001,2(1):248-254.
[9] LAYCOCK B,HALLEY P,PRATT S,et al.The chemomechanical properties of microbial polyhydroxy-alkanoates[J].Prog Polym Sci,2014,39(2):397-442.
[10] GAO X,CHEN J C,WU Q,et al.Polyhydroxy-alkanoates as a source of chemicals,polymers,and biofuels[J].Curr Opin Biotechnol,2011,22:768-774.
[11] LI R,ZHANG H X,QI Q S.The production of polyhydroxyalkanoates in recombinantEscherichiacoli[J].Bioresour Technol,2007,98(12):2313-2320.
[12] MENG D C,SHI Z Y,WU L P,et al.Production and characterization of poly(3-hydroxypropionate-co-4-h y d r o x y b u t y r a t e) with fully controllable structures by recombinantEscherichiacolicontaining an engineered pathway[J].Metab Eng,2012,14:317-324.
[13] LI Z J,SHI Z Y,JIAN J,et al.Production of poly(3-hydroxybutyrate-co-4-hydroxybutyrate) from unrelated carbon sources by metabolically engineeredEscherichiacoli[J].Metab Eng,2010,12:352-359.
[14] PARK S J,KANG K H,LEE H,et al.Propionyl-CoA dependent biosynthesis of 2-hydroxybutyrate containing p o l y h y d r o x y a l k a n o a t e s in metabolically engineeredEscherichiacoli[J].J Biotechnol,2013,165(2):93-98.
[15] RAI R,BOCCACCINI A R,KNOWLES J C,et al.The homopolymer poly(3-hydroxyoctanoate) as a matrix material for soft tissue engineering[J].J Appl Polym Sci,2011,122(6):3606-3617.
[16] REDDY M V,MOHAN S V.Influence of aerobic and anoxic microenvironments on polyhydroxyalkanoates (PHA) production from food waste and acidogenic effluents using aerobic consortia[J].Bioresour Technol,2012,103(1):313-321.
[17] LIU Q,LUO G,ZHOU X R,et al.Biosynthesis of poly(3-hydroxydecanoate) and 3-hydroxydodecanoate dominating polyhydroxyalkanoates byβ-oxidation pathway inhibitedPseudomonasputida[J].Metab Eng,2011,13:11-17.
[18] HU D,CHUNG A L,WU L P,et al.Biosynthesis and characterization of polyhydroxyalkanoate block copolymer P3HB-b-P4HB[J].Biomacromolecules,2011,12(9):3166-3173.
[19] MUHR A,RECHBERGER E M,SALERNO A,et al.Biodegradable latexes from animal-derived waste:biosynthesis and characterization of mcl-PHA accumulated byP.citronellolis[J].React Funct Polym,2013,73(10):1391-1398.
[20] WANG Q,TAPPEL R C,ZHU C,et al.Development of a new strategy for production of medium-chain-length polyhydroxyalkanoates by recombinantEscherichiacolivia inexpensive non-fatty acid feedstocks[J].Appl Environ Microbiol,2012,78(2):519-527.
[21] DOI Y,TAMAKI A,KUNIOKA M,et al.Production of copolyesters of 3-hydroxybutyrate and 3-hydroxyvalerate byAlcaligeneseutrophusfrom butyric and pentanoic acids[J].Appl Microbiol Biotechnol,1988,28(4):330-334.
[22] SUDESH K,ABE H,DOI Y.Synthesis,structure and properties of polyhydroxyalkanoates:biological polyesters[J].Prog Polym Sci,2000,25(10):1503-1555.
[23] WANG H,LI X,CHEN G Q.Production and characterization of homopolymer polyhydroxyheptanoate (P3HHp) by afadBAknockout mutantPseudomonasputidaKTOY06 derived fromP.putidaKT2442[J].Process Biochem,2009,44(1):106-111.
[24] REHM B H,STEINBüCHEL A.Biochemical and genetic analysis of PHA synthases and other proteins required for PHA synthesis[J].Int J Biol Macromol,1999,25:3-19.
[25] OJUMU T V,YU J,SOLOMON B O.Production of polyhydroxyalkanoates,a bacterial biodegradable polymers[J].Afr J Biotechnol,2004,3(1):18-24.
[26] MENG D C,SHEN R,YAO H,et al.Engineering the diversity of polyesters[J].Curr Opin Microbiol,2014,29:24-33.
[27] FU J,BIAN X,HU S,et al.Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J].Nat Biotechnol,2012,30:440-446.
[28] KOFFAS M,LEE K,ROBERGE C.Metabolic engineering[J].Annu Rev Biomed Eng,1999,1:535-557.
[29] ANDERSON J,DUEBER J E,LEGUIA M,et al.BglBricks:a flexible standard for biological part assembly[J].J Biol Eng,2010,4(1):1-12.
[30] GIBSON D G,YOUNG L,CHUANG R Y,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases[J].Nat Methods,2009,6(5):343-345.
[31] ENGLER C,KANDZIA R,MARILLONNET S.A one pot,one step,precision cloning method with high throughput capability[J].PLoS ONE,2008,3:e3647.
[32] ENGLER C,GRUETZNER R,KANDZIA R,et al.Golden gate shuffling:a one-pot DNA shuffling method based on type IIs restriction enzymes[J].PLoS ONE,2009,4:e5553.
[33] LI M Z,ELLEDGE S J.Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC[J].Nat Methods,2007,4(3):251-256.
[34] WANG R Y,SHI Z Y,CHEN J C,et al.Cloning large gene clusters fromE.coliusing in vitro single-strand overlapping annealing[J].ACS Synth Biol,2012,1(7):291-295.
[35] WANG R Y,SHI Z Y,CHEN J C,et al.Enhanced co-production of hydrogen and poly-(R)-3-hydroxybutyrate by recombinant PHB producingE.coliover-expressing hydrogenase 3 and acetyl-CoA synthetase[J].Metab Eng,2012,14:496-503.
[36] WANG R Y,SHI Z Y,GUO Y Y,et al.DNA fragments assembly based on nicking enzyme system[J].PLoS ONE,2013,8:e57943.
[37] ALPER H,FISCHER C,NEVOIGT E,et al.Tuning genetic control through promoter engineering[J].Proc Natl Acad Sci USA,2005,102:12678-12683.
[38] SALIS H M,MIRSKY E A,VOIGT C A.Automated design of synthetic ribosome binding sites to control protein expression[J].Nat Biotechnol,2009,27:946-950.
[39] CHEN Y J,LIU P,NIELSEN A A,et al.Characterization of 582 natural and synthetic terminators and quantification of their design constraints[J].Nat Methods,2013,10(7):659-664.
[40] WU H,WANG H,CHEN J,et al.Effects of cascadedvgbpromoters on poly(hydroxybutyrate) (PHB) synthesis by recombinantEscherichiacoligrown micro-aerobically[J].Appl Microbiol Biotechnol,2014,98:10013-10021.
[41] LI T,YE J,SHEN R,et al.Semirational approach for ultrahigh poly(3-hydroxybutyrate) accumulation inEscherichiacoliby combining one-step library construction and high-throughput screening[J].ACS Synth Biol,2016,5(11):1308-1317.
[42] TRIPATHI L,WU L P,MENG D,et al.Biosynthesis and characterization of diblock copolymer of p(3-hydroxypropionate)-block-p(4-hydroxybutyrate) from recombinantEscherichiacoli[J].Biomacromolecules,2013,14(3):862-870.
[43] ZHUANG Q Q,WANG Q,LIANG Q F,et al.Synthesis of polyhydroxyalkanoates from glucose that contain medium-chain-length monomers via the reversed fatty acidβ-oxidation cycle inEscherichiacoli[J].Metab Eng,2014,24:78-86.
[44] QI L S,LARSON M H,GILBERT L A,et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J].Cell,2013,152:1173-1183.
[45] LV L,REN Y L,CHEN J C,et a1.Application of CRISPRi for prokaryotic metabolic engineering involving multiple genes,a case study:controllable p(3HB-co-4HB) biosynthesis[J].Metab Eng,2015,29:160-168.
(责任编辑 荀志金)
ProgressinsyntheticbiologyofEscherichiacolitoproducepolyhydroxyalkanoates
ZHUANG Qianqian
(Shandong Provincial Key Laboratory of Microbial Engineering,School of Bioengineering,Qilu University of Technology,Jinan 250353,China)
As a rapidly developmental interdiscipline,synthetic biology offers powerful tools for microbial synthesis.Microbial cell factories can synthesize a variety of polyhydroxyalkanoates (PHA).The diversity of PHA can be explored and the cost of PHA production can be reduced using strategies of synthetic biology inEscherichiacoli.In addition,PHA yield can be increased.We review here the progress in production of PHA via strategies of synthetic biology inE.coliand indicate the research and application prospects of PHA.
Escherichiacoli;synthetic biology;polyhydroxyalkanoates
10.3969/j.issn.1672-3678.2017.06.006
2017-08-09
山东省自然科学基金(ZR2016CB03)
庄倩倩(1985—),女,山东济南人,博士,研究方向:微生物代谢调控、合成生物学,E-mail:zqq0608@126.com
Q819
A
1672-3678(2017)06-0038-06
