甲基营养菌代谢网络途径和代谢工程改造的研究进展
2017-12-06陈文静袁肖杰杨益明朱丽萍
杨 靖,陈文静,张 敏,袁肖杰,杨益明,朱丽萍,3,杨 松,3
(1.青岛农业大学 生命科学学院,山东 青岛 266109;2.青岛农业大学 山东省应用真菌重点实验室,山东 青岛 266109;3.青岛农业大学 青岛生物沼气环境微生物国际合作基地,山东 青岛 266109)
甲基营养菌代谢网络途径和代谢工程改造的研究进展
杨 靖1,2,陈文静1,2,张 敏1,2,袁肖杰1,2,杨益明1,2,朱丽萍1,2,3,杨 松1,2,3
(1.青岛农业大学 生命科学学院,山东 青岛 266109;2.青岛农业大学 山东省应用真菌重点实验室,山东 青岛 266109;3.青岛农业大学 青岛生物沼气环境微生物国际合作基地,山东 青岛 266109)
一碳化合物(甲醇或甲烷)高值化转化是当前生物工程领域的重要研究热点。甲基营养菌是一类以甲醇为碳源和能源生长的革兰氏阴性菌,随着基因组测序的开展和各类组学技术的发展,以扭脱甲基杆菌为代表的甲基营养菌的中心代谢网络途径和相关功能基因逐渐明晰。近年来,甲基营养菌中包括基因过表达、基因敲除整合等遗传操作工具的完善以及各种基因调控元件的开发,为甲基营养菌的底盘改造和异源途径优化表达提供了重要基础,国内外的研究推动了甲醇转化成多种高附加值产品。此外,人们渴望利用传统基因工程菌作为底盘构建合成型甲基菌,以期更高效实现甲醇催化转化。本文中,笔者综述了目前甲基营养菌,尤其是扭脱甲基杆菌的代谢网络特点、代谢工程改造策略与应用以及构建合成型甲基细菌这3个方面的研究进展,以期为甲基微生物催化转化的研究提供借鉴。
甲基营养菌;代谢工程;代谢网络途径;合成型甲基细菌;甲醇
甲基营养菌(methylotrophs)是能利用甲醇或甲烷等一碳化合物作为唯一碳源和能源的革兰氏阴性菌。甲基营养菌可以从植物叶片及根际、土壤、湖泊与海底沉积物等各种生境中分离得到。目前,已通过实验室培养条件分离到的不同种属甲基营养菌包括扭脱甲基杆菌(Methylobacteriumextorquens)、荚膜甲基球菌(Methylococcuscapsulatus)、甲醇芽孢杆菌(Bacillusmethanolicus)、耐辐射甲基杆菌(M.radiotolerans)和氯甲基杆菌(M.chloromethanicum)等[1]。其中,扭脱甲基杆菌M.extorquensAM1在1960年从甲胺的空气污染物中分离得到,是目前研究最多的甲基模式菌[2]。2009年,扭脱甲基杆菌全基因组测序诠释,其基因组全长6.88 Mb,包含1个5.51 Mb染色体,1个1.26 Mb大质粒和3个小质粒[3-4],继而包括基因过表达、基因敲除和基因整合等操作工具及多层次组学方法的相继开发[5],为在扭脱甲基杆菌中挖掘新基因和开展代谢工程改造奠定重要前期基础。
目前,生物燃料、精细化学品和药物分子等(如:烃类燃料、大宗有机酸和烯萜类衍生物)的微生物合成主要利用玉米淀粉等来源的葡萄糖作为碳源和能源,但是大规模化生产存在与人畜争粮争地的原料瓶颈问题[6]。近年来,随着代谢工程与合成生物技术发展,非传统碳源(如:甲醇、甲烷、CO2和CO)受到极大关注[6-8]。与葡萄糖相比,甲醇不但含有更多电子,为高还原度化学品合成提供更多还原力,而且可从沼气或合成气等多种可再生资源中获得,市场价格低廉,便于运输,供应不受季节和气候影响[6,9],因而甲基微生物合成路线具有明显发展优势,甲醇相对于传统碳源可以成为微生物合成路线的一种重要的非食品替代基质。2013年,美国能源部和欧盟分别启动ARPA-E(advanced research projects agency for energy)和PROMYSE(products from methanol by synthetic cell factories)创新专项,资助4 000多万美元,旨在推动甲醇/甲烷生物高值转化的研究[10-11]。
本文中,笔者主要综述了目前甲基营养菌,尤其是其模式菌扭脱甲基杆菌的代谢网络特点、代谢工程改造与应用以及构建合成型甲基细菌这3个方面的研究内容与进展,以期为甲基微生物催化转化的研究提供借鉴。
1 甲基营养菌的中心代谢网络
甲基营养菌(扭脱甲基杆菌)中心代谢网络如图1所示。由图1可知,它主要包括3个模块,一碳化合物(甲醇、甲胺等)氧化模块、一碳中间代谢物异化模块和一碳中间代谢物的同化模块。一碳化合物的氧化是利用甲醇脱氢酶氧化一碳底物,转化为甲醛。一碳化合物的异化过程是经过多步一碳传递,并最终将甲酸氧化为CO2,同时产生还原力。甲基营养菌根据同化模块的特点又分为α-变形菌类和γ-变形菌类,其中α-变形菌类甲基营养菌,以扭脱甲基杆菌作为代表,通过丝氨酸循环(serine cycle)耦合乙酰丙二酰辅酶A途径(ethylmalonyl-CoA pathway,EMCP )同化甲酸;而γ-变形菌类甲基营养菌以甲醇芽孢杆菌(Bacillusmethanolicus)为代表,通过核酮糖单磷酸途径(ribulose monophosphate pathway,RuMP)直接同化甲醛。

图1 扭脱甲基杆菌中心代谢网络及高值产品的合成Fig.1 Central metabolic network pathway and the synthesis of high value-addedproducts of Methylobacterium extorquens
1.1 一碳化合物的氧化模块
甲醇是甲基营养菌的主要一碳碳源,甲基营养菌细胞周质空间的甲醇脱氢酶(MDH)负责氧化甲醇。前期普遍认为扭脱甲基杆菌的甲醇脱氢酶是一种吡咯喹啉醌(PQQ)依赖性的酶,不需消耗还原力NAD(P)H,Ca2+结合到活性位点将甲醇催化氧化为甲醛,该甲醇脱氢酶是一个α2β2的四聚体结构,辅酶PQQ深埋在一个疏水口袋中,作为电子受体参与酶催化过程[12]。然而,最近研究发现甲醇脱氢酶除了包含上述MXAF型甲醇脱氢酶(MXAF-type MDH),还包括XOXF型甲醇脱氢酶(XOXF-type MDH)[13]。
扭脱甲基杆菌的基因组研究分析表明,MXAF类型甲醇氧化系统至少有25个基因参与MDH催化以及调控[14]。这些基因分为5个基因簇mxa、mxb、pqqABC/DE、pqqFG和mxc。mxa基因簇包括mxaFJGIRSACKLDEHB和位于其上游的mxaW基因,mxaF和mxaI分别编码MDH的α亚基(6.6×104)和β亚基(9.0×103),mxaG编码细胞色素C的结构蛋白[15],mxaJ、mxaR、mxaS、mxaD、mxaE和mxaH基因的功能未知,猜测可能与MDH的装配与稳定性有关[16],mxaACK和mxaL负责将Ca2+插入甲醇脱氢酶催化活性位点[17],mxaB是甲醇氧化相关基因的调控因子,mxaW的功能还不清楚,对该基因做突变也无任何表型,但在其上游存在一个甲醇诱导型启动子[18]。pqqABCDE和pqqFG构成一个基因簇,它与PQQ的合成有关[19]。mxbD、M和mxcQ、E这4个基因与甲醇氧化系统的转录调控有关[20-21]。
近年对甲基营养菌基因组分析发现,存在另一类重要的催化甲醇氧化的脱氢酶XOXF-type MDHs,XOXF型的甲醇脱氢酶同样依赖辅酶PQQ,但是以镧系元素(如:镧和铈)结合到活性位点催化甲醇氧化。宏基因组和蛋白质组数据表明[13],在环境样品中,XOXF型甲醇脱氢酶比MXAF型甲醇脱氢酶分布更广泛;系统发育分析表明,XOXF型甲醇脱氢酶至少分为5个不同的系统发育分支,而MXAF型甲醇脱氢酶只是其中的一小部分,所以MXAF型甲醇脱氢酶可能是在XOXF型甲醇脱氢酶基础上进化而来的[22]。XOXF型甲醇脱氢酶是由2个亚基组成的二聚体结构,通过晶体结构分析和密度泛函理论计算[23]发现:以镧系元素而不是Ca2+结合到活性位点,使得XOXF型甲醇脱氢酶具有更高的催化效率。Skovran等[24]发现扭脱甲基杆菌上XOXF型甲醇脱氢酶具有2个同源基因xoxF1和xoxF2,它们之间有90%的氨基酸相似性,与mxaF基因有50%相似性。在以甲醇为碳源、外加镧离子(La3+)时发现,xoxF1基因的单一突变导致扭脱甲基杆菌的部分生长缺陷,而xoxF2的突变没有明显的生长缺陷,因此,xoxF1发挥更为主要的作用;同时他们还发现:当以甲醇为碳源,加入Ca2+、不加La3+时,MXAF型甲醇脱氢酶将甲醇氧化成甲醛过程中,xoxF基因起到信号转导作用,xoxF突变菌株无法正常生长,故maxF基因的表达依赖于xoxf基因。Vu等[25]发现扭脱甲基杆菌以甲醇为碳源,外加La3+培养,当La3+浓度从25 nmol/L不断增加时,xoxF基因的启动子xox1的转录水平不断提高,在250 nmol/L的La3+浓度下,xox1达到最大表达量,由此发现:xox1是镧依赖型启动子,并对mxaF的启动子mxa的转录有明显抑制作用。此外,当以甲醇为碳源、不加La3+的情况下,xoxF2单突变体菌株生长基本不受影响,xoxF1单突变体菌株生长受到明显抑制,xoxF1和xoxF2双突变体菌株不能生长,三突变体菌株(敲除mxaF、xoxF1和xoxF2)也不能生长,但对三突变体培养体系外加镧系元素,菌株能够缓慢生长,这证明还可能存在另一种未知的醇脱氢酶,具有氧化甲醇的能力。Good等[26]进一步发现,当以甲醇为碳源、外加镧系元素时,乙醇脱氢酶(EXAF)发挥氧化甲醇的作用,实验数据表明,该乙醇脱氢酶是目前报道的最有效的PQQ-依赖型的乙醇脱氢酶,成为第一个非XOXF型的醇氧化系统中使用镧系元素作为辅助因子的脱氢酶。
1.2 一碳中间代谢物(甲醛)的异化模块
Vorholt等[27]在2000年发现:扭脱甲基杆菌中存在甲醛活化酶(FAE)催化甲醛转化为methylene-H4MPT,继而methylene-H4MPT脱氢酶(MTDAB)把methylene-H4MPT氧化为methenyl-H4MPT,同时产生1分子NAD(P)H为同化作用提供还原力[28];后经methenyl-H4MPT环化水解酶(MCH)转化为formyl-H4MPT,在甲酰-甲烷呋喃脱氢酶(formyl-MFR dehydrogenase, FCHABCD)作用下,水解formyl-H4MPT产生甲酸[29];最后由甲酸脱氢酶(FDH)彻底氧化甲酸,释放1分子CO2,并产生还原力NADH。扭脱甲基杆菌中存在编码甲酸脱氢酶的4个基因,对其中的3个基因突变后,突变株仍能在甲醇上生长,但有甲酸积累,对第4个基因进行突变后失去了生长能力,这说明了甲酸氧化对甲基营养菌在甲醇上生长的重要作用[30]。
1.3 一碳中间物(甲醛或甲酸)的同化模块
丝氨酸循环是最早在扭脱甲基杆菌中发现的同化模块,其一碳单元传递过程中首先消耗1分子ATP和1分子NADH生成formyl H4F,与甘氨酸在丝氨酸羟甲基转移酶(GlyA)催化作用下生成丝氨酸,进入丝氨酸循环,丝氨酸一部分转化成乙醛酸用于甘氨酸再生,另一部分则直接转化成乙酰辅酶A。在扭脱甲基杆菌中,丝氨酸循环的基因都在2个操纵子上转录,sga-hpr-mtdA-fch为qsc1操纵子,mtkA-mtkB-ppc-mclA为qsc2操纵子,且受QscR调控子的调控[31]。
丝氨酸循环的中间代谢产物与糖异生、三羧酸循环(TCA)共用,因此,乙醛酸的再生是保持循环正常持续所必须的,但是在扭脱甲基杆菌中缺少乙醛酸循环的关键酶异柠檬酸裂解酶(isocitratelyase),直到2007年,Erb等[32]发现扭脱甲基杆菌能够合成一系列以C5-二羧酸作为配体的辅酶A,提出乙酰丙二酰辅酶A途径(EMCP)实现乙醛酸的再生。2009年,Peyraud等[33]用13C标记代谢组测定了不同的辅酶A衍生物的生成顺序,进一步完整证明了EMCP途径和再生乙醛酸的功能。在EMCP代谢途径中,乙酰辅酶A在3个酶连续催化作用下生成巴豆酰辅酶A,在巴豆酰辅酶A羧化酶/还原酶(CCR)催化下生成乙基丙二酰辅酶A,这是EMCP途径中的第1个羧化反应,继而在乙基丙二酰辅酶A异构酶(EPI)和乙基丙二酰辅酶变位酶(ECM)催化下生成甲基琥珀酰辅酶A,再生成甲基延胡索酰辅酶A、甲基苹果酰辅酶A,随后裂解生成1分子丙酰辅酶A和1分子乙醛酸,乙醛酸再生进入丝氨酸循环,丙酰辅酶A与CO2发生羧化反应生成甲基丙酰辅酶A,这是EMCP途径中第2个羧化反应。最后生成琥珀酰辅酶A转化成苹果酸,进入丝氨酸循环。EMCP途径中的大部分酶都是与其他代谢途径共享的,只有乙基丙二酰辅酶变位酶(ECM)和巴豆酰辅酶A羧化酶/还原酶(CCR)是该途径所特有的[32,34],在EMCP代谢途径中包括两步NADPH消耗反应。2016年,笔者所在课题组的Cui等[35]在过表达ecm、phaA和mcmAB基因后发现:过表达ecm基因能够重新分配碳流量分布,降低合成PHB的代谢流,强化EMCP代谢通量,导致PHB降低4.5倍,并使中间代谢物3-羟基丙酰辅酶A提高1.6倍,使乙醛酸积累,超过丝氨酸循环的消耗能力将其释放到胞外,从而抑制细胞生长。由此可见,EMCP途径各个基因的协同调控才能促使甲基营养菌在甲醇上更好的生长。
甲基营养菌(如甲醇芽孢杆菌)还可以直接利用核酮糖单磷酸(RuMP)途径同化甲醛,核酮糖单磷酸途径由2种核心酶组成,即3-己酮糖-6-磷酸合成酶(HPS)和6-磷酸-3己糖异构酶(PHI)。HPS催化甲醛和核酮糖-5磷酸生成己酮糖-6-磷酸。PHI使己酮糖-6-磷酸异构化为果糖-6-磷酸[36-37]。这条途径产生1分子ATP和1分子NADH,因而从能量和还原力的角度看,该路径比丝氨酸循环途径具有更高效率,使得核酮糖单磷酸途径成为在异源宿主中构建一碳代谢网络的首选同化途径。
2 代谢工程改造甲基营养菌合成高值产品
虽然在1970年,甲醇用于工业化生产单细胞蛋白[36],但是直到近年,随着基因工程技术与组学技术的发展,在甲基营养菌中构建重组底盘将甲醇转化为各类高附加值产品(如:生物燃料、烯萜类化合物和有机酸)才备受关注,见表1。
在过去几十年中,生物法生产的二元羧酸产物是一个具有显著进步的新兴研究领域。2014年,Sonntag等[38]在扭脱甲基杆菌中利用EMCP途径中间代谢物作为前体物,通过异源表达硫酯酶(thioesterases)YCIA水解甲基琥珀酰辅酶A(mesaconyl-CoA)和中康酰辅酶A(methylsuccinyl-CoA)释放出辅酶A生成相应的二元羧酸、中康酸和(2S)-甲基琥珀酸,继而通过降低培养基中Na+浓度,实现中康酸和(2S)-甲基琥珀酸产量的提高,并且以敲除聚羟基脂肪酸酯合成酶(PHAC)的表达来阻止EMCP代谢流流向聚羟基丁酸酯(PHB)的合成,最终产物浓度增加5倍。此外,微调生长限制性金属钴浓度可以使甲基琥珀酰辅酶A和中康酰辅酶A在胞内大量积累(增加16倍),进而导致中康酸和(2S)-甲基琥珀酸的合成增加6倍,最终产量达到0.65 g/L,得率为17%(以1 g甲醇为参照)[46]。
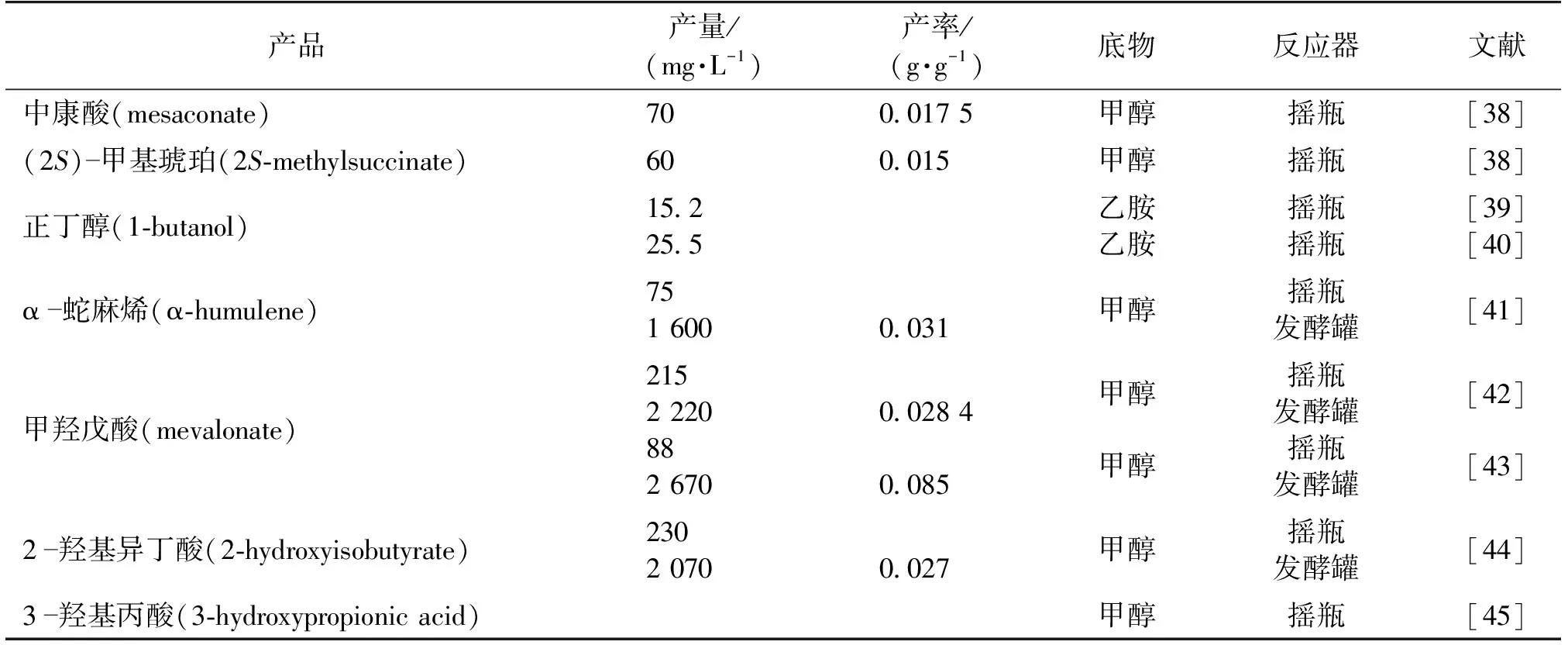
表1 源于甲醇高附加值产品
Hu等[39]和笔者所在课题组合作,在扭脱甲基杆菌利用EMCP途径的中间代谢产物巴豆酰辅酶A为前体物异源表达来自齿垢密螺旋体(Treponemadenticola)中的反式烯酰辅酶A还原酶(trans-enoyl-CoA reductase)和丙酮丁醇梭菌(Clostridiumacetobutylicum)中的醇脱氢酶(alcohol dehydrogenase),构建了合成正丁醇的异源途径(其中反式烯酰辅酶A可以催化巴豆酰辅酶A生成丁酰辅酶A,丁酰辅酶A在醇脱氢酶的催化下生成正丁醇);继而通过代谢工程调控ter和adhe2基因及上游croR基因表达、菌株适应性进化筛选及组学研究,在摇瓶中,正丁醇产量从15.2 mg/L提高到25.5 mg/L[40]。
Sonntag等[41]2015年在扭脱甲基杆菌中异源表达8个基因,利用甲醇从头合成类倍半萜烯-α-蛇麻烯。其中,构建来自黄粘球菌的异源甲羟戊酸(MVA)途径包括羟甲基戊二酰辅酶A合成酶(HMGS)、羟甲基戊二酰辅酶A还原酶(HMGR)、甲羟戊酸激酶(MVAK)、磷酸戊糖酸激酶(MVAK2)、焦磷酸戊糖醛酸脱羧酶(MVAD)和异戊烯焦磷酸异构酶(FNI),还有来自红球姜的α-蛇麻烯合成酶(ZSSI)结合来自酿酒酵母法呢基焦磷酸合成酶(ERG20)的表达,最后α-蛇麻烯基础产量达到18 mg/L。通过优化甲羟戊酸途径中α-蛇麻稀合成酶和FPP合成酶的核糖体结合位点序列,导致蛇麻烯产量增加3倍,进而利用类胡萝卜素合成缺陷突变株,产量又提高30%。在以甲醇为碳源的流加发酵培养条件下,最终产品质量浓度达到1.65 g/L。
2016年,清华大学邢新会课题组的Liang等[43]利用扭脱甲基杆菌EMCP途径的中间代谢物作为前体物,通过异源表达来自德国小蠊(Blattellagermanica)的基因hmgcs1和来自克氏锥虫(Trypanosomacruzi)的基因tchmgr,构建了甲羟戊酸途径。甲羟戊二酰辅酶A合成酶(HMGS)催化乙酰辅酶A和乙酰乙酰辅酶A生成羟甲基戊二酰辅酶A(HMG-CoA),继而在羟甲基戊二酰辅酶A还原酶(HMGR)催化下生成甲羟戊酸,获得基础产量是66 mg/L。在操纵子(MVH)中引入来自于罗尔斯通氏菌(Ralstoniaeutropha)的phaA,进而调控基因phaA表达水平,甲羟戊酸的产量增加到0.18 g/L,调节核糖体结合位点的强度,进一步修饰phaA基因表达水平,使得甲羟戊酸产量额外增加20%,为0.215 g/L,最终流加培养发酵产生的甲羟戊酸为2.22 g/L[42]。此外,他们通过一个辅助生物传感器的转录调控工程(SATRE)控制代谢流量的再分配来增加乙酰辅酶A通量,进一步增加了甲羟戊酸的产量。基于生物传感器转录调节库的高通量筛选,成功地分离了突变株(Q49),最终产品质量浓度达到2.67 g/L。
2017年,Rohde等[44]利用扭脱甲基杆菌聚-3-羟基丁酸(PHB)途径,通过异源表达表达来自塞内加尔芽孢杆菌(Bacillusmassiliosenegalensis) JC6特异性VB12依赖性的突变酶(RCM)催化(R)-3-羟基丁酰辅酶A((R)-3-hydroxybutyryl-CoA)生成2-羟基异丁酸(2-hydroxyisobutyric acid,2-HIBA),在流加培养发酵的条件下,2-HIBA产量可达到2.1 g/L。
2017年,笔者所在课题组的Yang等[45]在扭脱甲基杆菌中利用EMCP途径的乙酰辅酶A为前体物,通过异源表达来自橙色绿屈挠菌(Chloroflexusaurantiacus)基因mcr(该基因编码丙二酰辅酶A还原酶(malonyl-CoA)),转化甲醇合成3-羟基丙酸(3-hydroxypropionic acid,3-HP),并解析3-HP在合成过程中由指数期向稳定期过渡时快速降解的机制,为构建稳定而高效合成3-HP的工程菌株奠定前期基础。
3 构建合成型甲基菌
基于对甲基营养菌生理生化特性以及代谢途径改造的深入研究,人们渴望利用传统的基因工程菌作为底盘构建合成型甲基菌(synthetic m e t h y l o t r o p h s),一方面可以借助目前已经开发的各种合成生物学改造策略,另一方面,传统工程菌的生长和调控机制更加清晰,改造上具有更多优势。
Müller等[47]尝试在大肠杆菌中通过异源表达NAD依赖性的甲醇脱氢酶(MDH)和核酮糖单磷酸途径关键酶,构建甲醇氧化同化途径,构建核酮糖单磷酸循环需要表达的基因是编码3-己酮糖-6-磷酸合成酶的hps和编码6-磷酸-3-己糖异构酶的phi。通过对不同物种候选酶在大肠杆菌的体外和体内酶活进行筛选,发现来源于甲醇芽孢杆菌(Bacillusmethanolicus)的MDH、HPS和PHI在大肠杆菌中的异源表达效果最佳。在大肠杆菌表达这些酶后,其13C甲醇标记实验显示了40% 甲醇被利用转化成中心代谢物。
Witthoff等[48]尝试在谷氨酸棒状杆菌中通过异源表达来自甲醇芽孢杆菌中甲醇脱氢酶和核酮糖单磷酸途径关键酶HPS和PHI来实现改造谷氨酸棒状杆菌利用甲醇。重构底盘的谷氨酸棒杆菌显示在葡萄糖和甲醇混合培养基中,甲醇的平均消耗速率为(1.7±0.3) mmol/(L·h)。此外,13C甲醇标记实验同样证实细胞内多种代谢物,有3%~10%的量被13C标记,而谷氨酸棒状杆菌在ald和adhE双突变体的背景下,减少了甲醛氧化生成CO2的通量,细胞内代谢物13C的标记增加8%~25%。
2017年,Whitaker等[49]在大肠杆菌中异源表达来自嗜热脂肪芽孢杆菌(Bacillusstearothermophilus)的MDH组合和来自甲醇芽孢杆菌(Bacillusmethanolicus)的HPS和PHI,13C甲醇标记实验证实糖酵解途径(embden-meyerhof pathway,EMP)和柠檬酸循环途径(citric acid cycle pathway,TCA)的中间代谢物均被13C标记,并且进一步证明在大肠杆菌中合成黄烷酮柚皮素(flavanonenaringenin)的代谢流部分来源于底物甲醇。
4 结论与展望
甲基营养菌具有十分独特的一碳代谢途径和相关基因及酶系,这不但可拓展微生物代谢途径研究及发现新功能基因,而且同时为甲基营养菌基因工具开发和代谢工程改造、为将甲醇生物催化转生成高附加值产品提供重要理论与实践价值。目前,在扭脱甲基杆菌中构建异源途径,已成功实现将甲醇转生成中康酸、(2S)-甲基琥珀酸、正丁醇、α-蛇麻烯、甲羟戊酸、2-羟基异丁酸(2-HIBA)和3-羟基丙酸(3-HP)等多种高附加值产品,这些开创性研究为甲醇工业高值化的生物转化提供新策略。随着全球页岩气开采和沼气技术发展,甲醇基构建的底盘菌也可发展为甲烷基为中心的绿色催化转化细胞工厂。
目前,合成型甲基细菌还未能实现以甲醇作为唯一的碳源和能源进行生长、代谢与产品合成,究其原因可能是甲基营养菌和传统工程菌无论在代谢酶系、转录因子还是信号分子上都存在较大差异;此外,在传统工程菌中异源表达NAD依赖性的MDH具有可逆催化甲醛还原的特性[50],这会竞争一碳代谢流向下游同化途径。目前的研究证明,传统工程菌作为底盘改造成甲醇利用菌还存在一定的技术瓶颈,有待突破。未来,如何寻找更高效NAD依赖性的MDH,如何从基因组水平调控合成型甲基细菌的代谢网络以强化一碳氧化和同化过程,是获得高效利用一碳的合成型甲基细菌的新策略。
[1] CHISTOSERDOVA L,KALYUZHNAYA M G,LIDSTROM M E.The expanding world of methylotrophic metabolism[J].Annu Rev Microbiol,2009,63:477-499.
[2] KEMP M B,QUAYLE J R.Microbial growth on C1compounds:uptake of14C formaldehyde and14C formate by methane-grownPseudomonasmethanicaand determination of the hexose labelling pattern after brief incubation with14C methanol[J].Biochem J,1967,102(1):94-102.
[3] VUILLEUMIER S,CHISTOSERDOVA L,LEE M C,et al.Methylobacterium genome sequences:a reference blueprint to investigate microbial metabolism of C1 compounds from natural and industrial sources[J].PloS ONE,2009,4(5):e5584.
[4] CHISTOSERDOVA L,CHEN S W,LAPIDUS A,et al.Methylotrophy inMethylobacteriumextorquensAM1 from a genomic point of view[J].J Bacteriol,2003,185(10):2980-2987.
[5] OCHSNER A M,SONNTAG F,BUCHHAUPT M,et al.Methylobacteriumextorquens:methylotrophy and biotechnological applications[J].Appl Microbiol Biotechnol,2015,99(2):517-534.
[6] CLOMBURG J M,CRUMBLEY A M,GONZALEZ R.Industrial biomanufacturing:the future of chemical production[J].Science,2017,355:aag0804.
[7] GAO X,GAO F,LIU D,et al.Engineering the methylerythritol phosphate pathway in cyanobacteria for photosynthetic isoprene production from CO2[J].Energy Environ Sci,2016,9(4):1400-1411.
[8] LIEW F,HENSTRA A M,KPKE M,et al.Metabolic engineering ofClostridiumautoethanogenumfor selective alcohol production[J].Metab Eng,2017,40:104-114.
[9] WHITAKER W B,SANDOVAL N R,BENNETT R K,et al.Synthetic methylotrophy:engineering the production of biofuels and chemicals based on the biology of aerobic methanol utilization[J].Curr Opin Biotechnol,2015,33:165-175.
[10] The Advanced Research Projects Agency-Energy.Reducing emissions using methanotrophic organisms for transportation energy[EB/OL].[2017-08-01].http://arpa-e.energy.gov/?q=arpa-e-programs/remote.
[11] SINTE F.Promyse:products from methanol by synthetic cell factories[EB/OL].[2017-08-01].http://www.sintef.no/Projectweb/PROMYSE.
[12] KELTJENS J T,POL A,REIMANN J,et al.PQQ-dependent methanol dehydrogenases:rare-earth elements make a difference[J].Appl Microbiol Biotechnol,2014,98(14):6163-6183.
[13] CHISTOSERDOVA L.Modularity of methylotrophy,revisited[J].Environ Microbiol,2011,13(10):2603-2622.
[14] ZHANG M,LIDSTROM M E.Promoters and transcripts for genes involved in methanol oxidation inMethylobacteriumextorquensAM1[J].Microbiology,2003,149(4):1033-1040.
[15] ANDERSON D J,LIDSTROM M E.ThemoxFGregion encodes four polypeptides in the methanol-oxidizing bacteriumMethylobacteriumsp. strain AM1[J].J Bacteriol,1988,170(5):2254-2262.
[16] ANTHONY C.Methanol dehydrogenase,a PQQ-containing quinoprotein dehydrogenase[J].Subcell Biochem,2000,35:73-117.
[17] MORRIS C J,KIM Y M,PERKINS K E,et al.Identification and nucleotide sequences ofmxaA,mxaC,mxaK,mxaL,andmxaDgenes fromMethylobacteriumextorquensAM1[J].J Bacteriol,1995,177(23):6825-6831.
[18] SPRINGER A L,AUMAN A J,LIDSTROM M E.Sequence and characterization ofmxaB,a response regulator involved in regulation of methanol oxidation,and ofmxaW,a methanol-regulated gene inMethylobacteriumextorquensAM1[J].FEMS Microbiol Lett,1998,160(1):119-124.
[19] TOYAMA H,CHISTOSERDOVA L,LIDSTROM M E.Sequence analysis ofpqqgenes required for biosynthesis of pyrroloquinoline quinone inMethylobacteriumextorquensAM1 and the purification of a biosynthetic intermediate[J].Microbiology,1997,143(2):595-602.
[20] SPRINGER A L,CHOU H H,FAN W H,et al.Methanol oxidation mutants inMethylobacteriumextorquensAM1:identification of new genetic complementation groups[J].Microbiology,1995,141(11):2985-2993.
[21] SPRINGER A L,MORRIS C J,LIDSTROM M E.Molecular analysis ofmxbDandmxbM,a putative sensor-regulator pair required for oxidation of methanol inMethylobacteriumextorquensAM1[J].Microbiology,1997,143(5):1737-1744.
[22] NAKAGAWA T,MITSUI R,TANI A,et al.A catalytic role of XoxF1 as La3+-dependent methanol dehydrogenase inMethylobacteriumextorquensstrain AM1[J].PloS ONE,2012,7(11):e50480.
[23] SKOVRAN E,MARTINEZ-GOMEZ N C.Microbiology:just add lanthanides[J].Science,2015,348:862-863.
[24] SKOVRAN E,PALMER A D,ROUNTREE A M,et al.XoxF is required for expression of methanol dehydrogenase inMethylobacteriumextorquensAM1[J].J Bacteriol,2011,193(21):6032-6038.
[25] VU H N,SUBUYUJ G A,VIJAYAKUMAR S,et al.Lanthanide-dependent regulation of methanol oxidation systems inMethylobacteriumextorquensAM1 and their contribution to methanol growth[J].J Bacteriol,2016,198(8):1250-1259.
[26] GOOD N M,VU H N,SURIANO C J,et al.Pyrroloquinoline quinone ethanol dehydrogenase inMethylobacteriumextorquensAM1 extends lanthanide-dependent metabolism to multicarbon substrates[J].J Bacteriol,2016,198(22):3109-3118.
[27] VORHOLT J A,MARX C J,LIDSTROM M E,et al.Novel formaldehyde-activating enzyme inMethylobacteriumextorquensAM1 required for growth on methanol[J].J Bacteriol,2000,182(23):6645-6650.
[28] MARTINEZ-GOMEZ N C,NGUYEN S,LIDSTROM M E.Elucidation of the role of the methylene-tetrahydromethanopterin dehydrogenase MtdA in the tetrahydromethanopterin-dependent oxidation pathway inMethylobacteriumextorquensAM1[J].J Bacteriol,2013,195(10):2359-2367.
[29] POMPER B K,VORHOLT J A.Characterization of the formyltransferase fromMethylobacteriumextorquensAM1[J].Eur J Biochem,2001,268(17):4769-4775.
[30] CHISTOSERDOVA L,CROWTHER G J,VORHOLT J A,et al.Identification of a fourth formate dehydrogenase inMethylobacteriumextorquensAM1 and confirmation of the essential role of formate oxidation in methylotrophy[J].J Bacteriol,2007,189(24):9076-9081.
[31] KALYUZHNAYA M G,LIDSTROM M E.QscR-mediated transcriptional activation of serine cycle genes inMethylobacteriumextorquensAM1[J].J Bacteriol,2005,187(21):7511-7517.
[32] ERB T J,BERG I A,BRECHT V,et al.Synthesis of C5-dicarboxylic acids from C2-units involving crotonyl-CoA carboxylase/reductase:the ethylmalonyl-CoA pathway[J].Proc Natl Acad Sci USA,2007,104(25):10631-10636.
[33] PEYRAUD R,KIEFER P,CHRISTEN P,et al.Demonstration of the ethylmalonyl-CoA pathway by using13C metabolomics[J].Proc Natl Acad Sci USA,2009,106(12):4846-4851.
[34] ERB T J,RETEY J,FUCHS G,et al.Ethylmalonyl-CoA mutase fromRhodobactersphaeroidesdefines a new subclade of coenzyme B12-dependent acyl-CoA mutases[J].J Biol Chem,2008,283(47):32283-32293.
[35] CUI J,GOOD N M,HU B,et al.Metabolomics revealed an association of metabolite changes and defective growth inMethylobacteriumextorquensAM1 overexpressingecmduring growth on methanol[J].PloS ONE,2016,11(4):e0154043.
[36] YURIMOTO H,KATO N,SAKAI Y.Genomic organization and biochemistry of the ribulose monophosphate pathway and its application in biotechnology[J].Appl Microbiol Biotechnol,2009,84(3):407-416.
[37] KATO N,YURIMOTO H,THAUER R K.The physiological role of the ribulose monophosphate pathway in bacteria and archaea[J].Biosci Biotechnol Biochem,2006,70(1):10-21.
[38] SONNTAG F,BUCHHAUPT M,SCHRADER J.Thioesterases for ethylmalonyl-CoA pathway derived dicarboxylic acid production inMethylobacteriumextorquensAM1[J].Appl Microbiol Biotechnol,2014,98(10):4533-4544.
[39] HU B,LIDSTROM M E.Metabolic engineering ofMethylobacteriumextorquensAM1 for 1-butanol production[J].Biotechnol Biofuels,2014,7(1):156.
[40] HU B,YANG Y M,BECK D A,et al.Comprehensive molecular characterization ofMethylobacteriumextorquensAM1 adapted for 1-butanol tolerance[J].Biotechnol Biofuels,2016,9:84.
[41] SONNTAG F,KRONER C,LUBUTA P,et al.EngineeringMethylobacteriumextorquensfor de novo synthesis of the sesquiterpenoid alpha-humulene from methanol[J].Metab Eng,2015,32:82-94.
[42] ZHU W L,CUI J Y,CUI L Y,et al.Bioconversion of methanol to value-added mevalonate by engineeredMethylobacteriumextorquensAM1 containing an optimized mevalonate pathway[J].Appl Microbiol Biotechnol,2016,100(5):2171-2182.
[43] LIANG W F,CUI L Y,CUI J Y,et al.Biosensor-assisted transcriptional regulator engineering forMethylobacteriumextorquensAM1 to improve mevalonate synthesis by increasing the acetyl-CoA supply[J].Metab Eng,2017,39:159-168.
[44] ROHDE M T,TISCHER S,HARMS H,et al.Production of 2-hydroxyisobutyric acid from methanol byMethylobacteriumextorquensAM1 expressing (R)-3-hydroxybutyryl coenzyme A-isomerizing enzymes[J].Appl Environ Microbiol,2017,83(3):e02622-16.
[45] YANG Y M,CHEN W J,YANG J,et al.Production and degradation of 3-hydroxypropionic acid in engineeredMethylobacteriumextorquensAM1[J].Microbial Cell Fact,2017,16:179.
[46] SONNTAG F,MULLER J E,KIEFER P,et al.High-level production of ethylmalonyl-CoA pathway-derived dicarboxylic acids byMethylobacteriumextorquensunder cobalt-deficient conditions and by polyhydroxybutyrate negative strains[J].Appl Microbiol Biotechnol,2015,99(8):3407-3419.
[47] MULLER J E,MEYER F,LITSANOV B,et al.EngineeringEscherichiacolifor methanol conversion[J].Metab Eng,2015,28:190-201.
[48] WITTHOFF S,SCHMITZ K,NIEDENFUHR S,et al.Metabolic engineering ofCorynebacteriumglutamicumfor methanol metabolism[J].Appl Environ Microbiol,2015,81(6):2215-2225.
[49] WHITAKER W B,JONES J A,BENNETT R K,et al.Engineering the biological conversion of methanol to specialty chemicals inEscherichiacoli[J].Metab Eng,2017,39:49-59.
[50] PRICE J V,CHEN L,WHITAKER W B,et al.Scaffoldless engineered enzyme assembly for enhanced methanol utilization[J].Proc Natl Acad Sci USA,2016,113(45):12691-12696.
(责任编辑 荀志金)
Advancesinmetabolicnetworkpathwaysandmetabolicengineeringofmethylotrophicbacteria
YANG Jing1,2,CHEN Wenjing1,2,ZHANG Min1,2,YUAN Xiaojie1,2,YANG Yiming1,2,ZHU Liping1,2,3,YANG Song1,2,3
(1.School of Life Sciences,Qingdao Agricultural University,Qingdao 266109,China; 2.Shandong Province Key Laboratory of Applied Mycology,Qingdao Agricultural University,Qingdao 266109,China; 3.Qingdao International Center on Microbes Utilizing Biogas,Qingdao Agricultural University,Qingdao 266109,China)
Efficient conversion of a carbon compound (methanol or methane) is of important research interest in the field of bioengineering.Methylotrophs are gram negative organisms using C1 compounds such as methane and methanol as the sole carbon and energy source.With the development of genome sequencing and various omics technologies,the central metabolic network pathway and related functional genes of methylotrophs,represented byMethylobacteriumextorquens,are clearly shown.More recently,the improvement of genetic manipulation tools such as gene overexpression and gene knockout,and various gene regulatory elements,provide important basis for methylotrophic chassis modification and optimization of heterologous expression.Researches promoted the conversion of methanol into a variety of value-added products.In addition,people are eager to use the traditional genetic engineering bacteria as a chassis to build synthetic methylotrophs,in order to achieve more efficient methanol catalytic conversion.In this paper,the characteristics of metabolic network,metabolic engineering strategyand the construction of synthetic methylotrophs are summarized.
methylotrophs;metabolic engineering;metabolic network pathways;synthetic m e t h y l o t r o phs;methanol
10.3969/j.issn.1672-3678.2017.06.002
2017-08-08
国家自然科学基金联合基金重点培育项目(U1462109);山东省重点研发计划(2016GSF121010);山东省自然科学基金面上项目(ZR2013CM024)
杨 靖(1991—),女,山东枣庄人,研究方向:甲基微生物代谢网络调控与代谢工程改造;杨 松(联系人),教授,E-mail:yangsong1209@163.com
Q78;Q819
A
1672-3678(2017)06-0009-08
