5种鸟类FoxP2的分子进化分析
2017-10-16王青青陆文康丁恒武刘必融阚显照
王青青, 陆文康, 王 萍, 蒋 澜, 吴 璇, 王 莹, 章 勤, 丁恒武, 刘必融, 阚显照
(安徽师范大学 生命科学学院 生物信息研究所, 芜湖 241000)
5种鸟类FoxP2的分子进化分析
王青青, 陆文康, 王 萍, 蒋 澜, 吴 璇, 王 莹, 章 勤, 丁恒武, 刘必融, 阚显照
(安徽师范大学 生命科学学院 生物信息研究所, 芜湖 241000)
FoxP2是第一个被发现的和人类言语相关的基因。该基因在鸟类中也存在,在发育、成长、鸣曲学习等方面起着关键性作用。研究从NCBI和Ensembl中提取5种鸟类及相近物种FoxP2的序列,探讨FoxP2的序列结构特点和进化速率。以人和小鼠为外类群,对5种鸟类和两种爬行类进行系统发育树的构建。采用最大简约法、最大似然法和贝叶斯法进行分析,结果表明5种鸟类构成很强的单系群。结果将为鸟类FoxP亚家族的进化研究提供有用的信息。
FoxP2; 序列特征; 进化速率; 系统发育
AbstractFoxP2 is the first gene involved in human speech. This gene also exists in birds and plays pivotal roles in development, adulthood, and avian vocal learning. The sequences ofFoxP2 in five avian species and their related species were retrieved from GenBank and Ensembl. The sequence characterization, evolutionary rate ofFoxP2 were analyzed. The phylogenetic analyses of five avian species and two reptile species, with two mammalian species as outgroup, were conducted. Using maximum parsimony (MP),maximum likelihood (ML), and Bayesian inference (BI) methods based on the nucleotide sequence ofFoxP2, the monophyly of five avian species was confirmed. Our results may provide useful information about the evolution of FoxP subfamily in birds.
KeywordsFoxP2; sequence characterization; evolutionary rate; phylogeny
FOX家族(Forkhead box family,叉头框家族)是一类从低等真核生物、爬行类、鸟类到哺乳类都广泛存在的转录因子,属于“螺旋-转角-螺旋”类蛋白的一个亚群。大量研究表明FOX蛋白在胚胎发育、细胞凋亡、细胞免疫、细胞代谢、细胞衰老等生物学过程中发挥作用[1-6]。FoxP2是人类发现的第一个言语相关基因,位于第7号染色体长臂的31位点,属于FOX基因家族、FoxP亚家族。近年来,人们对于FoxP2对鸣禽神经回路发育和出生后的鸣唱学习进行了一系列研究[7-11]。然而,关于FoxP2在鸟类中的分子进化研究较少。本文选取了基因组注释较完善的5种鸟类[12-16]作为研究对象,对其基因组结构与特征、基因进化速率、系统发育关系进行分析,为鸟类FoxP亚家族的进一步研究提供有用的信息。
1 材料与方法
1.1 数据来源
FoxP2的序列来源于公共基因数据库NCBI(www.ncbi.nlm.nih.gov)和国际基因组数据库Ensembl (www.ensembl.org),见表1。本研究选取了基因组注释较完善的5种鸟类作为研究对象,另选取两种爬行类和两种哺乳类进行FoxP2的比较研究。物种拉丁名参照整合分类信息系统ITIS(Integrated Taxonomic Information System, www.itis.gov)。
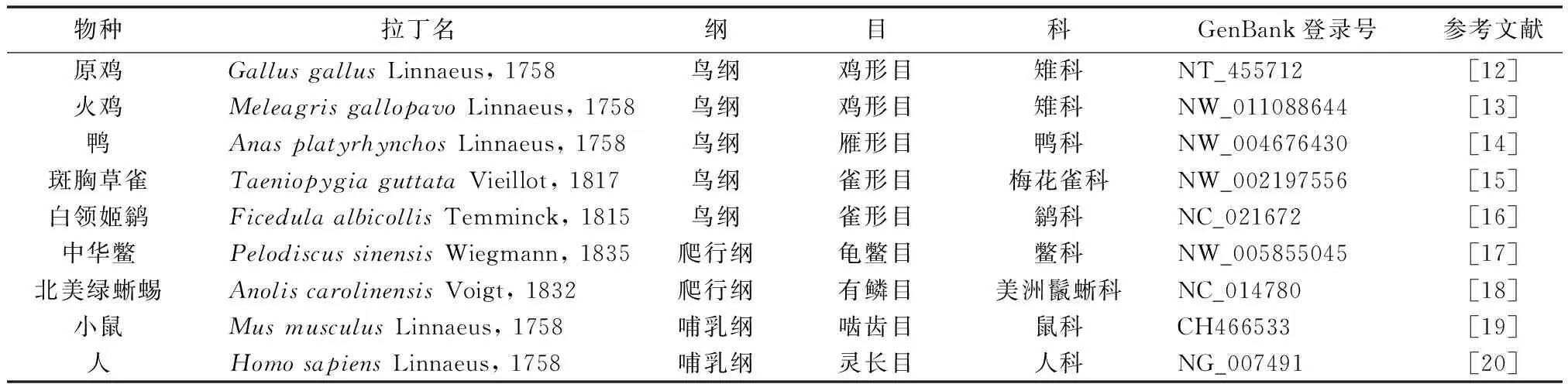
表1 本研究物种的来源及FoxP2的GenBank登录号
1.2 分析方法
1.2.1 基因的结构与特征分析
利用Genscan在线分析平台(http://genes.mit.edu/GENSCAN.html)对从GenBank或Ensembl数据库获取的FoxP2序列进行外显子和内含子分区。以家鸡FoxP2的外显子和内含子序号为参照进行基因区域的编号。使用BioEdit 7.1.3统计基因的碱基组成;碱基偏选使用的公式为:AT skew = (A-T)/(A+T), GC skew=(G-C)/(G+C)。
1.2.2 基因序列的比对及碱基替代模型的分析
通过MAFFT 7 在线分析平台(mafft.cbrc.jp/alignment/software/)对核苷酸和氨基酸进行序列比对。使用ModelGenerator V. 0.851软件对核苷酸替代最适模型进行分析,通过Bayesian Information Criterion(BIC)统计后估算。
1.2.3 基因进化速率分析
本研究中,由于绝大多数基因区域为内含子,所以采用核苷酸多样性指数(π值)来探讨基因进化速率的快慢。π值表示两条核苷酸序列之间每位点发生变异的平均数。我们构建了两组数据用以分析:1)选取5种鸟类作为材料,探讨FoxP2在鸟类内部的核苷酸多样性;2)选取5种鸟类和两种爬行类动物作为材料,探讨FoxP2在鸟类、爬行类组合的核苷酸多样性。编码区及17种内含子数据作为研究对象,采用DnaSP v5.10.01软件。对于内含子13,设置Sliding Window长度为100 sites,Step size为25 sites;对于其他16个内含子及编码区,Sliding Window长度为300 sites。
1.2.4 系统发育树的构建
最大简约法 (MP) 分析,使用PAUP*4.0b10软件。采用启发式 (heuristic) 搜索最大简约树 (MP),序列添加方式选用100次的随机分类群重复,树等分与重连分支交换法 (TBR)获取系统树。自展法(bootstrap)重复检验1000次,用以分析系统发育树拓扑结构的可靠性。
最大似然法(ML)分析,使用RaxML GUI v.1.3.1软件。核苷酸替代模型采用GTRCAT,ML + slow bootstrap,run 10次,通过1000次重复的自展法评估ML树上分支的可靠性。
贝叶斯法(BI)分析,使用MrBayes 3.2.2软件。对不同的基因区域,由于进化速率不同,采取分段计算。核苷酸替代最适模型采用ModelGenerator V. 0.851分析的结果。4条马尔科夫链 (Markov Chain Monte Carlo, MCMC) 共运行10 000 000代,每100代抽样一次。25%的树被摒弃 (burnin), 剩下的75%的树用来推测支持率大于50%的一致树和后验概率 (Posterior probability, PP)。所有的马尔科夫链运算都重复2次,以确保得到可靠的后验概率。
2 结果与分析
2.1 5种鸟类FoxP2的结构与特征
5种鸟类FoxP2全长的长度从338 773 bp (火鸡) 到 414 549 bp (白领姬鹟),见表2。17个内含子中,内含子1~4和内含子17的序列较长,均在50 kb以上。从A+T百分含量看,编码区明显低于内含子。以家鸡为例,编码区AT%为52.1,17个内含子AT%范围为56.0(内含子6)到 69.8(内含子13)。
我们对本研究9个物种FoxP2编码区的核苷酸和其翻译的氨基酸序列进行了比对,分别得到2229 bp(核苷酸)和795Aa(氨基酸)的序列矩阵。表3为本研究9个物种FoxP2编码区氨基酸序列突变位点(不包括氨基酸的插入和缺失)。从表3可以看出,序列矩阵的383位的缬氨酸(V)为本研究中鸟类特有的氨基酸,42位的酪氨酸(T)、262位的天冬酰胺(N)、276位的丝氨酸(S)为本研究中鸟类、爬行类所共有的氨基酸。
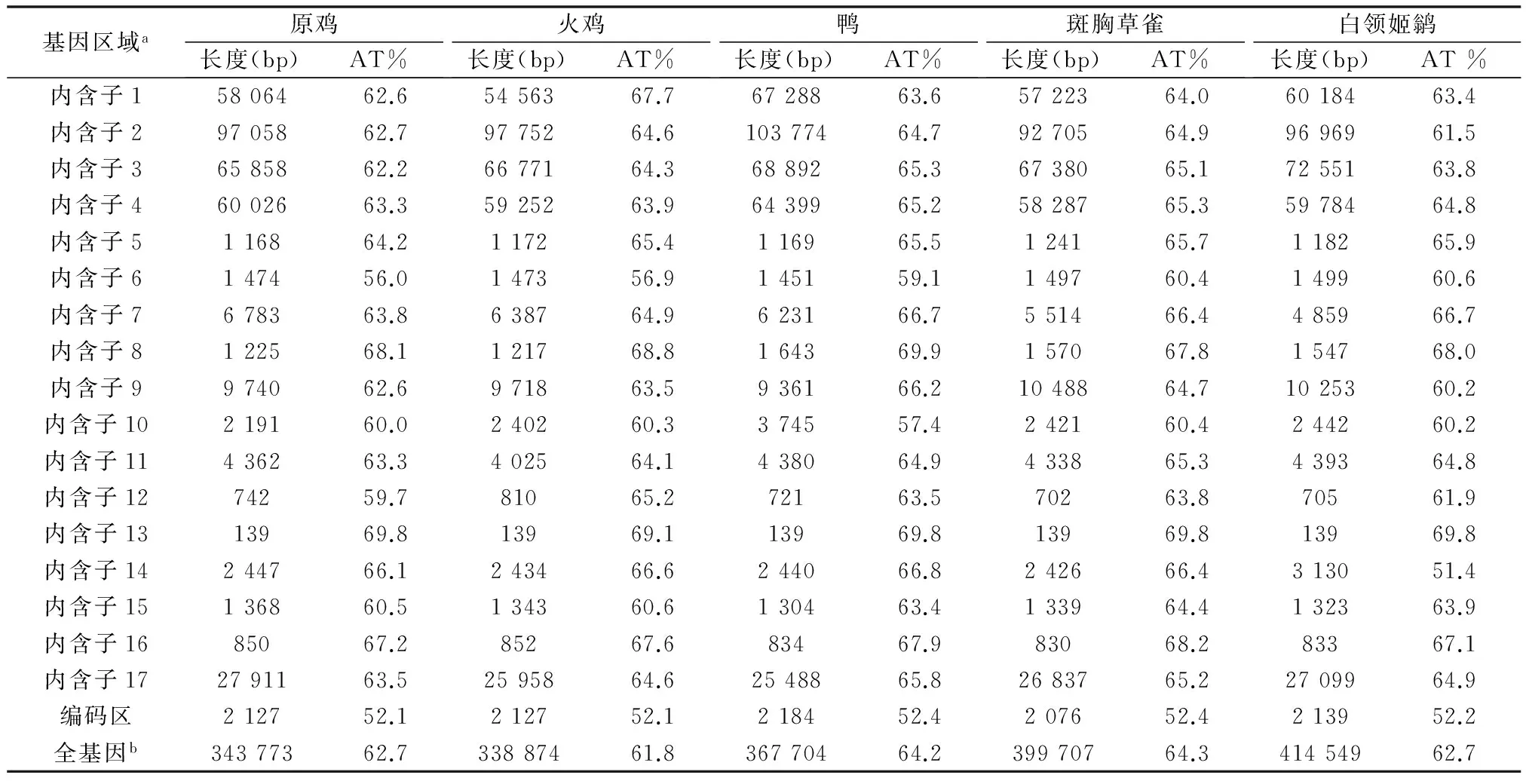
表2 5种鸟类FoxP2的分区、长度及AT含量
A:鸟类FoxP2区域的划分是以原鸡的外显子和内含子为参照,选择与其相对应的序列同源区域;编码区的划分是以ensembl数据库相对应的CDS数据为准。b:全基因包括所有内含子、编码区、5′非翻译区和3′非翻译区
表3 9物种FoxP2编码区氨基酸序列突变位点
Table 3 The mutation sites of amino acid sequences ofFoxP2 CDS in nine species

从碱基偏选看,所有的内含子和编码区显示明显的正AT skew值(图1)。除白领姬鹟内含子14外,其余的内含子和编码区均显示明显的负GC skew值(图1)。白领姬鹟内含子14共有3130 bp,从1399 bp位置开始,有696个G重复碱基,这是造成其GC skew正值的原因。
2.2 FoxP2的进化速率分析
在5种鸟类内部,内含子13的π值最低,为0.015 83;编码区的π值次之,为0.019 74;内含子12最高,为0.237 3(图2)。内含子13的长度在5种鸟类中高度保守,非常短,均为139 bp,且核苷酸多样性低。说明在5种鸟类中,内含子13最为保守,内含子12变异最大。

图1 5种鸟类FoxP2的碱基偏选
在鸟类和爬行类组合中,编码区的π值最低,为0.035 54,内含子14次之(0.056 94)。内含子13的π值为倒数第3(0.118 34)。内含子1变异最大,π值为0.383 5;内含子12次之(0.358 42)。
2.3 系统发育分析
经过Mafft软件序列比对,我们得到了用于系统发育分析的数据矩阵:1)CDS序列长度为2229 bp;2)内含子9序列长度为1505 bp;3)内含子11序列长度为4893 bp;4)内含子14为2321 bp;5)CDS和内含子整合序列组长度为10 948 bp。用Bayesian Information Criterion(BIC)法确定Foxp2的CDS序列和内含子9的最佳模型为HKY+G,内含子11为TVM+G,内含子14为TrN+G。系统发育树图的枝长和拓扑结构来自最大似然图。人和小鼠作为外类群,其余物种作为内类群。节点上的数字按顺序分别表示MP、ML的bootstrap支持率及BI法的后验概率。用MP、ML和BI 3种方法对5种数据组进行分析,均得到相同或相似的拓扑结构图,不同之处是节点的支持率(或后验概率)及枝长(图3~7)。在所有的系统发育分析中,5种鸟类均构成很强的单系群。

图2 5种鸟类FoxP2的核苷酸多样性
图中黑色实心框表示FoxP2在5种鸟类中的核苷酸多样性π值,红色实心框表示其在鸟类和爬行类组合(共7个物种)中核苷酸多样性π值在3个内含子分别构建的系统发育树图中(图4~6),中华鳖和5种鸟类构成姐妹群的拓扑结构相同,但支持率和后验概率不同。对于内含子9,MP和ML的bootstrap值分别为54和53,BI后验概率为0.78;对于内含子11,相对应的MP、ML、BI的值分别为100、100和0.98;对于内含子14,与之对应的值分别为96、76和0.97。其他系统发育关系,3个内含子用3种方法得出的结果非常相似。

图3 基于FoxP2编码序列构建的系统发育树
对于CDS和内含子整合序列组得到的树图,所有节点的支持率和后验概率都非常高,MP、ML和BI的值分别为100、100和1.00(图7)。
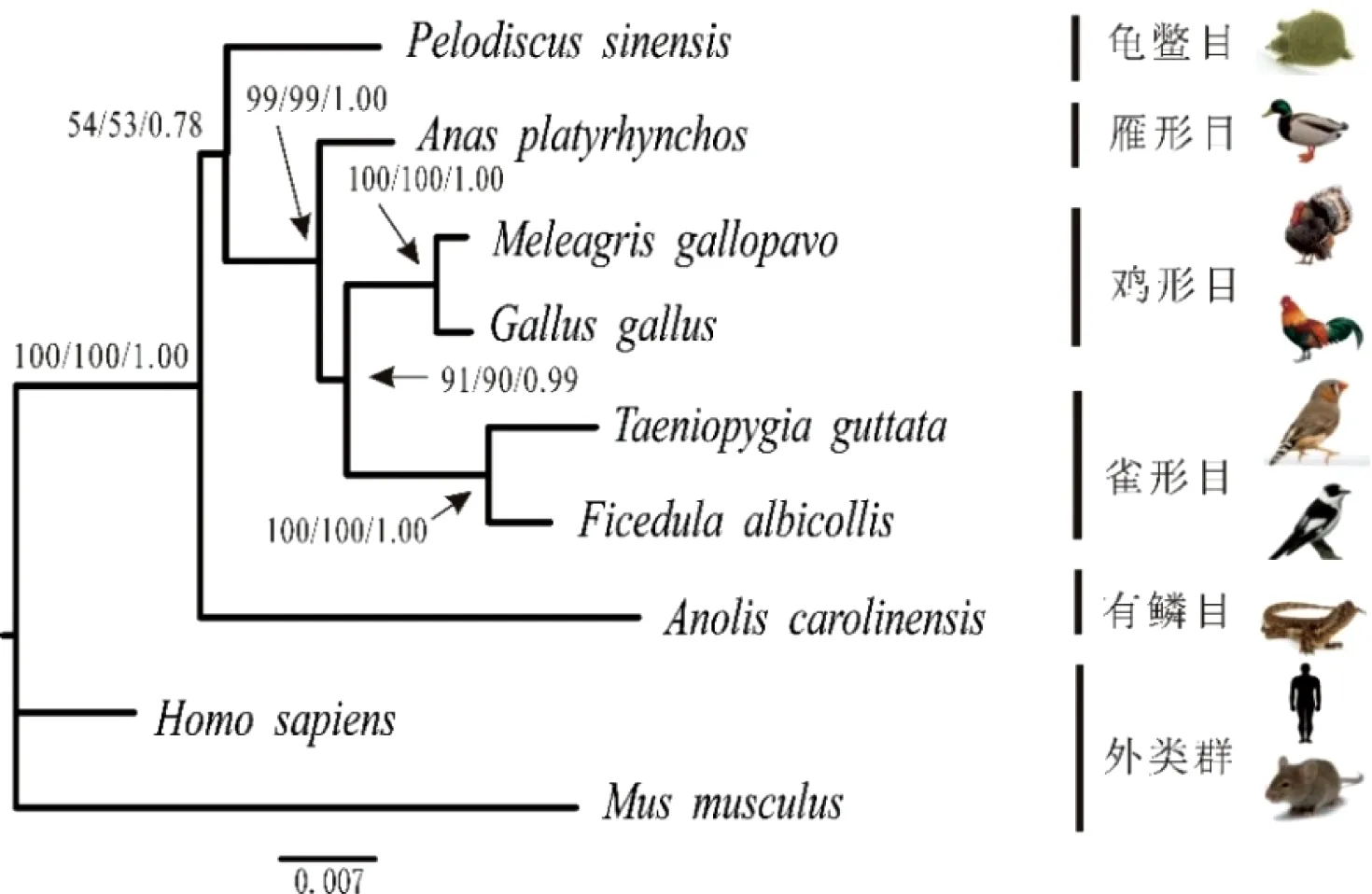
图4 基于FoxP2的内含子9序列构建的系统发育树
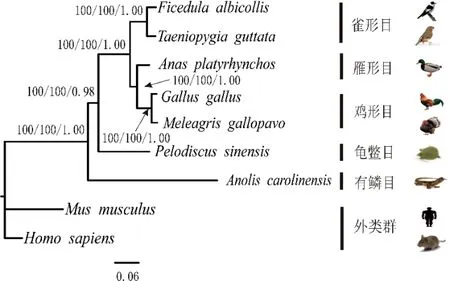
图5 基于FoxP2的内含子11序列构建的系统发育树
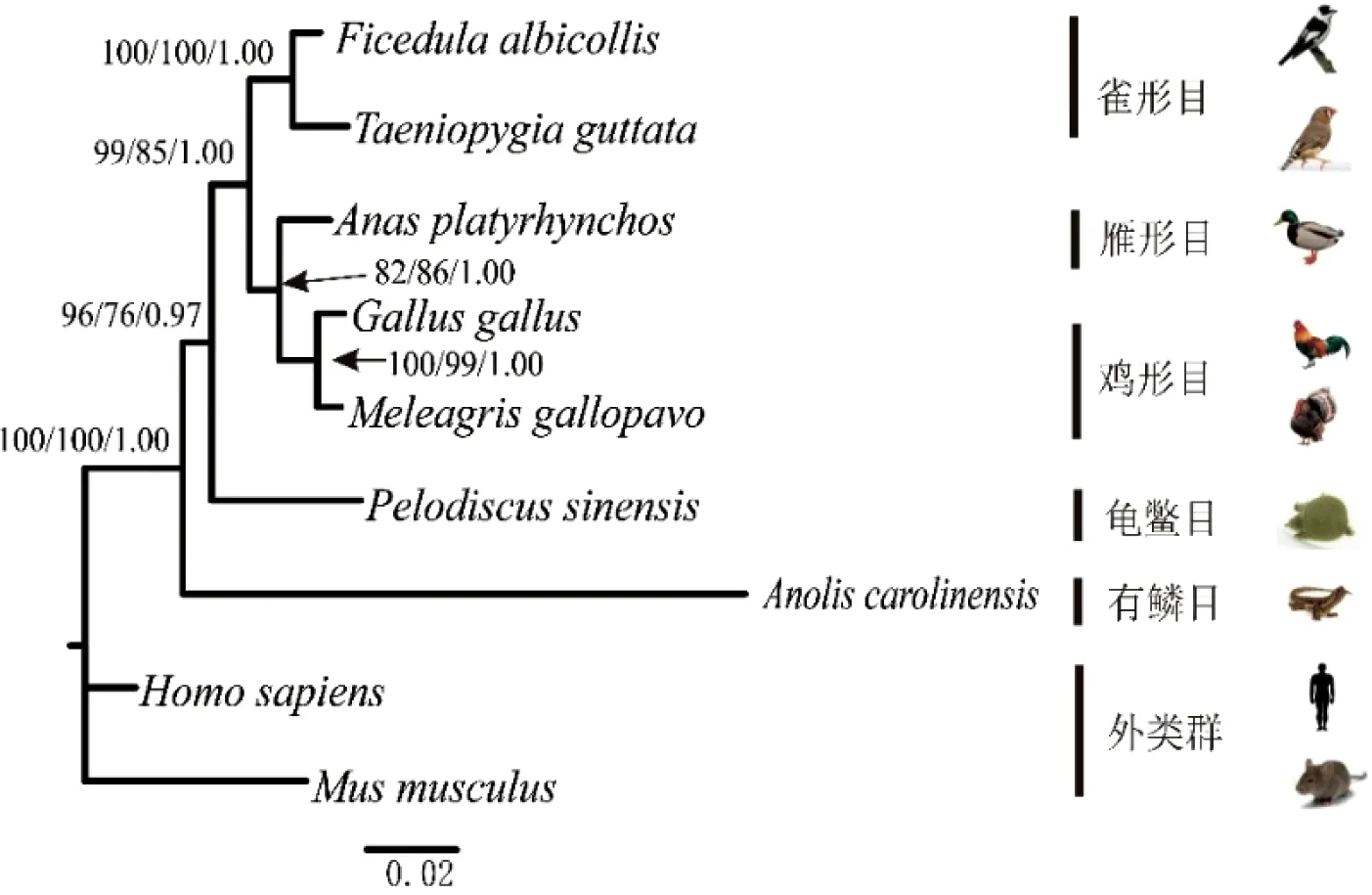
图6 基于FoxP2的内含子14序列构建的系统发育树
3 讨论
原鸡[12]和火鸡[13]的编码区核苷酸序列有14个碱基替代,但氨基酸序列完全相同。鸭[14]和原鸡的编码核苷酸序列相比,有一个54 bp 的碱基插入,此外还有25个位点发生碱基替代,不过,这25个位点的突变均是同义替代。和哺乳类、爬行类编码区氨基酸序列相比,383位的异亮氨酸突变为缬氨酸(V),5种鸟类共享这个突变位点。和人相比,Ser79Thr突变为原鸡、火鸡、鸭共有,这三者均属于鸡雁小纲;Asp80Glu突变为斑胸草雀[15]和白领姬鹟[16]共有,这二者属于鸟类最大的目——雀形目。在本研究的5种鸟类中,鸡雁小纲和雀形目为姐妹群关系。

图7 基于FoxP2的编码区和内含子9、11、14序列构建的系统发育树
此外,内含子在不同鸟类之间的序列变异较大。例如,火鸡内含子1长度为54 563 bp,鸭内含子1长度比火鸡约长12.5 kb。在所有内含子中,内含子17的长度最长,范围从25 488 bp(鸭)到27 911 bp(原鸡)。内含子13长度最小,5种鸟类均为139 bp。
在编码区序列的系统发育分析中,有鳞目的北美绿蜥蜴[18]位于本研究内类群中的基部位置,中华鳖[17]和5种鸟类构成姐妹群,但3种分析方法的支持率均很低(MP和ML的bootstrap值分别为49和44,BI后验概率为0.55),见图3。5种鸟类聚成一支,获得了较高的支持率(MP和ML的bootstrap值分别为97和99,BI后验概率为1.00)。火鸡和原鸡聚成一支,和鸭构成姐妹群,这样鸡形目和雁形目共同组成鸡雁小纲,获得较高的支持率(MP和ML的bootstrap值分别为96和97,BI后验概率为1.00)。此外,斑胸草雀和白领姬鹟互为姐妹群,聚成一支,见图3。对于CDS和内含子整合序列组得到的树图,能够很好地反映物种进化的历史,这说明FoxP2具有较强的系统发育意义,可以作为鸟类进化研究理想的分子标记。
[1]MORRIS G, FANUCCHI S. A key evolutionary mutation enhances DNA binding of the FOXP2 forkhead domain[J]. Biochemistry, 2016, 55(13): 1959-1967.
[2]MOZZI A, FORNI D, CLERICI M, et al. The evolutionary history of genes involved in spoken and written language: beyond FOXP2 [J]. Scientific reports, 2016, 6: 22157.
[3]ESTRUCH S B, GRAHAM S A, DERIZIOTIS P, et al. The language-related transcription factor FOXP2 is post-translationally modified with small ubiquitin-like modifiers [J]. Scientific Reports, 2016, 6: 20911.
[4]CESARIO J M, ALMAIDHAN A A, JEONG J. Expression of forkhead box transcription factor genesFoxp1 andFoxp2 during jaw development [J]. Gene Expr Patterns, 2016, 20(2): 111-119.
[5]CASTELLUCCI G A, MCGINLEY M J, MCCORMICK D A. Knockout of Foxp2 disrupts vocal development in mice [J]. Scientific Reports, 2016, 6: 23305.
[6]SIN C, LI H, CRAWFORD D A. Transcriptional regulation byFOXP1,FOXP2, andFOXP4 dimerization [J]. J Mol Neurosci, 2015, 55 (2): 437-448.
[7]MENDOZA E, TOKAREV K, DURING D N, et al. Differential coexpression ofFoxP1,FoxP2, andFoxP4 in the Zebra Finch (Taeniopygiaguttata) song system [J]. J Comp Neurol, 2015, 523(9):1318-1340.
[8]HESTON J B, WHITE S A. Behavior-linkedFoxP2 regulation enables zebra finch vocal learning [J]. J Neurosci, 2015, 35(7): 2885-2894.
[9]HARA E, PEREZ J M, WHITNEY O, et al. NeuralFoxP2 andFoxP1 expression in the budgerigar, an avian species with adult vocal learning [J]. Behav Brain Res, 2015, 283: 22-29.
[10]WOHLGEMUTH S, ADAM I, SCHARFF C. FoxP2 in songbirds [J]. Curr Opin Neurobiol, 2014, 28: 86-93.
[11]WHITE S A, FISHER S E, GESCHWIND D H et al. Singing mice, songbirds, and more: models for FOXP2 function and dysfunction in human speech and language [J]. J Neurosci, 2006, 26(41): 10376-10379.
[12]HILLIER L D W, MILLER W, BIRNEY E, et al. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution [J]. Nature, 2004, 432(7018): 695-716.
[13]DALLOUL R A, LONG J A, ZIMIN A V, et al. Multi-platform next-generation sequencing of the domestic turkey (Meleagrisgallopavo): genome assembly and analysis [J]. PLoS Biol, 2010, 8(9): 1000475.
[14]HUANG Y, LI Y, BURT DW, et al. The duck genome and transcriptome provide insight into an avian influenza virus reservoir species [J]. Nature Genetics, 2013,45(7): 776-783.
[15]WARREN W C, CLAYTON D F, ELLEGREN H, et al. The genome of a songbird [J]. Nature, 2010, 464(7289): 757-762.
[16]ELLEGREN H, SMEDS L, BURRI R, et al. The genomic landscape of species divergence in Ficedula flycatchers [J]. Nature, 2012, 491(7426): 756-760.
[17]WANG Z, PASCUAL-ANAYA J, ZADISSA A, et al. The draft genomes of soft-shell turtle and green sea turtle yield insights into the development and evolution of the turtle-specific body plan [J]. Nature Genetics, 2013, 45(6): 701-706
[18]ALFOLDI J, DI PALMA F, GRABHERR M, et al. The genome of the green anole lizard and a comparative analysis with birds and mammals [J]. Nature, 2011, 477(7366): 587-591.
[19]MURAL R J, ADAMS MD, MYERS E W, et al. A comparison of whole-genome shotgun-derived mouse chromosome 16 and the human genome [J]. Science, 2002,296(5573):1661-1671
[20]STROUD J C, WU Y, BATES D L, et al. Structure of the forkhead domain of FOXP2 bound to DNA [J]. Structure. 2006, 14(1): 159-166.
Molecular evolution ofFoxP2infiveavianspecies
WANGQing-qing,LUWen-kang,WANGPing,JIANGLan,WUXuan,WANGYing,ZHANGQing,DINGHeng-wu,LIUBi-rong,KANXian-zhao
(The Institute of Bioinformatics, College of Life Sciences, Anhui Normal University, Wuhu 241000, China)
Q951.3;Q959.7
A
2095-1736(2017)05-0029-05
2016-08- 11;
2016-08-20
安徽高校自然科学研究重大项目(KJ2016SD22);安徽省高等教育振兴计划重大教学改革项目(2015zdjy035);安徽师范大学研究生教育教学改革研究重点项目(2015yjg017zd)
王青青,硕士研究生,研究方向为鸟类基因组学,E-mail:wangqingqing@ahnu.edu.cn
阚显照,教授,博士生导师,研究方向为动物分子系统与进化,E-mail:xianzhao@ahnu.edu.cn
doi∶10.3969/j.issn.2095-1736.2017.05.029
