烟酰胺腺嘌呤二核苷酸(NAD)合成途径和水稻叶片早衰
2017-09-23陈平吴立文王忠伟张宇郭龙彪
陈平 吴立文 王忠伟 张宇,2 郭龙彪,*
(1中国水稻研究所, 杭州 310006;2广西瑞特种子有限责任公司, 南宁 530003;*通讯联系人, E-mail: guolongbiao@caas.cn)
烟酰胺腺嘌呤二核苷酸(NAD)合成途径和水稻叶片早衰
陈平1吴立文1王忠伟1张宇1,2郭龙彪1,*
(1中国水稻研究所, 杭州 310006;2广西瑞特种子有限责任公司, 南宁 530003;*通讯联系人, E-mail: guolongbiao@caas.cn)
叶片是植物进行光合作用的主要场所,其衰老由内源遗传发育信号和外界环境胁迫所启动,是一个非常复杂有序的调控过程。烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NAD)是脱氢酶的辅酶,在糖酵解、糖异生、三羧酸循环以及呼吸链等代谢中发挥着不可替代的作用。最新研究表明,水稻NAD生物合成参与调控沉默信息调控因子Sirtuins的生物活性、组蛋白H3K9去乙酰化、植物激素茉莉酸(JA)和叶片衰老。本文综述了有关水稻叶片衰老的细胞生理特征、Sirtuins酶活、NAD生物合成以及水稻早衰的OsSRT1-NAD调控途径和OsSRT1-MeOH-JA调控途径,以期阐明水稻叶片衰老的分子机理及其调控途径,为高产育种提供相应的理论参考。
水稻;叶片衰老;NAD合成途径;茉莉酸;正沉默信息调控因子
植物自然衰老是伴随着年龄增长而发生的细胞程序化死亡过程[1-3],叶片衰老是最常见的现象之一,它受内外因素的共同调控。内部因素主要包括植物生长发育过程中遗传因子和内源激素的改变。目前,已报道的水稻叶片衰老相关基因超过132个[4],其中与叶片持绿、早衰相关基因40余个,图位克隆的叶片早衰功能基因21个。这些基因主要参与调控叶绿体发育与叶绿素降解、蛋白质合成与降解及转运、细胞程序性死亡以及激素调控途径。内源激素如细胞分裂素、生长素、赤霉素有延迟叶片衰老作用,而乙烯、脱落酸、茉莉酸及其衍生物、水杨酸、油菜素内酯促进叶片衰老,这些内源激素之间协同拮抗,共同调控了植物叶片的衰老[5]。外部因素包括各种外界因子、机械损伤以及病原菌的侵害等非生物和生物所造成的胁迫[6-8]。在植物衰老过程中,细胞结构、激素水平、生理生化代谢以及基因表达等都发生变化,过程极其复杂。早期的营养胁迫假说、激素平衡假说以及程序性细胞死亡理论等在一定程度上对衰老机理作出了较为合理的解释[9]。
最新研究发现,烟酰胺腺嘌呤二核苷酸NAD(nicotinamide adenine dinucleotide)的生物合成参与调控水稻叶片衰老[10-11]。NAD及其相应的还原型态(NADH),即我们通常所说的辅酶Ⅰ,是生物体内细胞能量代谢所必需的小分子化合物。NAD+/NADH是电子载体,参与糖酵解、糖异生、三羧酸循环以及细胞呼吸链中各种酶促氧化还原反应[12-14]。NAD(P)(H)的生物合成途径,包括起始合成途径(the de novo pathway)和补救途径(the salvage pathway)[15,16],影响生物的信号转导、发育调控和抗逆性。NAD+/NADH比例(NAD水平)直接控制着细胞的节律、衰老、抗性和细胞保护作用等重大生命过程。细胞内联系代谢与衰老过程的酶是依赖NAD的去乙酰化酶Sirtuins(又称正沉默信息调控因子)。其去乙酰化酶活性对NAD水平的依赖,能够传递到细胞内影响代谢速率,从而延长细胞寿命。在水稻中,Wu等[11]最先发现NAD生物合成基因(OsNaPRT1)参与调控了NAD合成途径、Sirtuins酶活、组蛋白H3K9去乙酰化表观遗传和叶片早衰。最近,Fang等[10]揭示了水稻茉莉酸(jasmonic acid,JA)含量的控制基因OsPME1,参与调控Sirtuins酶活、去乙酰化,负调控水稻叶片衰老。
本文综述了有关植物叶片衰老细胞的生理特征、NAD生物合成、Sirtuins酶活以及水稻早衰的OsSRT1-NAD和OsSRT1-MeOH-JA调控途径的最新研究进展,以期阐明植物叶片衰老的分子机理及其调控途径,同时为水稻高产育种提供相应的参考。
1 叶片衰老的发育过程和特征
植物从生根发芽开始,在其生长发育的各个阶段中均伴随着各种器官的衰老和死亡。叶片从形成到发育成熟,直至衰老死亡,是一个不可避免的自然现象[7]。叶片衰老表现出有序的细胞结构变化和生理生化方面的变化(图1),主要表现为:叶绿素的降解、叶绿体结构的改变与解体、液泡的解体、细胞质膜和液泡膜的解体、核酸和蛋白含量的降低与降解、营养物质的转移等,最终导致叶肉细胞的死亡和叶片功能的丧失[6-7,17-18]。发育引起的叶片自然衰老是一个渐进有序而又极其复杂的调控过程,涉及一系列内外因素的协同响应,而活性氧ROS或糖被认为是衰老的信号因子[19]。
细胞结构变化:叶片衰老过程中细胞结构变化最初发生在叶绿体上,由于叶绿素的降解快于合成,叶片在形态上表现为黄化。叶片的变化表现为气孔导度降低、蒸腾速率下降、光合速率降低、叶片的运输能力下降、叶片中淀粉积累、叶片对光能的利用率下降[9,20]。而与基因表达相关的线粒体和细胞核结构会一直保持完整,直到衰老。叶片衰老至最后阶段时,液泡会崩裂,染色质会浓缩,原生质膜的完整性缺失导致细胞内平衡破坏,最终细胞死亡。
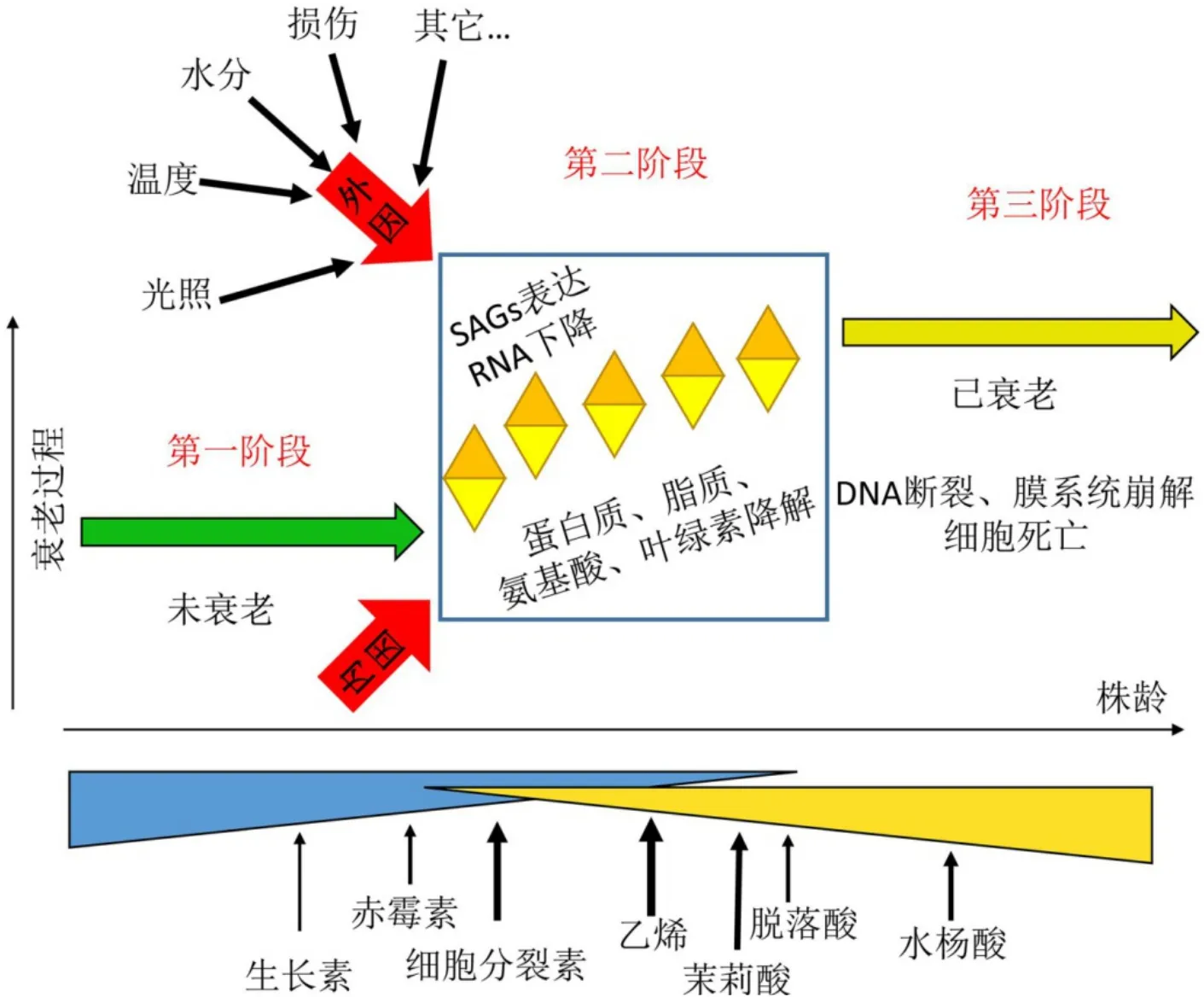
图1 植物叶片衰老过程Fig. 1. Leaf senescence process in plant.
生理生化典型特征:叶片衰老的生理生化典型特征表现为核酸含量降低,蛋白质含量下降,膜脂过氧化程度加重和细胞保护酶活性下降,光合作用功能衰退、细胞内部激素平衡破坏。RNA不断降解,但DNA含量和脱氧核糖核酸酶(DNase)活性无明显变化。所有细胞中多核糖体和核糖体的含量降低,rRNA和tRNA的合成也降低,内肽酶和氨肽酶活性明显上升。叶片衰老时,细胞内自由基产生及清除的动态平衡被破坏,自由基不断积累,从而加剧细胞膜脂过氧化。部分小分子抗氧化物及保护酶系统的数量和活性明显降低,如超氧化物歧化酶、抗坏血酸过氧化物酶、过氧化氢酶和谷胱甘肽过氧化物酶含量以及抗坏血酸、谷胱甘肽、甘露醇和类黄酮含量减少,作为细胞膜脂氧化产物的丙二醛(MDA)含量增加[6]。呼吸速率在叶片衰老初始阶段快速下降,然后急剧上升,随之迅速下降。
2 NAD生物合成途径
NAD和NADP是细胞代谢途径中多种酶的辅酶,参与细胞很多代谢反应。NADH或更准确地说NADH+H+是NAD的还原形式。NAD是脱氢酶的辅酶,如乙醇脱氢酶,用于氧化乙醇。中间产物会将脱下的氢传递给NAD,使其变为还原态NADH+H+。NADP为烟酰胺腺嘌呤二核苷酸磷酸(简称:辅酶Ⅱ),是另一种极为重要的核苷酸类辅酶,它是NAD中与腺嘌呤相连的核糖环系2′-位的磷酸化衍生物,参与多种合成代谢反应,如脂类、脂肪酸和核苷酸的合成。这些反应中需要NADPH作为氢供体,NADPH是NADP+的还原形式。植物叶绿体中,光合作用光反应电子传递链的最后一步即是以NADP+为原料,经过铁氧还蛋白-NADP+还原酶的催化而产生NADPH。产生的NADPH接下来在碳反应(暗反应)中被用于二氧化碳的同化。因此,NAD与NADP实际上是通过氧化和还原型的转换来为生物提供能量,从而实现其辅酶功能。
NAD起始合成途径和补偿途径:鉴于NAD(P)能够通过初级和次级代谢精确调控细胞内的动态平衡,所有组织合成NAD(P)都需要经过一个以色氨酸或者天冬氨酸为起始物的起始合成过程,植物以天冬氨酸为起始物[15]。先由其氧化酶和喹啉酸合成酶将天冬氨酸和二羟基丙酮催化生成喹啉酸,而后喹啉酸在喹啉酸磷酸核糖转移酶的催化作用下生成NaMN(烟酸单核苷酸),NaMN在NMNAT(烟酰胺单核苷酸腺苷酰基转移酶)的作用下生成NaAD(烟酸腺嘌呤二核苷酸),NaAD在NAD合成酶的催化下最终生成NAD(图2)。
除了氧化还原反应,NAD还是ADP-核糖、多聚ADP核糖基化、蛋白质脱乙酰作用等反应的底物,在这些反应中,NAD被分解得到烟酰胺(NIM),被称为NAD补偿途径或再循环途径。NAD和NADP是植物机体中的电子传输和代谢进程所需的基本辅助因子,能够调控代谢酶类的相关功能[22]。NAD用于呼吸作用中ATP的产生,而NADP则应用于还原性的生物合成反应中。在逆境下,胞质中会通过磷酸戊糖途径生成NADP的还原型(NADPH),参与调控钙调蛋白的NADK的活性和ROS活性氧含量。
3 依赖NAD的Sirtuins酶活
正沉默信息调控因子Sirtuins属于去乙酰化酶家族Ⅲ类蛋白,是唯一的组蛋白去乙酰化酶(deactyalses, HDACs),高度保守。Sirtuins蛋白含有一个保守的NAD结合和催化结构域,又称为Sirtuin核心域,但在N-末端和C-末端结构域存在差异。它们有特殊的底物,存在于各类细胞器中。Sirtuins酶活需要NAD激活,共同参与早衰和老化相关病害的新陈代谢活动。由于NAD是酶促反应必需的,所以,Sirtuins活性直接与细胞内的新陈代谢关联。Sirtuins脱乙酰化反应分两步骤。
第一,在乙酰化赖氨酸(Acetyl-lysine)参与下,Sirtuins切割NAD,产生烟酰胺(NAM)和含羰基碳的ADP-核糖;第二,通过脱乙酰作用,将Acetyllysine的乙酰基转移到ADP-核糖上,同时与酶紧连的醋酸进攻羰基碳产生O-acetyl-ADP核糖[23]。Sirtuins就是经过这样的一个过程将Acetyl-lysine的乙酰基转移到相关蛋白中使其乙酰化,从而激活相关蛋白的活性,调控相关生理过程(图2-B)。
有趣的是,生成的烟酰胺是一种Sirtuins的内源性抑制剂,通过NAD补救途径,烟酰胺磷酸核糖转移酶(Nampt)将烟酰胺转化为烟酰胺单核苷酸(NMN),随后,烟酰胺单核苷酸腺苷酰转移酶(NMNAT)再将NMN转化为NAD(图2)。NAD和它的合成途径中的一些衍生物能够调控复杂的细胞内进程,如通过NAD依赖的去乙酰化作用调控基因的转录活性[24-27]。NAD会被沉默信息调控因子Sir2和多聚ADP核糖聚合酶分解为烟酰胺。沉默信息调控因子Sir2家族酶类是NAD依赖的组蛋白去乙酰化酶,能够沉默染色质上的关键基因位点[28,29]。除此之外,NAD还参与营养胁迫的生物合成和生物钟周期调控[30]。
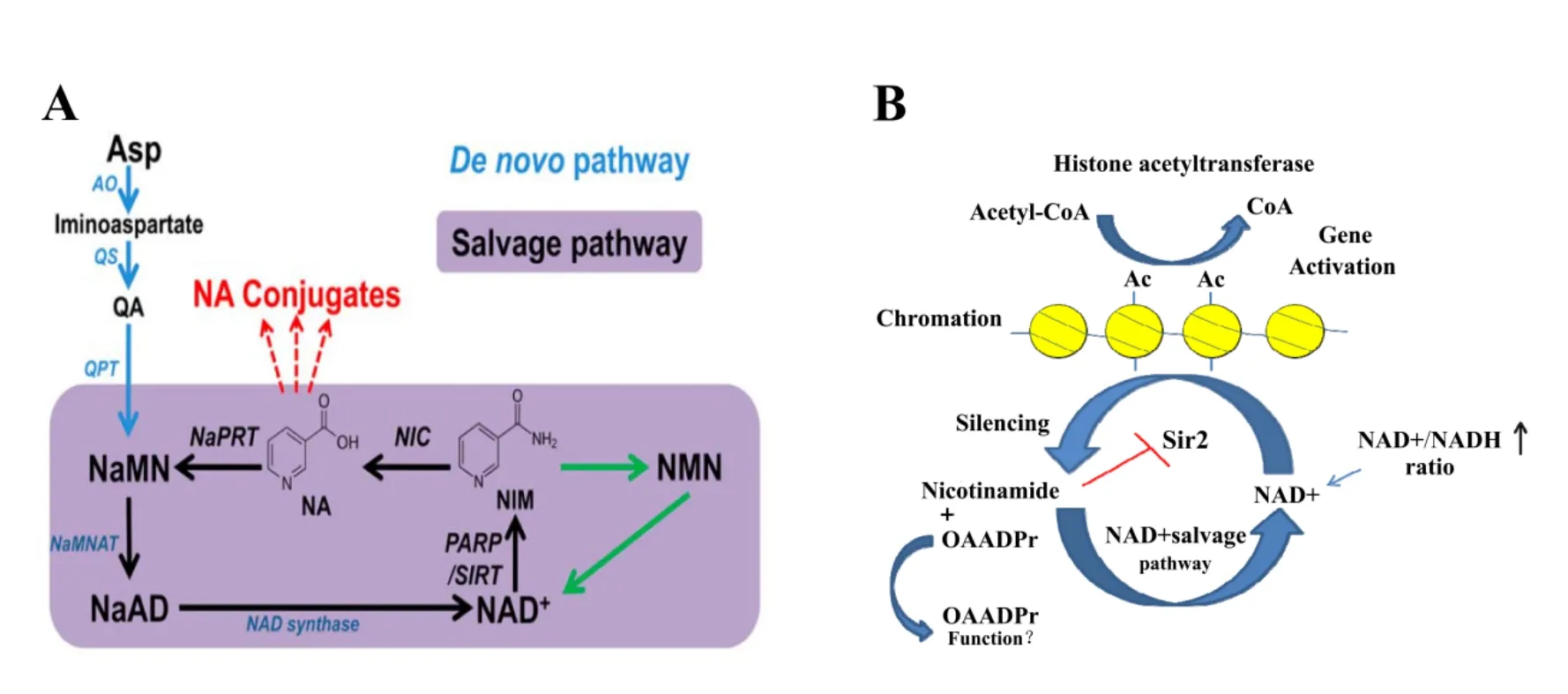
图2 植物中NAD的代谢途径[21]Fig. 2. Metabolism of NAD in plants[21].
Sir2家族蛋白分为四大类,植物中已发现的Sir2蛋白较少,拟南芥中发现有两个Sir2类蛋白编码基因(命名为AtSRT1和AtSRT2)分别属于Sir2蛋白家族四大类中的两类[31]。水稻中同样也发现了两个Sir2类基因(OsSRT1和OsSRT2),分别属于第四类和第二类,通过序列分析,比对拟南芥和水稻中的两个Sir2家族基因,发现AtSRT1和OsSRT1属于同一个组蛋白去乙酰化酶家族并且表现出很高的序列相似性,但是AtSRT2与OsSRT1的序列相比,则表现出高度的差异性,说明两者可能行使完全不同的功能[31](图3)。已有的研究表明拟南芥AtSRT2显示出通过抑制水杨酸的生物合成在植物基础防御病原体PstDC3000中发挥负面作用,感染病原菌后,AtSRT2的表达水平则会下降[32];而敲除AtSRT2能够增强植物对PstDC3000的抗性以及提高PR1等病原相关抗性基因的表达水平。此外,研究人员发现,AtSRT2能够通过调控组蛋白H4K8位点的乙酰化水平调控盐胁迫下的种子萌发过程,主要通过减少盐胁迫下植物DNA的损伤以及NAD+的积累行使基因功能[33]。在水稻中,已有研究表明OsSRT1通过直接去乙酰化组蛋白H3亚基上第9位的赖氨酸位点,抑制转座子类、代谢类和细胞凋亡类基因的表达[34-35]。烟酰胺无论是在体内还是体外的环境,都能有效地抑制SIRT1介导的p53蛋白的去乙酰化过程,影响Sir2蛋白的酶活性, 从而调控细胞的衰老进程[36]。
4 水稻早衰的OsSRT1-NAD调控途径
大部分生物体都是通过补偿途径,再利用NAD分解形成的烟酰胺去合成新的NAD,以维持体内NAD的平衡[37]。之前已有报道NAD合成途径中的一些关键基因在生物学方面的重要性,包括NMNAT、AtNIC1、NADK2等[16,38-39]。在拟南芥中,早衰基因onset-of-leaf-death5(old5)编码喹啉酸合成酶,参与调控NAD生物合成、SIR2蛋白酶活、ROS含量和SAGs基因表达,该基因突变后导致早期老化[16,40]。Liu等[41]报道了水稻中9 个与NADPH氧化酶基因同源的基因Rice respiratory burst oxidase homolog(Osrboh),它们参与调控ROS活性氧含量和细胞死亡。Wu等[11]利用一个水稻叶片早衰相关的叶尖枯突变体lts1(leaf tip senescence 1),借助图位克隆的手段分离了LTS1基因,该基因编码NAD补偿合成途径中的一个关键限速酶——烟酸磷酸核糖转移酶(OsNaPRT1)。揭示了水稻早衰的OsSRT1-NAD调控途径,阐明了异常的烟酸转化为烟酸单核苷酸的过程和如何加速水稻叶片的衰老过程。OsNaPRT1的突变体叶片叶肉细胞中积累大量淀粉粒,同时表现出明显的DNA片段化和过氧化氢产物的积累;叶片中烟酸和烟酰胺的含量较野生型明显增多,而NAD含量减少,烟酰胺的积累抑制了去乙酰化酶OsSRTs基因的表达,导致了组蛋白H3K9位乙酰化水平的上升;OsSRTs基因的表达量下调诱导了衰老相关基因(SAGs)的转录激活,使衰老相关基因的表达量出现不同程度的上调,从而启动植株叶片的过早衰老,最终导致了叶尖枯萎的表型(图3)。
水稻中包含两个SIR2家族编码基因:OsSRT1(Os04g0271000)和OsSRT2(Os12g0179800),且这两个基因分属于SIR2家族的两个不同分类[31]。NAD补偿合成途径的扰乱,导致水稻植株中烟酰胺含量增加和总NAD含量减少。研究发现,降低植株体内烟酰胺含量能有效提高OsSRT1和OsSRT2基因表达量。Huang等[34]报道水稻中OsSRT1主要作用于组蛋白H3K9位点的去乙酰化,参与淀粉积累、抑制转座子和细胞凋亡相关基因的表达;水稻SIR2类基因能维持基因组稳定性和避免植物细胞生长过程中DNA的损伤[35,42]。Wu等[11]通过OsNaPRT1基因的图位克隆和NAD合成途径分析,也证实了OsSRT1和OsSRT2基因表达的下调能够影响水稻组蛋白H3K9位点去乙酰化和衰老相关基因的表达量,最终导致了水稻叶片细胞的衰老。水稻早衰的OsSRT1-NAD调控途径中的关键限速基因的克隆和功能解析有利于探究一种新的延缓水稻叶片衰老的方法,进而为水稻增产提供新思路。
5 水稻早衰的OsSRT1-MeOH-JA调控途径
茉莉酸类物质(JAs)广泛存在于多种植物体内,是一种来源于亚麻酸的氧化脂类植物激素,由丙二烯氧化物合酶(AOS)、丙二烯氧化物酶(AOC)、OPDA reductases(OPR)和acyl CoA oxidases(ACX)等酶催化合成,可以作为一个oxylipin信号分子。茉莉酸甲酯 (methyl jasmonate,MeJA)和茉莉酸异亮氨酸 (jasmonoyl-isoleucine,JA-Ile)由茉莉酸羧基甲基转移酶(JMT)和jasmonoyl异亮氨酸共轭合成酶(JAR)催化茉莉酸获得[44-45]。茉莉酸及其衍生物是第一个被证实的促进衰老的激素[43]。在拟南芥茉莉酸受体coi1突变体中过表达COI1基因,能恢复黑暗诱导引起的叶片衰老,证实JA通过依赖COI1的途径抑制二磷酸核酮糖羧化酶活化酶(rubisco activase, RCA)表达, 进而诱导叶片的衰老[46]。拟南芥中碱性螺螺旋-环-螺旋((bHLH)转录因因子IIIe亚组组的MYC2、MYYC3和MYCC4和IIId亚组的bHLH003、bHLH13、bbHLH14之间间的拮抗作用用可以调控JAA诱导的叶片衰衰老[47]。miRR319通过对对TCP(Teossinte branched/Cyycloidea/PCFF)转录因子以及NAP(NAC-llike activatedd by AP3/PI)家家族的调控改改变植物叶片内内JA含量和AABA含量,从从而正调控植植物叶片的衰老老过程[48-50]。水稻叶片衰衰老负调控因因子OsDOS是一一个CCCH类类型的锌指蛋蛋白[51],通过过调控发育信号号和茉莉酸信号通路的整合合,延缓水稻稻叶片衰老的发发生。
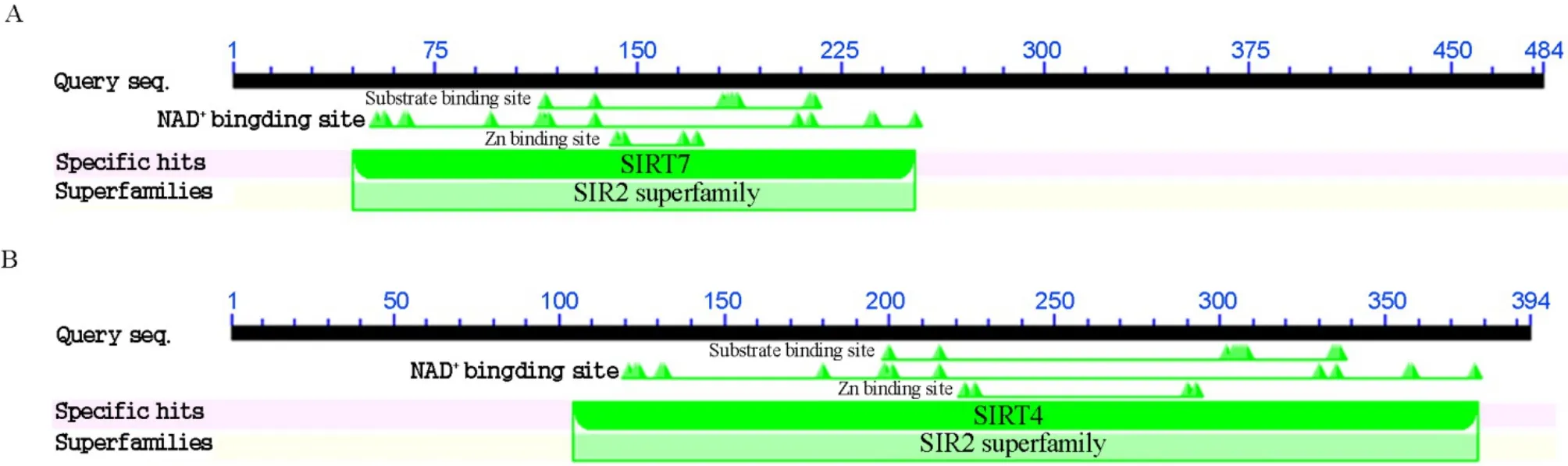
图3 OsSRT1和OsSRT2蛋白保守序列对比分析Fig. 3. Comparative analysis of conserved sequences of OsSRT1 and OsSRT2 proteins.
最新的的研究表明,甲甲醇-茉莉酸级级联反应及其其表观遗传调控控对水稻叶片的衰老起着重重要作用。在在叶片衰老过程程中,其他各各种代谢产物物发生系列反反应,包括甲醇的的积累[52]。在在叶片中,甲醇醇主要来源于于果胶的去甲基基化,这个过程程被果胶甲基基转移酶(PMMTs)、果胶甲酯酶酶(PMEs)和果果胶甲酯酶抑抑制剂(PMEIs)协同控制[52-544]。甲醇可以以作为一种信信号分子在植植物-草食动物相相互作用中起起作用,从而提提高产量和促促进成熟。Fang等[10]对国内外221份水稻稻种质中的茉茉莉酸含量变化化进行了代谢谢组学分析研究,随后基于于该数据进行了了全基因组关联分析,获得得了控制茉莉莉酸含量量的候选基因因OsPME1和和OsTSD2,后续的基因因沉默默和超表达研研究表明甲醇醇-茉莉酸级级联反应途径径在调调控水稻叶片片衰老中发挥挥重要作用。并揭示了OsSSRT1通过组组蛋白H3K9去去乙酰化来降降低OsPME1转录录水平,抑制甲醇-茉莉酸酸代谢生物合合成基因的表表达。。综上,水稻稻OsSRT1通通过表观遗传传修饰负调控控甲醇醇-茉莉酸合成成基因的表达达,进而负调调控水稻叶片片衰老老。水稻OsSRT1-甲醇-茉茉莉酸调控途途径和表观遗遗传调调控衰老的生生物学途径如如图4所示。
6 展望
NAD通过过起始合成途途径和补偿途途径两个途径径参与与生物的重要要生命活动,是重要的调调控因子,除除了在在氧化还原反反应、能量代代谢、信号传传导过程中,发挥挥着重要的作作用外,还作为为ADP-核糖糖、多聚ADPP核糖糖基化、蛋白白质脱乙酰作作用等反应的的底物,能够够调控控代谢酶类的的相关功能[222]。在逆境环环境下,通过过磷酸酸戊糖途径生生成的NADDPH,参与调调控钙调蛋白NAADK的活性和和ROS活性氧氧含量。NADPH还通过过参与与ABA相关关的生理过程程、气孔运动动以及生物和和非生生物逆境来调调控衰老[13-155]。大量研究究证实NADD参与了水稻生长发育、叶绿体发育和胁迫反应[30,55],PI3K调控NADPH氧化酶活性影响种子发芽[41]。水稻体内NAD合成途径受阻导致其叶部提前衰老,对这一途径中关键限速基因的克隆和功能解析有利于探究一种新的延缓水稻叶片衰老的方法。因此,调控NAD合成途径关键基因的表达应该是未来提高水稻产量的一个有效策略。
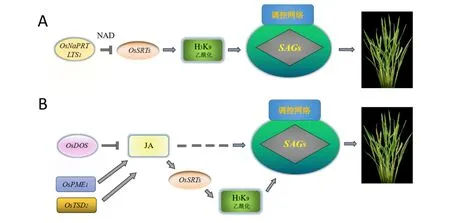
图4 水稻早早衰的NAD、JJA和SRT1调控途径Fig. 4. Regulaation pathwayss of NAD, JA aand SRT1 invoolved in leaf sennescence in ricce.
叶片衰老是一个极其复杂的过程,大量研究表明外界环境因子,内源激素、遗传因子和表观遗传等因素对水稻叶片衰老都会有影响,有的还涉及多层次的基因调控网络[42,56,57]。近几年,越来越多的研究发现表观遗传修饰也参与叶片衰老的调控,包括乙酰化、甲基化和染色质结构修饰,表观调控突变体的衰老变异,以及衰老叶片SAGs组蛋白修饰的衰老特异改变等。特别是水稻SIR2家族蛋白的乙酰化,参与调控叶片衰老。但由于叶片衰老机理的复杂性,水稻叶片衰老表观遗传研究还刚刚起步,SIR2如何延迟衰老、保持适当持绿和增产之间的分子机制,以及特异表观遗传因子和调控网络还有待深入研究。
虽然在农作物生产中,早衰水稻品种每天可减少产量2%。杂交稻多数组合存在早衰现象,早衰与其库源矛盾较大、根系活力低,供N量、叶绿素、可溶性蛋白质含量及SOD活性降低有关[8]。所以,选育抗早衰水稻品种具有重要的实践意义。随着现代生物信息学和蛋白质组学等技术的不断发展和进步,作物衰老的分子机理越来越明晰,我们能更好地利用和改善植物叶片衰老,比如外源喷施负调控因子细胞分裂素和生长素、通过基因工程定点激活衰老负相关因子和抑制衰老正相关因子等延长农作物的生长期,延缓叶片衰老。适当增加JA,既能影响ABA含量和叶片衰老,又能增强农作物对外部环境胁迫的响应能力,增加水稻产量。我们也可利用衰老频度检测水稻衰老过程[58]、导入持绿基因或SIR2长寿因子[59]延长功能叶片持绿期以提高作物产量,为抗衰老作物品种的遗传改良提供新思路。例如,我们了解了水稻早衰的OsSRT1-NAD调控途径,通过相关基因表达和底物生成物的含量变化,可以适当提前或推迟水稻熟期,控制早衰,达到增产目的。针对这一特点,我们可以根据生产需求采取以下措施。在水稻后期,适当增加水稻长寿因子OsSRTs含量或烟酰胺含量,可以抑制组蛋白H3K9位乙酰化和衰老相关基因(SAGs)的表达,适当延缓水稻叶片的衰老;相反,如果水稻后期产生贪青现象,就要适当减少水稻植株内OsSRTs含量,增加H3K9位乙酰化,促使叶片中的养分及时地运输到籽粒,达到增产的效果。类似地,生产中,也可通过调节水稻植株内茉莉酸含量,调节减缓衰老时间,确保水稻增产的效果。
[1] Gan S, Amasino R M. Making sense of senescence (molecular genetic regulation and manipulation of leaf senescence). Plant Physiol, 1997, 113(2): 313-319.
[2] Noodén L D. The phenomena of senescence and aging. Senescence & Aging in Plants, 1988, 1-50.
[3] Noodén L D. 12-Whole plant senescence. Senescence & Aging in Plants, 1988, 3(3): 391-439.
[4] 赵春德, 张迎信, 刘群恩, 余宁, 朱爱科, 程式华, 曹立勇. 水稻叶片衰老分子机制研究进展. 分子植物育种, 2015, 13(3): 680-688. 2008, 30(5): 757-765. Zhao C D, Zhang Y X, Liu Q E, Yu N, Zhu A K, Cheng S H, Cao L Y. Research advance on molecular regulation mechanism of leaf senescence in rice. Mol Plant Breeding, 2015, 13(3): 680-688. (in Chinese with English abstract)
[5] 段娜, 贾玉奎, 徐军, 陈海玲, 孙鹏. 植物内源激素研究进展. 中国农学通报, 2015, 31(2): 159-165. Duan N, Jia Y K, Xu J, Chen H L, Sun P. Research progress on plant endogenous hormone. Chin Agric Sci Bull, 2015, 13(3): 680-688. (in Chinese with English abstract)
[6] Guo Y F, Gan S S. Leaf senescence: Signals, execution, and regulation. Curr Top Dev Biol, 2005, 71: 83-112.
[7] Lim P O, Kim H J, Nam H G. Leaf senescence. Annu Rev Plant Biol, 2007, 58: 115-136.
[8] Gregersen P L, Culetic A, Boschian L, Krupinska K. Plant senescence and crop productivity. Plant Mol Biol, 2013, 82(6): 603-622.
[9] 孙玉莹, 毕京翠, 赵志超, 程治军, 万建民. 作物叶片衰老研究进展. 作物杂志, 2013(4): 11-19. Sun Y Y, Bi Y C, Zhao Z C, Chen Z J, Wan J M. The advancement on leaf senescence in crops. Crops, 2013, (4): 11-19. (in Chinese)
[10] Fang C, Zhang H, Wan J, Wu Y, Li K, Jin C, Chen W, Wang S, Wang W, Zhang H, Zhang P, Zhang F, Qu L, Liu X, Zhou D X, Luo J. Control of leaf senescence by a MeOH-Jasmonates cascade that is epigenetically regulated by OsSRT1 in rice. Mol Plant, 2016, 9(10): 1366-1378.
[11] Wu L , Ren D , Hu S, Li G, Dong G, Jiang L, Hu X, Ye W, Cui Y, Zhu L, Hu J, Zhang G, Gao Z, Zeng D, Qian Q,Guo L. Mutation of OsNaPRT1 in the NAD salvage pathway leads to withered leaf tips in rice. Plant Physiol, 2016, 171(2): 1085-1098.
[12] Berger F, Ramirez-Hernandez M H, Ziegler M. The new life of a centenarian: signalling functions of NAD(P). Trends Biochem Sci, 2004, 29(3): 111-118.
[13] Hunt L, Lerner F, Ziegler M. NAD-new roles in signalling and gene regulation in plants. New Phytol, 2004, 163(1): 31-44.
[14] Noctor G, Queval G, Gakiere B. NAD(P) synthesis and pyridine nucleotide cycling in plants and their potential importance in stress conditions. J Exp Bot, 2006, 57(8): 1603-1620.
[15] Katoh A, Uenohara K, Akita M, Hashimoto T. Early steps in the biosynthesis of NAD in Arabidopsis start with aspartate and occur in the plastid. Plant Physiol, 2006, 141(3): 851-857.
[16] Wang G D, Pichersky E. Nicotinamidase participates in the salvage pathway of NAD biosynthesis in Arabidopsis. Plant J, 2007, 49(6): 1020-1029.
[17] Buchananwollaston V. The molecular biology of leaf senescence. J Exp Bot, 1997, 48(307): 181-199.
[18] Gan S. Mitotic and postmitotic senescence in plants. Sci Aging Knowl Environ, 2003, 2003(38): 7.
[19] Zhang H, Zhou C. Signal transduction in leaf senescence. Plant Mol Biol, 2013, 82(6): 539-545.
[20] Rao Y, Yang Y, Xu J, Li X, Leng Y, Dai L, Huang L, Shao G, Ren D, Hu J, Guo L, Pan J, Zeng D. EARLY SENESCENCE1 encodes a SCAR-LIKE PROTEIN2 that affects water loss in rice. Plant Physiol, 2015, 169(2): 1225-1239.
[21] Joakim R, Anders W. Nicotinate O-glucosylation is an evolutionarily metabolic trait important for seed germination under stress conditions in Arabidopsis thaliana. Plant Cell, 2015, 27(7): 1907-1924.
[22] Ziegler M. New functions of a long-known molecule. Eur J Biochem, 2000, 267(6): 1550-1564.
[23] Tanner K G, Landry J, Sternglanz R, Denu J M. Silent information regulator 2 family of NAD-dependent histone/protein deacetylases generates a unique product, 1-O-acetyl-ADP-ribose. Proc Natl Acad Sci USA, 2000, 97(26): 14178-14182.
[24] Imai S, Armstrong C M, Kaeberlein M, Guarente L. Transcriptional silencing and longevity protein Sir2 is an NAD+-dependent histone deacetylase. Nature, 2000, 403(6771): 795-800.
[25] North B J, Marshall B L, Borra M T, Denu J M, Verdin E. The human Sir2 ortholog, SIRT2, is an NAD+-dependent tubulin deacetylase. Mol Cell, 2003, 11(2): 437-444.
[26] Sánchez J P, Duque P, Chua N H. ABA activates ADPR cyclase and cADPR induces a subset of ABA-responsive genes in Arabidopsis. Plant J, 2004, 38(3): 381-395.
[27] De Block M, Verduyn C, De Brouwer D, Cornelissen M. Poly(ADP-ribose) polymerase in plants affects energy homeostasis, cell death and stress tolerance. Plant J, 2005, 41(1): 95-106.
[28] Frye R. Phylogenetic classification of prokaryotic and eukaryotic Sir2-like proteins. Biochem Bioph Res Co, 2000, 273(2): 793-798.
[29] Moazed D. Enzymatic activities of Sir2 and chromatin silencing. Curr Opin Cell Biol, 2001, 13(2): 232-238.
[30] Sakuraba Y, Rahman M L, Cho S H, Kim Y S, Koh H J, Yoo S C, Paek N C. The rice faded green leaf locus encodes protochlorophyllide oxidoreductase B and is essential for chlorophyll synthesis under high light conditions. Plant J, 2013, 74(1): 122-133.
[31] Pandey R, Müller A, Napoli C A, Selinger D A, Pikaard C S, Richards E J, Bender J, Mount D W, Jorgensen R A. Analysis of histone acetyltransferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes. Nucleic Acids Res, 2002, 30(23): 5036-5055.
[32] Wang C, Gao F, Wu J, Dai J, Wei C, Li Y. Arabidopsis putative deacetylase AtSRT2 regulates basal defense by suppressing PAD4, EDS5 and SID2 expression. Plant Cell Physiol, 2010, 51(8): 1291-1299.
[33] 钟理. 拟南芥组蛋白去乙酰化酶基因家族分析及AtSRT2在盐胁迫下调控种子萌发的分子机制. 北京:中国农业科学院, 2015. Zhong L. Analysis of histone deacetylase gene families in Arabidopsis thaliana and the molecular mechanism of AtSRT2 controlling seed germination under salt stress. Beijing: Chinese Academy of Agricultural Sciences, 2015. (in Chinese with English abstract)
[34] Huang L, Zhou D X. Down-regulation of a SILENT INFORMATION REGULATOR2-related histone deacetylase gene, OsSRT1, induces DNA fragmentation and cell death in rice. Plant Physiol, 2007, 144(3): 1508-1519.
[35] Zhong X, Zhang H, Zhao Y, Sun Q, Hu Y, Peng H, Zhou D. The rice NAD+-dependent histone deacetylase OsSRT1 targets preferentially to stress- and metabolism-related genes and transposable elements. PLoS ONE, 2013, 8(6): e66807.
[36] Schmidt M T, Smith B C, Jackson M D, Denu J M. Coenzyme specificity of Sir2 protein deacetylases: Implications for physiological regulation. J Biol Chem, 2004, 279(38): 40122-40129.
[37] Ashihara H, Stasolla C, Yin Y, Loukanina N, Thorpe T A. De novo and salvage biosynthetic pathways of pyridine nucleotides and nicotinic acid conjugates in cultured plant cells. Plant Sci, 2005, 169(1): 107-114.
[38] Hashida S N, Takahashi H, Kawai-Yamada M, Uchimiya H. Arabidopsis thaliana nicotinate/nicotinamide mononucleotide adenyltransferase (AtNMNAT) is required for pollen tube growth. Plant J, 2007, 49(4): 694-703.
[39] Hashida S N, Hideyuki, Uchimiya H. The role of NAD biosynthesis in plant development and stress responses. Ann Bot, 2009, 103(6): 819-824.
[40] Schippers J H M, Nunes-Nesi A, Apetrei R, Hille J, Fernie A R, Dijkwel P P. The Arabidopsis onset of leaf death5 mutation of quinolinate synthase affects nicotinamide adenine dinucleotide biosynthesis and causes early ageing. Plant Cell, 2008, 20(10): 2909-2925. [41] Liu J, Zhou J, Xing D. Phosphatidylinositol 3-kinase plays a vital role in regulation of rice seed vigor via altering NADPH oxidase activity. PLoS ONE, 2012, 7(3): e33817.
[42] Huang Q N, Shi Y F, Zhang X B, Song L X, Feng B H, Wang H M, Xu X, Li X H, Guo D, Wu J L. Single base substitution in OsCDC48 is responsible for premature senescence and death phenotype in rice. J Integr Plant Biol, 2016, 58(1): 12-28.
[43] Ueda J, Kato J. Isolation and identification of a senescence-promoting substance from wormwood (Artemisia absinthium L.). Plant Physiol, 1980, 66(2): 246-249.
[44] Kim J H, Woo H R, Kim J, Lim P O, Lee I C, Choi S H, Hwang D, Nam H G. Trifurcate feed-forward regulation of age-dependent cell death involving miR164 in Arabidopsis. Science, 2009, 323(5917): 1053-1057.
[45] Wakuta S, Suzuki E, Saburi W, Matsuura H, Nabeta K, Imai R, Matsui H. OsJAR1 and OsJAR2 are jasmonyl-L-isoleucine synthases involved in wound- and pathogen-induced jasmonic acid signalling. Biochem Bioph Res Commun, 2011, 409(4): 634-639.
[46] Shan X Y, Wang J X, Chua L L, Jiang D A, Peng W, Xie D X. The role of Arabidopsis Rubisco activase in jasmonate-induced leaf senescence. Plant Physiol, 2011, 155(2): 751-764.
[47] Qi T, Wang J, Huang H, Liu B, Gao H, Liu Y, Song S, Xie D. Regulation of jasmonate-induced leaf senescence by antagonism between bHLH subgroup IIIe and IIId factors in Arabidopsis. Plant Cell, 2015, 27(6): 1634-1649.
[48] Zhou Y, Huang W, Liu L, Chen T, Zhou F, Lin Y. Identification and functional characterization of a rice NAC gene involved in the regulation of leaf senescence. BMC Plant Biol, 2013, 13(1): 1-13.
[49] Liang C Z, Wang Y Q, Zhu Y N, Tang J Y, Hu B, Liu L C, Ou S J, Wu H K, Sun X H, Chu J F, Chu C C. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice. Proc Natl Acad Sci USA, 2014, 111(27): 10013-10018.
[50] Chen X, Wang Y, Lv B, Li J, Luo L, Lu S, Zhang X, Ma H, Ming F. The NAC family transcription factor OsNAP confers abiotic stress response through the ABA pathway. Plant Cell Physiol, 2014, 55(3): 604-619.
[51] Kong Z, Li M, Yang W, Xu W, Xue Y. A novel nuclear-localized CCCH-type zinc finger protein, OsDOS, is involved in delaying leaf senescence in rice. Plant Physiol, 2006, 141(4): 1376-1388.
[52] Kang K, Park S, Natsagdorj U, Kim Y S, Back K. Methanol is an endogenous elicitor molecule for the synthesis of tryptophan and tryptophan-derived secondary metabolites upon senescence of detached rice leaves. Plant J, 2011, 66(2): 247-257.
[53] Krupková E, Immerzeel P, Pauly M, Schmülling T. The TUMOROUS SHOOT DEVELOPMENT2 gene of Arabidopsis encoding a putative methyltransferase is required for cell adhesion and co-ordinated plant development. Plant J, 2007, 50(4): 735-750.
[54] Körner E, von Dahl C C, Bonaventure G, Baldwin I T. Pectin methylesterase NaPME1 contributes to the emission of methanol during insect herbivory and to the elicitation of defence responses in Nicotiana attenuata. J Exp Bot, 2009, 60(9): 2631-2640.
[55] Brachmann C B, Sherman J M, Devine S E, Cameron E E, Pillus L, Boeke J D. The SIR2 gene family, conserved from bacteria to humans, functions in silencing, cell cycle progression, and chromosome stability. Gene Dev, 1995, 9(23): 2888-2902.
[56] Sun F, Qi W W, Qian X Y, Wang K J, Yang M F, Dong X X, Yang J S. Investigating the role of OsPDCD5, a homolog of the mammalian PDCD5, in programmed cell death by inducible expression in rice. Plant Mol Biol Rep, 2012, 30(1): 87-98.
[57] Singh S, Giri M K, Singh P K, Siddiqui A, Nandi A K. Down-regulation of OsSAG12-1 results in enhanced senescence and pathogen-induced cell death in transgenic rice plants. J Biosciences, 2013, 38(3): 583-592.
[58] 李木英, 石庆华, 郑伟, 潘晓华, 谭雪明. 杂交稻后期叶片早衰特征及其与叶片N含量和根系活力关系初探.江西农业大学学报, 2008, 30(5): 757-765. Li M Y, Shi Q H, Zheng W, Pan X H, Tan X M. A preliminary study on relationship between leaf premature senescence characteristic and leaf N content, root activity in hybrid rice during grain filling stage. Acta Agric Univ Jiangxi, 2008, 30(5): 757-765.
[59] Lin S J, Defossez P A, Guarente L. Requirement of NAD and SIR2 for life-span extension by calorie restriction in Saccharomyces cerevisiae. Science, 2000, 289(5487): 2126-2128.
Nicotinamide Adenine Dinucleotide(NAD) Biosynthesis Pathway and Leaf Senescence in Rice
CHEN Ping1, WU Liwen1, WANG Zhongwei1, ZHANG Yu1,2, GUO Longbiao1,*
(1China National Rice Research Institute, Hangzhou 310006, China;2Guangxi Rate Seed Co. Ltd, Nanning 530003, China;*Corresponding author, E-mail: guolongbiao@caas.cn)
Leaf is an important photosynthetic organ in rice, and its senescence is initiated by the endogenous genetic and developmental signals and the external environmental stress. Regulation of leaf senescence is a very complicated and delicate process in plants. NAD (nicotinamide adenine dinucleotide), a coenzyme of dehydrogenase, plays an irreplaceable role in glycolysis, gluconeogenesis, Krebs cycle, respiratory chain and other metabolism. The latest researches showed that the NAD biosynthesis in rice is involved in the regulation of the enzyme activity of silencing gene Sirtuins, histone H3K9 deacetylation, plant hormone jasmonic acid (JA) and leaf senescence. This paper highlights cell physiological characteristics, enzyme activity of Sirtuins, NAD biosynthesis of plant leaf senescence, and the OsSRT1-NAD and OsSRT1-MeOH-JA regulation pathways of leaf senescence in rice. It is beneficial to elucidation of the molecular mechanism and regulation pathways of leaf senescence, and application of high yield breeding in rice.
Oryza sativa; leaf senescence; NAD biosynthesis pathway; jasmonic acid; silent information regulator
Q755; S511.01
A
1001-7216(2017)05-0447-10
2017-03-08;修改稿收到日期:2017-06-06。
国家自然科学基金资助项目(31461143014); 国家科技支撑计划资助项目(2015BAD01B02-2); 中国博士后科学基金资助项目(2016T90155)。
