水稻淡绿叶基因PGL11的鉴定与精细定位
2017-09-23涂政军邹国兴黄李超陈龙代丽萍高易宏冷语佳朱丽张光恒胡江任德勇高振宇董国军陈光郭龙彪钱前曾大力
涂政军 邹国兴 黄李超 陈龙 代丽萍 高易宏 冷语佳 朱丽 张光恒胡江 任德勇 高振宇 董国军 陈光 郭龙彪 钱前,* 曾大力,*
(1中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;2江西省农业科学院 水稻研究所, 南昌 330200;#共同第一作者;*通讯联系人, E-mail: dalizeng@126.com; qianqian188@hotmail.com)
水稻淡绿叶基因PGL11的鉴定与精细定位
涂政军1,#邹国兴2,#黄李超1陈龙1代丽萍1高易宏1冷语佳1朱丽1张光恒1胡江1任德勇1高振宇1董国军1陈光1郭龙彪1钱前1,*曾大力1,*
(1中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;2江西省农业科学院 水稻研究所, 南昌 330200;#共同第一作者;*通讯联系人, E-mail: dalizeng@126.com; qianqian188@hotmail.com)
【目的】叶片是水稻进行光合作用的主要场所,叶片颜色的变化与水稻的生长发育直接相关。发掘水稻叶色突变体,是水稻功能基因组学研究的重要遗传基础。【方法】利用EMS诱变日本晴获得一个能稳定遗传的淡绿叶突变体,暂命名为pgl11(pale green leaf 11)。在不同生育期测定野生型与突变体的叶绿素含量。在苗期,取野生型与突变体叶片进行叶绿体结构的透射电镜观察。在分蘖期,测定野生型与突变体的光合参数并观察气孔结构。在成熟期,测定野生型和pgl11的主要农艺性状。以pgl11为母本,南京6号为父本构建相应的F2群体,采用图位克隆的方法,对该基因进行定位。【结果】从苗期开始,突变体pgl11的每一片新叶均表现为淡绿色,叶绿素含量显著降低,叶绿体发育异常。随着叶片的生长,叶色由淡绿逐渐转绿,至抽穗期时叶绿素含量亦无明显差异。pgl11还表现光合速率、气孔导度明显下降,胞间CO2浓度上升。扫描电镜观察发现,突变体pgl11的气孔发育异常。与野生型相比,突变体的农艺性状如株高、剑叶宽、二次枝梗数、每穗粒数、粒长、粒宽、千粒重以及结实率等均显著降低。对叶绿素合成、光合作用以及质体发育相关基因的表达量测定表明,突变体pgl11中参与叶绿体转录和翻译相关基因的表达量显著升高,而叶绿素合成和光合作用相关基因的表达量显著下降。遗传分析表明,该突变表型受一对隐性核基因控制。通过图位克隆的方法将该基因定位于第1染色体上的C6和C8标记之间,物理距离约为110 kb。【结论】该定位区间内未见有叶色相关基因报道,推测PGL11基因可能是一个新的水稻叶色基因。
水稻;淡绿叶;叶绿体;遗传分析;基因定位
叶绿素是光合器官的重要组成部分,在植物生长发育过程中起关键作用[1]。除了为植物提供绿色的外观之外,叶绿素在光合作用中负责接收捕获光能,同时驱动电子通过电子传递链转移到光合反应中心[2],完成光能转化为化学能的过程[3]。水稻叶绿素含量的增加可以提高光合作用率[4],增加光合有机物的积累,并最终提高水稻的产量[5-6]。叶色相关基因的突变,会引起叶绿素的含量及叶绿体结构发生变化,最终形成白化、黄化、条纹、浅绿和斑点等类型的突变[7]。目前,在水稻[8-9]、拟南芥[10]、玉米[11]、棉花[12],小麦[13]、油菜[14]等多种植物叶均发现有叶色突变体的存在。
迄今为止,已有不少于80个水稻叶色突变体得到鉴定,且多个已被克隆。其中包括NTRC[15]、OsClpP5[16]、SGR[17]、OsDVR[18]、OsChlH/I/D[19-20]、YGL1[21]、OsCAO1[22]、OsCAO2[23]、YSA[24]、OsRpoTp[25]、OsCHR4[26]、OsPPR1[27]以及OsPDS[28]等。这些基因克隆和功能分析丰富了水稻叶色相关遗传调控机理的阐述,水稻叶色突变体的表型一般是受核基因或细胞质基因控制,其中,以隐性核基因控制的情况居多[29-31]。根据水稻叶色突变体的形成机制可分为叶绿素代谢途径受阻[22,23]、叶绿体发育进程受阻[9,25]、光敏色素调控受阻[32]、血红素反馈调节紊乱等[33],而以叶绿素合成途径的研究报道最为深入。通过对双子叶植物(如拟南芥)的研究发现,高等植物叶绿素生物合成通常需要通过一系列复杂的反应[34],参与叶绿素合成的27个基因及其编码的酶都已被鉴定[35]。水稻中目前也已克隆9个编码叶绿素合成途径相关酶的基因,分别为编码镁离子螯合酶三个D、I、H小亚基的OsChlD、OsChlI和OsChlH基因[19,20]、编码联乙烯还原酶的OsDVR基因[18]、编码NADPH-原叶绿素酸酯氧化还原酶A和B的OsPORA和OsPORB基因[36,37]、编码叶绿素合酶的YGL1基因[21]以及编码叶绿素酸酯a加氧酶的OsCAO1和OsCAO2基因[22,23]。这些基因的突变都会对水稻叶色及生长产生影响。
目前,对叶色突变体形成的分子机制的研究仍然不是十分清楚,主要是因为其形成过程十分复杂,牵涉到多方面的生理生化反应。与叶绿体发育相关基因的突变不仅直接影响叶绿体的发育,同时也会间接影响叶绿素的合成。水稻叶色基因V2的突变导致叶绿体的分化受到抑制,尤其在叶片发育早期破坏了叶绿体的转录/翻译机制,最终造成叶绿体发育迟滞,叶绿素含量降低,表现出白化表型[38]。V3和St1分别编码核糖核酸还原酶RNR的大、小亚基,其突变体植株的核糖核苷酸还原酶活性显著下降,导致与质体发育及光合作用相关基因的表达量发生了改变,进而影响叶绿体的发育[39]。三角状五肽重复区蛋白(PPR)在水稻叶绿体发育过程中起着重要的作用,OsPPR1是第一个报道的参与水稻叶绿体形成的PPR蛋白。抑制该基因的表达,植株表现出缺绿的突变表型,甚至还会出现白化致死现象[27]。
本研究从粳稻日本晴EMS突变体库中筛选到一个能够稳定遗传的淡绿叶突变体,暂命名为pgl11。从表型、生理、遗传和基因表达等方面分析了pgl11的特征,图位克隆将pgl11定位在第1染色体上110 kb的物理距离。这些结果将为最终分离PGL11基因,阐明其遗传调控机理奠定基础。
1 材料与方法
1.1 突变体来源及种植
EMS诱变粳稻品种日本晴,经多代自交性状稳定,获得淡绿叶突变体pgl11。pgl11与培矮64S和南京6号杂交获得的F1及F2群体用于基因的遗传分析与精细定位。所有材料(包括野生型日本晴)均种植于中国水稻研究所富阳和海南试验基地。
1.2 农艺性状调查
在水稻抽穗期测定野生型和突变体pgl11的剑叶长、剑叶宽,在完熟期测定野生型和突变体pgl11的株高、分蘖数、穗长、一次枝梗数、二次枝梗数、每穗粒数、粒长、粒宽、千粒重与结实率等主要农艺性状,每个性状10次重复,并采用t检验法分析突变体与野生型之间的差异显著性。
1.3 叶绿素含量的测定取相同生长时期野生型和突变体的叶片0.05 g,浸入5 mL的丙酮提取液(v95%丙酮∶v无水乙醇=2∶1)中,在26℃黑暗条件下充分浸提24 h[40]。利用 DU800UV/Vis分光光度计分别测定三个不同波长(470 nm、645 nm和663 nm)下浸提液的吸光度值,根据公式[41]计算出对应的叶绿素a、叶绿素 b及类胡萝卜素含量,每个样品设置3个重复,结果取平均值。
1.4 光合参数的测定
水稻分蘖盛期,在晴好天气的上午9点到11点时间段内,用便携式光合测定仪Li-6400到田间对野生型及突变体的剑叶进行光合测定。选取位于种植小区中间部分且长势基本一致的野生型和突变体pgl11植株,分别测定其净光合速率、气孔导度、胞间CO2浓度和蒸腾速率等参数,测定部位为剑叶中上部,共测定5个重复。结果取平均值。
1.5 叶片的扫描电镜观察
为了观察叶绿体的结构,分别取野生型和突变体幼苗的新出叶,剪成小片,于2.5%的戊二醛溶液中抽真空。固定后分别用 50%、70%、80%、95%和100%的乙醇和丙酮进行逐级脱水,最后用环氧化树脂包埋,样品经过切片、醋酸铀染色后,在透射电子显微镜下进行观察[42]。
1.6 突变体pgl11的遗传分析与基因定位
当年夏季,将突变体pgl11与培矮64和南京6号杂交,同年冬季,将杂交种F1种植于海南陵水基地,并观察两个组合的F1表型,自交获得F2种子。次年夏季将这两个F2群体种植于浙江杭州富阳实验基地,并分别调查野生型表型和突变体表型植株的数目,考查性状的分离比并通过卡方检验验证,完成遗传分析。
利用本实验室保存的均匀分布于12条染色体的232对引物对双亲和F1进行多态性筛选,从中获得136对多态性的标记用于pgl11的连锁分析和初定位。并利用籼稻9311序列(http://www.rise. genomics.org.cn)和粳稻日本晴(NPB)序列(http://rgp.dna.affrc.go.jp),在目标区间开发多态性标记,进一步扩大pgl11与南京6号的F2群体,进行精细定位。
1.7 RNA的提取及定量RT-PCR
利用Qiagen公司的总RNA提取试剂盒(RNeasy plant mini kit)对野生型和突变体pgl11的总RNA进行提取,并以RNase-free DNaseⅠ处理过的总RNA为模板,采用ToYoBo公司的cDNA合成试剂盒进行反转录。利用Applied Biosystems公司的ABI7900对野生型和突变体中相关基因的表达量进行测定。以基因Ubiqutin作为内参基因。10 μL RT-PCR体系包括cDNA模板1 μL,2×SYBR qPCR Mix(ToYoBo) 5 μL,正反引物(10 μmol/L)各0.5 μL,ddH2O补足至10 μL。RT-PCR扩增程序如下:95℃下预变性1.5 min;95℃下10 s,60℃下30 s,72℃下20 s,共40个循环。以2-ΔΔCT法计算各基因的相对表达量[43]。RT-PCR中相关基因检测所用引物见表1。

表1 实时定量PCR引物Table 1. Primers used in real-time PCR.
2 结果与分析
2.1 突变体pgl11表型及农艺性状分析
在自然生长条件下,突变体pgl11从出芽开始叶片即表现淡绿(图1-A),随着叶片的发育,新叶从叶尖到叶基部逐渐变绿(图1-B),到抽穗期叶片颜色完全恢复绿色(图1-C)。调查结果显示,突变体的抽穗期较野生型推迟一周,其农艺性状除分蘖数、剑叶长、穗长及一次枝梗数未发生明显变化外,株高、剑叶宽、二次枝梗数、每穗粒数、粒宽、千粒重以及结实率等性状均存在极显著差异(表2)。
2.2 突变体pgl11叶绿素含量的变化
由于突变体pgl11叶片颜色在发育过程中出现转绿现象,因此,我们对野生型和突变体pgl11苗期、分蘖期和抽穗期叶片的叶绿素含量进行了测定。结果表明,在苗期突变体叶绿素含量显著下降;到分蘖期,二者差异减小;抽穗期时,野生型与突变体的叶绿素含量则无明显差异。其中,突变体pgl11苗期的叶绿素a、叶绿素b及类胡萝卜素含量分别较野生型下降了77.25%、80.06%和51.88%(图2-A)。在分蘖期,突变体pgl11的叶绿素a、叶绿素b及类胡萝卜素含量则分别降低了22.2%、28.7%和12.5%(图2-B)。而到了抽穗期,野生型与突变体pgl11的光合色素含量差异不显著(图2-C)。
2.3 突变体pgl11的光合参数分析

图1 野生型和突变体pgl11在不同生育期的表型Fig. 1. Phenotypes of the wild type(WT) and the pgl11 mutant at various growth stage.
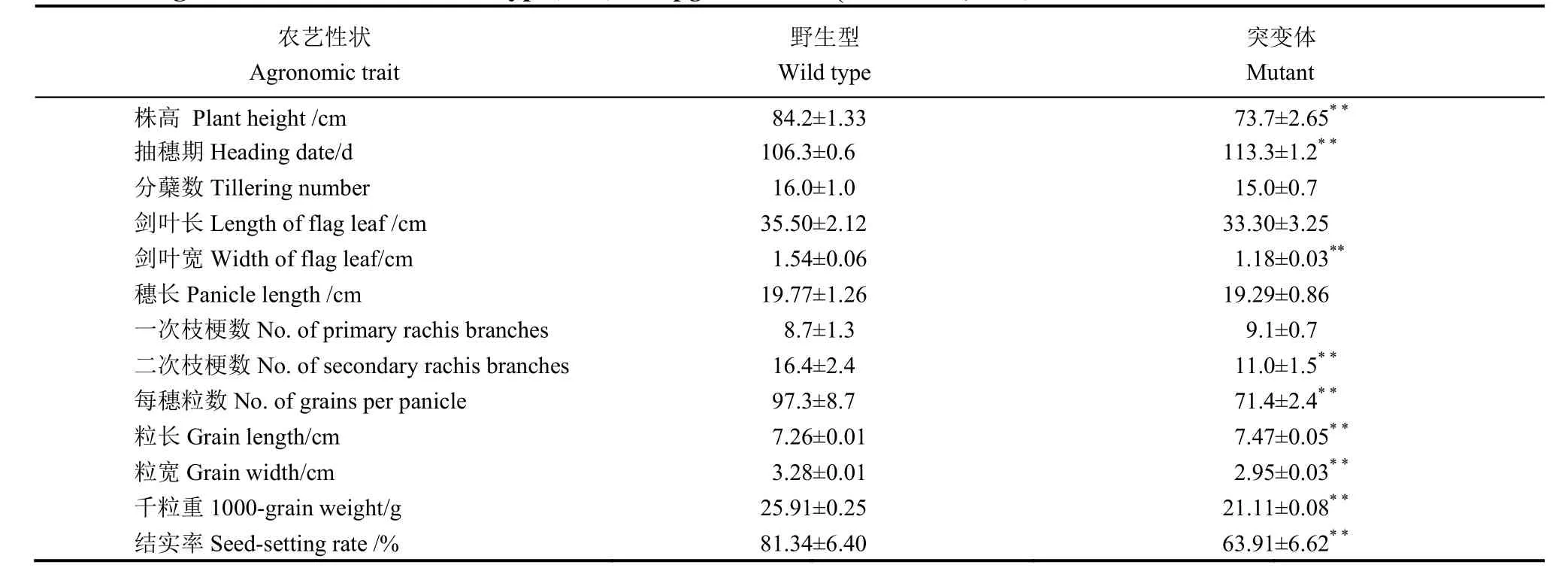
表2 野生型WT和突变体pgl11的基本农艺性状(平均数±标准差, n=3)Table 2.Agronomic traits of the wild type(WT) and pgl11 mutant(Mean±SD, n=3).
为了分析突变体pgl11光合作用是否受到影响,我们对分蘖期野生型和突变体pgl11剑叶的净光合速率、气孔导度、胞间CO2浓度和蒸腾速度等4个光合指标进行测定。结果表明,突变体除了胞间CO2浓度有所增加外,其余3个参数均出现显著下降。其中,突变体pgl11剑叶净光合速率、蒸腾速率和气孔导度分别下降了约32.3%、34.3%和21.9%,差异均达极显著水平,而胞间CO2浓度上升了16.84%(表3)。扫描电镜观察野生型和突变体pgl11叶片气孔结构发现,突变体pgl11的气孔收缩并呈带状下陷,且下陷的气孔口周围没有硅质化乳突,部分气孔发育不完全(图3)。说明PGL11突变后导气孔发育异常,光合作用受到明显影响,主要体现在胞间CO2浓度显著提高,而净光合速率和气孔导度显著降低。

表3 分蘖盛期野生型和突变体pgl11的剑叶光合参数(平均数±标准差, n=5)Table 3.Photosynthetic parameters of flag leaf of wild type(WT) and pgl111 mutant at the tillering stage(Mean±SD, n=5).
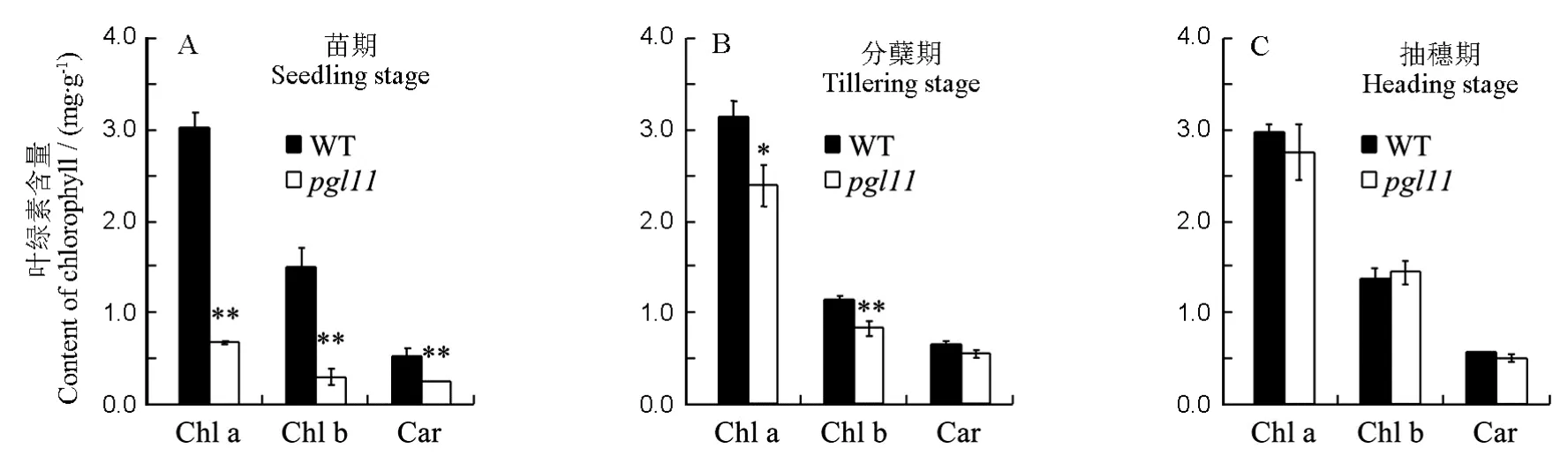
图2 野生型与突变体pgl11的叶绿素含量比较Fig. 2. Comparison of chlorophyll contents between WT and pgl11.
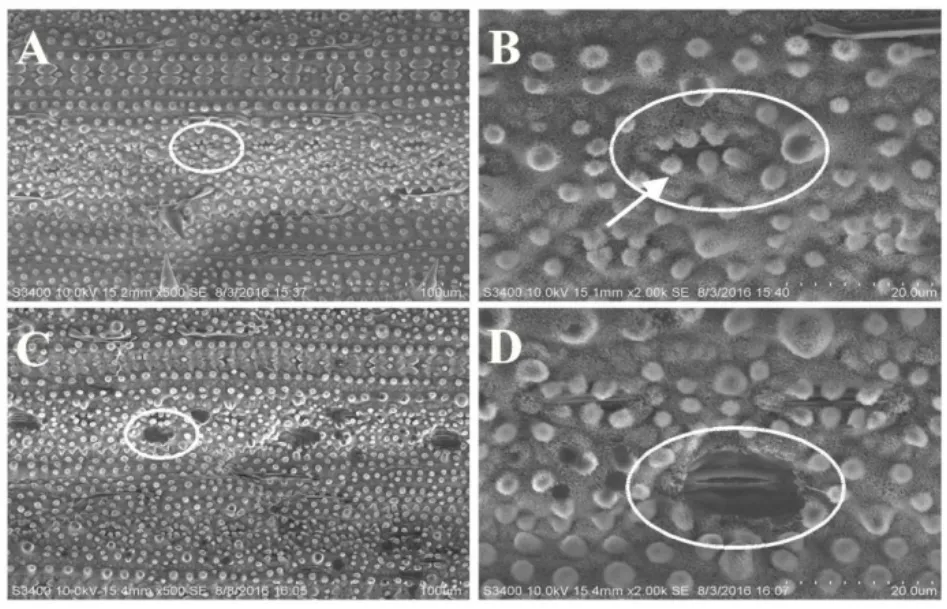
图3 分蘖盛期野生型和突变体pgl11叶片气孔结构Fig. 3. Leaf stomatal structure of wild type(WT) and pgl11 at the tillering stage.
2.4 叶绿体结构的显微观察
为了进一步确定突变体pgl11叶绿体发育是否异常,我们分别对野生型与突变体pgl11的4叶期叶片进行透射电镜观察。结果表明,野生型叶肉细胞中叶绿体数目较多且体积较大,基质较浓厚,基粒片层垛叠正常,具有正常的片层结构(图4-A,B),而突变体pgl11中叶肉细胞中的叶绿体数目明显减少且体积减小,没有正常的基粒垛叠及成形的淀粉粒堆积,甚至有的细胞中没有叶绿体结构(图4-C,D)。该结果表明PGL11基因突变影响了水稻叶肉细胞中叶绿体结构的正常发育。
2.5 PGL11的遗传分析和基因定位
为了进一步明确突变体pgl11的遗传特性,将突变体pgl11分别与籼稻品种培矮64和南京6号进行杂交。结果表明,突变体pgl11与培矮64的F1为正常表型,F2分离出的正常表型和淡绿叶表型植株分别为413株和141株,经卡方检验符合3∶1(χ2=0.0602<;与南京6号杂交后,F1为正常表型,F2分离出正常表型和淡绿叶表型植株分别为504株和167株,经卡方检验符合3∶1(χ2=0.0045<(表4),表明突变体pgl11的淡绿叶表型受一对隐性核基因控制。
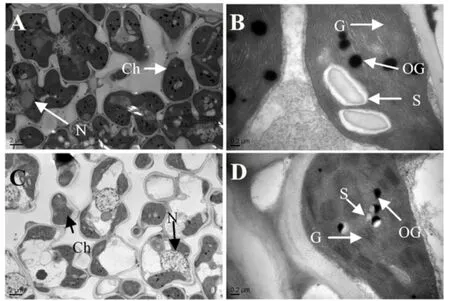
图4 野生型和突变体pgl11叶片叶绿体的超微结构. Fig. 4. Chloroplast ultrastructure of wild type and pgl11.
利用本实验室保存的均匀分布在水稻12条染色体上的232对SSR和STS标记对双亲和F1进行多态性分析,从中获得136对多态性的标记用于pgl11的初定位。根据连锁分析,将目标基因初步定位在第1染色体短臂上的M1和M2两个标记之间。为了对PGL11进行精细定位,我们扩大pgl11与南京6号的F2群体,并挑选出1 328株有突变表型的单株。根据水稻粳稻品种日本晴及籼稻品种93-11的序列差异,在M1与M2之间开发了多个在双亲具有多态性的标记(表5)。根据染色体步移的方法,最终将PGL11定位于标记C6和C8之间,物理距离约为110 kb的范围内,此区间包含2个BACs(AP000836和AP000837)(图5)。该范围内共有10个开放阅读框,包括8个编码假定蛋白、1个编码表达蛋白的基因及1个已被克隆的基因。已被克隆的基因为一个编码MYB结构域蛋白的CSA基因。
2.6 叶绿素合成、叶绿体发育及光合作用相关基因的表达
由于突变体叶片表现出明显的淡绿叶表型, 且色素含量及光合速率均下降,叶绿体结构也表现出异常。因此,叶绿素合成、光合作用及叶绿体发育相关基因的表达可能会受到一定程度的影响。利用实时荧光定量PCR对突变体pgl11及野生型中与叶绿素合成相关基因HEMA1(谷氨酰-tRNA还原酶)、CHLH(镁离子螯合酶)、PORA(NADPH-原叶绿素酸酯氧化还原酶a)、PORB(NADPH-原叶绿素酸酯氧化还原酶b)、CAO1(叶绿素酸酯氧化酶)、光合作用相关基因LhcP2(捕光复合蛋白)、LhcB1/B4(叶绿素a/b结合光捕获蛋白)、叶绿体发育过程相关基因PsaA(光系统Ⅰ叶绿素脱辅基蛋白)、PsbA(光系统Ⅱ醌结合蛋白)、Rps15(核糖体小亚基蛋白15)、OsSig2A(质体RNA聚合酶)的表达水平进行测定(图6)。结果表明,与野生型相比,突变体的叶绿素代谢相关基因CHLH和HEMA的表达量明显上升,而PORA、PORB及CAO1的表达水平显著下降。光合作用相关基因LhcB1、LhcP2的表达量无明显变化,而LhcB4的表达量则显著降低。叶绿体发育相关基因PsaA、PsbA的表达量显著降低。与叶绿体转录/翻译相关基因Rps15和OsSig2A的表达量则显著上升。这些结果表明,PGL11的突变导致与叶绿素合成、光合作用以及叶绿体发育相关基因的表达发生明显改变。
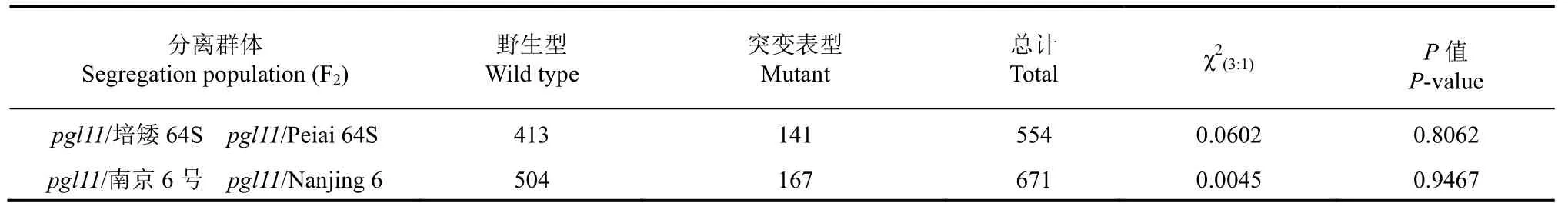
表4 突变体基因PGL11的遗传分析Table 4.Genetic analysis of PGL11.
表5 本研究中精细定位所用引物Table 5.Primers used for fine mapping in the study.
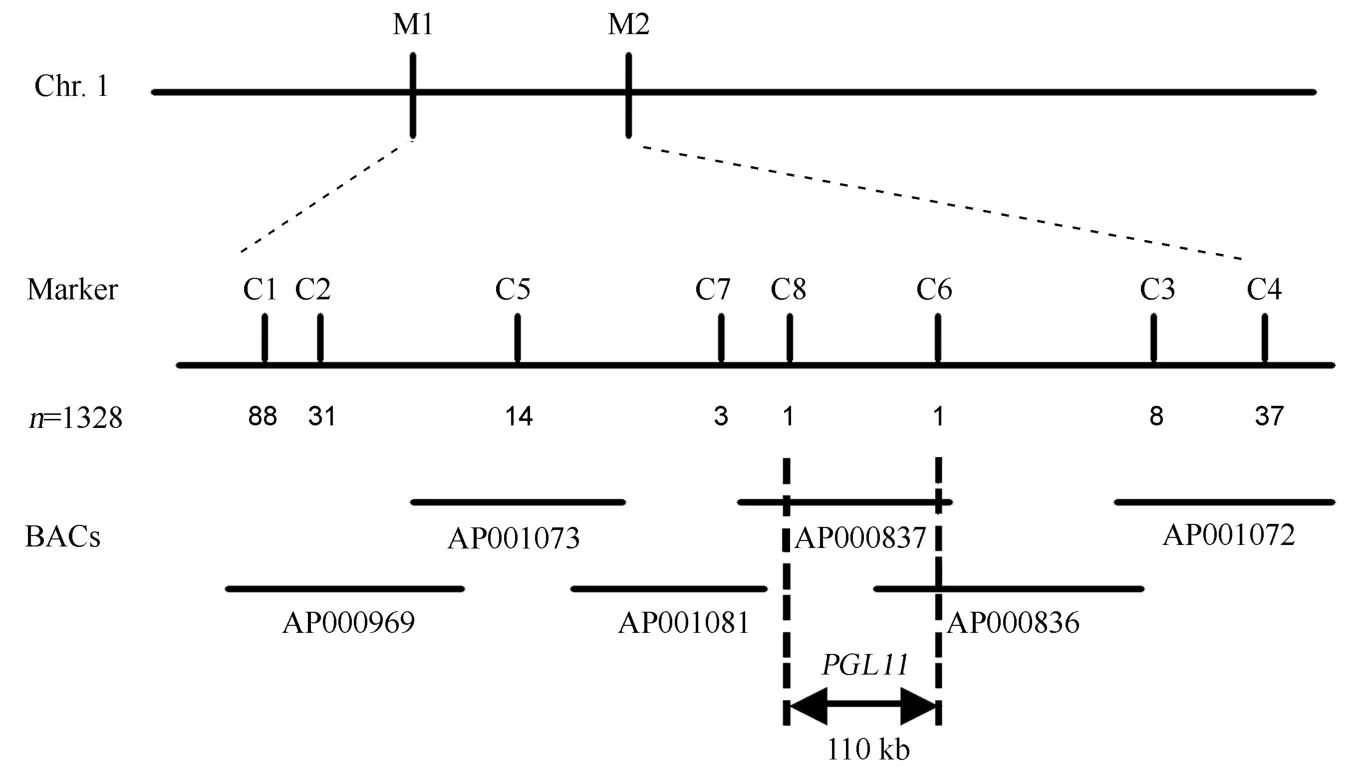
图5 PGL11基因的分子定位Fig. 5. Molecular mapping of PGL11.
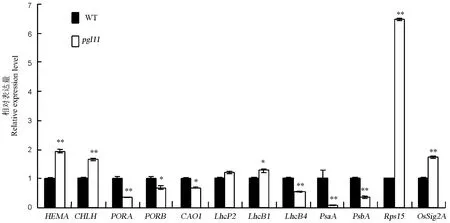
图6 野生型(WT)与突变体pgl11中叶绿素合成、光合作用及叶绿体发育相关基因的表达Fig. 6. Expression analysis of genes associated with chlorophyll biosynthesis, photosynthesis and chloroplast development in the wild type(WT) and pgl11 mutant.
3 讨论
水稻是一种重要的单子叶模式植物,随着水稻全基因组测序的完成,通过水稻叶色突变体来研究水稻的功能基因组学越来越受到大家的关注。叶色突变体是水稻突变体中十分常见且容易通过观察辨别的突变类型,是研究水稻叶绿体功能、叶绿素代谢及高光效育种的重要的遗传材料。在已经报道的水稻叶色突变体中,存在着许多转绿类型的突变体。如突变体v4在3叶期表现出白化表型,叶绿体发育异常,4叶期后,叶片则逐渐变绿[44]。白化突变体ysa在3叶期前生长白化叶,之后叶片逐渐变绿,直到6叶期叶片完全恢复成正常绿色[24]。YGL1基因编码叶绿素合成酶,水稻黄绿叶突变体ygl1生长发育前期叶片为黄色,中期叶色开始转绿,后期叶色则接近野生型[21]。与以上突变体表型相似,本研究的pgl11突变体也是一个典型的水稻转绿类型的突变体。生长前期,叶片淡绿甚至黄化,中期叶片逐渐转绿,后期叶片恢复为正常绿色。
基因PGL11定位于水稻第1染色体上,而在前人的研究中,水稻第1染色体上已克隆的叶色基因只有4个,分别是NYC1[45]、WLP1[46]、YGL8[47]和OsHAP3A[48]。NYC1编码叶绿素b还原酶基因。NYC1的突变造成叶绿素降解途径受抑制,突变体nyc1的叶片在衰老时仍然保持绿色。WLP1编码一个叶绿体核糖体L13蛋白,是叶绿体核糖体的主要组成成分。低温条件下,wlp1突变体叶绿素合成受阻,在4叶期前叶片白化,4叶期以后转为绿色,且wlp1的株高较野生型更高。YGL8基因编码镁原卟啉Ⅸ单酯环化酶(MgPME)的一个催化亚基。ygl8突变体在幼苗期、分蘖期和抽穗期均表现出明显的黄绿叶表型,叶片中叶绿素含量降低,生长后期叶片逐渐转绿,抽穗期迟滞,分蘖数、千粒重与结实率等产量性状显著降低。OsHAP3A编码一个CAAAT-box结合复合体HAP3亚基。反义表达OsHAP3A的植株叶绿体发育异常,叶绿素含量降低,叶绿体降解,叶片出现浅绿色表型。其中只有ygl8突变体在整个生育期的表型与pgl11较为相似,但二者在染色体上的位置完全不同。进一步将基因PGL11定位在标记C6与C8之间,遗传距离约为110 kb。该范围内共含有10个开放阅读框。其中已被克隆的基因为一个编码MYB结构域蛋白的CSA基因[49]。CSA是一个在水稻雄性生殖发育过程中调控糖分配的重要的转录因子,该基因突变最终导致植株雄性不育。另外,LOC_Os01g16730编码一个C端催化域包含蛋白质,该蛋白属于类泛素(ulp1)蛋白酶家族,主要参与细胞内蛋白质的降解;LOC_Os01g16750预测为编码一个黄素单氧酶,参与水稻IAA的生物合成;LOC_Os01g16770预测编码一个扩增前体蛋白,参与细胞的扩增;LOC_Os01g16840预测编码一个微线蛋白sm70;LOC_Os01g16800编码一个表达蛋白;另外4个为编码转座蛋白的基因。在前人的研究中,在该区间内尚未报道过与控制pgl11突变表型相似的基因,这进一步说明了PGL11可能是一个控制水稻叶色的新基因。
本研究的突变体材料pgl11的叶绿体结构异常,结合其叶色的变化,推测可能是由于其叶绿体发育过程与野生型不同,突变体的叶绿体可能经历了前质体到白色体再到叶绿体这样一个较长的发育过程[50],所以突变体在生长前期,叶绿体发育缓慢、滞后。到生长后期,叶绿体逐渐发育完全,叶色开始转绿。同时我们在转录水平分析突变基因对植株的影响。RT-PCR结果表明,叶绿素合成途径的前期参与基因HEMA、CHLH的表达量上升,而合成后期的基因PORA、PORB、CAO1的表达量则显著下降,产生这一结果的原因可能是基因突变引起了这个过程的某一个酶的活性改变,从而影响了叶绿素合成的进程,这与叶绿素含量的测定值相吻合。质体内存在两种类型的RNA酶:质体编码的RNA聚合酶(PEP)与核编码的RNA聚合酶(NEP)。核基因OsSig2A编码质体RNA聚合酶的一个调控亚基,参与质体的转录调控。质体基因Rps15编码叶绿体核糖体S15小亚基,负责质体翻译过程中的识别功能。结果表明,基因OsSig2A与rps15的表达水平显著上升,特别是基因rps15,其表达水平增加了约5.4倍,表明基因PGL11的突变影响质体的转录和翻译过程。叶绿体编码的基因PsaA、PsbA、LhcB1/B4及LhcP2与叶绿体的光合器官形成有关。这些基因的表达能够激活叶绿体内光合器官的光合作用能力,促进叶绿体的发育成熟。除LhcB1表达水平未发生明显变化外,其他4个基因的表达水平均显著降低,这也很好地解释了突变体叶绿体发育的异常及光合速率的下降。
突变体pgl11不仅有叶色的变化,也有株高、剑叶宽、二次枝梗数、每穗粒数、粒宽、千粒重以及结实率等性状的变化,说明基因PGL11的突变是一因多效的。pgl11植株的抽穗期较野生型晚约一周,可能是由于基因突变使水稻的叶色变浅,叶绿体发育异常,光合速率下降,导致源器官的能量供给不足,从而引起抽穗期的延后。气孔是植物体与外部环境进行水和气体交换的重要通道,能够维持植物体的温度、代谢稳定及光合作用的正常进行。突变体pgl11叶片的气孔形态与结构异常,局部的气孔出现成片的下陷,这些气孔周围没有任何的乳突结构。气孔的发育直接影响叶片中的碳同化进程,这可能是其光合作用下降的重要原因。本研究的突变体材料可为研究水稻叶片叶绿素合成、叶绿体发育及提高水稻光合效率与产量提供重要的种质资源,同时为该基因的最终克隆提供参考。
[1] Zhen X H, Xu J G, Shen W J, Zhang X J, Zhang Q J, Lu C G, Chen G X, Gao Z P. Photosynthetic characteristics of flag leaves in rice white stripe mutant 6001 during senescence process. Rice Sci, 2014, 21(6): 335-342.
[2] Tanaka A, Tanaka R. Chlorophyll metabolism. Curr Opin Plant Biol, 2006, 9(3): 248-255.
[3] Fromme P, Melkozernov A, Jordan P, Krauss N. Structure and function of photosystem: Ⅰ. Interaction with its soluble electron carriers and external antenna systems. FEBS Lett, 2003, 555: 40-44.
[4] Huang J L, Qin F, Zang G C, Kang Z H, Zou H Y, Hu F, Yue C L, Li X Y, Wang G X. Mutation of OsDET1 increases chlorophyll content in rice. Plant Sci, 2013, 210: 241-249.
[5] Bansal U, Saini R, Kaur A. Genetic variability in leaf area and chlorophyll content of aromatic rice. Int Rice Res Notes, 1999, 24: 21.
[6] Mitchell P L, Sheehy J E. Supercharging rice photosynthesis to increase yield. New Phytol, 2006, 171: 688-693.
[7] Gustafsson Å. The plastid development in various types of chlorophyll mutations. Hereditas, 2010, 28(3-4): 483-492.
[8] Kusumi K, Mizutani A, Nishimura M, Iba K. A virescent gene V1 determines the expression timing of plastid genes for transcription/translation apparatus during early leaf development in rice. Plant J, 1997, 12(6): 1241-1250.
[9] Sugimoto H, Kusumi K, Tozawa Y, Yazaki J, Kishimoto N, Kikuchi S, Iba K. The virescent-2 mutation inhibits translation of plastid transcripts for the plastid genetic system at an early stage of chloroplast differentiation. Plant Cell Physiol, 2004, 45(8): 985-996.
[10] Nakanishi H, Nozue H, Suzuki K, Kaneko Y, Taguchi G, Hayashida N. Characterization of the Arabidopsis thaliana mutant pcb2 which accumulates divinyl chlorophylls. Plant Cell Physiol, 2005, 46(3): 467-473.
[11] Roussell D L, Thompson D L, Pallardy S G, Miles D, Newton K J. Chloroplast structure and function is altered in the NCS2 maize mitochondrial mutant. Plant Physiol, 1991, 96: 232-238.
[12] Alberte R S, Hesketh J D, Hofstea G, Thornber J P, Naylor A W, Bernard R L , Brim C , Endrizzi J, Kohel R J. Composition and activity ot the photosynthetic apparatus in temperature-sensitive mutants of higher plants. Proc Natl Acad Sci USA, 1974, 71(6): 2414-2418.
[13] Guardi M T, Kucera T, Briantais J M, Hodges M. Decreased photosystemⅡ core phosphorylation in a yellow-green mutant of wheat showing monophasic fluorescence induction curve. Plant Physiol, 1995, 109: 1059-1068.
[14] 肖华贵, 杨焕文, 饶勇, 杨斌, 朱英, 张文龙. 甘蓝型油菜黄化突变体的叶绿体超微结构、气孔特征参数及光合特性. 中国农业科学, 2013, 46(4): 715-727. Xiao H G, Yang H W, Rao Y, Yang B, Zhu Y, Zhang W L. Analysis of chloroplast ultrastructure, stomatal characteristic parameters and photosynthetic characteristics of chlorophyll-reduced mutant in Brassica napus L. Sci Agric Sin, 2013, 4: 715-727. (in Chinese with English abstract)
[15] Pérezruiz J M, Spínola M C, Kirchsteiger K, Moreno J, Sahrawy M, Cejudo F J. Rice NTRC is a high-efficiency redox system for chloroplast protection against oxidative damage. Plant Cell, 2006, 18(9): 2356-2368.
[16] Tsugane K, Maekawa M, Takagi K, Takahara H, Qian Q, Eun C H, Iida S. An active DNA transposon nDart causing leaf variegation and mutable dwarfism and its related elements in rice. Plant J, 2006, 45(1): 46-57.
[17] Jiang H W, Li M R, Liang N T, Yan H B, Wei Y B, Xu X L, Liu J, Xu Z F, Chen F, Wu G J. Molecular cloning and function analysis of the stay green gene in rice. Plant J, 2007, 52(2): 197-209.
[18] Wang P R, Gao J X, Wan C M, Zhang F T, Xu Z J, Huang X Q, Sun X Q, Deng X J. Divinyl chlorophyll(ide) a can be converted to monovinyl chlorophyll(ide) a by a divinyl reductase in rice. Plant Physiol, 2010, 153(3): 994-1003.
[19] Goh C H, Satoh K, Kikuchi S, Kim S C,Ko S M, Kang H G, Jeon J S, Kim C S, Park Y. Mitochondrial activity in illuminated leaves of chlorophyll-deficient mutant rice (OsCHLH) seedlings. Plant Biotechnol Rep, 2010, 4(4): 281-291.
[20] Zhang H T, Li J J, Yoo J H, Yoo S C, Cho S H, Koh H J, Seo H S, Paek N C. Rice Chlorina-1 and Chlorina-9 encode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development. Plant Mol Biol, 2006, 62(3): 325-337.
[21] Wu Z M, Zhang X, He B, Diao L P, Sheng S L, Wang J L, Guo X P, Su N, Wang L F, Jiang L, Wang C M, Zhai H Q, Wan J M. A Chlorophyll-deficient rice mutant with impaired chlorophyllide esterification in chlorophyll biosynthesis. Plant Physiol, 2007, 145(1): 29-40.
[22] Lee S, Kim J H, Yoo E S, Lee C H, Hirochika H, An G. Differential regulation of chlorophyll a oxygenase genes in rice. Plant Mol Biol, 2005, 57(6): 805-818.
[23] Yang Y L, Xu J, Huang L C, Leng Y J, Dai L P, Rao Y C, Chen L, Wang Y Q, Tu Z J, Hu J, Ren D Y, Zhang G H, Zhu L, Guo L B, Qian Q, Zeng D L. PGL, encodingchlorophyllide a oxygenase 1, impacts leaf senescence and indirectly affects grain yield and quality in rice. J Exp Bot, 2016, 67(5): 1297-1310.
[24] Su N, Hu M L, Wu D X, Wu F Q, Fei G L, Lan Y, Chen X L, Shu X L, Zhang X, Guo X P, Cheng Z J, Lei C L, Qi C K, Jiang L, Wang H Y, Wan J M. Disruption of a rice pentatricopeptide repeat protein causes a seedlingspecific albino phenotype and its utilization to enhance seed purity in hybrid rice production. Plant Physiol, 2012, 159(1): 227-238.
[25] Kusumi K, Yara A, Mitsui N, Tozawa Y, Iba K. Characterization of a rice nuclear-encoded plastid RNA polymerase gene OsRpoTp. Plant Cell Physiol, 2004, 45: 1194-1201.
[26] Zhao C F, Xu J M, Chen Y, Mao C Z, Zhang S L, Bai Y H, Jiang D A, Wu P. Molecular cloning and characterization of OsCHR4, a rice chromatinremodeling factor required for early chloroplast development in adaxial mesophyll. Planta, 2012, 236(4): 1165-1176.
[27] Kodiveri M G, Kim E S, Cho H, Chung Y Y, Kodiveri M G. OsPPR1, a pentatricopeptide repeat protein of rice is essential for the chloroplast biogenesis. Plant Mol Biol, 2005, 58(3): 421-433.
[28] Fang J, Chai C L, Qian Q, Li C L, Tang J Y, Sun L, Huang Z J, Guo X L, Sun C H, Liu M, Zhang Y, Lu Q T, Wang Y Q, Lu C M, Han B, Chen F, Cheng Z K, Chu C C. Mutations of genes in synthesis of the carotenoid precursors of ABA lead to pre-harvest sprouting and photo-oxidation in rice. Plant J, 2008, 54(2): 177-189.
[29] 钱前, 朱旭东, 曾大力, 张小惠, 严学强, 熊振民. 细胞质基因控制的新特异材料白绿苗的研究. 作物品种资源, 1996(4): 11-12. Qian Q, Zhu X D, Zeng D L, Zhang X H, Yan X Q, Xiong Z M. Study on the new special material white-green seedlings controlled by cytoplasmic gene. China Seeds, 1996(4): 11-12. (in Chinese with English abstract)
[30] Wang F H, Wang G X, Li X Y, Huang J, Zheng J. Geredity, physiology and mapping of a chlorophyll content gene of rice(Oryza sativa L.). Plant Physiol, 2008, 165: 324-330.
[31] 谢戎, 朱发云, 何光华, 邓锡洪, 左永树, 杨正林, 吴丽君. 水稻两用系温敏叶绿素自然突变体的初步研究.西南农业学报, 1995, 8: 124-128. Xie R, Zhu F Y, Deng X H, Zuo Y S, Yang Z L, Wu L J.A preliminary study on the temperature sensitive chlorophyll natural mutant in dual-purpose genic male sterile rice. Southwest China J Agric Sci, 1995, 8: 124-128.
[32] Huq E, Al-Sady B, Hudson M, Kim C, Apel K, Quail P H. Phytochrome-interacting factor 1 is a critical bHLH regulator of chlorophyll biosynthesis. Science, 2004, 305: 1937-1941.
[33] Terry M J, Kendrick R E. Feedback inhibition of chlorophyll synthesis in the phytochrome chromophoredeficient aurea and yellow-green-2 mutants of tomato. Plant Physiol, 1999, 119: 143-152.
[34] Beale S I. Green genes gleaned. Trends Plant Sci, 2005, 10: 309-312.
[35] Nagata N, Tanaka R, Satoh S, Tanaka A. Identification of a vinyl reductase gene for chlorophyll synthesis in Arabidopsis thaliana and implications for the evolution of Prochlorococcus species. Plant Cell, 2005, 17(1): 233-240.
[36] Sakuraba Y, Rahman M L, Cho S H, Kim Y S, Koh H J, Yoo S C, Paek N C. The rice faded green leaf locus encodes protochlorophyllide oxidoreductase B and is essential for chlorophyll synthesis under high light conditions. Plant J, 2013, 74(1): 122-133.
[37] Yang Q S, He H, Li H Y, Tian H, Zhang J J, Zhai L G, Chen J D, Wu H, Yi G J, He Z H, Peng X X. NOA1 functions in a temperature-dependent manner to regulate chlorophyll biosynthesis and rubisco formation in rice. PLoS ONE, 2011, 6(5): e20015.
[38] Sugimoto H, Kusumi K, Noguchi K, Yano M, Yoshimura A, Iba K. The rice nuclear gene, VIRESCENT 2, is essential for chloroplast development and encodes a novel type of guanylate kinase targeted to plastids and mitochondria. Plant J, 2007, 52(3): 512-527.
[39] Yoo S C, Cho S H, Sugimoto H, Li J J, Kusumi K, Koh H J, Iba K, Paek N C. Rice virescent 3 and stripe 1 encoding the large and small subunits of ribonucleotide reductase are required for chloroplast biogenesis during early leaf development. Plant Physiol, 2009, 150(1): 388-401.
[40] 苏正淑, 张宪政. 几种测定植物叶绿素含量的方法比较. 植物生理学通讯, 1989(5): 77-78. Su Z S, Zhang X Z. Comparison of several methods for determining chlorophyll content of plants. Plant Physiol Commun, 1989(5): 77-78. (in Chinese with English abstract)
[41] Lichtenthaler H K. Chlorophylls and caroteniods: Pigments of photosynthetic biomembranes. Method Enzymol, 1987, 148: 350-382.
[42] 李超, 林冬枝, 董彦君, 叶胜海, 张小明. 一个水稻苗期温敏感白色条斑叶突变体的遗传分析及基因定位.中国水稻科学, 2010, 24(3): 223-227. Li C, Lin D Z, Dong Y J, Ye S H, Zhang X M. Genetic analysis and mapping of a thermo-sensitive white stripe leaf mutant at the seedling stage in rice(Oryza sativa). Chin J Rice Sci, 2010, 24(3): 223-227. (in Chinese with English abstract)
[43] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2001, 25: 402-408.
[44] Gong X D, Su Q Q, Lin D Z, Jiang Q, Xu J L, Zhang J H, Teng S, Dong Y J. The rice OsV4 encoding a novel pentatricopeptide repeat protein is required for chloroplast development during the early leaf stage under cold stress. J Integr Plant Biol, 2014, 56(4): 400-410.
[45] Kusaba M, Ito H, Morita R, Iida S, Sato Y, Fujimoto M, Kawasaki S, Tanaka R, Hirochika H, Nishimura M, Tanaka A. Rice NON-YELLOW COLORING1 is involved in light-harvesting complex II and grana degradation during leaf senescence. Plant Cell, 2007, 19(4): 1362-1375.
[46] Song J, Wei X J, Shao G N, Sheng Z H, Chen D B, Liu C L, Jiao G A, Xie L H, Tang S Q, Hu P S. The rice nuclear gene WLP1 encoding a chloroplast ribosome L13 protein is needed for chloroplast development in rice grown under low temperature conditions. Plant Mol Biol, 2014, 84(3): 301-314.
[47] Kong W Y, Yu X W, Chen H Y, Liu L L, Xiao Y J, Wang Y L, Wang C L, Lin Y, Yu Y, Wang C M, Jiang L, Zhai H Q, Zhao Z G, Wan J M. The catalytic subunit of magnesium-protoporphyrin IX monomethyl ester cyclase forms a chloroplast complex to regulate chlorophyll biosynthesis in rice. Plant Mol Biol, 2016, 92(1): 177-191.
[48] Miyoshi K, Ito Y, Serizawa A, Kurata N. OsHAP3 genes regulate chloroplast biogenesis in rice. Plant J, 2003, 36(4): 532-540.
[49] Zhu X L, Liang W Q, Cui X, Chen M J, Yin C S, Luo Z J, Zhu J Y, Lucas W J, Wang Z Y, Zhang D B. Brassinosteroids promote development of rice pollen grains and seeds by triggering expression of carbon starved anther, a MYB domain protein. Plant J, 2015, 82(4): 570-581
[50] 武立权, 尤翠翠, 柯建, 何清华. 叶色白化水稻突变体转绿中若干生理与叶绿体发育特型的研究. 热带作物学报, 2013, 34(6): 1115-1120. Wu L Q, You C C, Ke J, He Q H. Chloroplast development and physiological characteristics of green-revertible albino leaf color mutants in rice. Chin J Trop Crops, 2013, 34(6): 1115-1120. (in Chinese with English abstract)
Identification and Fine Mapping of Pale Green Leaf PGL11 in Rice
TU Zhengjun1,#, ZOU Guoxing2,#, HUANG Lichao1, CHEN Long1, DAI Liping1, GAO Yihong1, LENG Yujia1, ZHU Li1, ZHANG Guangheng1, HU Jiang1, REN Deyong1, GAO Zhenyu1, DONG Guojun1, CHEN Guang1, GUO Longbiao1, QIAN Qian1,*, ZENG Dali1,*
(1State Key Laboratory for Rice Biology, China National Rice Research Institute, Hangzhou 310006, China;2Rice Research Institute, Jiangxi Academy of Agricultural Sciences, Nanchang 330200, China;#These authors contributed equally to this work;*Corresponding author, E-mail: dalizeng@126.com; qianqian188@hotmail.com)
【Objective】 Leaf is the main site of photosynthesis in rice. The change of leaf color is directly related to the growth and development of rice. The research of leaf color mutants is an important genetic basis for the study of rice functional genomics. 【Method】We identified a pale green leaf mutant termed pgl11(pale green leaf 11) from japonica cultivar Nipponbare by ethyl methylsulfonate (EMS) treatment. The chlorophyll content of wild type(WT) and mutant was measured at different growth stages. At the seedling stage, the chloroplast structure of the leaves of the WT and the mutant were observed with a transmission electron microscopy. At the tillering stage, the photosynthetic parameters of WT and pgl11 were measured and the stomatal structure was observed. At the mature stage, the main agronomic traits of WT and pgl11 were determined. F2population derived from pgl11/Nanjing 6 was used to map this gene by position cloning approach.【Result】At seedling stage, every new leaf of the mutant pgl11 was pale green. As the leaves matured, the leaf color gradually turned green. Compared with wild type, the chlorophyll content in pgl11 decreased at seedling stage. However, there was no significant difference at heading stage. In addition, the photosynthetic rate and stomatal conductance of pgl11 were significantly decreased, but the intercellular CO2concentration was apparently increased. The
stomata was abnormal in pgl11. The agronomic traits including plant height, flag leaf width, secondary rachis branch number, grain number per panicle, grain length, grain width, 1000-grain weight and seed setting rate decreased significantly in pgl11 compared with wild type. The expression of genes associated with chloroplast transcription and translation were upregulated in pgl11, while the expression of chlorophyll synthesis and photosynthesis related genes were downregulated. Genetic analysis showed that the mutant phenotype was controlled by a single recessive nuclear gene, and PGL11 was mapped to a 110kb region between the markers C6 and C8 on the short arm of chromosome 1. 【Conclusion】It was suggested that PGL11 gene would be a putative novel pale green leaf gene.
rice; pale green leaf; chloroplast; genetic analysis; gene mapping.
Q755; S511.03
A
1001-7216(2017)05-0489-11
2017-04-11; 修改稿收到日期:2017-05-12。
中央级公益性科研院所专项;中国农科院科技创新工程资助项目;国家自然科学基金资助项目(31661143006,91435105)。
