水稻着丝粒特异组蛋白CENH3抗体的制备与应用
2017-09-23薛超张融郭瑞刘帅刘晓宇沈明晨邓世峰龚志云
薛超 张融 郭瑞 刘帅 刘晓宇 沈明晨 邓世峰 龚志云
(扬州大学 农学院/江苏省作物遗传生理国家重点实验室培育点/江苏省粮食作物现代产业技术协同创新中心/教育部植物功能基因组学重点实验室,江苏 扬州 225009;*通讯联系人,E-mail: zygong@yzu.edu.cn)
水稻着丝粒特异组蛋白CENH3抗体的制备与应用
薛超 张融 郭瑞 刘帅 刘晓宇 沈明晨 邓世峰 龚志云*
(扬州大学 农学院/江苏省作物遗传生理国家重点实验室培育点/江苏省粮食作物现代产业技术协同创新中心/教育部植物功能基因组学重点实验室,江苏 扬州 225009;*通讯联系人,E-mail: zygong@yzu.edu.cn)
【目的】着丝粒是真核生物染色体的基本功能元件之一,其功能是在细胞有丝分裂和减数分裂时期精确地调控染色体配对和分离并维持染色体的稳定。着丝粒结构是由DNA和蛋白质形成的一种复合体。着丝粒特异组蛋白(centromere-specific histone H3, CENH3)是功能着丝粒是否具有活性的最基本特征。所以制备CENH3的相关抗体是进行着丝粒结构与功能研究的前提条件之一。【方法】通过设计短肽进行兔免疫实验,制备了水稻着丝粒特异组蛋白CENH3兔源抗体,利用ELISA和蛋白免疫荧光(immunofluorescence, IF)等检测方法对抗体有效性进行了鉴定。【结果】ELISA检测显示制备的CENH3抗体有效稀释度为1: 40万,并且蛋白免疫荧光信号在水稻体细胞每条染色体的着丝粒区域均能检测到。同时,该抗体也可以应用于玉米等其他物种。通过染色质免疫沉淀(ChIP)技术获得与CENH3相结合的DNA分子,并进行PCR扩增和FISH定位分析,结果显示相应的ChIP-DNA位于水稻功能着丝粒区域。【结论】本研究制备的水稻CENH3兔源抗体能满足着丝粒研究中相关实验的要求,可进一步应用于着丝粒的结构与功能研究。
水稻;着丝粒特异组蛋白;染色质免疫沉淀;蛋白免疫荧光
着丝粒是真核生物染色体中重要的结构与功能元件, 是由DNA和蛋白质组成的复合体, 是真核生物有丝分裂和减数分裂过程中染色体正常分离和传递所必需的染色体区域[1]。每条染色体必须拥有正常的功能着丝粒, 以维持物种染色体组成的稳定性。目前, 已在拟南芥、水稻、玉米等植物的着丝粒进行了较深入的分子生物学研究,都表明了着丝粒的功能非常保守, 但真核生物各物种的着丝粒DNA序列却是高度变异的[2-4]。着丝粒DNA序列进化速率极高, 在不同物种中, 组成着丝粒的DNA序列是没有同源性的[5]。除芽殖酵母的着丝粒以外[6-7], 大多数生物的着丝粒均由高度重复的DNA序列构成, 并且在这些重复序列会间或插入一些逆转座子成分, 一起组成着丝粒特异DNA序列[8-12]。
相比之下, 着丝粒区域另外一个重要组成成分——着丝粒特异蛋白在真核生物中则相对保守。其中, 着丝粒特异组蛋白H3(centromere-specific histone H3, CENH3)是较早发现的一种重要蛋白,存在于真核生物活性着丝粒的核小体中, 是着丝粒形成的早期标志[13-15]。第一个鉴定出的CENH3是人类活性着丝粒上的CENP-A蛋白[16]。CENH3是功能着丝粒染色质最基本的特征, 与CENH3相连着丝粒染色质只包含一小部分的卫星DNA[17]。比如, 在栽培稻第8染色体的着丝粒中,与CENH3结合的卫星DNA(CentO)约为750 kb,即其功能着丝粒的大小约为750 kb。CENH3在着丝粒染色质的识别和保持中起关键作用[4], 决定动粒组装的位置和着丝粒的活性, 而且仅存在有活性的着丝粒中[18-23]。研究表明, 在果蝇和老鼠中, 当CENH3被破坏或者缺失时, 染色体不能正常传递[24-25]。在植物中,不同染色体着丝粒区域的DNA序列虽然差异很大,但是与CENH3结合的区域大小是一致的。如水稻中,通过CENH3的蛋白免疫荧光检测,12条染色体着丝粒区域的CENH3信号强度是一致的[26]。此外,在拟南芥、玉米和甘蔗等植物中也鉴定出CENH3,几乎存在于整个细胞周期中[15,27-28]。虽然着丝粒的功能很保守,但不同于H3的保守性,CENH3的快速进化导致不同物种之间具有一定的特异性。因此,对CENH3进行研究具有重要的意义。
真核生物的基因组DNA以染色质的形式存在。因此, 研究蛋白质与DNA在染色质环境下的相互关系是阐明真核生物基因表达机制的基本途径。染色质免疫沉淀技术(chromatin immunoprecipitation assay, ChIP)是目前研究体内DNA与蛋白质相互作用的主要方法之一, 其基本原理是在活细胞状态下固定蛋白质-DNA复合物, 并将其随机切断为一定长度范围内的染色质小片段,然后通过免疫学方法沉淀此复合体, 特异性地富集目的蛋白结合的DNA片段, 通过对目的片断的纯化与检测, 从而获得蛋白质与DNA相互作用的信息[29-33]。通过用CENH3抗体进行染色质免疫沉淀(ChIP)实验, 可以确定所检测的DNA序列是否属于着丝粒的功能DNA元件, 同时对于CENH3抗体发生免疫沉淀的染色质进行克隆与分析, 可获得功能着丝粒DNA序列[34]。ChIP实验表明, 拟南芥、玉米、水稻和甘蔗等植物通过卫星DNA和反转录转座子元件能与CENH3发生相互作用, 说明卫星DNA和反转录转座子元件CR(centromere- specific retrotransposon)是这些物种着丝粒的功能组成成分[35]。
水稻是世界上最重要的粮食作物之一, 同时也是单子叶植物的模式生物。近年来随着水稻全基因组测序的完成, 对水稻各染色体组中不同染色体着丝粒的组成也进行了深入分析。 其中, 栽培稻的着丝粒由串联重复序列组成。 重复单元为155 bp的CentO。目前, 定位分析水稻着丝粒的方法除了对CentO的荧光原位杂交(fluorescence in situ hybridization, FISH)分析外, 还可以利用CENH3抗体进行蛋白免疫分析。通过与水稻CENH3结合的蛋白免疫反应研究表明, 水稻每一着丝粒结合的CENH3是十分相近的, 说明不同染色体着丝粒功能DNA结合区域是特定的[26]。目前水稻CENH3抗体商品化产品很少,即使有也由于其特定的使用范围,价格比较昂贵,所以相关研究中的大多数抗体由国外实验室赠送,但一般情况下获得的途径比较麻烦,因此制备CENH3抗体对进一步开展水稻或其他物种着丝粒的相关研究具有重要意义。
本研究对水稻着丝粒特异组蛋白CENH3抗体进行制备, 并利用ELISA方法对抗体的效价进行测定, 通过蛋白免疫荧光进一步验证该抗体的有效性。利用ChIP技术分析CENH3抗体, 对沉淀下来的DNA进行PCR和FISH分析, 以明确该抗体应用于水稻着丝粒研究的可能性, 并且对该抗体是否能应用于其他物种进行了鉴定,为进一步利用和推广该抗体开展着丝粒相关研究奠定了实验基础。
1 材料与方法
1.1 试验材料
试验材料包括粳稻品种日本晴(Oryza sativa japonica, Nipponbare)、普通玉米(Zea mays)、小麦(Triticum aestivum)和大麦(Hordeum vulgare)种子。
1.2 水稻CENH3抗体的制备
本研究中通过人工设计合成CENH3的相关多肽, 对兔子进行7次免疫后, 取兔子全血进行纯化。得到的抗体通过ELISA方法检测效价。抗体的效价为大于NC值2倍且大于0.25时的OD450值, 当效价大于2时, 我们认为抗体是呈阳性的。ELISA方法检测具体步骤如下:
取纯化的CENH3蛋白以浓度为1 μg/mL包被酶标板, 每孔200 μg, 盖上盖子放入37℃温箱温育2 h。然后取出包被和封闭好的酶标板, 将封闭液甩入水池, 用自来水冲洗2次, 在毛巾上拍干6次。选用HIS抗体(用牛奶稀释, 1 mg/mL HIS抗体与牛奶的体积比为1∶8000)作为阳性对照, 阴性对照为5%牛奶,并进行稀释(稀释度分别为3125, 6250, 1.25万, 2.5万, 5万, 10万, 20万, 40万, 80万), 每孔100 μg, 盖上盖子放入37℃温箱温育1 h后, 取出酶标板, 将板内的一抗甩入水池, 自来水冲洗8遍, 拍干。将稀释好的二抗以每孔100 μg的量依次加入酶标板孔中, 盖上盖子放入37℃温箱温育45 min。从温箱取出二抗温育好的酶标板, 甩去二抗, 用自来水冲洗10次, 在吸水毛巾上拍干。将准备好的显色液按每孔100 μg的量依次加入酶标板孔中, 盖上盖子放入温箱,15 min后从温箱取出酶标板, 将2 mol/L硫酸以每孔50 μg的量依次加入酶标板孔中终止反应, 读取吸光值。
1.3 蛋白免疫荧光检测
根尖染色体制备: 采集新鲜根尖, 用4%多聚甲醛(w/v)室温固定30~40 min, 然后用1×磷酸盐缓冲液(8.0 g NaCl, 0.2 g KH2PO4, 2.9 g Na2HPO4, 0.2 g KCl, 双蒸水定容至1 L,调pH至7.4)清洗3次, 并保存在1×磷酸盐缓冲液中, 于4℃冰箱中保存备用。从磷酸盐缓冲液中取出准备好的根尖材料, 取1 mm左右根尖于载玻片上, 加1滴磷酸盐缓冲液, 加盖玻片压片, 压好的载玻片迅速放于液氮中, 冷冻1~2 min, 用刀片迅速揭去盖玻片, 晾干。
蛋白免疫荧光(immunofluorescence,IF)检测:向染色体制备好的载玻片上滴加30 μL含有R(兔源)-CENH3抗体的1×TNB溶液(0.1 mol/L Tris-HCl, pH 7.5; 0.15 mol/L NaCl; 0.5%阻断剂), 盖上盖玻片,放入湿盒内于37℃下保温4 h以上; 取出后用1×磷酸缓冲液洗片3次, 每次5 min, 加50 μL含有anti-Rabbit-488抗体(Invitrogen)的1×TNB溶液, 放入湿盒内于37℃避光保温1 h; 1×磷酸盐缓冲液洗片3次, 每次5 min, 晾干后, 加10 μL含有DAPI的抗褪色剂(Vector Laboratories, Inc.), 盖上盖玻片, 在荧光显微镜下观察拍照。
1.4 染色质免疫沉淀
1.4.1 细胞核的提取
染色体免疫共沉淀(ChIP)实验参照Nagaki等的实验方法[35]并有所优化。取8~10 d的日本晴幼苗叶片在液氮中研磨成粉状, 加等体积的抽提缓冲液M1(4 mL 0.1 mol/L磷酸钾, pH 7.0, 4 mL 1 mol/L NaCl, 31.25 μL β-巯基乙醇, 4.74 mL己二醇, 加ddH2O至40 mL)后放置在冰上, 于摇床上缓慢摇5 min。用灭菌后的Miracloth(EMD Chemicals, Inc.)过滤, 滤液在2410 r/min、4℃下离心10 min,去上清。加10 mL抽提缓冲液M2(3 mL 0.1 mol/L磷酸钾, pH 7.0, 3 mL 1 mol/L NaCl, 300 μL 1 mol/L氯化镁, 23.43 μL β-巯基乙醇, 3.54 mL己二醇, 130 μL Triton X-100, 加ddH2O至30 mL)混匀, 2410 r/min、4℃下离心10 min,去上清, 反复操作3次, 如沉淀中有杂质, 再重复上述操作。
1.4.2 细胞破碎
先加5 mL MNB缓冲液(2 mL 50%蔗糖, 0.5 mL 1 mol/L Tris-HCl, pH7.5, 40 μL 1 mol/L MgCl2, 10 μL 1 mol/L CaCl2, 加ddH2O至10 mL)对细胞核进行清洗, 于2410 r/min、4℃下离心10 min, 去上清。再加4 mL MNB缓冲液混匀, 分装成3管,每管1200 μL, 剩下的作为input, 在分装的3管中分别加1.5 U的MNase(Sigma)对DNA片段进行切割, 于37℃水浴锅中水浴10 min, 每2 min摇1次。加15 μL EDTA终止反应。于13 000 r/min、4℃下离心10 min,取上清, 用琼脂糖凝胶电泳检测切割结果。切割后的DNA片段大小约147 kb。1.4.3 染色体免疫共沉淀
加入特异性抗体CENH3及温育缓冲液[1 mL 1 mol/L NaCl, 400 μL 1 mol/L Tris-HCl, pH 7.5, 200 μL 0.5 mol/L EDTA, pH 8.0, 40 μL 0.1 mol/L PMSF, 200 μL蛋白酶抑制剂混合物(Complete Mini), 加ddH2O至20 mL], 密封、8℃下,上下颠倒孵育过夜, 然后再用Protein A beads(GE Healthcare)密封放置在8℃上下颠倒孵育2 h。通过不同浓度的盐溶液洗去与beads结合的非特异性染色质, 加400 μL 42℃预热的洗提缓冲液(500 μL 1 mol/L NaCl, 200 μL 1 mol/L Tris-HCl, pH 7.5, 100 μL 0.5 mol/L EDTA , pH 8.0, 500 μL 20% SDS, 加ddH2O补足至10 mL), 于65℃水浴锅水浴15 min, 每5 min摇1次, 于13 000 r/min、室温下离心5 min,取上清, 重复上次操作, 收集合并两次上清, 然后收集并纯化沉淀下来的ChIP-DNA。
1.4.4 ChIP-DNA的引物设计与PCR检测
设计的引物序列: CentO上游引物5′-TGC GATGTTTTCTACTGGAATC-3′; CentO下游引物5′-AAATCATGTTTTGGCTCTTTTT-3′。
以ChIP-DNA作为模板, 使用20 μL体系(Sangon Biotech)进行PCR扩增, 反应体系为: 1 μL ChIP-DNA模板, 2 μL 10×缓冲液(含Mg2+), 1.6 μL dNTPs(2.5 mmol/L), 上下游引物各2.5 μL, 0.2 μL Taq DNA聚合酶(5 U/μL), 加超纯水补足至20 μL。PCR扩增反应结束后取10 μL PCR产物经2%琼脂糖凝胶电泳, 凝胶成像系统观察电泳结果, 并拍照保存。
1.5 荧光原位杂交检测
1.5.1 根尖固定
取新鲜根尖置于2 mmol/L的8-羟基喹啉中,在20℃下处理2 h。用卡诺固定液(v甲醇∶v冰醋酸=3∶1)清洗3次后再用固定液室温固定24 h, 然后置于-20℃下备用。从固定液中取出固定好的根尖,用蒸馏水清洗3次。切取2 mm左右的白色根尖部位置于酶解液(2%纤维素酶与1%果胶酶), 37℃下酶解40 min。酶解完全的根尖经固定液清洗2次后, 用固定液再次固定5 min以上, 可置于-20℃下冷却待用。
1.5.2 染色体制备
将充分冰冻的根尖取出, 置于一张载玻片上,滴加适量固定液于根尖上。用镊子迅速敲碎根尖,再在载玻片上滴加少量固定液, 在酒精灯上点燃,使其充分燃烧, 斜置玻片晾干待用。
1.5.3 探针标记
把1 μg ChIP-DNA溶于16 μL双蒸水中。加入4 μL DIG-Nick-Translation Mix (Roche)混匀,离心。在15℃下反应90 min。停止反应后, 加入1 μL 0.5 mol/L EDTA(pH 8.0)终止反应。标记的探针-20℃下保存备用。
1.5.4 FISH检测
在制备好的载玻片上加入70%甲酰胺50 μL,盖上盖玻片。将玻片置于85℃的杂交炉中100 s。取出置于70%的冰酒精中, 脱色摇床5 min, 然后分别置于90%、100%酒精中脱色摇床各2 min, 取出晾干。配置杂交液(含ChIP-DNA探针)置于沸水中水浴5 min, 然后置于冰中5 min以上。每玻片滴加20 μL杂交液, 置于杂交盒中, 放入37℃恒温箱过夜处理。取出后用1×磷酸缓冲液脱色摇床3次, 每次5 min。FISH信号通过抗地高辛抗体偶联罗丹明偶联物(anti-digoxigenin-Rhodamine)检测, 染色体用DAPI反染。Olympus BX60镜检中获得清晰图像后, 以气冷式数码相机(CCD) (Olympus, DP80)摄像。
2 结果与分析
2.1 CENH3抗体的多肽合成与效价分析
根据NCBI的蛋白序列库得到CENH3的序列如下:
N′-MARTKHPAVRKSKAEPKKKLQFERSP RPSKAQRAGGGTGTSATTRSAAGTSASGTPR QQTKQRKPHRFRPGTVALREIRKFQKTTELLIP FAPFSRLVREITDFYSKDVSRWTLEALLALQE AAEYHLVDIFEVSNLCAIHAKRVTIMQKDMQ LARRIGGRRPW-C′。
设计近氨基端的14个氨基酸序列(AEPKKK LQFERSPR)进行多肽人工合成, 合成成功后利用该多肽进行兔免疫实验, 进行抗体制备。7次免疫后提取兔血清, 进行抗体效价的ELISA。由于HIS抗体分子量较小并且容易分离和纯化, 选其作为系统操作的阳性对照, 效价(即OD450值)为2.758;阴性对照为5%牛奶, 效价为0.075。血清抗体稀释度为3125, 6250, 1.25万, 2.5万, 5万, 10万, 20万, 40万, 80万时, 效价分别为2.491, 2.671, 2.758, 2.665, 2.652, 2.685, 2.457, 2.034, 1.084。可见,当血清抗体稀释度为1∶80万时, OD450值小于2, 该浓度下的抗体呈阴性, 抗体灵敏度较低; 在稀释度为1∶40万以内的效价都大于2, 抗体灵敏性较好。因此制备的血清抗体其有效的稀释度要控制在1∶40万以内。
2.2 CENH3蛋白免疫荧光分析
蛋白免疫荧光分析是检测着丝粒相关蛋白抗体是否有效的主要手段之一。通过观察信号在染色体上的位置和强弱来判断抗体的使用价值。
我们利用通过ELISA分析合格的CENH3抗体, 对水稻材料日本晴的根尖细胞进行蛋白免疫荧光检测(图1), 其中染色体用含DAPI抗褪色剂反染, 如红色区域所示; 绿色信号为CENH3蛋白信号, 在细胞有丝分裂间期、前期、中期和后期时期信号都明显可见。在有丝分裂间期看不出着丝粒区域所在位置, 但是染色质经碱性染料DAPI染色, 染色较深的区域是异染色质区域(图1-A1中箭头所示), 染色较浅的区域是常染色质区域, 而着丝粒就位于异染色质区域。可以看出, CENH3信号都出现在异染色质区域(图1-A3中箭头所示); 在有丝分裂前期(图1-B), 染色体形态分明, 可以清晰的辨识出染色体的主缢痕, 即着丝粒区域(图1-B1中箭头所示), 其中,共有24个CENH3的信号并且均位于染色体的着丝粒区域; 有丝分裂中期(图1-C), 染色体并排分布在赤道板上, 在中期结束后染色体将加倍, 而CENH3的信号也将随着着丝粒而加倍。细胞分裂后期(图1-D), 染色体随着着丝点被拉向两级, CENH3信号均位于子细胞的一侧(如图1-D3所示)。在所观察的有丝分裂的各个时期的细胞中, 均能观察到明显可见的CENH3的信号, 表明制备的抗体可用于蛋白免疫荧光分析。
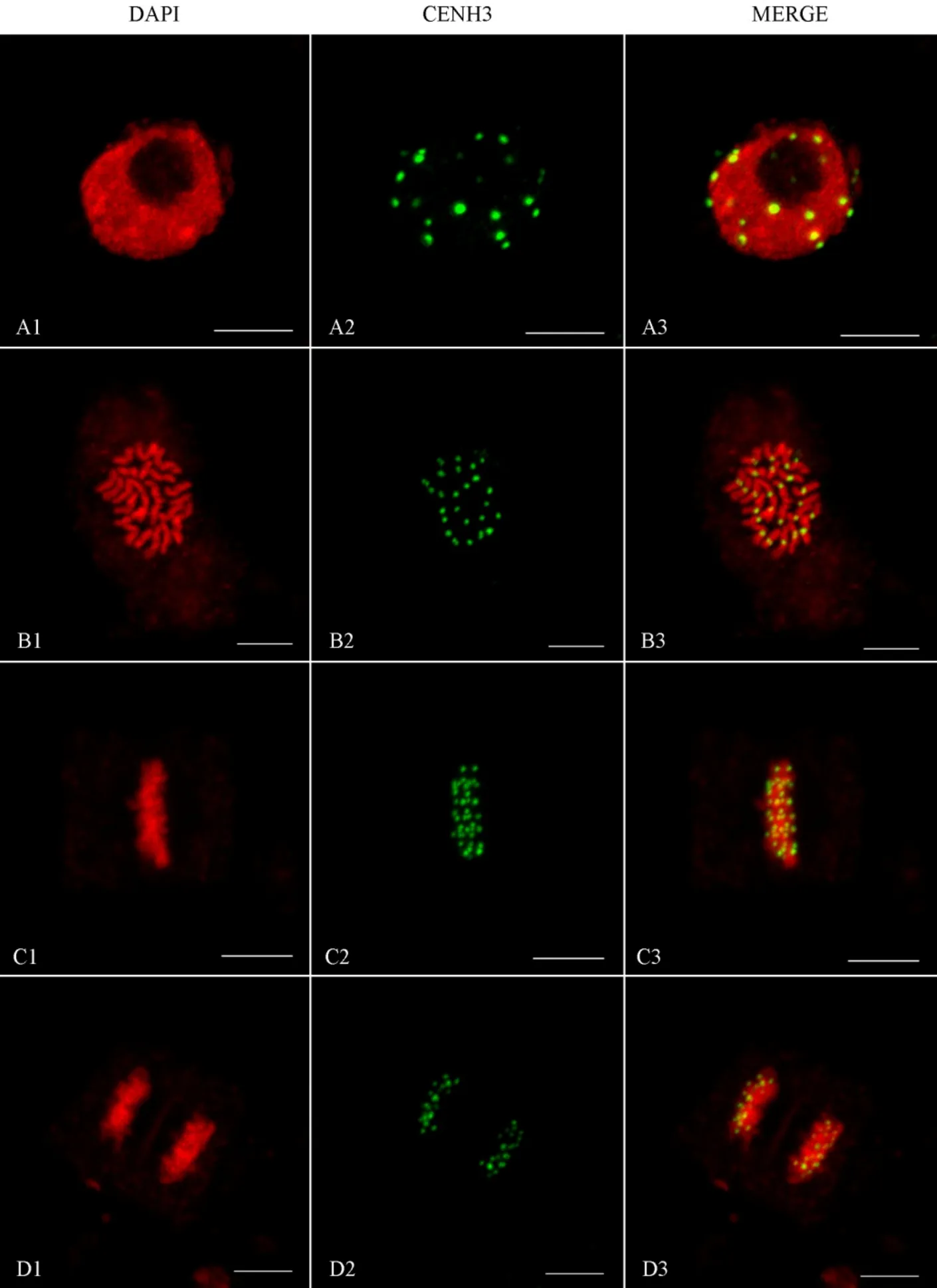
图1 水稻日本晴根尖细胞CENH3抗体的蛋白免疫荧光信号检测分析Fig. 1. Immunofluorescence analysis of CENH3 antibody in somatic root tip cells of Nipponbare.
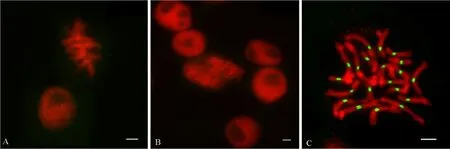
图2 大麦(A)、小麦(B)和玉米(C)根尖细胞CENH3抗体的蛋白免疫荧光信号检测分析Fig. 2. Immunofluorescence analysis of CENH3 antibody in somatic root tip cells of barley(A), wheat(B) and maize(C).
同时, 为了验证制备的CENH3抗体是否在其他作物上也有效,分别对小麦、大麦和玉米的根尖染色体进行了蛋白免疫荧光检测(图2)。在小麦和大麦的根尖染色体上并没有检测到CENH3信号, 但是在玉米上能够观察到明显的CENH3信号, 并且也位于着丝粒区域。说明本研究制备的CENH3抗体在玉米中的蛋白免疫荧光分析有效。
2.3 ChIP-DNA的PCR分析
由于水稻着丝粒是由蛋白和DNA组成的复合体, 所以分析与着丝粒蛋白相结合DNA序列的特征是进行着丝粒功能和结构分析的重要内容之一。近年来发展的染色质免疫共沉淀(ChIP)方法是研究蛋白与DNA互作的主要手段。为了进一步分析本研究中所获得的着丝粒蛋白CENH3抗体是否能应用于相关的ChIP分析, 我们利用该抗体进行ChIP-PCR验证该DNA是否来源于水稻着丝粒特异DNA序列。
通过分别对基因组DNA(Input), 阴性对照(Mock)和ChIP-DNA设置两个重复组, 以水稻着丝粒特异DNA序列CentO设计引物对进行PCR扩增(图3)。以阳性对照(基因组DNA)为模板扩增出相关条带, 由于水稻着丝粒特异DNA序列CentO是重复序列, 所以扩增出3个条带, 而阴性对照(Mock)几乎没有相应条带扩增出。而由CENH3抗体所获得的ChIP-DNA序列为模板可以扩增出相应条带, 并与阳性对照相吻合。说明该ChIP-DNA来源于水稻着丝粒特异DNA序列。
2.4 ChIP-DNA的FISH定位分析
染色体荧光原位杂交(FISH)是验证重复序列在染色体上具体位置的重要方法之一。为了进一步验证CENH3抗体的ChIP-DNA是否位于着丝粒区域, 我们将ChIP-DNA标记成FISH探针, 进行荧光原位杂交。根据FISH的检测结果(图4), 红色信号为染色体, 绿色信号为ChIP-DNA的杂交信号, 其中ChIP-DNA的信号位于染色体着丝粒区域, 进一步说明该ChIP-DNA位于水稻着丝粒区域, 是与着丝粒特异组蛋白CENH3特异结合的DNA序列。
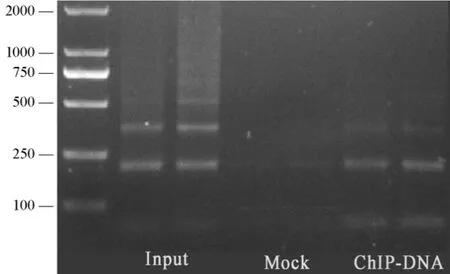
图3 CENH3抗体免疫沉淀DNA的PCR结果Fig. 3. PCR results of ChIP-DNA immunoprecipitated with CENH3 antibody.
3 讨论
本研究中制备的CENH3抗体, 通过效价的测定和蛋白免疫荧光检测, 都证明了CENH3抗体的有效性, 蛋白荧光信号存在于水稻细胞各时期的着丝粒上。同时, 对制备的CENH3抗体进行染色质免疫沉淀, 得到ChIP-DNA, 通过FISH定位分析进一步证明制备的CENH3抗体可用于ChIP分析。
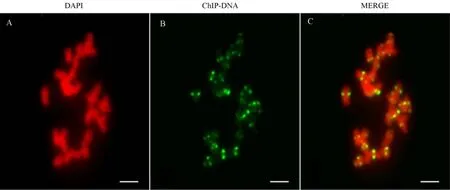
图4 日本晴根尖细胞ChIP-DNA的荧光原位杂交信号检测结果Fig. 4. FISH analysis of ChIP-DNA in somatic root tip cells of Nipponbare.
CENH3是组蛋白H3的变体, 存在于真核生物的功能着丝粒中[18]。近年来, 在玉米、水稻和拟南芥中都鉴别出植物的CENH3。对于着丝粒特异组蛋白CENH3的定位分析重要的方法之一就是利用对应的抗体进行蛋白免疫荧光检测。目前,可用于蛋白免疫荧光分析的抗体很多, 但是这些抗体不一定可以用到ChIP分析上, 这一方面和抗体的制备有关系, 另一方面是由于抗体的复杂性导致非特异性反应较多, 从而导致实验结果无法判断, 所以对于制备的抗体需要进一步的纯化。此前, 我们实验室使用的CENH3抗体都是购买或赠与的国外制备抗体, 获得途径繁琐或研究成本较高, 所以很有必要通过自己制备有效可用的CENH3抗体。本研究中, 我们制备的水稻CENH3抗体, 先后通过ELISA方法测定效价, 蛋白免疫荧光进行检测, 都证实制备的CENH3抗体是有效的。同时, 进一步的ChIP分析和FISH定位分析证明由该抗体所免疫共沉淀获得的DNA序列是属于水稻功能着丝粒区域。确定制备的CENH3抗体具有较好的特异性, 可应用于与着丝粒结构与功能分析的相关实验中, 对进一步解析水稻CENH3及着丝粒功能关系的研究提供了便利条件。另外,该抗体还可以应用于玉米等其他物种,
为进一步推广该抗体的使用提供可能。
[1] Allshire R C, Karpen G H. Epigenetic regulation of centromeric chromatin: Old dogs, new tricks? Nat Rev Genet, 2008, 9: 923-937.
[2] Malik H S, Henikoff S. Major evolutionary transitions in centromere complexity. Cell, 2009, 138: 1067-1082. [3] Henikoff S, Ahmad K, Malik H S. The centromere paradox: Stable inheritance with rapidly evolving DNA. Science, 2001, 293: 1098-1102.
[4] Sullivan B A, Blower M D, Karpen G H. Determining centromere identity: Cyclical stories and forking paths. Nat Rev Genet, 2001, 2: 584-596.
[5] Lefrancois P, Auerbach R K, Yellman C M, Roeder G S, Snyder M. Centromere-like regions in the budding yeast genome. PLoS Genet, 2013, 9: e1003209.
[6] Clarke L. Centromeres: proteins, protein complexes, and repeated domains at centromeres of simple eukaryotes. Curr Opin Genet&Dev, 1998, 8: 212-218.
[7] Cheeseman I M, Drubin D G, Barnes G. Simple centromere, complex kinetochore: Linking spindle microtubules and centromeric DNA in budding yeast. J Cell Biol, 2002, 157: 199-203.
[8] Schueler M G, Higgins A W, Rudd M K, Gustashaw K, Willard H F. Genomic and genetic definition of a functional human centromere. Science, 2001, 294: 109-115.
[9] Sun X, Le H D, Wahlstrom J M, Karpen G H. Sequence analysis of a functional Drosophila centromere. Genome Res, 2003, 13: 182-194.
[10] Ananiev E V, Phillips R L, Rines H W. Chromosomespecific molecular organization of maize (Zea mays L.)centromeric regions. Proc Natl Acad Scie USA, 1998, 95: 13073-13078.
[11] Kamm A, Galasso I, Schmidt T, Heslop-Harrison J S. Analysis of a repetitive DNA family from Arabidopsis arenosa and relationships between Arabidopsis species. Plant Mol Biol, 1995, 27: 853-862.
[12] Cheng Z, Dong F, Langdon T, Ouyang S, Buell C R, Gu M, Blattner F R, Jiang J. Functional rice centromeres are marked by a satellite repeat and a centromere-specific retrotransposon. Plant Cell, 2002, 14: 1691-1704.
[13] Yoda K, Ando S, Morishita S, Houmura K, Hashimoto K, Takeyasu K, Okazaki T. Human centromere protein A (CENP-A) can replace histone H3 in nucleosome reconstitution in vitro. Proc Natl Acad Sci USA, 2000, 97: 7266-7271.
[14] Cleveland D W, Mao Y, Sullivan K F. Centromeres and kinetochores: From epigenetics to mitotic checkpoint signaling. Cell, 2003, 112: 407-421.
[15] Talbert P B, Masuelli R, Tyagi A P, Comai L, Henikoff S. Centromeric localization and adaptive evolution of an Arabidopsis histone H3 variant. Plant Cell, 2002, 14: 1053-1066.
[16] Earnshaw W C, Rothfield N. Identification of a family of human centromere proteins using autoimmune sera from patients with scleroderma. Chromosoma, 1985, 91: 313-321.
[17] Jin W, Melo J R, Nagaki K, Talbert P B, Henikoff S, Dawe R K, Jiang J. Maize centromeres: Organization and functional adaptation in the genetic background of oat. Plant Cell, 2004, 16: 571-581.
[18] Nagaki K, Terada K, Wakimoto M, Kashihara K, Murata M. Centromere targeting of alien CENH3s in Arabidopsis and tobacco cells. Chrom Res, 2010, 18: 203-211.
[19] Kurumizaka H, Horikoshi N, Tachiwana H, Kagawa W. Current progress on structural studies of nucleosomes containing histone H3 variants. Curr Opin Struct Biol, 2013, 23: 109-115.
[20] Palmer D K, O'Day K, Wener M H, Andrews B S, Margolis R L. A 17-kD centromere protein (CENP-A) copurifies with nucleosome core particles and with histones. J Cell Biol, 1987, 104: 805-815.
[21] Bui M, Dimitriadis E K, Hoischen C, An E, Quenet D, Giebe S, Nita-Lazar A, Diekmann S, Dalal Y. Cellcycle-dependent structural transitions in the human CENP-A nucleosome in vivo. Cell, 2012, 150: 317-326.
[22] Malvezzi F, Litos G, Schleiffer A, Heuck A, Mechtler K, Clausen T, Westermann S. A structural basis for kinetochore recruitment of the Ndc80 complex via two distinct centromere receptors. EMBO J, 2013, 32: 409-423.
[23] Hori T, Shang W H, Takeuchi K, Fukagawa T. The CCAN recruits CENP-A to the centromere and forms the structural core for kinetochore assembly. J Cell Biol, 2013, 200: 45-60.
[24] Blower M D, Karpen G H. The role of Drosophila CID in kinetochore formation, cell-cycle progression and heterochromatin interactions. Nat Cell Biol, 2001, 3: 730-739.
[25] Howman E V, Fowler K J, Newson A J, Redward S, MacDonald A C, Kalitsis P, Choo K H. Early disruption of centromeric chromatin organization in centromere protein A (Cenpa) null mice. Proc Natl Acad Sci USA, 2000, 97: 1148-1153.
[26] Nagaki K, Cheng Z, Ouyang S, Talbert P B, Kim M, Jones K M, Henikoff S, Buell C R, Jiang J. Sequencing of a rice centromere uncovers active genes. Nat Genet, 2004, 36: 138-145.
[27] Zhong C X, Marshall J B, Topp C, Mroczek R, Kato A, Nagaki K, Birchler J A, Jiang J, Dawe R K. Centromeric retroelements and satellites interact with maize kinetochore protein CENH3. Plant Cell, 2002, 14: 2825-2836.
[28] Nagaki K, Murata M. Characterization of CENH3 and centromere-associated DNA sequences in sugarcane. Chrom Res, 2005, 13: 195-203.
[29] van Lente F, Jackson J F, Weintraub H. Identification of specific crosslinked histones after treatment of chromatin with formaldehyde. Cell, 1975, 5: 45-50.
[30] Jackson V. Studies on histone organization in the nucleosome using formaldehyde as a reversible crosslinking agent. Cell, 1978, 15: 945-954.
[31] Jackson V, Chalkley R. A new method for the isolation of replicative chromatin: selective deposition of histone on both new and old DNA. Cell, 1981, 23: 121-134.
[32] Jackson V, Chalkley R. Use of whole-cell fixation to visualize replicating and maturing simian virus 40: Identification of new viral gene product. Proc Natl Acad Sci USA, 1981, 78: 6081-6085.
[33] Solomon M J, Varshavsky A. Formaldehyde-mediated DNA-protein crosslinking: A probe for in vivo chromatin structures. Proc Natl Acad Sci USA, 1985, 82: 6470-6474.
[34] Lee H R, Zhang W, Langdon T, Jin W, Yan H, Cheng Z, Jiang J. Chromatin immunoprecipitation cloning reveals rapid evolutionary patterns of centromeric DNA in Oryza species. Proc Natl Acad Sci USA, 2005, 102: 11793-11798.
[35] Nagaki K, Talbert P B, Zhong C X, Dawe R K, Henikoff S, Jiang J. Chromatin immunoprecipitation reveals that the 180-bp satellite repeat is the key functional DNA element of Arabidopsis thaliana centromeres. Genetics, 2003, 163: 1221-1225.
Preparation and Application of Centromere-Specific Protein CENH3 Antibody in Rice
XUE Chao, ZHANG Rong, GUO Rui, LIU Shuai, LIU Xiaoyu, SHEN Mingchen, DENG Shifeng, GONG Zhiyun*
(Jiangsu Key Laboratory of Crop Genetics and Physiology/Jiangsu Co-Innovation Center for Modern Production Technology of Grain Crops/Key Laboratory of Plant Functional Genomics of the Ministry of Education, Yangzhou University, Yangzhou 225009, China;*Corresponding author, E-mail: zygong@yzu.edu.cn)
【Objective】 The centromeres of eukaryotic chromosomes are essential for cell division and inheritance of genetic information. The centromere is a complex composed of centromeric proteins and DNA motifs. In particular, a centromere-specific histone 3 variant, referred to CENH3, is a key centromere-specific protein for centromeric chromatin. So the preparation of CENH3 antibody is necessary for the research of centromeric structure and function.【Method】We designed a short-peptide for the rabbit immune experiment and prepared CENH3 antibody in rice. ELISA and protein immunofluorescence(IF) assay were performed to detect the effectiveness and availability of CENH3 antibody. 【Result】 The effective dilution of CENH3 antibody was detected at 1:400 000. Through IF analysis, obvious signals were detected in the centromeric region of each chromosome in rice and maize. DNA associated with CENH3 was selectively immunoprecipitated in chromatin immunoprecipitation assay(ChIP). After PCR amplification and FISH(fluorescence in situ hybridization) analysis, the results showed ChIP-DNA was located in functional centromere in rice. 【Conclusion】 These results demonstrated that CENH3 antibody prepared in this study can meet the requirement of related experiments in the research of centromere, and the antibody was prepared successfully.
rice; centromere-specific histone; chromatin immune-precipitation assay; immunofluorescence
Q786; S511.01
A
1001-7216(2017)05-0475-08
2017-03-24; 修改稿收到日期:2017-05-18。
国家自然科学基金面上项目(3137021);江苏省“青蓝工程”项目。
