水稻硝酸盐转运蛋白基因OsNPF7.9在氮素积累和转运中的功能研究
2017-09-23冯慧敏陆宏王汉卿李昕玥
冯慧敏 陆宏 王汉卿 李昕玥
(南京农业大学 资源与环境科学学院,南京 210095;*通讯联系人,E-mail:huimin.feng@njau.edu.cn)
水稻硝酸盐转运蛋白基因OsNPF7.9在氮素积累和转运中的功能研究
冯慧敏*陆宏 王汉卿 李昕玥
(南京农业大学 资源与环境科学学院,南京 210095;*通讯联系人,E-mail:huimin.feng@njau.edu.cn)
【目的】植物NPF家族成员具有转运硝酸盐、小肽等的功能。对水稻OsNPF7.9基因的功能研究能够为水稻氮素高效利用的分子机制提供理论基础。【方法】利用不同的生物软件对OsNPF7.9蛋白的生物学信息进行了预测分析;用OsNPF7.9基因启动子融合GUS报告基因的转基因水稻进行OsNPF7.9的组织定位观察;利用半定量RT-PCR和pOsNPF7.9::GUS转基因水稻分析OsNPF7.9受氮素调控特征;构建了OsNPF7.9的超表达水稻株系,并进行生理指标测定。【结果】OsNPF7.9是细胞质膜蛋白,有12个跨膜结构域,在第6~7个结构域之间有一个大的亲水环。组织定位结果显示OsNPF7.9在根、叶片、根茎结合处和花中都有表达。RT-PCR和pOsNPF7.9::GUS转基因水稻的GUS染色结果都表示,OsNPF7.9的表达不受氮素形态和浓度的影响。OsNPF7.9基因超表达后,不仅显著增加水稻的地上部硝酸盐含量以及根系向地上部的硝酸盐转运,而且增加了地上部的总氮浓度、总氮积累以及根系向地上部的总氮转运。【结论】OsNPF7.9参与硝酸盐从根系向地上部的转运,并且OsNPF7.9超表达可以提高水稻氮素积累和转运能力。
水稻;氮素;OsNPF7.9;硝酸盐转运蛋白
水稻是重要的粮食作物,氮素是植物生长发育必需的大量营养元素之一。在我国,低氮肥利用造成大量氮肥流失,进而带来严重的环境问题。水稻基因组测序完成,为水稻功能组学的研究提供有力的生物信息学背景[1-3]。因此,开展水稻氮素高效利用的分子机制研究,进而培育水稻氮素高效利用的品种有着重大的意义。
植物根系从土壤中吸收的矿质氮源主要有铵态氮(NH4+)和硝态氮(NO3-)。植物NO3-运输系统分为高亲和系统(HATS)和低亲和系统(LATS)[4-5],这两个系统都由NO3-转运蛋白来执行。NPF(原名为NRT1/PTR)以及NRT2/NAR2(NAR2是NRT2的辅助蛋白)家族编码NO3-转运蛋白,其中,NRT1负责LATS硝酸盐的吸收和转运,PTR负责转运含氮化合物比如寡肽和氨基酸,NRT2/NAR2家族成员负责HATS硝酸盐的吸收和转运[6-7]。拟南芥和水稻NPF家族分别有53和80多个成员,NPF家族中的很多基因的功能已经得到鉴定[7-8]。在拟南芥中,AtNPF6.3(AtCHL1/AtNRT1.1)属于双亲和NO3-转运蛋白,也是响应NO3-信号的受体[8-13]。AtNPF7.3(AtNRT1.5)负责NO3-从根部向地上部运输,而且和逆境的响应有关[14-15];AtNPF7.2 (AtNRT1.8)参与NO3-木质部运输,调节拟南芥对镉的耐性[16]。此外,AtNPF3.1被发现能够转运亚硝酸盐和赤霉素(GA)[17-19]。OsNPF8.2 (OPTR9)超表达后促进水稻生长,提高水稻氮素利用效率和产量[20];OsNPF2.4和OsNPF2.2参与NO3-从根系向地上部运输[21-22];OsNPF7.2被定位在液泡膜, OsNPF7.2敲除导致水稻在外界高NO3-条件下生长受阻[23]。目前,有关其他水稻OsNPF成员的研究还尚未见报道。
水稻OsNPF7.9的功能研究未有报道。我们从水稻日本晴中克隆了OsNPF7.9的启动子序列和ORF序列,并通过不同的方法研究了OsNPF7.9蛋白的跨膜结构和亚细胞定位,OsNPF7.9基因的表达特征和组织定位,以及OsNPF7.9超表达水稻的硝酸盐吸收转运的生理特征,以期为进一步研究OsNPF7.9参与水稻氮素高效利用的分子机制提供理论基础和有效指导。
1 材料与方法
1.1 材料
试验材料武运粳7号为本实验繁种保存。
1.2 试验方法
1.2.1 进化树和跨膜结构分析
NPF家族基因进化树绘制:在Aramemnon上(http://aramemnon.botanik.uni-koeln.de/index.ep)及NCBI(https://www.ncbi.nlm.nih.gov/)数据库中找出已明确基因功能的NPF家族成员的氨基酸序列,通过Clustal Omega (http://www.ebi.ac.uk/Tools/msa/ clustalo/)在线软件的聚类比较,再由MEGA5.1软件绘制完成NPF家族基因进化树,最后用软件iTOL美化进化树(http://itol.embl.de/)。
利用HmmTop_v2在线(http://www.enzim.hu/ hmmtop/html/submit.html)分析OsNPF7.9的跨膜结构,并运用TMRPre2D(http://biophysics.biol.uoa.gr/ TMRPres2D/download.jsp)绘制跨膜结构图。
1.2.2 亚细胞定位
根据OsNPF7.9基因序列(LOC_Os02g46460)和构建eGFP融合表达载体的规则设计PCR引物(金斯瑞公司合成),上游引物序列5'-AAAGCTTGAG CAAGAAATGTCTGGAACG-3',下游引物序列为5'-AGAATTCCTTGTCGTGCTAAACC-3',引物两端酶切位点分别为SpeⅠ和SacⅠ。水稻cDNA为模板扩增基因,连到克隆载体pMD19-T(TaKaRa公司)上,经过测序验证正确后用限制性内切酶HindⅢ和EcoRⅠ(TaKaRa公司)将目的片段切下来分别连接到中间载体pSAT6-EGFP-N1上,经验证正确后用限制性内切酶PI-PspⅠ将含有eGFP基因和OsNPF7.9的DNA片段切下来连接到表达载体pRCS2-ocs-nptⅡ,即为OsNPF7.9::eGFP。提取水稻原生质体和转化[24],将5 μg的OsNPF7.9::eGFP载体质粒加入200 μL原生质体中,12~16 h后,先用膜蛋白染料FM™4-64给原生质体染色2 min,然后利用激光共聚焦显微镜(Leica SP8)分别在560 nm波长和450~490 nm波长下激发膜蛋白染料和eGFP发出荧光,拍照记录。
1.2.3 pOsNPF7.9::GUS转基因水稻获得及组织定位观察
根据OsNPF7.9启动子序列设计引物,上游引物序列5'-TTAATTAAATCTCTAAGGGACTAA CACA-3',下游引物序列5'-GGCGCGCCCATTTC TTGCTCCTCCTCG-3',引物两端酶切位点分别为PacⅠ和AscⅠ。以水稻gDNA为模板,扩增出一段长1651 bp的启动子,并将启动子连接到克隆载体pMD19-T上,经测序验证序列完全正确后将启动子连接到表达载体PS1aG-3上,形成载体pOsNPF7.9::GUS。将pOsNPF7.9::GUS载体转入农杆菌,用农杆菌浸染法转入日本晴水稻基因组中,参考Ai等[25]的方法,获得T1代水稻种子。
种子发芽后,用正常营养液培养10 d,苗期取根系、叶片和根茎结合处;将水稻苗用土壤培养至抽穗扬花期,取花器官。采样后立即将样品浸入GUS染液,37℃培养箱过夜,在体式显微镜下观察拍照。叶片用70%酒精脱色后再观察拍照。

表1 本研究所用的半定量RT-PCR引物序列Table 1. Primers for semi-RT PCR in the study.
1.2.4 半定量PCR检测基因表达
提取水稻总RNA,经反转录成cDNA(TaKaRa公司反转录试剂盒),用OsNPF7.9基因和OsActin半定量引物扩增,引物序列、退火温度和循环数列于表1。
1.2.5 OsNPF7.9超表达水稻获得及分子检测
根据OsNPF7.9 ORF序列设计引物,上游引物序列为5'-AACTAGTGAGCAAGAAATGTCTGGA ACG-3',下游引物序列5'-AGAGCTCCGGAGAC TTGTCGTGCTAAACC-3',引物两端酶切位点分别为SpeⅠ和SacⅠ。连到克隆载体pMD19-T (TaKaRa公司)上,经过测序验证后将目的片段酶切下来分别连接到表达载体pTCK303,测序正确后,将载体转入农杆菌,用农杆菌浸染法转入武运粳7号基因组中,转基因方法参考Ai等[25],获得T1代种子。将T1代连续繁种获得T3代植株,提取水稻gDNA,用Southern印迹法[24]检测插入基因的拷贝数。提取T3代植株总RNA,半定量RT-PCR检测OsNPF7.9基因的表达,参考步骤1.2.4。
1.2.6 水稻培养
水稻种子武运粳7号野生型及T4代转基因株系OE1和OE2,经15% H2O2消毒30 min,蒸馏水洗净后放入37℃培养箱黑暗培养3 d催芽,到第2叶完全展开时,移入含有50 mg/L潮霉素的水溶液中筛选1周,再挑选长势良好、形态一致的苗,移至6 L的周转箱,每箱12穴,每穴2株苗,用IRRI营养液 (1.25 mmol/L NH4NO3, 0.35 mmol/L K2SO4, 1 mmol/L CaCl2·2H2O, 1 mmol/L MgSO4·7H2O, 0.5 mmol/L Na2SiO3·9H2O, 20 mmol/L Fe-EDTA, 20 mmol/L H3BO3, 9 mmol/L MnCl2·4H2O, 0.32 mmol/L CuSO4·5H2O, 0.77 mmol/L ZnSO4·7H2O, 0.39 mmol/L Na2MoO4·2H2O) 培养,调pH值为5.5,每3 d更换一次。每组实验每个株系3个技术重复,2次生物学重复。
将水稻植株放进105℃烘箱杀青30 min,75℃下烘干至恒重,粉碎后称取0.05 g样品,用H2SO4-H2O2法对样品进行消化后使用流动分析仪(型号为AA3)测定样品中的总氮。取水稻植株干样0.03 g置于10 mL离心管中,加5 mL去离子水后将装有样品的离心管放在100℃沸水中煮30 min,等试管中的溶液冷却后吸取1 mL上清稀释到10 mL,用AA3型连续性流动分析仪测溶液中NO3--N的含量。
2 结果与分析
2.1 植物NPF家族成员进化树分析
OsNPF7.9位于水稻第2染色体上,cDNA全长序列为2240 bp,编码610个氨基酸,有5个外显子和4个内含子。在植物中,有约45个NPF成员已被报道有硝酸盐、亚硝酸盐、小肽或者激素转运的功能[6-8]。利用生物学软件Clustal Omega和MEGA5.1对水稻、拟南芥、黄瓜和苜蓿等植物的这45个NPF成员进行进化树分析(图1)。结果发现水稻的OsNPF7.9与拟南芥的AtNPF7.2 (AtNRT1.8)和AtNPF7.3(AtNRT1.5)进化关系最密切。在水稻OsNPF家族成员中,OsNPF7.9与OsNPF7.1、OsNPF7.2、OsNPF7.3(OsPTR6)和OsNPF7.4 (OsPTR5)进化关系较密切。
2.2 OsNPF7.9亚细胞水平定位分析
跨膜结构预测软件分析结果表明(图2),OsNPF7.9蛋白含有12次跨膜域,N端在细胞内,而且在第6和第7跨膜结构域之间有一个由99个氨基酸组成的亲水环。为了进一步确认OsNPF7.9的亚细胞定位,将OsNPF7.9融合GFP蛋白,并转化水稻原生质体。图3结果显示,阳性对照eGFP表达在整个细胞中,而OsNPF7.9和GFP的融合蛋白只表达在细胞质膜上,而且与膜定位染料FM64
结果完全重合,表明OsNPF7.9定位在细胞质膜上。
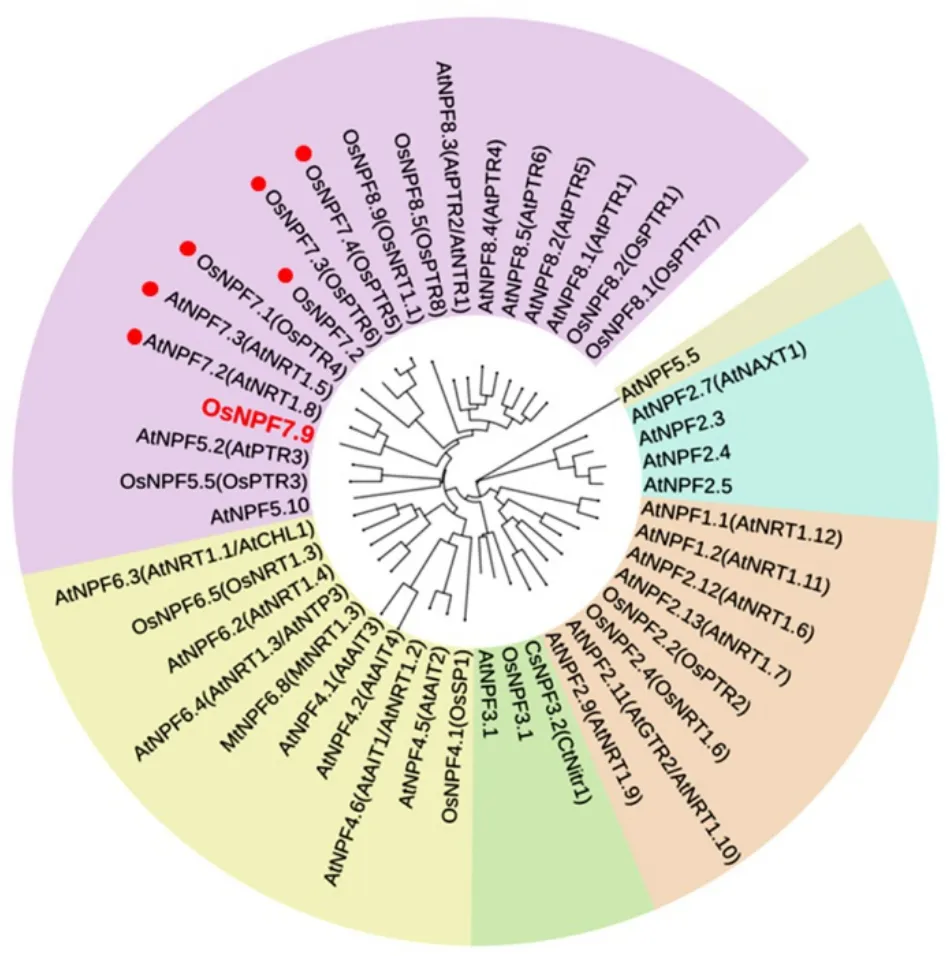
图1 植物NPF家族进化树分析Fig. 1. Phylogenetic of NPF in plants.
2.3 OsNPF7.9在不同组织中的表达
为研究OsNPF7.9在不同组织中的表达,构建了OsNPF2.4启动子融合GUS报告基因的载体,并得到转基因水稻pOsNPF2.4::GUS。将14 d大小的转基因水稻幼苗不同部位进行GUS染色检测,发现OsNPF2.4在根尖没有GUS表达(图4-A),主要在主根成熟区和侧根有强烈GUS表达(图4-B),同时在叶片和根茎结合处检测到GUS活性(图4-C,D);OsNPF7.9在花药和种子外壳的盾片上也有表达(图4-E,F)。
2.4 OsNPF7.9在不同形态氮素下的表达水平
为了研究OsNPF7.9受氮素调控模式,将水稻幼苗缺氮后分别供应0.2 mmol/L NO3-、5 mmol/L NO3-、0.2 mmol/L NH4+和5 mmol/L NH4+,结果显示,在根部OsNPF7.9的表达水平不受氮素形态和浓度的调控(图5-A)。对pOsNPF7.9::GUS转基因水稻进行不同氮素形态和浓度的处理,和RT-PCR结果一致,转基因水稻根系的GUS活性不受氮素形态和浓度的影响(图5-B)。
2.5 OsNPF7.9超表达水稻的获得和分子鉴定
通过转基因手段将pUbi-OsNPF7.9转化日本晴,得到了OsNPF7.9的超表达转基因水稻苗60株,经过GUS染色和PCR扩增鉴定,30株转基因苗是阳性苗,转基因苗的阳性率为50%。从中随机挑选两个株系,命名为OE1和OE2,用OsNPF7.9特异的半定量引物对野生型和超表达株系mRNA水平进行分析,发现OE1和OE2的表达水平都显著提高(图6-A)。Southern blot结果表明,OE1和OE2是插入位点不同的两个单拷贝株系(图6-B)。

图2 OsNPF7.9跨膜结构分析Fig. 2. Transmembrane topology of OsNPF7.9.

图3 OsNPF7.9水稻原生质体亚细胞定位Fig. 3. Sub-celluar localization of OsNPF7.9 in rice protoplast.
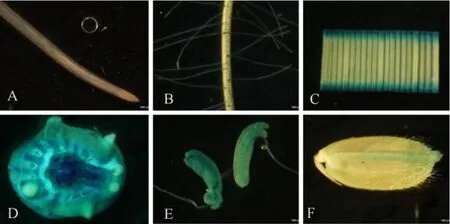
图4 OsNPF7.9启动子融合GUS转基因水稻的组织定位Fig. 4. Tissue expression of pOsNPF7.9::GUS transgenic rice.
2.6 OsNPF7.9超表达对水稻生长和硝酸盐吸收转运的影响
为了研究OsNPF7.9过表达后对水稻生长、硝酸盐吸收和转运的影响,我们将OE1和OE2在含有1.25 mmol/L NH4NO3的营养液中培养30 d。结果发现,OE1和OE2地上部长势优于野生型水稻(图 7-A),进一步结果显示(图7-B),两个超表达株系地上部的干质量比野生型增加约31%和27%,而地下部没有显著差异。如图7-C显示,与野生型相比,超表达株系OE1和OE2地上部NO3-浓度分别提高了26%和17%,但是地下部分别降低了17%和19%。通过计算地上部和地下部含量的比值发现,与野生型相比,OE1和OE2的比值都显著提高(图7-D)。这表明OsNPF7.9超表达可以促进地上部的生长,并且OsNPF7.9超表达虽然降低了根系NO3-含量,但是提高了水稻从根系向地上部的转运,进而提高了地上部量。
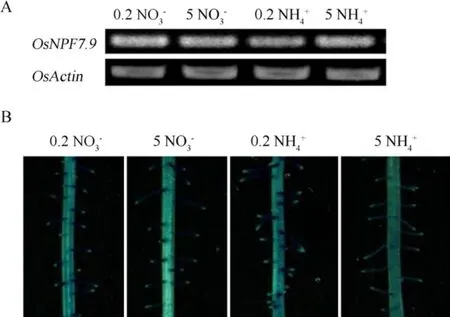
图5 OsNPF7.9在不同形态氮素和不同浓度下的表达Fig. 5. Expression of OsNPF7.9 supplied with various forms of nitrogen at different N concentrations.
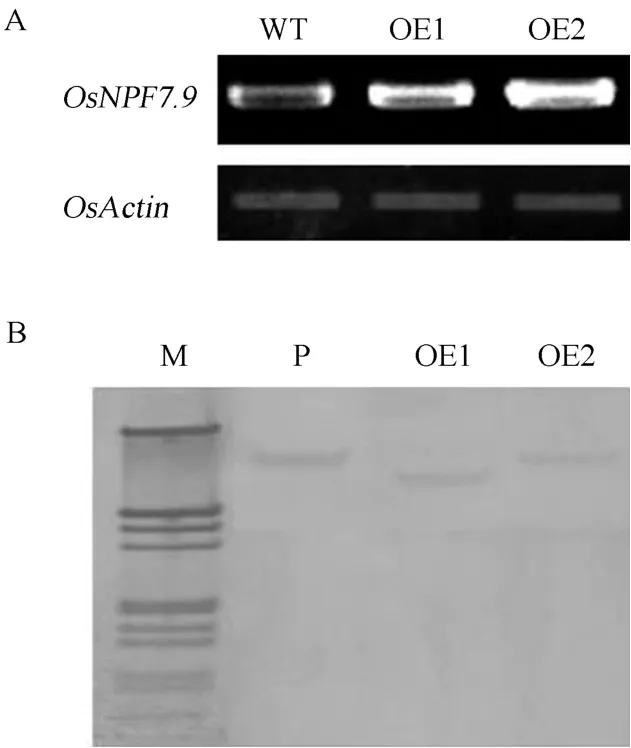
图6 OsNPF7.9超表达水稻的分子鉴定Fig. 6. Identification of OsNPF7.9-overexpressing rice.
2.6 OsNPF7.9超表达对水稻氮素积累和转运的影响
进一步测定超表达株系和野生型的总氮,如图8-A显示,和野生型相比,OE1和OE2在地上部的总氮浓度显著提高,而地下部没有显著差异。根据干质量和总氮浓度计算得到的总氮积累,OE1和OE2和总氮积累比野生型高55%和60%(图8-B)。同时,OE1和OE2地上部和地下部总氮积累的比率与野生型相比显著提高(图8-C)。这说明OsNPF7.9超表达不仅对维持水稻生长具有重要作用,而且能够提高水稻氮素积累和转运。
3 讨论
水稻OsNPF家族中有80个成员基因,迄今为止已报道了多个基因具有参与硝酸盐或者小肽的转运的功能[6,8]。本研究中,OsNPF7.9与拟南芥AtNPF7.2和AtNPF7.3在进化树上的亲缘关系较近(图1),AtNPF7.2和AtNPF7.3已被发现具有硝酸盐转运的功能[14,16]。
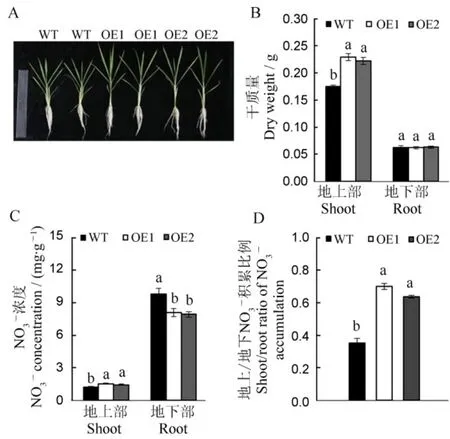
图7 OsNPF7.9超表达水稻的生长和含量Fig. 7. Growth andconcentration in the OsNPF7.9 overexpressing rice.
植物NPF家族蛋白一般由450~600个氨基酸构成,有12次跨膜结构域,并且在第6~7个跨膜结构域之间有一个大的亲水环[26,27],而OsNPF7.9的跨膜结构具有NPF家族蛋白的特征(图2)。已有研究发现AtNPF7.2和AtNPF7.3是细胞质膜蛋白[14,16],我们将OsNPF7.9定位于细胞质膜上(图3)。
OsNPF7.9在根系的表达不受氮素形态和浓度的调控(图5),属于组成型表达。在水稻中,有些NPF家族成员都显示不受氮素形态和浓度的调控,如OsNPF8.9(OsNRT1.1)[28]和OsNPF2.4[21]。OsNPF7.9组织定位结果表明该基因在根系、叶片和根茎结合处都有表达(图4),这与已报道的OsNPF7.9和OsNPF2.4组织定位结果比较相似,这两个基因也在根系和叶片中都有表达[21,22]。
OsNPF7.9超表达能提高水稻地上部生物量,虽然根系NO3-浓度降低,但是地上部NO3-浓度提高,而根系向地上部总氮和硝酸盐的转运都提高了(图7),推测可能因为根系向地上部转运了更多的硝酸盐导致根系硝酸盐浓度降低。同时OsNPF7.9超表达提高了水稻地上部的总氮浓度和总氮积累,而地下部的这些指标都没有变化。我们计算了地下部向地上部的氮素转运,也发现OsNPF7.9超表达提高了水稻氮素从根系向地上部的转运(图8)。拟南芥AtNPF7.3基因只在根系靠近木质部的中柱鞘细胞表达,而且敲除该基因后木质部伤流液和地上部硝酸盐含量都显著下降,说明AtNPF7.3参与了根系木质部硝酸盐的装载[14]。而另外一个基因AtNPF7.2在拟南芥根系和地上部的所有木质部导管的薄壁细胞都有表达,而且敲除基因后木质部伤流液中硝酸盐含量提高了[16]。在进化树上OsNPF7.9与AtNPF7.3以及AtNPF7.2的亲缘关系较近,推测OsNPF7.9具有类似的功能,能够连接地下部和地上部硝酸盐的运输。
综上所述,OsNPF7.9是位于细胞膜上的蛋白,可能参与硝酸盐从根系向地上部的转运,而且OsNPF7.9超表达可以提高氮素积累和转运能力。

图8 OsNPF7.9超表达水稻的总氮浓度、积累和转运Fig. 8. Total N concentration, accumulation and transport of OsNPF7.9 overexpression rice.
[1] Feng Q, Zhang Y, Hao P, Wang S, Fu G, Huang Y, Li Y, Zhu J, Liu Y, Hu X, Jia P, Zhang Y, Zhao Q, Ying K, Yu S, Tang Y, Weng Q, Zhang L, Lu Y, Mu J, Lu Y, Zhang L S, Yu Z, Fan D, Liu X, Lu T, Li C, Wu Y, Sun T, Lei H, Li T, Hu H, Guan J, Wu M, Zhang R, Zhou B, Chen Z, Chen L, Jin Z, Wang R, Yin H, Cai Z, Ren S, Lv G, Gu W, Zhu G, Tu Y, Jia J, Zhang Y, Chen J, Kang H, Chen X, Shao C, Sun Y, Hu Q, Zhang X, Zhang W, Wang L, Ding C, Sheng H, Gu J, Chen S, Ni L, Zhu F, Chen W, Lan L, Lai Y, Cheng Z, Gu M, Jiang J, Li J, Hong G, Xue Y, Han B. Sequence and analysis of rice chromosome 4. Nature, 2002, 420(6912): 316-320.
[2] Sasaki T, Matsumoto T, Yamamoto K, Sakata K, Baba T, Katayose Y, Wu J, Niimura Y, Cheng Z, Nagamura Y, Antonio B A, Kanamori H, Hosokawa S, Masukawa M, Arikawa K, Chiden Y, Hayashi M, Okamoto M, Ando T, Aoki H, Arita K, Hamada M, Harada C, Hijishita S, Honda M, Ichikawa Y, Idonuma A, Iijima M, Ikeda M, Ikeno M, Ito S, Ito T, Ito Y, Ito Y, Iwabuchi A, Kamiya K, Karasawa W, Katagiri S, Kikuta A, Kobayashi N, Kono I, Machita K, Maehara T, Mizuno H, Mizubayashi T, Mukai Y, Nagasaki H, Nakashima M, Nakama Y, Nakamichi Y, Nakamura M, Namiki N, Negishi M, Ohta I, Ono N, Saji S, Sakai K, Shibata M, Shimokawa T, Shomura A, Song J, Takazaki Y, Terasawa K, Tsuji K, Waki K, Yamagata H, Yamane H, Yoshiki S, Yoshihara R, Yukawa K, Zhong H, Iwama H, Endo T, Ito H, Hahn J H, Kim H I, Eun M Y, Yano M, Jiang J, Gojobori T. The genome sequence and structure of rice chromosome 1. Nature, 2002, 420(6913): 312-316.
[3] Yang J L, Li Y Y, Zhang Y J, Zhang S S, Wu Y R, Wu P, Zheng S J. Cell wall polysaccharides are specifically involved in the exclusion of aluminum from the rice root apex. Plant Physiol, 2008, 146(2): 602-611.
[4] Parker J L, Newstead S. Molecular basis of nitrate uptake by the plant nitrate transporter NRT1.1. Nature, 2014, 507(7490): 68-72.
[5] Krapp A, David L C, Chardin C, Girin T, Marmagne A, Leprince A S, Chaillou S, Ferrario-Méry S, Meyer C, Daniel-Vedele F. Nitrate transport and signalling in Arabidopsis. J Exp Bot, 2014, 65(3): 789-798.
[6] Léran S, Varala K, Boyer J C, Chiurazzi M, Crawford N, Daniel-Vedele F, David L, Dickstein R, Fernandez E, Forde B, Gassmann W, Geiger D, Gojon A, Gong J M,Halkier B A, Harris J M, Hedrich R, Limami A M, Rentsch D, Seo M, Tsay Y F, Zhang M, Coruzzi G, Lacombe B. A unified nomenclature of NITRATE TRANSPORTER 1/PEPTIDE TRANSPORTER family members in plants. Trends Plant Sci, 2014, 19(1): 5-9.
[7] Xu G, Fan X, Miller A J. Plant nitrogen assimilation and use efficiency. Annu Rev Plant Biol, 2012, 63(1): 153-182.
[8] Wang Y Y, Hsu P K, Tsay Y F. Uptake, allocation and signaling of nitrate. Trends Plant Sci, 2012, 17(8): 458-467.
[9] Tsay Y F 1, Schroeder J I, Feldmann K A, Crawford N M. The herbicide sensitivity gene CHL1 of Arabidopsis encodes a nitrate-inducible nitrate transporter. Cell, 1993, 72(5): 705-713.
[10] Liu K H, Huang C Y, Tsay Y F. CHL1 is a Dual-affinity nitrate transporter of Arabidopsis involved in multiple phases of nitrate uptake. Plant Cell, 1999, 11(5): 865-874.
[11] Liu K H, Tsay Y F. Switching between the two action modes of the dual-affinity nitrate transporter CHL1 by phosphorylation. EMBO J, 2003, 22(5): 1005-1013.
[12] Guo F Q, Wang R, Chen M, Crawford N M. The Arabidopsis dual-affinity nitrate transporter gene AtNRT1.1 (CHL1) is activated and functions in nascent organ development during vegetative and reproductive growth. Plant Cell, 2001, 13(8): 1761-1778.
[13] Ho C H, Lin S H, Hu H, Tsay Y F. CHL1 functions as a nitrate sensor in plants. Cell, 2009, 138(6): 1184-1194.
[14] Lin S H, Kuo H F, Canivenc G, Lin C S, Lepetit M, Hsu P K, Tillard P, Lin H L, Wang Y Y, Tsai C B, Gojon A, Tsay Y F. Mutation of the Arabidopsis NRT1.5 nitrate transporter causes defective root-to-shoot nitrate transport. Plant Cell, 2008, 20(9): 2514-2528.
[15] Chen C Z, Lv X F, Li J Y, Yi H Y, Gong J M. Arabidopsis NRT1.5 is another essential component in regulation of nitrate reallocation and stress tolerance. Plant Physiol, 2012, 159 (4): 1582-1590.
[16] Li J Y, Fu Y L, Pike S M, Bao J, Tian W, Zhang Y, Chen C Z, Zhang Y, Li H M, Huang J, Li LG, Schroeder J I, Gassmann W, Gong J M. The Arabidopsis nitrate transporter NRT1.8 functions in nitrate removal from the xylem sap and mediates cadmium tolerance. Plant Cell, 2010, 22(5):1633-1646.
[17] Pike S, Gao F, Kim M J, Kim S H, Schachtman D P, Gassmann W. Members of the NPF3 transporter subfamily encode pathogen-inducible nitrate/nitrite transporters in grapevine and Arabidopsis. Plant Cell Physiol, 2014, 55(1):162-170.
[18] Tal I, Zhang Y, Jørgensen M E, Pisanty O, Barbosa I C, Zourelidou M, Regnault T, Crocoll C, Olsen C E, Weinstain R, Schwechheimer C, Halkier B A, Nour-Eldin H H, Estelle M, Shani E. The Arabidopsis NPF3 protein is a GA transporter. Nat Commun, 2016, 7: 11486.
[19] David L C, Berquin P, Kanno Y, Seo M, Daniel-Vedele F, Ferrario-Méry S. N availability modulates the role of NPF3.1, a gibberellin transporter, in GA-mediated phenotypes in Arabidopsis. Planta, 2016, 244(6): 1315-1328.
[20] Fang Z, Xia K, Yang X, Grotemeyer M S, Meier S, Rentsch D, Xu X, Zhang M. Altered expression of the PTR/NRT1 homologue OsPTR9 affects nitrogen utilization efficiency, growth and grain yield in rice. Plant Biotechnol J, 2013, 11(4): 446-458.
[21] Xia X, Fan X, Wei J, Feng H, Qu H, Xie D, Miller A J, Xu G. Rice nitrate transporter OsNPF2.4 functions in low-affinity acquisition and long-distance transport. J Exp Bot, 2015, 66(1): 317-331.
[22] Li Y, Ouyang J, Wang Y Y, Hu R, Xia K, Duan J, Wang Y, Tsay Y F, Zhang M. Disruption of the rice nitrate transporter OsNPF2.2 hinders root-to-shoot nitrate transport and vascular development. Sci Rep, 2015, 5: 9635.
[23] Hu R, Qiu D, Chen Y, Miller A J, Fan X, Pan X, Zhang M. Knock-Down of a tonoplast localized low-affinity nitrate transporter osnpf7.2 affects rice growth under high nitrate supply. Front Plant Sci, 2016, 7: 1529.
[24] Tang Z, Fan X, Li Q, Feng H, Miller A J, Shen Q, Xu G. Knockdown of a rice stellar nitrate transporter alters long-distance translocation but not root influx. Plant Physiol, 2012, 160(4): 2052-2063.
[25] Ai P, Sun S, Zhao J, Fan X, Xin W, Guo Q, Yu L, Shen Q, Wu P, Miller A J, Xu G. Two rice phosphate transporters, OsPht1;2 and OsPht1;6, have different functions and kinetic properties in uptake and translocation. Plant J, 2009, 57(5): 798-809.
[26] Tsay Y F, Chiu C C, Tsai C B, Ho C H, Hsu P K. Nitrate transporters and peptide transporters. FEBS Lett, 2007, 581(12): 2290-2300.
[27] Parker J L, Newstead S. Molecular basis of nitrate uptake by the plant nitrate transporter NRT1.1. Nature, 2014, 507(7490): 68-72.
[28] Fan X, Feng H, Tan Y, Xu Y, Miao Q, Xu G. A putative 6-transmembrane nitrate transporter OsNRT1.1b plays a key role in rice under low nitrogen. J Integr Plant Biol, 2016, 58(6): 590-599.
Function Analyses of Rice Nitrate Transporter Gene OsNPF7.9 in Nitrogen Accumulation and Transport
FENG Huimin*, LU Hong, WANG Hanqing, LI Xinyue
(College of Resources and Environmental Sciences, Nanjing Agricultural University, Nanjing 210095, China;*Corresponding author, E-mail: huimin.feng@njau.edu.cn)
【Objective】 NPF family in plants can transport nitrate and peptide, etc. Functional analyses of OsNPF7.9 can lay a theoretical basis for studying molecular mechanism of high nitrogen use-efficiency.【Method】 Bioinformatics of OsNPF7.9 was predicted with different biological softwares. The OsNPF7.9 promoter-GUS transgenic rice was obtained for analyzing the spatial expression pattern of OsNPF7.9. And the expression patterns of OsNPF7.9 under different N supply levels were observed using semi-quantitative RT-PCR and pOsNPF7.9::GUS transgenic rice. OsNPF7.9 overexpressing rice was obtained, and their different physiological indexes were measured. 【Result】 OsNPF7.9 is localized on plasma membrane with 12 transmembrane domains and a big hydrophilic loop between the 6th and the 7th transmembrane domain. Tissue expression pattern showed that OsNPF7.9 was expressed in roots, leaves, root-shoot junction and flowers. Both RT-PCR and GUS staining of pOsNPF7.9::GUS transgenic rice indicated that the expression of OsNPF7.9 was not affected by N concentration and form. Overexpression of OsNPF7.9 could significantly increase not only nitrate concentration in shoots and roots-shoots nitrate ratio; but also total nitrogen concentration and accumulation in shoots, and roots-shoots total nitrogen ratio. 【Conclusion】OsNPF7.9 participated nitrate transport from roots to shoots, and overexpression of OsNPF7.9 could increase N accumulation and transport from roots to shoots.
rice; nitrogen; OsNPF7.9; nitrate transporter
Q786; S511.01
A
1001-7216(2017)05-0457-09
2017-03-15; 修改稿收到日期:2017-04-06。
国家自然科学基金资助项目(31401938);中央高校基本科研业务费专项资金资助项目(Y0201500014,Y0201600366)。
