控制水稻金黄色颖壳与节间基因OsCAD2的图位克隆
2017-09-23王宏张迎信孙廉平孟帅徐鹏吴玮勋程式华曹立勇沈希宏中国水稻研究所浙江省超级稻研究重点实验室水稻生物学国家重点实验室杭州311401共同第一作者通讯联系人mailcaoliyongcaascnxihongshen126com
王宏 张迎信 孙廉平 孟帅 徐鹏 吴玮勋 程式华 曹立勇 沈希宏(中国水稻研究所/浙江省超级稻研究重点实验室/水稻生物学国家重点实验室,杭州 311401;共同第一作者;通讯联系人, E-mail: caoliyong@caas.cn,xihongshen@126.com)
控制水稻金黄色颖壳与节间基因OsCAD2的图位克隆
王宏#张迎信#孙廉平 孟帅 徐鹏 吴玮勋 程式华 曹立勇*沈希宏*
(中国水稻研究所/浙江省超级稻研究重点实验室/水稻生物学国家重点实验室,杭州 311401;#共同第一作者;*通讯联系人, E-mail: caoliyong@caas.cn,xihongshen@126.com)
【目的】水稻色素不仅对其自身生长发育有重要生理作用,而且在水稻育种、农副产品改良等方面运用比较广泛。对水稻色素相关基因进行表型分析和基因定位,为进一步研究色素代谢途径及调控机理奠定基础。【方法】利用EMS诱变粳稻长粒粳(CLJ),在突变体库内筛选到一个金黄色颖壳与节间突变体gh881;在成熟期,测定野生型与gh881的主要农艺性状;将gh881与野生型及中恢8015杂交,观察BC1F1及F1植株表型,并对BC1F2及F2表型分离进行卡方检验,对gh881进行遗传分析;利用F2群体和图位克隆的方法对gh881突变基因进行定位;采用qPCR检测颖壳颜色相关基因在gh881与野生型不同发育时期的幼穗、节间以及剑叶叶鞘的相对表达量。【结果】与野生型相比,突变体的颖壳与节间均呈金黄色;除单株有效穗数外,gh881突变体的株高、每穗总粒数及实粒数、结实率和千粒重等性状均极显著降低。遗传分析和基因定位结果表明,gh881的突变表型受1对隐性核基因控制,位于第2染色体短臂,并最终将该基因精细定位于标记FH-13和RH-25之间,物理距离约33.2 kb,该区域中包含4个开放阅读框(ORFs)。【结论】序列分析结果表明,发现其中一个编码肉桂醇脱氢酶(CAD)的基因OsCAD2(Os02g0187800)的 3 563 bp处发生了1个单碱基突变(G转换为A),导致该基因编码区的第297位氨基酸由甘氨酸突变为天冬氨酸,由此认为该突变体为OsCAD2基因单碱基突变的新等位基因。qRT-PCR结果表明,突变体的节间中OsCAD2相对表达量极显著下调,而在剑叶叶鞘及穗部则基本都是极显著增加,其他相关基因也发生显著变化,证实OsCAD2是木质素代谢中的重要基因,且可能与其他相关基因存在反馈调节。
水稻;色素;突变体;颖壳;节间;基因定位
水稻是世界上最重要的粮食作物之一,为人类提供了超过21%的能量[1],而植物色素的组成及代谢不仅对水稻生长发育具有重要意义,而且对水稻农副产品再利用具有重要价值。植物色素是植物体内次级代谢产物之一,其主要成分是类黄酮,而水稻颜色的变化一般认为是类黄酮化合物成分与代谢的影响[2,3]。水稻中,金黄色颖壳与节间颜色的变化同时受类黄酮化合物和木质素的成分与代谢共同影响[4-8]。这两类物质合成都是由苯丙氨酸在苯丙氨酸裂解酶(phenylalanine ammonia-lyase, PAL)催化形成肉桂酸开始的,肉桂酸经肉桂酸-4-羟基化酶(cinnamate 4-hydroxylase, C4H)催化形成香豆酸。此后,类黄酮化合物和木质素类物质分别以不同的代谢途径合成并转化。一方面,香豆酸可进一步形成香豆酰辅酶A,并与丙二酰辅酶A经查耳酮合酶(chalcone synthase, CHS)催化形成查耳酮,一部分查耳酮直接作为黄铜的前体物质,另一部分可经查耳酮异构酶(chalcone isomerase, CHI)催化形成黄烷酮以作为异黄酮的前体物质[9];另一方面,香豆酸可在一系列关键酶的作用下形成木质素前体物质香豆醇(p-coumaryl alcohol)、松柏醇(coniferyl alcohol)和芥子醇(sinapyl alcohol),进而形成木质素[10]。
与类黄酮化合物与木质素代谢途径相关且引起水稻颖壳颜色变化的已克隆的基因主要有3个。位于水稻第3染色体上、黄酮类化合物代谢途径中的一个重要基因查耳酮异构酶(chalcone isomerase,CHI)基因OsCHI,在Dasheng转座子插入突变体gh1中,OsCHI基因完全不表达,导致gh1颖壳和节间中总类黄酮物质含量剧增,表现为颖壳与节间均为金黄色[4]。褐色颖壳抑制因子IBF1编码一个包含Kelch重复序列结构域F-box蛋白,在类黄酮化合物生物合成过程中,作为抑制剂,能够抑制褐色素在水稻颖壳沟纹中沉积[5]。肉桂醇脱氢酶(cinnamyl-alcohol dehydrogenase, CAD)基因OsCAD2具有强脱氢酶活性,是参与木质素前体物质松柏醇(coniferyl alcohol)和芥子醇(sinapyl alcohol)的合成所必需的,主要负责水稻茎秆木质素单体生物合成,该基因定位在第2染色体上,在gh2中,由于OsCAD2突变,使得木质素含量和组成变化,从而出现水稻金黄色颖壳与节间表型[6-8]。此外,通过水稻全基因组预测和图位克隆的方法,已陆续报道了gh3[11]、gh5[12]和gh6[13]等金黄色颖壳与节间突变体,但相关基因尚未克隆。
此外,类黄酮化合物和木质素代谢途径中的相关基因通过调控这两种物质的成分与代谢,还可影响水稻正常的生长发育。水稻4-香豆酸:辅酶A连接酶基因Os4CL3,作为单木质醇类和黄酮类生物合成中苯丙素代谢途径中的一个关键酶,抑制Os4CL3 表达导致植株变矮、木质素含量显著降低并伴随水稻育性降低;可能是木质素合成受阻,导致花粉囊发育异常引起[14]。另一个苯丙氨酸裂解酶基因OsPAL4,处于类黄酮化合物和木质素代谢途径上游,直接影响水稻的广谱抗病性,其突变可增加植株对白叶枯病、纹枯病和稻瘟病的敏感性[15]。另外,OsMYB48转录因子作为木质素调控途径中的正调控子,当该基因表达量上升后,能够一定程度上导致木质素结构基因上调[16]。
水稻颖壳颜色作为谷粒的一个重要性状之一,不仅可作为最为直观的形态学标记[17],制种上用于色选,而且对该类基因调控类黄酮化合物和木质素合成与代谢过程的分子机理研究具有十分重要的生物学意义。本研究对一个金黄色颖壳与节间突变体gh881进行了表型鉴定及其目的基因的分离与定位研究,同时利用qPCR对该基因及水稻木质素与黄酮类物质代谢相关基因进行表达分析,以充分认识该基因对颖壳、节间及剑叶叶鞘发育的影响,为进一步明确该基因功能奠定基础。
1 材料与方法
1.1 试验材料
常规品种粳稻长粒粳经1%甲基磺酸乙脂(EMS)诱变后,在其M2群体中鉴定出稳定遗传的兼有金黄色颖壳与节间表型的突变体gh881。
1.2 遗传分析及定位群体的构建
2015年春季于海南陵水收获突变体种子,当年夏季种植于浙江富阳,以gh881为母本,与野生型和籼稻中恢8015分别杂交;冬季于海南陵水种植BC1F1、F1,观察BC1F1、F1植株和穗部表型并混合收获;2016年夏于浙江富阳种植BC2F2、F2群体,于抽穗期观察植株表型并统计野生型与突变型的分离比例。BC1F1、F1、BC2F2、F2群体用于遗传分析及基因定位。所有材料的种植管理均参照常规。
1.3 表型及两亲本间重要农艺行性状的考查
表型的考查在分离群体BC2F2中进行,在成熟期目测考查颖壳与节间颜色。于孕穗期,在gh881与野生型植株中,观察颖壳、节间及剑叶叶鞘颜色的动态变化;完熟期,随机各取10株,主要考查包括千粒重、单株有效穗数、每穗总粒数、每穗实粒数和株高等农艺性状。利用SAS软件一般线性模型进行两者间农艺性状的差异显著性检验[18]。
1.4 DNA的提取
于抽穗期,取长粒粳、gh881、中恢8015以及F2群体植株中944个隐性单株的叶片,并随机选取10株显性植株的适量叶片,采用改良的CTAB法提取全基因组DNA[19]。
1.5 目的基因的精细定位及候选基因分析
基因定位采用传统的图位克隆方法。首先,对本实验室519对SSR公共引物及166对InDel标记检测gh881与中恢8015之间基因组DNA的多态性。随机选取F2群体中10个隐性单株及10个显性单株的DNA等量混合,形成隐性单株DNA混池和显性单株DNA混池;并选取出均匀分布在水稻12条染色体的分子标记对隐性单株DNA混池和显性单株DNA混池进行PCR扩增,在隐性单株DNA混池中检测出发生连锁的分子标记,再利用77个隐性单株验证这些连锁标记以确定这些分子标记与突变位点的连锁关系,初步定位目的基因的候选区间。在此基础上,基于网站Gramene(http://www. gramene.org/) 公布的籼稻93-11和粳稻日本晴全基因组序列,对初定位区间内的序列进行比对,寻找两者之间的插入/缺失(InDel)位点,利用软件Primer Premier 5.0设计InDel引物并筛选双亲间具有多态性的标记(表1),对目的基因进行精细定位。
基于网站水稻基因组注释工程数据库(http://rapdb.dna.affrc.go.jp)预测精细定位区间内的所有开放阅读框(open reading frame, ORF),根据其全长序列,设计测序引物,采用高保真DNA聚合酶KOD-Plus-Neo分别扩增gh881突变体与野生型开放阅读框的基因组序列,并结合序列比对(MegAlign,DNASTAR-Lasergene 6)结果与测序峰图(SeqMan,DNASTAR-Lasergene 6)对比,确定突变位点,进一步确定目的基因。
1.6 木质素与类黄酮化合物代谢相关基因表达分析
采用qPCR检测颖壳颜色相关基因在gh881与野生型不同发育时期的幼穗、节间以及剑叶叶鞘的时空表达差异。利用TIANGEN植物组织总RNA提取试剂盒分别提取WT与突变体gh881不同时期幼穗、节间和剑叶叶鞘的总RNA,采用QIAGEN去基因组DNA试剂盒(RNase-Free DNase Set)去除基因组DNA后,采用实时定量PCR逆转录试剂盒(ReverTra Ace qPCR RT Master Mix,TOYOBO)进行第1链cDNA的合成。最后,采用TAKARA SYBR qPCR Mix的反应体系在Roche Light Cycle 480上进行qPCR。内参基因为OsActin1(登录号为LOC_Os03g08020)。针对黄酮类化合物与木质素生物合成途径,我们选取其中的几个关键酶基因及一个转录因子利用qPCR进行表达分析,检测目标基因包括:OsMYB48、Os4CL、OsCHI[4]、OsCAD2[6]和OsPAL4[15]。

表1 本研究所用的引物Table 1. Primers used in this study.
2 结果与分析
2.1 突变体gh881表型特征
成熟期,野生型长粒粳的谷粒颖壳为正常的淡黄色,节间也表现为正常的浅绿色;但突变体gh881的谷粒颖壳与节间均呈现金黄色(图1)。在主要农艺性状方面,除单株有效穗数与野生型没有显著差异外,突变体gh881株高、结实率、每穗总粒数、每穗实粒数和千粒重等主要农艺性状均极显著降低(表2),可能由于突变体内颖壳和节间颜色变化导致其正常的光合作用受到影响,从而影响了正常的生长发育和产量形成。
2.2 突变体gh881表型发育进程分析

图1 突变体gh881的表型分析Fig. 1. Phenotype analysis of mutant gh881.

表2 野生型与gh881的主要农艺性状比较(平均值±标准差, n=10)Table 2. Comparison of main agronomic traits between gh881 and its wild type(WT)(Mean±SD, n=10).

图2 gh881与野生型(WT)各个时期穗部、节间以及剑叶叶鞘颜色的变化与对比Fig. 2. Panicle, internode and flag leaf sheath color in various developmental periods of gh881 and its wild type(WT).
参照程式华等[20]水稻稻穗分化发育各期形态特征的鉴别方法,分别观察野生型和gh881突变体抽穗前15 d(S1)、抽穗前7 d(S2)、抽穗前3 d左右(S3)以及始穗期(S4)和开花当日(S5)五个时期的小穗、节间以及剑叶叶鞘的颜色变化情况(图2)。结果发现,野生型穗部颜色变化正常,从S1到S5期,由白色逐渐转变为正常的青绿色(图2-A),而在gh881中,S1时期的穗部颜色与野生型并无明显差异,均为乳白色(图2-A);自S2时期开始,突变体的穗部颜色开始转变为黄色,而这个差异首先在芒上开始出现(图2-A,图2-B-Ⅰ、Ⅱ,颜色变化黑色箭头所示);至S3时期,在野生型颖壳颜色由白转绿的过程中(图2-A-S3,图2-B-Ⅴ,Ⅵ),突变体gh881的颖壳则表现为一种黄绿中间色(图2-B-Ⅲ、Ⅳ);此过程一直延续至始穗期,gh881穗部(包括颖壳、芒以及枝梗)基本都变为金黄色(图2-A),仅穗底部的部分小穗仍在进行由白转黄的转变(图2-B-Ⅶ、Ⅷ);至开花当日,穗部基本全部抽出,突变体的穗部颜色全部都转换为金黄色(图2-A),此后整个颖壳都表现为更明显的金黄色。

表3 gh881位点的遗传分析Table 3. Segregation of gh881 locus.
在此转变过程中,野生型剑叶叶鞘颜色变化一直处于由基部的白色转为浅绿色、再向青绿色转变(图2-C);但突变体gh881的剑叶叶鞘在S1到S3等3个时期则是由基部的白色转为淡黄色、并逐步转变为金黄色(图2-C),而在始穗期和开花期又逐渐变为淡黄色(图2-C)。而整个过程中,野生型的节间都呈现为浅绿色,gh881节间则为金黄色(图2-D)。
2.3 金黄色颖壳与节间基因的遗传分析和图位克隆
针对以上表型,分别观察BC1F1、F1、植株和BC2F2和F2分离群体内颖壳和节间的表型并统计其分离比,发现BC1F1、F1植株的颖壳颜色和节间以及剑叶叶鞘颜色均与野生型相同,而在gh881分别与野生型和中恢8015杂交所得的BC2F2和F2群体中各观察1000个单株,野生型和突变型的分离比例经卡方检验结果显示(表3),该突变表型符合3∶1分离比,表明该金黄色颖壳与节间表型受1对隐性核基因控制。
对实验室内519对SSR引物[21]与166对InDel引物[22]进行gh881与中恢8015之间的多态性筛选,共获得160对均匀分布在水稻12条染色体且在双亲间具有多态性的分子标记,利用这些标记对的突变型DNA混池和野生型DNA混池进行连锁,发现两池间在第2染色体短臂的分子标记RM4355和RM12729间发生了偏分离,紧接着利用77个单株对这两个标记进行连锁验证并确定了该初定位区间,遗传距离约9.9 cM。进一步在此区间内开发了9对多态性InDel分子标记(表1),利用944个隐性单株,最终将GH881位点锁定在FH-13和RH-25之间,物理距离约33.2 kb(图3-A)。
2.4 候选基因分析
基于水稻基因组注释工程数据库(http://rapdb.dna.affrc.go.jp)所公布的日本晴(Nipponbare)全基因组序列,对上述锁定的33.2 kb区间内所预测的的4个开放阅读框(ORFs)进行序列分析(图3-A),对这四个ORF设计测序引物进行PCR扩增其基因全长序列,经测序比对发现,其中第3个ORF(Os02g0187800)编码一个肉桂醇脱氢酶(cinnamyl-alcohol dehydrogenase),是合成木质素前体物质必不可少的,该基因突变可导致水稻金黄色颖壳与节间表型[6];该基因包含4个外显子和3个内含子,编码363个氨基酸;gh881突变体在其第4外显子的第3 563位碱基处发生了一个单碱基的转换(G→A)(图3-B、C),从而导致其第297位氨基酸由原来的甘氨酸(Gly)突变为天冬氨酸(Asp)(图3-C)。因此,推测gh881的突变表型是由OsCAD2基因(Os02g0187800)单碱基突变所致。
2.5 木质素和类黄酮化合物代谢相关基因的表达分析
通过qPCR 检测野生型和突变体中木质素和类黄酮化合物代谢相关基因的表达。与野生型相比,突变体中包括OsCAD2在内的5个基因在S1时期的幼穗中的表达量均显著上调(图4-A);S2和S3时期,除OsMYB48表现为显著下调外,其他基因均显著上调,尤其OsPAL4的表达量上调极为显著,分别高达9.4倍和3.6倍(图4-B、C);至S4时期,OsPAL4的表达量仍呈现较高的上调模式,OsMYB48则表现为显著下调,而其他基因没有显著差异(图4-D)。在突变体的节间中则呈现不同的表达模式,OsCHI的表达显著上调、OsMYB48 无显著差异外,其他基因均显著下调(图4-E);在其剑叶叶鞘中5个基因均呈极显著上调模式(图4-F)。可见,突变体中OsCAD2基因的隐性突变会引起其植株体内木质素与类黄酮化合物代谢相关基因在不同发育时期幼穗和组织中表达模式的改变,而且此效应在剑叶叶鞘中最为显著。
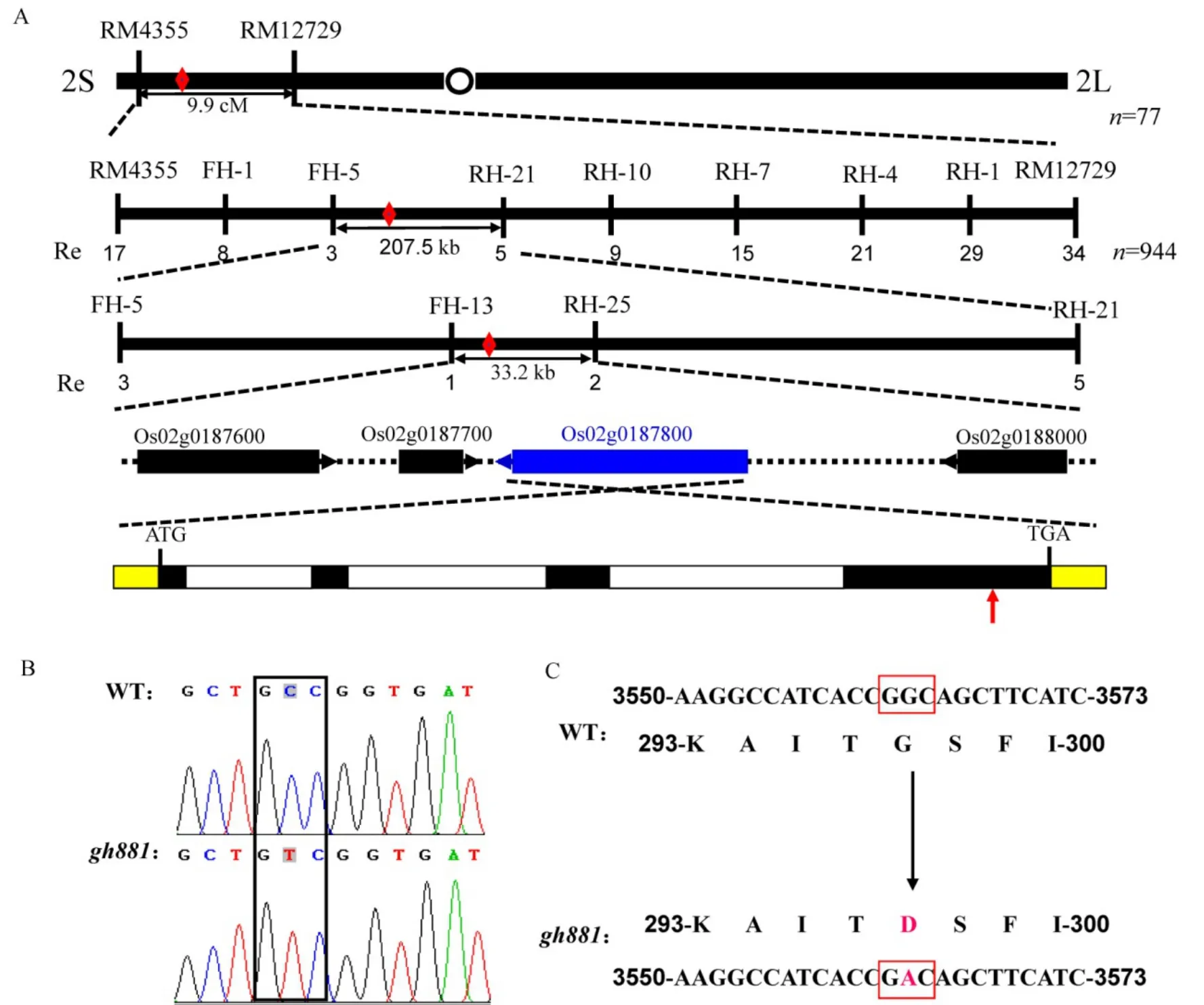
图3 gh881突变基因的图位克隆Fig. 3. Map-based cloning of the gh881 mutant gene.
3 讨论
本研究针对一个由长粒粳化学突变而来的金黄色颖壳与节间突变体gh881,对其开展表型分析和突变位点的基因定位研究。结果表明,该表型受1对隐性核基因控制,位于第2染色体短臂的一个肉桂醇脱氢酶基因OsCAD2的第4外显子内发生了一个单碱基的转换(G→A),此结果符合EMS化学诱变常出现(G→A或C→T)[23]的诱变结果;该单碱基突变导致其第297个氨基酸由甘氨酸转变为天冬氨酸Asp。而甘氨酸是非极性脂肪族类氨基酸,疏水性强,天冬氨酸则是极性R基带负电荷的氨基酸,亲水性强,还易形成氢键,由于两种不同性质的氨基酸之间的变化,可能导致CAD蛋白空间三维构象变化及其底物结合位点的变化,从而使得其蛋白功能显著下调。因此,我们认为OsCAD2单碱基的转换突变是导致gh881金黄色颖壳与节间以及剑叶叶鞘表型的原因。
水稻金黄色颖壳和节间突变体gh2是由于OsCAD2基因编码的蛋白序列内第185位甘氨酸突变为天冬氨酸,其所在突变位点仅包含2个结构域[聚酮化合物合酶和烯酰还原酶结构域和NAD(P)-结合域][24-27],导致其CAD酶活性降低、且检测不到SAD活性,使得对-羟基苯基木质素、愈创木基木质素和紫丁香木基木质素几乎均显著减少,出现金黄色颖壳和节间,但是并不影响突变体的正常生长发育[6]。而本研究中的突变体gh881的隐性突变导致其第297位氨基酸同样由甘氨酸突变为天冬氨酸,但该位点存在于多个结构域中,不仅包括聚酮化合物合酶和烯酰还原酶结构域,还包括NAD(P)-结合域和C-末端醇脱氢酶结构域[24-27]。因此,其效应比gh2中的突变更强,这也是本研究中的gh881突变体不仅表现为颖壳、节间和剑叶叶鞘的金黄色,其结实率、千粒重等产量性状也受到了严重影响,可见OsCAD2基因不同位点的突变不仅会引起水稻植株组织器官转为金黄色,还会影响其正常的生长发育(表2)。
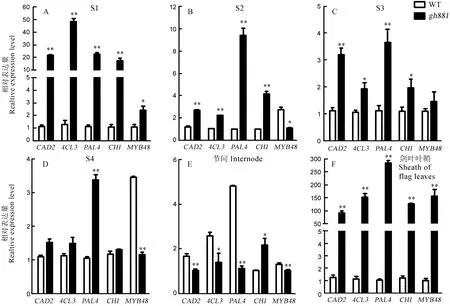
图4 qPCR检测野生与gh881中相关基因的表达Fig. 4. Expression analysis of genes associated with golden hull and lignin metabolic pathway by qPCR in WT and the gh881 mutant.
结合qPCR分析,我们观察了gh881穗部发育4个时期、节间及剑叶叶鞘颜色的变化。首先,所检测的木质素及类黄酮化合物相关基因在穗部、节间及剑叶叶鞘均有表达,且集中在幼穗发育早期,而其表达量在突变体内的更早时期就出现了上调,说明木质素的积累可能先于gh881穗部表型的变化,这与前人研究结果一致[8]。其次,在幼穗发育的早期(S1)这些基因的上调表达量差异最为显著、S2及S3表达量差异逐渐缩小;至抽穗期(S4),除OsPAL4外,其他基因的表达量与野生型基本无差异,gh881穗部的金黄色表型在S4时期基本稳定也与此趋势基本一致。另外,由于gh881中基因OsCAD2突变,该基因在节间与剑叶叶鞘表达量分别是显著下调和极显著上调,且导致木质素和类黄酮化合物代谢途径中相关基因表现为同样的趋势,其中,在剑叶叶鞘中影响效应最为显著,可见该基因突变能够影响节间与剑叶叶鞘木质素和类黄酮物质的代谢。突变体中OsCAD2基因突变会引起其植株体内木质素合成基因与类黄酮化合物代谢相关基因表达模式的改变(OsPAL4始终极显著变化以及转录因子OsMYB48表达量变化等),进一步证明OsCAD2在水稻植物色素相关物质尤其是木质素类物质合成与代谢中具有重要作用,且可能反馈调节其他相关基因表达。
木质素是水稻次级代谢产物之一,其含量不仅影响水稻对旱涝、细菌或病毒等生物与非生物胁迫的抗性[28-30],还影响其农副产品属性,如其秸秆可以作为饲料、造纸和生物燃料的制备等[31,32]。本研究针对突变体gh881的表型分析和OsCAD2基因的定位,为进一步研究其参与木质素代谢及调控机理奠定了坚实基础;也为进一步结合分子标记辅助选择技术选育适当木质素含量的水稻新品种以增强水稻新品种的抗逆性、改善水稻农副产品属性,提供了优异的种质资源。
[1] Fitzgerald M A, McCouch S R, Hall R D. Not just a grain of rice: the quest for quality. Trends Plant Sci, 2009, 14: 133-139.
[2] Robards K, Antolovich M. Analytical chemistry of fruit bioflavonoids. Analyst, 1997, 122(2): 11-34.
[3] Furukawa T, Maekawa M, Oki T, Suda I, Iida S, Shimada H, Takamure I, Kadowak K. The Rc and Rd genes are involved in proanthocyanidin synthesis in rice pericarp. Plant J, 2007, 49(1): 91-102.
[4] Hong L, Qian Q, Tang D, Wang K, Li M, Cheng Z K. A mutation in the rice chalcone isomerase gene causes the golden hull and internode 1 phenotype. Planta, 2012, 236(1): 141-151.
[5] Shao T, Qian Q, Tang D, Chen J, Li M, Cheng Z K, Luo Q. A novel gene IBF1 is required for the inhibition of brown pigment deposition in rice hull furrows. Theor Appl Genet, 2012, 125: 381-390.
[6] Zhang K W, Qian Q, Huang Z J, Wang Y Q, Li M, Hong L L, Zeng D L, Gu M H, Chu C C, Cheng Z K. Gold hull and internode2 encodes a primarily multifunctional cinnamyl-alcohol dehydrogenase In rice. Plant Physiol, 2006, 140 (3): 972-983.
[7] Tobias C M, Chow E K. Structure of the cinnamylalcohol dehydrogenase gene family in rice and promoter activity of a member associated with lignification. Planta, 2005, 220(5): 678-688.
[8] Hirano K, Aya K, Kondo M, Okuno A, Morinaka Y, Matsuoka M. OsCAD2 is the major CAD gene responsible for monolignol biosynthesis in rice culm. Plant Cell Rep, 2012, 31(1): 91-101.
[9] Doxin R A, Paiva N L. Stress-induced pheny lpropanoid metabolism. Plant Cell, 1995, 7: 1085-1097.
[10] Humphreys J M, Chapple C. Rewriting the lignin roadmap. Curr Opin Plant Biol, 2002, 5(3): 224-229.
[11] Iwata N, Omura T. Linkage studies in rice plants (Oryza sativa L.) on some mutants derived from chronic gamma irradiation. J Fac Agric Kyushu Univ, 1977, 21: 117-127.
[12] 李亮杰, 周海鹏, 占小登, 楚宗丽, 程式华, 曹立勇.一个水稻金黄色颖壳和节间基因的遗传定位. 中国水稻科学, 2008, 22(4): 432-434. Li L J, Zhou H P, Zhan X D, Chu Z L, Cheng S H, Cao L Y. Genetic mapping of a gold hull and internode gene in rice (Oryza sativa). Chin J Rice Sci, 2008, 22(4): 432-434.
[13] 李秋圆, 叶胜海, 张迎迎, 马晓静, 梅杰, 金庆生, 何祖华, 董彦君, 张小明. 一个水稻金黄色颖壳与节间基因的定位. 核农学报, 2012, 26(7): 983-987. Li Q Y, Ye S H, Zhang Y Y, Ma X J, Mei J, Jin Q S, He Z H, Dong Y J, Zhang X M. Molecular mapping of a gold hull and internode gene in Oryza sativa L. J Nucl Agric Sci, 2012, 26(7): 983-987.
[14] Gui J, Shen J, Li L. Functional characterization of evolutionarily divergent 4-coumarate: Coenzyme A ligases in rice. Plant Physiol, 2011, 157(2): 574-586.
[15] Tonnessen B W, Manosalva P, Lang J M, Baraoidan M, Bordeos A, Mauleon R, Oard J, Hulbert S, Leung H, Leach J E. Rice phenylalanine ammonia-lyase gene OsPAL4 is associated with broad spectrum disease resistance. Plant Mol Biol, 2015, 87(3): 273-286.
[16] 石坚. OsSHN1和OsMYB48参与水稻木质素合成调控的研究. 武汉: 华中农业大学, 2014. Shi J. Regulation of lignin biosynthesis by OsSHNl and OsMYB48 in rice. Wuhan: Huazhong Agricultural University, 2014.
[17] Zeng D L, Qian Q, Dong G J. Zhu X D, Dong F G, Teng S, Gong L B, Cao L Y, Cheng S H, Xiong Z M. Development of isogenic lines of morphological markers in indica rice. Acta Bot Sin, 2003, 45: 1116-1120.
[18] SAS Institute Inc, SAS / STAT User's Guide. Cary, NC, USA: SAS Institute, 1999.
[19] Rogers S O, Bendich A J. Extraction of DNA from milligram amounts of fresh, herbarium and mummified plant tissues. Plant Mol Biol, 1985, 5: 69-76.
[20] 程式华, 曹立勇, 占小登. 杂交水稻制种技术. 北京:金盾出版社, 2005. Cheng S H, Cao L Y, Zhan X D. Seed Production Technology of Hybrid Rice. Beijing: Jindun Press, 2005. [21] Orjuela J, Garavito A, Bouniol M, Arbelaez J D, Moreno L, Kimball J, Wilson G, Rami J F, Tohme J, McCouch SR, Lorieux M. A universal core genetic map for rice. Theor Appl Genet, 2010, 120: 563-572.
[22] Li Z, Zhang Y, Liu L, Liu Q, Bi Z, Yu N, Cheng S, Cao L. Fine mapping of the lesion mimic and early senescence 1 (lmes 1) in rice (Oryza sativa L.). Plant Physiol Biochem, 2014, 80: 300-307.
[23] Bökel C. EMS screens: From mutagenesis to screening and mapping. Meth Mol Biol, 2008, 420: 119-138.
[24] Mulder NJ, Apweiler R, Attwood T K, Bairoch A, Bateman A, Binns D, Bradley P, Bork P, Bucher P, Cerutti L, Copley R, Courcelle E, Das U, Durbin R, Fleischmann W, Gough J, Haft D, Harte N, Hulo N, Kahn D, Kanapin A, Krestyaninova M, Lonsdale D, Lopez R, Letunic I, Madera M, Maslen J, McDowall J, Mitchell A, Nikolskaya A N, Orchard S, Pagni M, Ponting C P, Quevillon E, Selengut J, Sigrist C J A, Silventoinen V, Studholme D J, Vaughan R, Wu C H. InterPro, progress and status in 2005. Nucleic Acids Res, 2005, 33: D201-D205.
[25] Bateman A, Coin L, Durbin R, Finn RD, Hollich V, Griffiths-Jones S, Khanna A, Marshall M, Moxon S, Sonnhammer E, Studholme D J Y, Eddy S. The Pfam protein families database. Nucleic Acids Res, 2004, 32: D138-D141.
[26] Madera M, Vogel C, Kummerfeld SK, Chothia C, Gough J. The SUPERFAMILY database in 2004: Additions and improvements. Nucleic Acids Res, 2004, 32: D235-D239.
[27] Gough J, Karplus K, Hughey R & Chothia C. Assignment of homology to genome sequences using a library of hidden Markov models that represent all proteins of known structure. J Mol Biol, 2001, 313(4): 903-919.
[28] Liu L, Shang-Guan K, Zhang B, Liu X, Yan M, Zhang L, Shi Y, Zhang M, Qian Q, Li J, Zhou Y. Brittle Culm1, a COBRA-Like protein, functions in cellulose assembly through binding cellulose microfibrils. PLoS Genet, 2013, 9: e1003704.
[29] Kawasaki T, Koita H, Nakatsubo T, Hasegawa K, Takahashi H, Umemura K, Umezawa T, Shimamoto K. Cinnamoyl-CoA reductase, a key enzyme in lignin biosynthesis, is an effector of small GTPase Rac in defense signaling in rice. PNAS, 2006, 103(1): 230-235.
[30] Lee D K, Jung H, Jang G, Jeong J S, Kim Y S, Do Choi Y, Kim J K. Overexpression of the OsERF71 transcription factor alters rice root structure and drought resistance. Plant Physiol, 2016, 172(1): 575-588.
[31] Kong X, Xie J, Wu X, Huang Y, Bao J. Rapid prediction of acid detergent fiber, neutral detergent fiber, and acid detergent lignin of rice materials by near-infrared spectroscopy. J Agric Food Chem, 2005, 53: 2843-2848. [32] Reddy N, Yang Y. Biofibers from agricultural byproducts for industrial applications. Trends Biotechnol, 2005, 23: 22-27.
Map-Based Cloning of OsCAD2 Regulating Golden Hull and Internode in Rice
WANG Hong#, ZHANG Yingxin#, SUN Lianping, MENG Shuai, XU Peng, WU Weixun, CHENG Shihua, CAO Liyong*, SHEN Xihong*
(China National Rice Research Institute/State Key Laboratory of Rice Biology/Key Laboratory for Zhejiang Super Rice Research, Hangzhou 311401, China;#These authors contributed equally to this work;*Corresponding author, E-mail: caolycgf@mail.hz.zj.cn, xihongshen@126.com)
【Objective】Rice pigments not only have important physiological effects on their own development, but also have been widely used in rice breeding, by-product improvement, and so on. Phenotypic analysis and gene mapping of pigment-related genes could lay the foundation for the further study of rice pigments metabolism and regulation mechanism.【Method】A golden hull and internode rice mutant, gh881, was isolated from an elite japonica cultivar Changlijing (CLJ) using ethyl methane sulphonate(EMS) mutagenesis strategy;the main agricultural traits of gh881 and its wild type(WT) were measured at mature stage; we crossed gh881 with WT and ZH8015, respectively, followed by observation of F1and BC1F1plants phenotype, and we made genetic analysis using Chi-square test in BC1F2population and gene mapping using map-based cloning in F2population; the related-genes expression of young panicles, internodes and flag leaf sheaths of gh881 and WT at various stages were studied by qPCR. 【Result】Compared with wild type, gh881 exhibited golden hull and internode at the mature stage. Except for no significant difference in the number of panicles per plant, there were significant reduction in plant height, the seed-setting rate, the number of spikelets per panicle, the number of grains per panicle and 1000-grain weight between WT and gh881. Genetic analysis and gene fine-mapping results suggested that gh881 was controlled by a single recessive gene and the mutant gene was mapped to a region of 33.2 kb in which four open reading flames (ORFs) existed on the short arm of chromosome 2 between markers FH-13 and RH-25. 【Conclusion】Sequencing analysis revealed that a single base mutation (G to A) occurred at the site 3563 bp of OsCAD2 (Os02g0187800) encoding a Cinnamyl-Alcohol Dehydrogenase (CAD), which led to a G297D mutation (codon GGC to GAC). This implied that gh881 might carry a novel allele of OsCAD2. The quantitative real-time PCR analysis showed that the relative expression level of OsCAD2 decreased significantly in the internode, while it almost increased significantly in the flag leaf sheath and panicle, and the related-genes also almost changed significantly. These results demonstrated that OsCAD2 is an important gene involved in lignin metabolic pathway, and may regulate other related-genes expression feedback.
rice; pigment; mutant; hull; internode; gene mapping
Q343.5;S511.032
A
1001-7216(2017)05-0465-10
2016-12-23; 修改稿收到日期:2017-02-08。
国家863计划资助项目(2014AA10A603);浙江省粮食新品种选育重大科技专项(2016C02050-1);中国农业科学院基本科研业务费专项(Y2016PT34);国家公益性农业科技研究专项(201403002);中国农业科学院创新工程资助项目(CAAS-ASTIP-2013-CNRRI)。
