假交替单胞菌遗传操作系统的研究进展
2017-07-05羊燕燕裘娟萍余志良
羊燕燕,裘娟萍,余志良
(浙江工业大学 生物工程学院,浙江 杭州 310014)
假交替单胞菌遗传操作系统的研究进展
羊燕燕,裘娟萍,余志良
(浙江工业大学 生物工程学院,浙江 杭州 310014)
假交替单胞菌能产生多种胞外活性物质,赋予其重要的基础研究和应用研究价值.目前已有多株假交替单胞菌完成基因组测序.基因组序列分析揭示不同的假交替单胞菌具有产生不同活性代谢产物的能力.假交替单胞菌遗传操作体系欠成熟,这极大地限制了假交替单胞菌的研究与应用.主要对近十多年来假交替单胞菌遗传操作系统研究进展进行综述,包括感受态制备,穿梭载体、自杀敲除载体及筛选标记的成功应用,以期为假交替单胞菌遗传操作系统的发展提供参考.
假交替单胞菌;遗传操作;接合转移
假交替单胞菌(Pseudoalteromonas)在分类学上属于细菌域、变形菌门、γ-变形菌纲、交替单胞菌目、交替单胞菌科、假交替单胞属[1],是一类好氧异养型、不产芽胞、具有鞭毛的革兰氏阴性细菌,能产胞外多糖[2]、色素[3]、抗菌溶菌物质[4]和胞外酶(如淀粉酶[5]、琼胶酶[6])等多种活性物质,这些活性物质有抗菌溶菌、裂解藻类和降解琼脂等多种生物学功能,在养殖业的生物防治[7]、赤潮治理[8],在抗菌抗肿瘤新型海洋药物的开发[9-10]及工业生产[11]等方面具有广阔的应用前景.
生物体内代谢途径控制着细胞活性物质的产生,了解和掌握活性物质的代谢途径及调控机理对活性物质的工业化生产十分重要.因此,构建一套适用于假交替单胞菌的遗传操作系统(利用基因工程、蛋白质工程等技术[12])是开发利用假交替单胞菌活性物质极为重要的基础工作.目前,部分假交替单胞菌全基因组序列已测定,这为遗传操作体系的构建奠定了基础,但丰富的耐多药抗性基因、药物流出泵及遗传修饰体系的存在导致假交替单胞菌遗传操作还不是很成功[13],这限制了在分子水平对该类细菌的改造,从而影响了对假交替单胞菌产生活性物质的机制及调控机理的深入研究.遗传操作是指在体外将目的DNA片段连接到载体上,形成重组DNA,并将其转入宿主细胞进行表达或沉默宿主特定基因的过程,涉及到的操作主要有感受态制备、载体构建、转化方法和筛选标记的选择等.经过十多年研究,假交替单胞菌遗传操作系统研究已有一定进展,笔者主要从感受态制备、转化方法、载体构建和筛选标记的选择等方面对其遗传操作体系的研究进展进行概述.
1 感受态细胞的制备
细胞感受态是指细菌具遗传转化的能力,即通过理化方法诱导细胞,使其处于最适摄取和容纳外来DNA的生理状态.目前鲜有假交替单胞菌感受态制备的相关研究报道.Kurusu等[14]将培养至对数生长期的Pseudoalteromonassp. PS1M3离心取菌体,用10 mL含7 mmol/L N-(2-羟乙基)哌嗪-N-(2-乙磺酸)和252 mmol/L蔗糖(pH 7.0)的缓冲液洗涤、重悬菌体两次,最后将菌体用同种缓冲液悬浮成100 μL体积的感受态细胞.Zhao等[15]将培养至对数生长期的细胞离心取菌体,用预冷的ddH2O洗涤、重悬菌体两次,最后用100 μL含20 mmol/L葡萄糖的ddH2O重悬,成功制备Pseudoalteromonassp. Bsi20429感受态细胞.此外,笔者借鉴普通大肠杆菌和枯草芽孢杆菌感受态制备条件,在感受态制备过程中使用高渗溶液作为电击缓冲液以保护嗜盐的假交替单胞菌.取Pseudoalteromonassp. R3对数生长期细胞,用10 mL含0.5 mol/L山梨醇、0.5 mol/L的甘露醇和体积分数为10%的甘油的预冷缓冲液A洗涤、重悬R3菌体两次,用10 mL含0.5 mol/L海藻糖、0.5 mol/L山梨醇、0.5 mol/L的甘露醇和体积分数为10%的甘油的缓冲液B洗涤一次,最后用100 μL缓冲液B重悬,制备R3感受态细胞.加复苏培养基复苏2 h后,涂布平板培养,结果平板上有305 个菌落生长,说明该方法可以用于制备R3感受态细胞.
2 假交替单胞菌遗传操作载体
2.1 穿梭载体
穿梭载体是将两个亲缘关系不同的复制子整合在一个质粒上,以期能在两个不同的菌种中实现复制和异源表达.目前,与假交替单胞菌有关的穿梭载体都是大肠杆菌-假交替单胞菌系列,包括CloneQ,pPACE1,pWD和pOriT-4Em.
2.1.1 质粒CloneQ
2001年,Tutino等[16]在游海假交替单胞菌PseudoalteromonashaloplanktisTAC 125中发现内源质粒pMtBL,大小为4 081 bp.将其最小复制单元片段ARS克隆至大肠杆菌表达载体pGEM-T上,构建了4.7 kb的大肠杆菌-假交替单胞菌穿梭载体CloneQ,并利用该穿梭载体在嗜冷假交替单胞菌中异源表达了α-淀粉酶基因.
2.1.2 质粒pPACE1
2001年,Kurusu等[14]在Pseudoalteromonassp. PS1M3中获得了大小为3.1 kb的内源质粒pPS1M3.在该质粒上插入大肠杆菌pHSG99载体的复制起始(ori)和氯霉素、红霉素抗性基因,获得了大小为7.0 kb的大肠杆菌-假交替单胞菌穿梭载体pPACE1.研究者将该穿梭载体成功导入到消除了内源质粒的Pseudoalteromonassp. PS1M13菌中,实现了氯霉素、红霉素双抗重组菌的稳定传代[15].
2.1.3 质粒pWD系列
Zhao等[15]从Pseudoalteromonassp. Bsi20429中获得内源性质粒pSM429,测序发现该质粒包含4个开放阅读框.将4个开放阅读框随机组合克隆至pUC19质粒,同时加入大肠杆菌pBR322质粒复制起始(pBR322 origin)和氯霉素抗性标记,构建了含有不同开放阅读框的系列大肠杆菌-假交替单胞菌穿梭载体(pWD1~pWD6).将不同穿梭载体电转化至已消除内源性质粒的Pseudoalteromonassp. Bsi20429中,结果发现4号开放阅读框是pSM429质粒的最小复制单元,而1号和2号开放阅读框在pSM429质粒稳定中起重要作用.随后,研究者利用构建的穿梭载体pWD2,在Pseudoalteromonassp. Bsi20429中成功表达了红霉素抗性基因和纤维素降解酶基因.Wang等[13]发现将pWD2穿梭载体导入到9株不同假交替单胞菌中的转化效率为10-6~10-3.
2.1.4 pOriT-4Em
Yu等[17]将接合转移起始oriT、质粒pUZ8002在E.coliDH5α中复制的元件phage f1及pSM429质粒上的repA复制起始连接到pWD2,构建了大小为6 702 bp的大肠杆菌-假交替单胞菌穿梭载体pOriT-4Em,研究者将其导入Pseudoalteromonassp. SM9913,转化效率为1.8×10-3.
2.2 自杀性敲除质粒
自杀质粒的性质是复制时需要一种特殊蛋白,即协助复制起始的蛋白,大多数细菌不产生这种蛋白质,因此当自杀质粒进入受体细胞时,不能以游离的方式复制,只能整合到受体细胞染色体上随着宿主染色体的复制而复制,自杀质粒的这个特点常用于基因的敲除.
将构建的基因缺失片断克隆至自杀质粒,在抗性选择压力下,利用缺失基因两端的同源片断,自杀质粒被迫整合到基因组上发生第1轮单交换.再在第2轮反筛选压力下得到发生第2轮单交换的敲除子.因此自杀性敲除质粒一般包括5个元件:1) 能启动在供体菌复制而不能启动在受体菌复制的复制起始[17-22];2) 用于第1轮正向筛选的抗性标记;3) 用于第2轮反筛选的标记;4) 与染色体上欲敲除基因上下游具有一定同源性的融合片段;5) 用于可诱导供体菌和受体菌接合转移的转移酶基因tra和接合转移起始oriT[13,17].目前报道的关于假交替单胞菌基因敲除的自杀质粒均包括以上5个元件.自杀敲除载体的图谱归纳见图1,两轮同源重组敲除示意图归纳见图2.
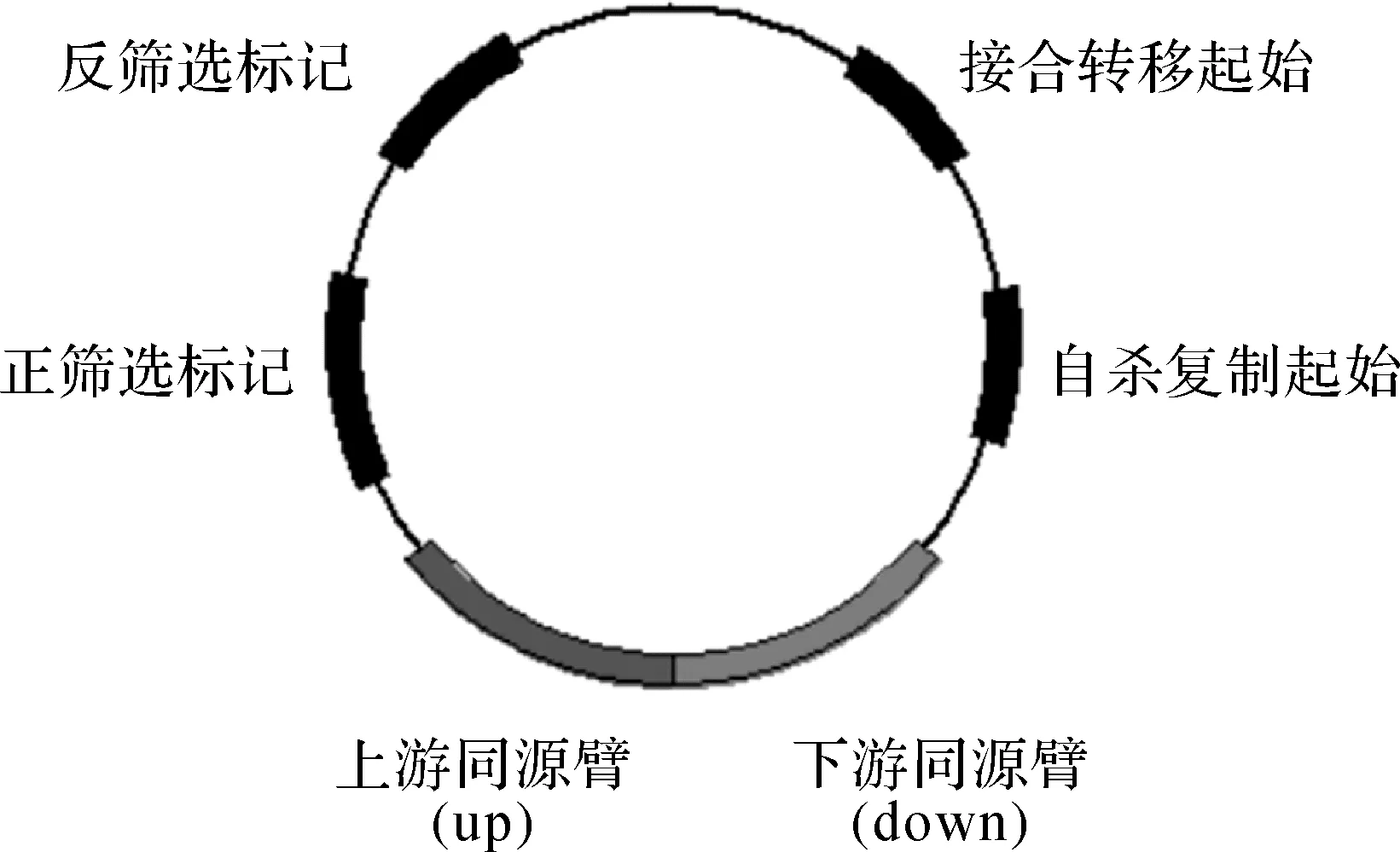
图1 自杀敲除载体示意图Fig.1 The map of suicide knock-out vector

图2 两轮同源重组敲除示意图[13]Fig.2 Schematic of the two rounds recombination event
2.2.1 质粒pVS
Parrill等[18]将广泛宿主克隆载体pJB3[23]上oriT位点克隆至表达载体pGEM-7z,构建pGEM7Z-oriT质粒;再以克隆载体pKSS[24]为模板获得pheSGly294反筛选标志,克隆至pPM13质粒[25],获得pPM13-pheSGly294质粒,并以此质粒为模板克隆P13嗜冷启动子及pheSGly294基因至pGEM7Z-oriT质粒,最终构建了自杀载体pVS.Parrilli等[26-27]利用这个自杀质粒,通过β-内酰胺酶(betalactamase,blaM)和pheSGly294基因作为正负筛选标记,构建了假交替单胞菌TAC125的蛋白酶分泌途径中gspE基因缺失的突变株(P.haloplanktisTAC125::VSgspE),导致突变株TAC125胞外蛋白酶活性降低,从而避免表达的外源重组蛋白被降解.
2.2.2 质粒pMT
Yu等[17]酶切假交替单胞菌-大肠杆菌的穿梭载体pOriT-4Em,将repA片段移除,加入Ery抗性基因和sacB反筛选标记,构建了自杀穿梭敲除载体pMT,将其导入Pseudoalteromonassp. SM9913中,敲除了菌株SM9913中编码糖基转移酶基因epsT和与鞭毛运动性有关调控蛋白基因及2个极生鞭毛蛋白丝基因.
2.2.3 质粒pK18mobsacB-Ery和pK18mobsacB-Cm
Wang等[13]将红霉素Ery基因和氯霉素Cm基因克隆到自杀质粒pK18mobsacB[28]中,构建了pK18mobsacB-Ery和pK18mobsacB-Cm敲除质粒,成功敲除了PseudoalteromonasrubraDSM6842中灵菌红素生物合成基因pigM-K,Pseudoalteromonassp. SM9913中编码生物膜和运动性基因bsmA,PseudoalteromonaslipolyticaSCSIO04301中编码尿黑酸1,2-双脱氧酶基因hmgA和Pseudoalteromonassp. SCSIO11900 中编码鞭毛马达蛋白基因fliFG[13].此外,pAK405[29]和pCVD442[30]也常用于革兰氏阴性菌的基因敲除,但至今还没有用于假交替单胞菌的成功报道.综上,已成功运用的穿梭载体都是将特定的假交替单胞菌的复制元件与大肠杆菌的复制起始克隆入载体,因此是特定假交替单胞菌专用的穿梭载体.而自杀敲除载体是假交替单胞菌及其他种属通用的敲除载体.
3 重组DNA转化方法
有效的转化方法是实现外源DNA导入受体细胞的基础[31].细菌常用的转化方法有:自然转化、电转化、热激转化和种间接合转移等.目前成功应用于假交替单胞菌遗传操作的转化方法主要有电转化和接合转移,特别是接合转移成功案例更多.因此接合转移是假交替单胞菌通用的转化方法,而电转化方法比较局限,可能是部分假交替单胞菌专用.
3.1 接合转移及成功案例
接合转移是指两细菌细胞直接接触,供体细胞中的F质粒将其携带的遗传信息转移到受体细胞的过程.F质粒通过接合转移进入受体菌依赖于接合转移起始oriT和转移区tra[32],tra区编码一个DNA松弛酶,识别、结合到oriT位点,切开质粒的一条链,并共价结合到转移链的5′端形成单链DNA,游离的单链DNA通过细胞膜IV型分泌系统(Type 4 secretion system,T4SS)进入宿主菌后形成双链DNA并在宿主中稳定传代.由于转移的片段是单链DNA,故可以减少受体菌限制修饰系统对它的降解,提高转化效率[32].
Tutino等[16]在2001年将外源敲除质粒通过接合转移的方式导入P.haloplanktisTAC125,成功构建胞外蛋白酶活性降低的P.haloplanktisTAC125突变株.2004年,Duilio等[20]通过接合转移将带有P.haloplanktisA23的α-淀粉酶的重组质粒在P.haloplanktis中实现了重组蛋白的异源表达.Egan等[33-34]通过接合转移将Mini-Tn10转座子随机插入假交替单胞菌基因组中,成功构建色素和抗真菌物质突变体库.此外,2010年本实验室在产L-氨基酸氧化酶的调控机理研究中也通过接合转移成功将Mini-Tn10转座子随机插入Pseudoalteromonassp. Rf-1基因组上[35].
3.2 电转化及成功案例
相对于接合转移,假交替单胞菌电转化方法研究较少.电转化是利用脉冲电场提高细胞膜的通透性,使细胞膜上形成纳米孔大小的微孔,将DNA转移到受体细胞中[36].2001年,Kurusu等[14]首次将电转化应用于假交替单胞菌中,7 mmol/L N-(2-羟乙基)哌嗪-N-(2-乙磺酸)和252 mmol/L蔗糖组成pH 7.0的电击缓冲液,在电压2 000 V/cm、电阻200 Ω、电容量50 μF的电击条件下,成功将他们自己构建的穿梭载体pPACE1导入消除了内源性质粒的Pseudoalteromonassp. PS1M13中,实现了重组菌的稳定传代.2011年,Zhao等[15]以含有20 mmol/L葡萄糖双蒸水溶液为电击缓冲液,成功将质粒pWD2电击导入已消除内源性质粒的Pseudoalteromonassp. Bsi20429中,并成功表达了质粒携带的红霉素抗性基因和纤维素降解酶基因[15].此后,Wang等[13]利用载体pWD2,尝试通过电转化的方法在其他假交替单胞菌中进行基因敲除,但因转化效率太低,基因敲除并未成功.
4 转化子选择标记
合适有效的筛选标记有助于快速获得目的转化子.已报道可用于假交替单胞菌遗传操作的筛选有正向筛选(抗性筛选)和两轮同源重组敲除的反向筛选.
4.1 正向筛选
抗生素抗性标记是常用的转化子筛选方法.筛选原理是在抗生素平板上只有发生转化而获得外源抗性基因的转化子才能在该种抗生素选择压力下生长,未发生转化的细胞不能生长.
4.1.1 青霉素类抗性标记
Parrilli等[26-27]等在研究构建P.haloplanktisTAC125突变株工作中将羧苄青霉素(carbenicillin,Car)、氨苄青霉素(ampicillin,Amp)作为抗性筛选标记,在30 μg/mL Car和100 μg/mL Amp抗性平板上实现了P.haloplanktisTAC125重组子的筛选.
4.1.2 氯霉素、红霉素抗性标记
2015年,Wang等[13]对9株假交替单胞菌进行抗生素抗性研究,发现9株亲株对25 μg/mL红霉素(erythromycin,Ery)和30 μg/mL氯霉素(chloramphenicol,Cm)均敏感,在基因敲除研究中以Ery为标记获得转化子P.rubraDSM 6842,P.lipolyticaSCSIO 04301.以Cm为标记获得转化子P. sp. SM 9913,P. sp. SCSIO 11900.
4.1.3 正向筛选的缺点
由于假交替单胞菌存在丰富的耐多药抗性基因和药物流出泵,抗生素抗性筛选在假交替单胞菌中易失效[13].因此,选择合适的抗性筛选标记非常重要.
4.2 反向筛选
反向筛选一般适用于两轮同源重组的遗传操作体系的第2轮筛选.反筛选标记是指在第1轮正向筛选的基础上增加1轮筛选压力,第1轮转化子在相应反筛选物质的平板上不能生长,只有发生第2轮基因重组,将反筛选标记丢失的转化子才能生长.常见的反筛选标记有:sacB基因(编码果聚糖蔗糖酶)[13,37]和pheSGly294基因(编码苯丙氨酸-tRNA合酶基因的突变体)[27,37].反筛选图示归纳见图3.

图3 反筛选图示Fig.3 Schematic of counter select
4.2.1sacB反筛选标记
sacB反筛选的原理:sacB编码蔗糖果聚糖酶,具有蔗糖水解酶和果糖转移酶双重活性,蔗糖水解酶水解蔗糖生成葡萄糖和果糖,果糖转移酶催化果糖合成果聚糖[38],果聚糖对革兰氏阴性菌具毒害作用,毒害机制还不清楚[39].当培养基中含有质量分数10%的蔗糖时,蔗糖果聚糖酶催化蔗糖水解并最终合成果聚糖,导致菌体死亡,故带有sacB基因的第1轮转化子在质量分数10%蔗糖平板上不生长而发生第2轮重组后将sacB基因丢失的转化子能生长.
4.2.2pheSGly294反筛选标记
pheSGly294反筛选的原理:当pheS编码的Phe-tRNA合酶的第294位氨基酸突变成甘氨酸后,Phe-tRNA合酶的α亚基发生突变,突变后的酶对底物特异性会降低,使得有毒的苯丙氨酸类似物氯苯丙氨酸(pCl-Phe)也能被利用,从而致死宿主菌[37,40].因此往培养基中添加氯苯丙氨酸,带有pheSGly294基因第1轮转化子不生长,只有发生第2轮重组将pheSGly294基因丢失的转化子能生长.
4.2.3 反向筛选的缺点
后续研究发现sacB和pheSGly294作为反筛选标记存在缺点[29].sacB反筛选的缺点是会发生高频率的对果聚糖有抗性的自发突变事件,使得第1轮重组子也能在质量分数10%的蔗糖平板上生长;而pheSGly294反筛选的缺点是培养基中pCl-Phe的添加对受体细胞具损伤作用.
5 结 论
近15年来,随着假交替单胞菌内源性质粒的发现、最小复制单元的分离和测序,假交替单胞菌遗传操作体系的研究已取得较大进展,包括电转化感受态的制备,穿梭质粒、敲除质粒和筛选标记的成功应用.但假交替单胞菌遗传操作体系还存在可以改进或完善的地方,比如,自杀敲除载体复制起始位点可能不够严谨;两轮同源重组敲除的反筛选标记有局限性,它们或在第2轮反筛选添加浓度下,第1轮突变子依旧生长,或在低于第2轮反筛选10倍添加浓度下依然能致死宿主菌.近来,Julia等[29]为了避免鞘酯单胞菌中sacB,pheSGly294两个反筛选标记存在的缺陷,使用了rpsL(链霉素反筛选标记)作为反筛选标记,成功实现了鞘酯单胞菌Sphingomonassp. Fr1中编码响应调节子基因phyR、选择性σ因子σEcfG、番茄红素环化酶基因crtY和八氢番茄红素脱氢酶基因crtI的敲除.这个反筛选标记有望在假交替单胞菌中应用.另外,转化方式的局限极大地限制了假交替单胞菌遗传操作体系的应用,摸索行之有效的转化体系极为迫切.近年来兴起的以RNA为向导的Cas9[41]的敲除手段也有望运用于假交替单胞菌.随着分子生物学和基因工程技术的进步,通过针对性的基因修改,探究假交替单胞菌活性代谢产物的代谢途径和调控机理,将为假交替单胞菌来源的活性产物实现工业化生产提供理论基础[42].
[1] 于子超. 海洋假交替单胞菌适应海冰环境的机制及其遗传操作体系的建立[D]. 山东:山东大学,2014.
[2] GEESEY G G. Microbial exopolymers: ecological and economic considerations[J]. Asm news, 1982, 48: 9-14.
[3] HOLMSTRÖM C, KJELLEBERG S. MarinePseudoalteromonasspecies are associated with higher organisms and produce biologically active extracellular agents[J]. FEMS microbiology ecology, 1999, 30(4): 285-293.
[4] GAUTHIER M J.Alteromonasrubrasp. nov. a new marine antibiotic-producing bacterium[J]. International journal of systematic bacteriology, 1976, 26(4): 459-466.
[5] 席宇,朱大恒,刘红涛,等. 假交替单胞菌及其胞外生物活性物质研究进展[J]. 微生物学通报,2005,32(3):108-112.
[6] DOBRETSOV S, QIAN P Y. Effect of bacteria associated with the green alga ulva reticulata on marine micro and macrofouling[J]. Biofouling, 2002, 18(3): 217-228.
[7] YU M, TANG K, LIU J, et al. Genome analysis ofPseudoalteromonasflavipulchraJG1 reveals various survival advantages in marine environment[J]. BMC genomics, 2013, 14(1): 707-718.
[8] LONG R A, QURESHI A, FAULKNER D J, et al. 2-n-Pentyl-4-quinolinol produced by a marineAlteromonassp. and its potential ecological and biogeochemical roles[J]. Applied and environmental microbiology, 2003, 69(1): 568-576.
[9] EGAN S, HOLMSTRÖM C, KJELLEBERG S.Pseudoalteromonasulvaesp. nov. a bacterium with antifouling activities isolated from the surface of a marine alga[J]. International journal of systematic & evolutionary microbiology, 2001, 51(4): 1499-1504.
[10] AUSTIN B. Novel pharmaceutical compounds from marine bacteria[J]. Journal of applied bacteriology, 1989, 67(5): 461-470.
[11] 孙星. 养殖文蛤溶藻弧菌、颉颃菌的筛选与鉴定及其颉颃物质的研究[D]. 南京:南京农业大学, 2012.
[12] 汪钊, 黄美娟, 高亮,等. 化学酶法合成D-泛解酸内酯的研究进展[J]. 发酵科技通讯, 2016,45(4): 193-198.
[13] WANG P X, YU Z C, LI B Y, et al. Development of an efficient conjugation-based genetic manipulation system forPseudoalteromonas[J]. Microbial cell factories, 2015, 14(1): 11-21.
[14] KURUSU Y, YOSHIMURA S, TANAKA M, et al. Genetic transformation system for a psychrotrophic deep-sea bacterium: isolation and characterization of a psychrotrophic plasmid[J]. Marine biotechnology, 2001, 3(2): 96-99.
[15] ZHAO D L, YU Z C, LI P Y, et al. Characterization of a cryptic plasmid pSM429 and its application for heterologous expression in psychrophilicPseudoalteromonas[J]. Microbial cell factories, 2011, 10(1): 30-41.
[16] TUTINO M L, DUILIO A, PARRILLI R, et al. A novel replication element from an Antarctic plasmid as a tool for the expression of proteins at low temperature[J]. Extremophiles, 2001, 5(4): 257-264.
[17] YU Z C, ZHAO D L, RAN L Y, et al. Development of a genetic system for the deep-sea psychrophilic bacteriumPseudoalteromonassp. SM9913[J]. Microbial cell factories, 2014, 13(1): 1-9.
[18] GIULIANI M, PARRILLI E, PEZZELLA C, et al. A novel strategy for the construction of genomic mutants of the Antarctic bacteriumPseudoalteromonashaloplanktisTAC125[J]. Methods in molecular biology, 2012, 824: 219-233.
[19] DAS A, XIE Y H. Replication of the broad-host-range plasmid RK2: isolation and characterization of a spontaneous deletion mutant that can replicate inAgrobacteriumtumefaciensbut not inEscherichiacoli[J]. Molecular genetics and genomics, 1995, 246(3): 309-315.
[20] DUILIO A, TUTINO M L, MARINO G. Recombinant protein production in antarctic gram-negative bacteria[J]. Methods in molecular biology, 2004, 267(267): 225-237.
[21] THOMAS C M. Complementation analysis of replication and maintenance functions of broad host range plasmids RK2 and RP1[J]. Plasmid, 1981, 5(3): 277-291.
[22] SAXENA M, ABHYANKAR M, BASTIA D. Replication initiation at a distance[J]. Journal of biological chemistry, 2010, 285(8): 5705-5712.
[23] BLATNY J M, BRAUTASET T, WINTHERLARSEN H C, et al. Construction and use of a versatile set of broad-host-range cloning and expression vectors based on the RK2 replicon[J]. Applied & environmental microbiology, 1997, 63(2): 370-379.
[24] KAST P. pKSS-a second-generation general purpose cloning vector for efficient positive selection of recombinant clones[J]. Gene, 1994, 138(1/2): 109-114.
[25] PAPA R, RIPPA V, SANNIA G, et al. An effective cold inducible expression system developed inPseudoalteromonashaloplanktisTAC125[J]. Journal of biotechnology, 2007, 127(2): 199-210.
[26] PARRILLI E, CUSANO A M, GIULIANI M, et al. Cell engineering ofPseudoalteromonashaloplanktisTAC125: construction of a mutant strain with reduced exo-proteolytic activity[J]. Microbial cell factories, 2006, 5(1): 1-3.
[27] PARRILLI E, VIZIO D D, CIRULLI C, et al. Development of an improvedPseudoalteromonashaloplanktisTAC125 strain for recombinant protein secretion at low temperature[J]. Microbial cell factories, 2008, 7(1): 1-10.
[29] KACZMARCZYK A, VORHOLT J A, FRANCEZCHARLOT A. Markerless gene deletion system forSphingomonads[J]. Applied & environmental microbiology, 2012, 78(10): 3774-3777.
[30] PHILIPPE N, ALCARAZ J P, COURSANGE E, et al. Improvement of pCVD442, a suicide plasmid for gene allele exchange in bacteria[J]. Plasmid, 2004, 51(3): 246-255.
[31] 王飞雁,张松涛,黄奇洪,等. 超嗜热古菌遗传操作系统研究进展[J]. 微生物学报,2009,49(11):1418-1423.
[32] SMILLIE C, PILAR GARCILLAN-BARCIA M, VICTORIA FRANCIA M, et al. Mobility of plasmids[J]. Microbiology & molecular biology reviews mmbr, 2010, 74(3): 434-452.
[33] EGAN S, JAMES S, HOLMSTRÖM C, et al. Correlation between pigmentation and antifouling compounds produced byPseudoalteromonastunicata[J]. Environmental microbiology, 2002, 4(8): 433-442.
[34] FRANKS A, EGAN S, HOLMSTRÖM C, et al. Inhibition of fungal colonization byPseudoalteromonastunicataprovides a competitive advantage during surface colonization[J]. Applied & environmental microbiology, 2006, 72(9): 6079-6087.
[35] YU Z, WANG J, LIN J, et al. Exploring regulation genes involved in the expression of L-amino acid oxidase inPseudoalteromonassp. Rf-1[J]. Plos one, 2015, 10(3): e0122741.
[36] 钟卫鸿. 基因工程技术[M]. 北京:化学工业出版社, 2007.
[37] REYRAT J M, PELICIC V, GICQUEL B, et al. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis[J]. Infection & immunity, 1998, 66(9): 4011-4017.
[38] 高翔. 小麦族植物果聚糖合成酶基因克隆及功能验证[D]. 北京:中国农业科学院, 2010.
[39] STEINMETZ M. Analysis genetique desacB, gene de structure d'une enzyme secreetee, la levanesaccharase deBacillussubtilismarburg[J]. Mol.gen.genet, 1983, 191: 138-144.
[40] KAST P, HENNECKE H. Amino acid substrate specificity ofEscherichiacoli, phenylalanyl-tRNA synthetase altered by distinct mutations[J]. Journal of molecular biology, 1991, 222(1): 99-124.
[41] CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 197-217.
[42] 魏爱英, 葛玉萍. L-赖氨酸的代谢途径到工业生产[J]. 发酵科技通讯, 2011, 40(2): 31-34.
(责任编辑:朱小惠)
Advances in genetic manipulation onPseudoalteromonas
YANG Yanyan, QIU Juanping, YU Zhiliang
(College of Biotechnology and Bioengineering, Zhejiang University of Technology, Hangzhou 310014, China)
Pseudoalteromonaswas ubiquitous in environments and can produce diverse bioactive compounds. So far, severalPseudoalteromonasisolates have been sequenced and the bioinformatics analysis has revealed their abilities to produce different bioactive compounds. However, the lack of genetic manipulation system ofPseudoalteromonasgreatly restricted their applications and studies at molecular level. Here, we describe the advances in genetic manipulation onPseudoalteromonas, including the preparation of competent cells, the successful application of shuttle vectors, suicide knockout vectors and screening markers, which provide the reference for the development of genetic manipulation system inPseudoalteromonas.
Pseudoalteromonas; genetic manipulation; conjugation
2017-03-23
国家自然科学基金资助项目(31670114);浙江省自然科学基金资助项目(LY16C010002);国家海洋经济创新发展区域示范项目(12PYY001SF08)
羊燕燕(1992—),女,浙江磐安人,硕士研究生,研究方向为应用微生物学,E-mail: 2111405109@zjut.edu.cn. 通信作者:余志良副教授,E-mail: zlyu@zjut.edu.cn.
Q933
A
1674-2214(2017)02-0100-07
