调控脂肪酸合成植物转录因子的研究进展
2017-07-05闵文莉曹喜涛季更生张国政
闵文莉,曹喜涛,2,季更生,2,张国政,2
(1.江苏科技大学 生物技术学院,江苏 镇江 212018;2. 中国农业科学院 蚕业研究所,江苏 镇江 212018)
调控脂肪酸合成植物转录因子的研究进展
闵文莉1,曹喜涛1,2,季更生1,2,张国政1,2
(1.江苏科技大学 生物技术学院,江苏 镇江 212018;2. 中国农业科学院 蚕业研究所,江苏 镇江 212018)
转录因子是植物特有的一类调节蛋白,对植物生长发育及抗逆性等有重要的作用,尤其是调控脂肪酸合成与油脂积累的转录因子,对生产生物能源起到关键作用,利用转录因子提高作物产油量已逐渐受到青睐,为未来获得高产油脂的优良作物作铺垫.对与脂肪酸合成相关的几种主要转录因子进行了概述,阐释其结构、功能及研究进展等,并对转录因子的应用进行了展望,以期为更多转录因子的结构和功能机制的研究提供基础.
脂肪酸;油脂积累;转录因子
生物柴油由脂肪酸酯类组成,是一种洁净的生物燃料[1],植物油是生物柴油的原料,所以植物油成为柴油替代品的潜能很大.如今对于如何提高油料作物含油量的研究很多,周丹等[2]已对植物油脂合成途径、关键基因及合成调控进行了概述,种子中丰富的油脂在工农业中有重要的经济价值.根据国际能源机构(IEA, US)的报道,目前运输所消耗的能源占据全球能源的23%,并且此数据仍然保持增长趋势.据评估,至2030年仅来自运输行业所产生的温室气体至少增长20%.为了维护能源供应平衡以及控制温室效应所带来的环境危害,IEA倡导开发高能源效率的新能源.据Buijs等[3]报道,高级生物燃料丁醇、倍半萜类化合物和脂肪酸乙酯会逐渐取代化石燃料.高级生物燃料因其环保、安全及可持续利用的特性,逐渐受到人们的青睐,有望成为未来运输业的主要能源.
转录因子是植物体中环境胁迫应答反应所激发产生的一类蛋白,也称为反式作用因子,其通过功能结构域与启动子顺式作用元件或其他转录因子发生特异性相互作用以激活或抑制转录[4].近年来,植物转录因子的研究已成为热点之一,调控脂肪酸合成与油脂积累的转录因子更是备受青睐,这些转录因子分属各个家族,包括AP2家族、B3家族、DOF家族、HAP3/CBP和CHD3等.转录因子并不单独作用,而是形成代谢调控网络,共同发挥调控脂肪酸合成的作用.
1 调控脂肪酸合成的转录因子
1.1 WRI1转录因子
转录因子WRINKLED(WRI1)是由Focks和Benning[5]在拟南芥中首次发现并命名,与野生型拟南芥相比,WRI1基因突变使得种皮褶皱,种子含油量减少80%.WRI1蛋白含有两个AP2/EREBP结构域,是APETALA2(AP2)家族一员,其具体结构尚不清楚,但已经有了相关的研究进展.通过拟南芥、油棕及其他许多物种中WRI1蛋白的序列对比,Ma等[6]发现了一段短小的、非常保守的蛋白序列“VYL”,对此段序列进行定点诱变研究,发现作物出现含油量减少的表型,此研究可以表明“VYL”蛋白序列在许多植物的种子或非种子组织中,对于植物合成油脂起到关键作用.Subroto等[7]建立了油棕WRI1转录因子的cDNA文库,克隆出的WRI1基因片段长1 014 bp,并且两个克隆产物仅有8 个氨基酸的差异;在鳄梨果皮中,Murphy等[8]发现WRI1的3 种同源基因在三脂酰甘油积累阶段高度表达,系统进化树分析表明WRI1基因家族较为保守.这些研究均可以表明不同植物中WRI1转录因子的序列相对保守,在调控脂肪酸合成与油脂积累的过程中有重要的作用.
WRI1转录因子的功能已得到验证,WRI1蛋白主要在脂肪酸的合成和糖酵解后期发挥调控作用[9-11].目前已有许多学者对WRI1转录因子的调控机制进行了研究:Pouvreau等[12]发现从拟南芥克隆出的WRI1转录因子直接与AW-box结合,而AW-box正位于脂肪酸合成相关基因的上游区域;Fukuda等[10]通过启动子分析验证了其他转录因子也结合到AW-box上,与WRI1共同调控编码质粒乙酰辅酶A羧化酶的基因,而乙酰辅酶A羧化酶催化过程是脂肪酸合成的限速步骤.因此,AW-box是WRI1与其他转录因子协同调控的桥梁,在WRI1发挥作用的过程中有着重要的作用.据文献报道:WRI1转录因子不仅可以调控脂肪酸的合成,还在植物光合作用中发挥一定作用,通过与GT1-element或GCC-box单独结合,或同时与二者结合,将脂肪酸合成与光合作用过程整合在一起,提高了植物含油量[13].
目前对于WRI1转录因子的应用主要集中于在作物中过表达以达到改良作物产油量的目的.研究者已从多种植物中克隆、验证了WRI1同源基因并进行了应用,包括玉米[12]、甘蓝型油菜[13]、油棕[7,14]、拟南芥[6,15]及鳄梨[8]等.An等[16]从拟南芥中克隆WRI1基因,在油料作物亚麻荠中过表达,得到的植株与野生型相比,产油量提高了14%,重组亚麻荠成为更有发展前景的作物用于生产生物燃料.为了进一步提高植物产油量,除了过表达WRI1基因外,对脂肪酸的代谢过程进行代谢改造逐渐成为一个研究热点.糖代谢与脂肪酸合成途径相竞争,这意味着碳流向脂肪酸的量减少.通过过表达促进脂肪酸合成的基因与敲除竞争途径的关键基因,就会获得高产脂肪酸的植株或微生物[17].WRI1,二酰甘油酰基转移酶(DGAT1-2)和油质蛋白(OLE1)共同过表达,以及ADP-葡萄糖焦磷酸化酶(AGPase)和过氧化物体ABC转运子(PXA1)的共抑制,使得油脂积累量显著提高[18].
1.2 DOF转录因子
DOF转录因子的全称为DNA-binding with one finger,是一种在N末端含有C2—C2锌指结构的植物特有的转录因子[19].DOF蛋白包含了N末端较为保守的DNA结合结构域和C末端较为多变的转录调控结构域,这是DOF蛋白在植物生长发育中发挥功能的两个主要结构域,识别的核心序列是AAAG,序列中任何突变都会影响DOF蛋白与DNA结合的特异性[20-21].DOF蛋白已从多种植物中发现并克隆,其中包括大豆、拟南芥、西红柿以及黄瓜[22]等,此外,已研究报道了DOF转录因子参与植物多种生理生化过程,如碳代谢[23]、种子发育[24]以及促进种子油脂含量的增加[25]等.
据报道,从西红柿中分离出的DOF转录因子可以增加西红柿中抗坏血酸的含量,并且增强植株对盐的抗性,虽然作用机制尚不清楚,但其功能可改良植物品种,改善人类饮食的营养成分[26].在香蕉中,DOF蛋白是一种转录抑制子,参与了果实成熟的过程,从香蕉中克隆25 个DOF同源基因,在果实成熟的不同阶段,各个DOF基因出现差异性表达,发挥不同的作用.研究发现,DOF转录因子与一个香蕉果实成熟过程中存在的潜在调控子ERF9相互作用,ERF9是转录激活子,相互作用后一起调控与果实成熟相关的10 种基因的表达,这为香蕉果实成熟的转录调控网络提供一个新视角[27].早在2007年,Wang等[28]就在拟南芥中异源表达大豆DOF转录因子,发现重组拟南芥中脂肪酸和脂质含量均增加,随后Ibáez Salazar等[25]克隆DOF基因,构建质粒,并转化进莱茵衣藻内,对重组莱茵衣藻进行分子分析,发现其与野生莱茵衣藻相比,脂肪酸含量提高大约两倍,这是因为DOF基因过表达的同时促进了脂肪酸合成的相关酶的表达,包括ACP还原酶-脂肪酸合酶的一个亚单位,这为使用莱茵衣藻生产生物燃料提供一个新方向,也证实了DOF转录因子参与了脂肪酸合成过程的调控.
1.3 FUS3转录因子
FUSCA3(FUS3)转录因子含有B3结构域,通过与RY元件结合,在植物中发挥调控种子发育与成熟的作用[29-30].据Elahi等[30]的研究报道,从甘蓝型油菜中克隆出FUS3基因,对其B3结构域下游区域进行突变,会导致种子含油量减少,促进脂肪酸合成的转录因子的表达也减少,这表明FUS3在脂肪酸合成的过程中有着必不可少的作用.许多关于FUS3的研究是以模式植物拟南芥作为研究对象的,在拟南芥中,FUS3基因的表达调控脱落酸和赤霉素的合成,而这两者在胚发育阶段控制细胞周期[31].此外,敲除FUS3基因C-端结构域会使得mRNA和蛋白质高水平积累,并降低脱落酸和赤霉素对植物影响的敏感性[32].据报道,FUS3的耦合子AKIN10通过减少其磷酸化的方式增加FUS3的稳定性,并且FUS3基因N末端对AKIN10的磷酸化有重要作用[33].
Elahi等[30]克隆出甘蓝型油菜中编码FUS3转录因子的基因,由927 个核苷酸组成,对FUS3转录因子的下游区域进行突变,会出现角果数及种子含油量均有所下降的表型,同时编码促进脂肪酸合成的转录因子LEC1,LEC2,BnABI3和WRI1的基因在表达水平上也降低.对BnFUS3进行靶向操作可提高甘蓝型油菜的产油量,使其成为重要的经济型油料作物.
ABI3,FUS3和LEC2通常称为AFL,虽然它们有各自特异的功能,但在种子成熟以及油脂合成等主要生理过程中功能相似,彼此相互调节,有着密不可分的关系[34].Roscoe等[35]研究了AFL发挥功能的机制,发现ABI3主要调控储存蛋白的含量,FUS3主要调控脂质的含量,此外LEC2和FUS3在种子胚的不同阶段发挥作用,突变其中一种转录因子,都会对种子的成熟发育及种子含油量产生不利影响.
1.4 LEC1及LEC1-LIKE 1(L1L)
拟南芥LEAFY COTYLEDON 1(LEC1)属于HAP3/CBP家族一员,是首次验证出的种子发育的调控子,是NF-YB蛋白家族的一员,而NF-YB蛋白是HAP3亚单位中的CCAAT-box结合因子.据文献报道,在种子胚的早期,LEC1在叶子由球形成长为弯曲叶子的阶段进行了表达[36];此外LEC1,LEC2和FUS3控制胚的发育,若其功能缺失,会诱导成熟叶子在胚中发育,最终形成毛状体和积累花青素[37].朱红林等[38]根据已报道的拟南芥LEC1序列设计引物,克隆了大豆中长度为850 bp的LEC1同源序列,测序结果显示大豆和拟南芥中的LEC1基因高度同源,有完整的阅读框.
LEC1已被验证在种子发育过程中能促进与脂肪酸合成相关基因的表达,在油菜中表达LEC1使得种子油脂含量增加7%~16%,若下调其表达量,含油量会减少9%~12%,但均未引起脂肪酸组成的变化,这表明LEC1引起了糖酵解、脂肪酸合成等过程中相关酶转录水平的变化,使得碳流向脂肪酸合成途径[39].据报道,在转基因油菜中过表达甘蓝型油菜LEC1及L1L转录因子,种子含油量明显增加,并且对植物其他生长性状没有不利影响;此外,LEC1在发育的种子及角果中提高了蔗糖合成相关基因的表达,使得蔗糖和果糖含量增加,碳源增加进而油脂含量增加,可说明LEC1促进碳向脂肪酸转化[40].
据Mu等[41]报道,在质粒脂肪酸合成途径中,LEC1过表达可促进其他基因的表达,这些基因编码超过58%已知的酶,其中包括脂肪酸合成中限速步骤中的关键酶乙酰辅酶A羧化酶;此外还促进糖酵解及油脂积累相关基因的表达.Ke等[42]在芝麻中建立表达序列标签,简称ESTs,编码调控脂肪酸合成的相关转录因子的同源基因,如LEC1,PICKLE (PKL)和WRI1等均已得到验证,这是在芝麻种子中首次发现.
1.5 转录因子的代谢调控网络
脂肪酸是合成生物燃料的基础,有很多基因参与其中进行调控(如表1所示),同时脂肪酸从头合成的过程中受到很多基因的共同调控,形成代谢调控网络(如图1所示).研究表明LEC1和LEC2等上游因子调控WRI1,ABI3和FUS3等转录因子基因表达,同时WRI1也对FUS3存在调控作用[48-49].LEC2的异位表达会诱导WRI1,ABI3和FUS3等多个基因RNA的积累,推断其与LEC1,FUS3和ABI3在种子的成熟过程中共同发挥调控作用.异位过表达LEC1同样激活了WRI1,因此推测在调控WRI1的表达上LEC1和LEC2可能以互不干扰的方式来调控WRI1基因的表达[48].
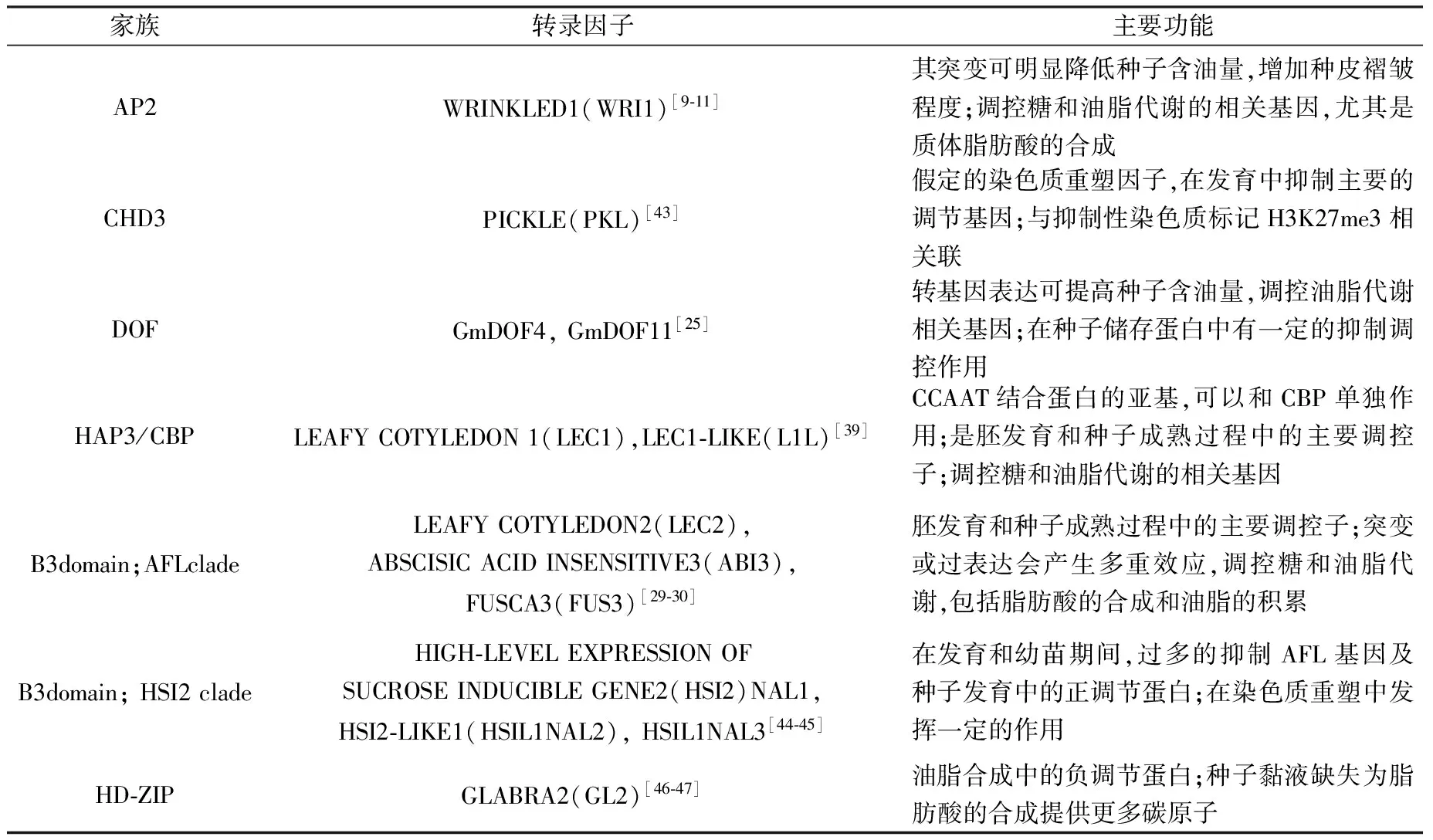
表1 调控脂肪酸合成与油脂积累的相关转录因子Table 1 Relevent transcription factors regulating fatty acid synthesis and oil accumulation
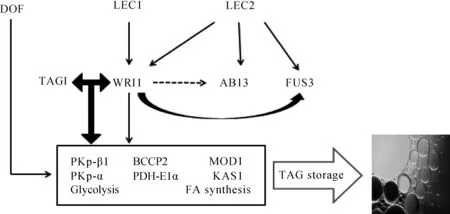
图1 以WRI1为中心的代谢调控网络Fig.1 Regulation networks of WRI1 transcription factor
油棕的非种子器官中,在没有发现LEC1,LEC2等表达的情况下WRI1大量表达,推测非种子器官中的WRI1蛋白可能与其他上游调控因子或独立调控脂肪酸合成.烟草中共表达WRI1和DGAT1时,发现糖酵解后期和脂肪酸的合成过程均上调,使得烟草叶子中三脂酰甘油的含量增加了100倍[50],这表明WRI1和DGAT1共同发挥了上调脂肪酸的功能.Baud等[51]研究发现转录因子WRI1的靶基因PKp-β1 (Plastidicpyruvate kinase beta subunit 1),BCCP2 (Biotin carboxyl-carrier protein 2)均与糖酵解与脂肪酸合成相关.同时,相关研究还表明WRI1与TAG1 (Triacylglycerol biosynthesis defect 1) 在油脂积累过程中存在协同作用.
2 结 论
尽管上述植物中的各类转录因子参与脂肪酸调控已被证实,但其主要应用集中在作物品质改良上,如拟南芥、玉米和油棕等,而关于其在微生物中应用的报道极少.提高油料作物的含油量是一个长期目标,并且脂肪酸代谢与许多其他代谢途径紧密相连,单一改造某个途径对提高脂肪酸的含量没有太大影响.若要获得高量脂肪酸,一方面需增加碳源向脂肪酸合成途径的流通,另一方面是要尽量阻断脂肪酸的消耗途径(如β-氧化),减少脂肪酸的消耗.调控脂肪酸合成相关的转录因子的研究为高产油脂做了铺垫,但目前仍有很多局限:1) 转录因子的来源有限,许多转录因子的结构特征尚不清楚,并且很多植物(如桑树)还未得到鉴定;2) 转录因子的调控网络不够完善,其下游靶基因及作用机制需要阐明,转录因子与哪些基因共同起作用也需要研究清楚,为更深入的研究铺平道路;3) 转录因子在微生物中的应用和探索都极少.若利用合成生物学的方法在微生物中构建脂肪酸合成的代谢通路,消除竞争途径,使更多碳源流向脂肪酸途径,获得的重组菌与植物相比,由于生长周期短、易于培养和不受季节气候的影响等优势可使脂肪酸进行大规模工业化生产,因此利用转录因子开发出高产油脂的微生物菌株更有应用前景.
[1] 徐元浩, 叶盛焱, 陈忠祥, 等. 生物柴油的实用性研究[J]. 武汉理工大学学报, 2005, 27(5): 90-93.
[2] 周丹, 赵江哲, 柏杨, 等. 植物油脂合成代谢及调控的研究进展[J]. 南京农业大学学报, 2012, 35(5): 77-86.
[3] BUIJS N A, SIEWERS V, NIELSEN J. Advanced biofuel production by the yeastSaccharomycescerevisiae[J]. Current opinion in chemical biology, 2013, 17(3): 480-488.
[4] 刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用[J]. 科学通报, 2000, 45(14): 1465-1474.
[5] FOCKS N, BENNING C. Wrinkled1: a novel, low-seed-oil mutant of Arabidopsis with a deficiency in the seed-specific regulation of carbohydrate metabolism[J]. Plant physiology, 1998, 118(1): 91-101.
[6] MA W, KONG Q, ARONDEL V, et al. Wrinkled1, a ubiquitous regulator in oil accumulating tissues from Arabidopsis embryos to oil palm mesocarp[J]. Plos One, 2013, 8(7): 68887.
[7] SUBROTO A P, UTOMO C, DARMAWAN C, et al. Isolation and characterization of oil palm wrinkled 1 (WRI1) gene[J]. Procedia chemistry, 2015, 14: 40-46.
[8] MURPHY D J. The dynamic roles of intracellular lipid droplets: from archaea to mammals[J]. Protoplasma, 2012, 249(3): 541-585.
[9] BAUD S, WUILLèME S, TO A, et al. Role of WRINKLED1 in the transcriptional regulation of glycolytic and fatty acid biosynthetic genes in Arabidopsis[J]. Plant journal for cell & molecular biology, 2009, 60(6): 933-947.
[10] FUKUDA N, IKAWA Y, AOYAGI T, et al. Expression of the genes coding for plastidic acetyl-CoA carboxylase subunits is regulated by a location-sensitive transcription factor binding site[J]. Plant molecular biology, 2013, 82(4): 473-483.
[11] MARCHIVE C, EACUTE, NIKOVICS K, et al. Transcriptional regulation of fatty acid production in higher plants: molecular bases and biotechnological outcomes[J]. European journal of lipid science & technology, 2014, 116(10): 1332-1343.
[12] POUVREAU B, BAUD S, VERNOUD V, et al. Duplicate maize wrinkled1 transcription factors activate target genes involved in seed oil biosynthesis[J]. Plant physiology, 2011, 156(2): 674-686.
[13] WU X L, LIU Z H, HU Z H, et al. BnWRI1 coordinates fatty acid biosynthesis and photosynthesis pathways during oil accumulation in rapeseed[J]. Journal of integrative plant biology, 2014, 56(6): 582-593.
[14] DUSSERT S, GUERIN C, ANDERSSON M, et al. Comparative transcriptome analysis of three oil palm fruit and seed tissues that differ in oil content and fatty acid composition[J]. Plant physiology, 2013, 162(3): 1337-1358.
[15] SANJAYA, DURRETT T P, WEISE S E, et al. Increasing the energy density of vegetative tissues by diverting carbon from starch to oil biosynthesis in transgenic Arabidopsis[J]. Plant biotechnology journal, 2011, 9(8): 874-883.
[16] AN D H, MICHUNG S. Overexpression of Arabidopsis WRI1 enhanced seed mass and storage oil content inCamelinasativa[J]. Plant biotechnology reports, 2015, 9(3): 137-148.
[17] YU A Q, JUWONO N K, FOO J L, et al. Metabolic engineering ofSaccharomycescerevisiaefor the overproduction of short branched-chain fatty acids[J]. Metabolic engineering, 2016, 34: 36-43.
[18] ZALE J, JUNG J H, KIM J Y, et al. Metabolic engineering of sugarcane to accumulate energy-dense triacylglycerols in vegetative biomass[J]. Plant biotechnology journal, 2015, 14(2): 661.
[19] SONG A, GAO T, LI P, et al. Transcriptome-wide identification and expression profiling of the DOF transcription factor gene family inChrysanthemummorifolium[J]. Frontiers in plant science, 2016, 164(7): 199.
[20] 郭晓芳, 严海燕. 植物中的Dof蛋白和Dof转录因子家族[J]. 植物生理学报, 2005, 41(4): 419-423.
[21] 徐慧妮, 王康, 李昆志. 植物Dof转录因子及其生物学功能[J]. 生物技术通报, 2010(1): 19-23.
[22] WEN C, CHENG Q, ZHAO L, et al. Identification and characterisation of Dof transcription factors in the cucumber genome[J]. Scientific reports, 2016(6): 23072.
[23] YANAGISAWA S. Dof1 and Dof2 transcription factors are associated with expression of multiple genes involved in carbon metabolism in maize[J]. Plant journal, 2000, 21(3): 281-288.
[24] WARD J M, CUFR C A, DENZEL M A, et al. The Dof transcription factor OBP3 modulates phytochrome and cryptochrome signaling in Arabidopsis[J]. Plant cell, 2005, 17(2): 475-485.
[26] CAI X, ZHANG C, SHU W, et al. The transcription factor SlDof22 involved in ascorbate accumulation and salinity stress in tomato[J]. Biochemical & biophysical research communications, 2016, 474(4): 736-741.
[27] FENG B H, HAN Y C, XIAO Y Y, et al. The banana fruit Dof transcription factor MaDof23 acts as a repressor and interacts with MaERF9 in regulating ripening-related genes[J]. Journal of experimental botany, 2016, 67(8): 2263-2275.
[28] WANG H W, ZHANG B, HAO Y J, et al. The soybean Dof-type transcription factor genes, GmDof4 and GmDof11, enhance lipid content in the seeds of transgenic Arabidopsis plants[J]. Plant journal, 2007, 52(4): 716-729.
[29] WANG F, PERRY S E. Identification of direct targets of FUSCA3, a key regulator of Arabidopsis seed development[J]. Plant physiology, 2013, 161(3): 1251-1264.
[30] ELAHI N, DUNCAN R W, STASOLLA C. Decreased seed oil production in FUSCA3Brassicanapusmutant plants[J]. Plant physiology & biochemistry, 2015, 96(8): 222.
[31] GAZZARRINI S, TSUCHIYA Y, LUMBA S, et al. The transcription factor FUSCA3 controls developmental timing in Arabidopsis through the hormones gibberellin and abscisic acid[J]. Developmental cell, 2004, 7(3): 373-385.
[32] LU Q S, PAZ J D, PATHMANATHAN A, et al. The C-terminal domain of FUSCA3 negatively regulates mRNA and protein levels, and mediates sensitivity to the hormones abscisic acid and gibberellic acid in Arabidopsis[J]. Plant journal, 2010, 64(1): 100-113.
[33] TSAI A Y, GAZZARRINI S. AKIN10 and FUSCA3 interact to control lateral organ development and phase transitions in Arabidopsis[J]. Plant journal for cell & molecular biology, 2012, 69(5):809-821.
[34] YAMAMOTO A. Cell-by-cell developmental transition from embryo to post-germination phase revealed by heterochronic gene expression and ER-body formation in Arabidopsis leafy cotyledon mutants[J]. Plant & cell physiology, 2014, 55(12): 2112-2125.
[35] ROSCOE T T, GUILLEMINOT J, BESSOULE J J, et al. Complementation of seed maturation phenotypes by ectopic expression of abscisic acid insensitive3, fusca3 and leafy cotyledon2 in Arabidopsis[J]. Plant & cell physiology, 2015, 56(6): 1887-1908.
[36] FATIHI A, BOULARD C, BOUYER D, et al. Deciphering and modifying LAFL transcriptional regulatory network in seed for improving yield and quality of storage compounds[J]. Plant science, 2016, 250: 198-204.
[37] SANTOSMENDOZA M, DUBREUCQ B, BAUD S, et al. Deciphering gene regulatory networks that control seed development and maturation in Arabidopsis[J]. Plant journal, 2008, 54(4): 608-620.
[38] 朱红林, 沙爱华, 符秀梅, 等. 转录调控基因GmLEC1的cDNA克隆及其植物表达载体的构建[J]. 华北农学报, 2009, 24(1): 64-68.
[39] ELAHI N, DUNCAN R W, STASOLLA C. Modification of oil and glucosinolate content in canola seeds with altered expression ofBrassicanapusLEAFY COTYLEDON1[J]. Plant physiology & biochemistry, 2016, 100: 52-63.
[40] TAN H, YANG X, ZHANG F, et al. Enhanced seed oil production in canola by conditional expression ofBrassicanapusLEAFY COTYLEDON1 and LEC1-LIKE in developing seeds[J]. Plant physiology, 2011, 156(3): 1577-1588.
[41] MU J, TAN H, ZHENG Q, et al. LEAFY COTYLEDON1 is a key regulator of fatty acid biosynthesis in Arabidopsis[J]. Plant physiology, 2008, 148(2): 1042-1054.
[42] KE T, DONG C, MAO H, et al. Analysis of expression sequence tags from a full-length-enriched cDNA library of developing sesame seeds (Sesamumindicum)[J]. BMC plant biology, 2011, 11(1): 180.
[43] HUANG F, ZHU Q H, ZHU A, et al. Mutants in the imprinted plckle related 2 gene, suppress seed abortion of fertilization independent seed class mutants and paternal excess interploidy crosses in Arabidopsis[J]. Plant journal, 2017,90(2):383-395.
[44] VEERAPPAN V, WANG J, KANG M, et al. A novel HSI2 mutation in Arabidopsis affects the PHD-like domain and leads to derepression of seed-specific gene expression[J]. Planta, 2012, 236(1): 1.
[45] SHARMA N, BENDER Y, BOYLE K, et al. High-level expression of sugar inducible gene2 (HSI2) is a negative regulator of drought stress tolerance in Arabidopsis[J]. BMC plant biology, 2013,13(1): 1-20.
[46] ZHU Y, DONG A, MEYER D, et al. Arabidopsis NRP1 and NRP2 encode histone chaperones and are required for maintaining postembryonic root growth[J]. Plant cell, 2006, 18(11): 2879-2892.
[47] ZHU Y, DONG A, SHEN W H. Histone variants and chromatin assembly in plant abiotic stress responses [J]. Biochimica et biophysica acta, 2011, 1819(3/4): 343-348.
[48] WANG H, YUN L. Developmental control of Arabidopsis seed oil biosynthesis[J]. Planta, 2007, 226(3): 773-783.
[49] BOURGIS F, KILARU A, CAO X, et al. Comparative transcriptome and metabolite analysis of oil palm and date palm mesocarp that differ dramatically in carbon partitioning[J]. Proceedings of the national academy of sciences, 2011, 108(30): 12527-12532.
[50] VANHERCKE T, TAHCHY A E, SHRESTHA P, et al. Synergistic effect of WRI1 and DGAT1 coexpression on triacylglycerol biosynthesis in plants[J]. Febs letters, 2013, 587(4): 364-369.
[51] BAUD S, LEPINIEC L. Regulation of de novo fatty acid synthesis in maturing oilseeds of Arabidopsis [J]. Plant physiology & biochemistry, 2009, 47(6): 448-455.
(责任编辑:朱小惠)
Researches of plant transcription factors involving in fatty acid synthesis
MIN Wenli1, CAO Xitao1,2, JI Gengsheng1,2, ZHANG Guozheng1,2
(1.College of Biotechnology, Jiangsu University of Science and Technology, Zhenjiang 212018, China;(2.The Sericultural Research Institute, Chinese Academy of Agricultural Sciences, Zhenjiang 212018, China)
Transcription factors are plant-specific regulatory proteins, which play essential roles in the physiological and biochemical processes, especially in the synthesis of fatty acid and accumulation of oil. Recently, the application of transcription factors for higher oil production in crops has attracted a great deal of attention. This article reviews the characteristics, functions and applications of key transcription factors involved in fatty acid synthesis and triacylglycerol biosynthesis, providing the basis for further study of structural-functional analysis of transcription factors.
fatty acid; oil accumulation; transcription factors
2017-03-03
江苏科技大学博士启动基金(1732921203)
闵文莉(1990—),女,江苏徐州人,硕士研究生,研究方向为植物分子生物学,E-mail: wenlimin188@163.com.
Q943.2
A
1674-2214(2017)02-0107-06
