基于突变阻滞扩增系统的实时荧光PCR技术检测幽门螺杆菌23S rRNA基因突变及其评价
2017-04-15沈维祥陈春峰张小燕郜恒骏
沈维祥,陈春峰,张小燕,郜恒骏
基于突变阻滞扩增系统的实时荧光PCR技术检测幽门螺杆菌23S rRNA基因突变及其评价
沈维祥,陈春峰,张小燕,郜恒骏
目的探索突变阻滞扩增系统(ARMS)联合实时荧光 PCR技术用于检测幽门螺杆菌 23S rRNA 基因突变的可行性。
突变阻滞扩增系统; 实时荧光 PCR; 基因突变
基因突变是人类各种遗传疾病和恶性肿瘤发生的根本原因,检测与遗传病及肿瘤发生有关的突变基因一直是医学遗传学和肿瘤学研究的热点,它对阐明遗传病和肿瘤发生的分子生物学基础及早期诊断具有重要意义。目前传统的基因检测方法有聚合酶链式反应-限制性片段长度多态性(polymerase chain reaction restriction fragment length polymorphism,PCR-RFLP)、单链构象多态性(single strand conformation polymorphism,PCR-SSCP)及荧光原位杂交(fluorescence in situ hybridization,FISH)技术[1-4]。近年来兴起的基因芯片、二代测序、变性高效液相色谱、生物质谱等基因分型技术,以其通量大和自动化程度高等特点,逐渐被广泛应用到疾病基因组学、药物基因组学等研究领域[5-7]。但上述这些检测技术操作繁琐且依赖昂贵的仪器设备,限制了其在临床检测方面的应用。本研究采用突变阻滞扩增系统(amplification refractory mutation system,ARMS)联合实时荧光PCR 技术,以幽门螺杆菌 23S rRNA 基因 2143位点的两种基因型(野生型为 AAA,突变型为AGA)为研究靶点,主要从 ARMS 引物的设计,引物浓度的选择,PCR 反应体系及条件的优化等方面进行基因分型特异性研究,希望在此基础上发展成为一种适合临床应用的基因突变检测方法。
1 材料与方法
1.1 材料
1.1.1 DNA 模板 幽门螺杆菌 23S rRNA 基因野生型质粒 DNA 模板(2143 位点为 AAA,简称AAA 模板)直接以正常人基因组 DNA 为模板构建,突变型质粒(简称 AGA 模板)采用点突变方法构建。野生型质粒和突变型质粒委托上海生工生物工程有限公司制备,所有构建的质粒 DNA 模板序列均经测序确认。
1.1.2 主要仪器和试剂 LightCycler 480 实时荧光 PCR 仪购自美国 Roche 公司;NanoDrop 2000超微量生物检测仪购自美国 ThermoFisher 公司;Taq 酶、dNTPs 等购自大连宝生物工程有限公司。
1.1.3 引物探针 根据已公布的 23S rRNA 基因的 2143 位点序列设计特异的 ARMS 上游引物以及一条公共的下游引物和探针(序列如表 1 所示),所有的引物和探针均由大连宝生物工程有限公司合成。
1.2 方法
1.2.1 ARMS-实时荧光 PCR 扩增曲线分析 PCR 体系含 10×PCR buffer,5 mmol/L MgCl2,0.2 mmol/L dNTPs,2 U Taq 酶,0.3 μmol/L 下游引物和 0.1 μmol/L 探针(表 1 中 R 和 P)。将上述组分混匀后,每管体积 100 μl 分装,分别加入表 1中的上游引物 F1 和 F2 各 0.3 μmol/L。然后每管再分装 4 小管,每小管最终体积 25 μl,使每条引物分别对两种质粒 DNA 模板(约 0.1 ng)和水(作为阴性对照)进行 PCR 扩增与结果分析。反应条件:94 ℃ 预变性 5 min;94 ℃ 变性 15 s,55 ℃ 退火 20 s,72 ℃ 延伸 30 s,扩增 45 个循环;在每一循环的退火温度收集荧光信号。
1.2.2 ARMS-实时荧光 PCR 反应体系及条件优化 ARMS-PCR 反应步骤改良:PCR 体系组成同1.2.1。反应条件:94 ℃ 预变性 5 min;94 ℃ 变性15 s,62 ℃ 延伸 30 s,扩增 45 个循环;在每一循环的延伸温度 62 ℃ 时收集荧光信号。
ARMS 引物浓度的优化:将 ARMS 引物(浓度 0.3 μmol/L)分别稀释 5、10 和 20 倍后,再进行实时荧光 PCR 检测。反应体系其他成分及反应条件不变。

表1 ARMS 引物设计列表Table 1 Sequence information of ARMS primers
镁离子浓度的优化:将镁离子(原浓度4 mmol/L)调整为 2.5 mmol/L 后,再进行实时荧光 PCR 检测。反应体系其他成分及反应条件不变。
1.2.3 ARMS-实时荧光 PCR 突变检测 PCR体系含 10×PCR buffer,2 mmol/L MgCl2,0.2 mmol/L dNTPs,2 U Taq 酶,0.3μmol/L 下游引物和 0.1 μmol/L 探针(表 1 中 AAA-R-primer 和AAA-probe)。将上述组分混匀后,每管体积 120 μl分装,分别加入表 1 中的上游引物各 0.03 μmol/L。然后每管再分装 5 小管,每小管最终体积 25 μl,使每条引物分别对 4 种质粒 DNA 模板(约0.1 ng)和水(作为阴性对照)进行 PCR 扩增与结果分析。反应条件:94 ℃ 预变性 5 min;94 ℃ 变性 15 s,62 ℃ 延伸 30 s,扩增 45 个循环;在每一循环的延伸温度 62 ℃ 时收集荧光信号。
2 结果
2.1 ARMS-实时荧光 PCR 扩增曲线分析
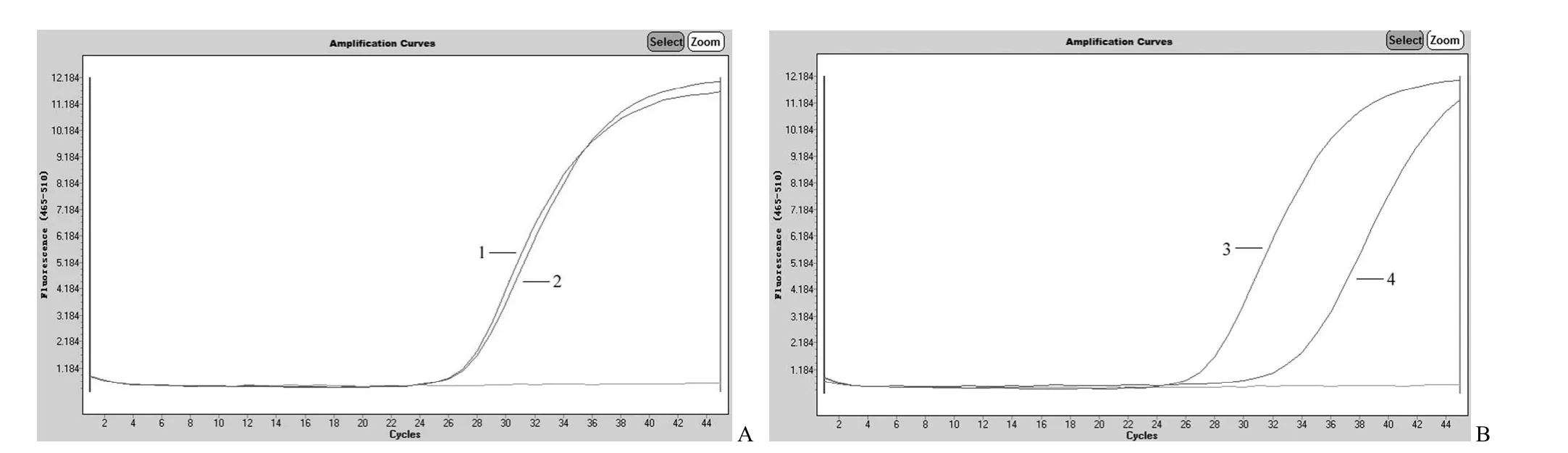
图1 ARMS-PCR 扩增曲线分析(A、B 图分别采用 F1、F2 引物和传统三步法 PCR 进行 ARMS-PCR 基因分型)Figure 1 Results of ARMS-PCR genotyping based on the basic three-step PCR protocol (A:Primer F1 with AAA and AGA templates; B:Primer F2 with AAA and AGA templates)
如图 1 所示,使用传统三步法 PCR 程序用于分别扩增 AAA 模板和 AGA 模板的两条引物 F1和 F2,其中 F1 是和模板序列完全互补的引物,而 F2 是在距离 3' 端的第(n-3)位点引入故意的错配,以 T 取代 G 的引物。使用 F1 引物体系分别扩增 AAA 模板和 AGA 模板,两条扩增曲线无明显差别(Ct 值分别为 26.56 和 26.61),表明 F1引物并不能区分 AAA 模板和 AGA 模板,见图1A。再使用 F2 引物体系分别扩增 AAA 模板和AGA 模板,结果见图 1B。F2 引物表现出一定的分辨能力,扩增 AGA 模板的曲线 Ct 值滞后(Ct值为 30.67),表明引入故意的错配能够发挥作用。同时,F2 引物体系也能够较好地扩增 AAA 模板(Ct 值为 26.58),表明引入故意的错配对 AAA模板的扩增影响较小。值得注意的是,F2 引物扩增 AGA 模板虽然出现了 Ct 值滞后,但并不能够完全消除这种非特异性扩增的现象。根据 F2 和F3 自身 Tm值的差别,对传统三步法 PCR 程序进行改良,即将传统退火和延伸步骤改为一步恒温62 ℃ 采集荧光信号,期望提高特异性引物与模板的匹配度,增加 ARMS 引物基因分型的特异性。
2.2 ARMS-实时荧光 PCR 反应体系优化结果
2.2.1 反应程序优化结果 对 PCR 循环进行改良,即将传统退火和延伸步骤改为一步恒温 62 ℃采集荧光信号,结果如图 2 所示。使用 F1 引物体系分别扩增 AAA 模板和 AGA 模板,两条扩增曲线也出现了 Ct 值的差异,两者的 Ct 值与传统三步法 PCR 程序产生的 Ct 值相比明显提前(Ct值分别为 16.58 和 22.19),表明改良的 PCR 程序有助于提高特异性引物与模板的匹配度,见图 2A。再使用 F2 引物体系分别扩增 AAA 模板和 AGA模板,结果见图 2B,产生的 Ct 值之差(△Ct 值)进一步增大,表明改良 PCR 程序有助于增加基因分型的特异性。图 2C 为使用 F3 引物体系分别扩增 AAA 模板和 AGA 模板,结果也显示出类似的趋势。尤其是采用 ARMS 引物与两步法 PCR 程序结合后,AAA 模板和 AGA 模板的荧光信号可以很清晰地区分,基因分型特异性大大增加。因此,研究人员进一步对 PCR 反应体系中的关键成分ARMS 引物和镁离子作优化,以期消除非特异性扩增的现象。
2.2.2 ARMS 引物浓度优化结果 将 F3 引物浓度稀释 5、10 和 20 倍后的结果表明,若稀释倍数太低,如图 3A 所示,对基因分型的结果改善不明显;若稀释倍数太高,如图 3C 所示,则造成目的产物扩增效率太低,不利于基因分型。总的来说本研究中所采用的 F2 ARMS 引物稀释 10 倍时可得到特异性较好的分型结果,见图 3B。
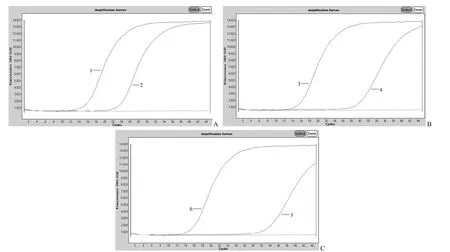
图2 ARMS-实时 PCR 基因分型 PCR 程序改良(A、B、C 图分别采用 F1、F2 和 F3 引物和改良两步法 PCR 进行ARMS-实时 PCR 基因分型)Figure 2 Results of ARMS-PCR genotyping based on the improved two-step PCR protocol (A:Primer F1 with AAA and AGA templates; B:Primer F2 with AAA and AGA templates; C:Primer F3 with AAA and AGA templates)

图3 ARMS-实时 PCR 基因分型引物浓度优化(A、B、C 图分别采用 0.05、0.03、0.015 μmol/L 的 F3 引物进行 ARMS-实时 PCR 基因分型)Figure 3 Results of ARMS-PCR genotyping based on the improved two-step PCR protocol with different concentrations of ARMS primer (A:0.05 μmol/L primer F3; B:0.03 μmol/L primer F3; C:0.015 μmol/L primer F3)

图4 ARMS-实时 PCR 基因分型镁离子浓度优化(A、B 图分别为 F2 和 F3 引物采用 2 mmol/L 镁离子浓度进行ARMS-实时 PCR 基因分型)Figure 4 Results of ARMS-PCR genotyping based on the improved two-step PCR protocol with 2 mmol/L concentration of Mg2+(A:Primer F2 with AAA and AGA templates; B:Primer F3 with AAA and AGA templates)

图5 ARMS-实时 PCR 基因分型(A、B 图分别为 F2 和 F3 引物采用优化后的体系以及 PCR 程序进行 ARMS-实时PCR 基因分型)Figure 5 Results of ARMS-PCR genotyping based on the final PCR protocol (A:Primer F2 with AAA and AGA templates; B:Primer F3 with AAA and AGA templates)
2.2.3 镁离子浓度优化 将 PCR 体系中的镁离子浓度降低,F2 引物和 F3 引物分别扩增完全匹配的模板(AAA 模板和 AGA 模板),Ct 值均表现出一定程度的滞后(Ct 值分别为 20.85 和21.03),但对于非特异性模板的扩增表现出明显的抑制现象,结果如图 4 所示。表明降低镁离子浓度能够抑制非特异性扩增,分型特异性大大增强。
2.3 ARMS-实时荧光 PCR 突变检测结果
经过上述对 ARMS-PCR 基因分型特异性的探讨后,采用在距离引物 3' 端的第(n-3)位点引入故意错配碱基的 ARMS 引物,并将该引物浓度稀释 10 倍(0.03 μmol/L),镁离子浓度调整为2 mmol/L,结合改良的两步法 PCR 程序进行基因分型研究。结果如图 5 所示,两种基因型均可以得到很好的区分,错配模板的影响能够降到最低,基本不影响基因分型结果的判定。
3 讨论
突变阻滞扩增系统(ARMS)也被称为等位基因特异性扩增法(allele-specific amplification,ASA),其通常与 PCR 技术相结合。在进行扩增反应时若 3' 端碱基对形成错配,根据 3' 端错配原则,链延伸反应就会因 3',5'-磷酸二酯键形成的障碍而受阻[8]。而只有特定等位基因的模板链存在条件下,链延伸才会正常进行。ARMS 引物设计是决定突变阻滞扩增系统检测特异性的关键。引物 3'末端碱基必须落在突变的位置上,该碱基可以是与野生型基因序列完全互补,而与突变型不匹配;也可以与突变型互补而与野生型不匹配。但 3' 端最后碱基必须在突变位置上,否则将不能很好地判断该位点的突变是否发生。本研究针对幽门螺杆菌在临床上出现耐药突变,在 23S rRNA 基因 2143 点存在两种基因型(野生型为 AAA,突变型为AGA),设计 ARMS 引物,以期通过荧光 PCR 技术区分这两种基因型,达到检测基因突变的目的。
值得注意的是,ARMS-PCR 对 SNP 的检出还依赖于防止引物与靶 DNA 错配时可能发生的延伸,在 ARMS 引物设计时,为了增加引物对 SNP检测的特异性和灵敏度,可人为地在引物 3' 末端引入第二个错配碱基,以阻止非完全匹配的延伸。本次研究即采用此种方法,在距离引物 3' 端的第(n-3)位点引入故意的错配。有国外研究表明,在所有的碱基对中 G∶T 错配相对容易[9]。据此,本研究的几条 ARMS 引物均引入该种类型的错配形式。图 1B 所示,引入第二个错配碱基,匹配型扩增和错配型扩增的 △Ct 值为 3~4。
此外,荧光 PCR 的反应体系与 ARMS 引物的特异性密切相关。因此对反应体系进行优化是十分必要的。一方面,可以添加 DMSO、甲酰胺或者甜菜碱等化学添加剂;另一方面,调整镁离子浓度、靶 DNA 浓度、Taq 酶的用量和退火温度等来提高特异性[10]。在本研究中,反应程序的改变和镁离子浓度的调整对于非特异性模板的扩增都有明显的抑制,匹配型扩增和错配型扩增的 △Ct 值均在10 以上,如图 2B、2C 和图 4 显示。虽然不能完全消除非特异性扩增,但在一定程度上增加了ARMS 引物的特异性,增强了其分型能力。
除了常规的退火温度和镁离子浓度的优化外,本研究对 ARMS 引物浓度对分型特异性的影响进行考察,结果表明:较低的 ARMS 引物浓度可以增加 PCR 反应的特异性,但与此同时会出现扩增效率的降低。如图 3C 所示,将引物稀释 20 倍后,正常的匹配型扩增 Ct 值为 32.26;而错配型扩增的 Ct 值虽然滞后明显,但两者 △Ct 值并未变化。所以必须找到一个平衡点,选择最佳的反应条件可以取得令人满意的分型效果。图 3B 表明,将引物稀释 10 倍后的 PCR 反应条件下,23S rRNA 基因的两种基因型得到了最大程度的区分。
本研究对基于突变阻滞扩增系统的实时荧光PCR 基因分型方法的特异性进行了深入探讨,实验结果表明,经优化后的两步法 ARMS-PCR 分型方法,是一种简便、准确、快速的基因分型方法,并有望在此基础上发展成为适合临床应用的突变检测方法。
[1]Fontana C,Favaro M,Pietroiusti A,et a1.Detection of clarithromycin-resistant Helicobacter pylori in stool sample.Clin Microbiol,2003,41(8):3636-3640.
[2]Rimbara E,Noguchi N,Yamaguchi T,et a1.Development of a highly sensitive method for detection of clarithromycin-resistant Helicobacter pylori from human feces.Curr Microbiol,2005,51(1):1-5.
[3]Morris JM,Reasonover AL,Bruce MG,et a1.Evaluation of seaFAST,a rapid fluorescent in situ hybridization test,for detection of Helicobacter pylori and resistance to clarithromycin in paraffin-embedded biopsy sections.J Clin Microbio,2005,43(7):3494-3496.
[4]Yilmaz O,Demiray E.Clinical role and importance of fluorescence in situ hybridization method in diagnosis of H pylori infection and determination of clarithromycin resistance in H pylori eradication therapy.World J Gastroenteml,2007,13(5):671-675.
[5]Ezzeldin H,Okamoto Y,Johnson MR,et al.A high-throughput denaturing high-performance liquid chromatography method for the identification of variant alleles associated with dihydropyrimidine dehydrogenase deficiency.Anal Biochem,2002,306(1):63-73.
[6]Posteraro P,Bmnca G,Sangllinetti M,et a1.Rapid detection of clarithromycin resistance in Helicobacter pylori using a PCR-based denaturing HPLC assay.J Antimicmb Chemother,2006,57(1):71-78.
[7]Griffin TJ,Smith LM.Single-nucleotide polymorphism analysis by MALDI-TOF mass spectrometry.Trends Biotechnol,2000,18(2):77-84.
[8]Newton CR,Graham A,Heptinstall LE,et al.Analysis of any point mutation in DNA.The amplification refractory mutation system (ARMS).Nucleic Acids Res,1989,17(7):2503-2512.
[9]Cotton RG.Detection of single base changes in nucleic acids.Biochem J,1989,263(1):1-10.
[10]Cheng J,Zhang Y,Li Q.Real time PCR genotyping using displacing probes.Nucleic Acids Res,2004,32(7):e61.
Detection of Helicobacter Pylori 23S rRNA gene mutation by using the quantitative real-time PCR technology based on the amplification refractory mutation system
SHEN Wei-xiang,CHEN Chun-feng,ZHANG Xiao-yan,GAO Heng-jun
ObjectiveTo evaluate the feasibility of detection of the 23S rRNA gene mutation of Helicobacter pylori in gastric mucosa by using the amplification refractory mutation system (ARMS) combined with real-time PCR.MethodA kind of plasmids containing two different genotypes in 2143 site of 23S rRNA gene (which wild type is AAA and mutants are AGA) was used as model to study the specificity of ARMS primers-based real-time PCR genotyping.Based on designing different primers and optimizing the real-time fluorescent PCR system,the ARMS combined with quantitative real-time PCR method could be used to detection of the 23S rRNA gene mutation of Helicobacter pylori.ResultThe concentrations of ARMS primers and magnesium ion were reduced by harboring additional mismatch at the vicinity of the 3' end of the ARMS primers,and the specificity of the ARMS primer was enhanced by using a 2-step PCR protocol,and then a clear genotype results were achieved.ConclusionThe amplification refractory mutation system combined with quantitative real-time PCR is proved to be a rapid,simple,cost effective,and reliable genotyping method.It can be applicable to genotyping of a variety of genes and has the potential to apply in clinical diagnosis.
Amplification refractory mutation system; Real-time PCR; Gene mutation
GAO Heng-jun,Email:xiaoyan_zhang@shbiochip.com
上海市浦东新区科技发展基金(PKJ2015-S)
201203 上海,生物芯片上海国家工程研究中心
郜恒骏,Email:xiaoyan_zhang@shbiochip.com
2016-12-30
10.3969/j.issn.1673-713X.2017.02.004
方法以幽门螺杆菌 23S rRNA 基因 2143 位点的两种基因型(野生型为 AAA,突变型为 AGA)质粒 DNA 为研究模型,设计 ARMS 引物同时对实时荧光 PCR 体系进行优化,对突变阻滞扩增系统联合实时荧光 PCR 技术的基因分型特异性进行评价。
结果在与模板匹配的 ARMS 引物 3' 端附近故意引入错配碱基,降低该 ARMS 引物浓度和镁离子浓度,并采用两步 PCR循环的反应程序,使 ARMS 引物的特异性增强,能够得到清晰的基因分型结果。
结论这种基于突变阻滞扩增系统的实时荧光 PCR 技术,实验设计简便,特异性较好,并有望在此基础上发展成为适合临床应用的突变检测方法。
Author Affiliations:National Engineering Center for Biochip,Shanghai 201203,China
