明悉引物设计 参透PCR技术
2019-03-22孙娟
孙 娟
(首都师范大学附属回龙观育新学校 北京 102208)
关键字 PCR 引物设计 题型解析
PCR(Polymerase Chain Reaction)是一种人工合成DNA的聚合酶链式反应,PCR技术是高中生物学选修3《现代生物科技专题》模块的重点和难点之一,其基本原理和过程如图1所示。
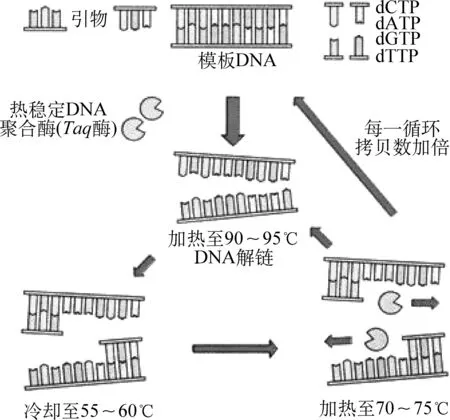
图1 PCR原理反应示意图[1]
在教学实践中,教师一般采用动画演示或者绘图的方式阐释PCR的原理和过程。但是,学生在解决PCR实际问题时仍然会有很多困惑,其中最为突出的问题是PCR的引物设计。引物设计是PCR的核心,学生既可以通过Primer Premier等软件去设计引物,也可以通过考题去明悉引物设计的意义,从而深刻理解PCR的原理和过程。
PCR除了能完成常规的DNA扩增,还可以通过巧妙的引物设计实现基因定点突变、添加酶切位点、特定片段的拼接(搭桥PCR)等功能。本文结合具体题目明悉引物设计的多种方法,从而对不同的PCR进行深度解析。
1 常规PCR的引物设计和选择
PCR的引物包括两条,这两条引物能够分别与待扩增片段(序列已知)的两条链碱基互补配对,而DNA的两条新链延伸方向相反,这与DNA的空间结构为反向平行双螺旋的特点是一致的。在常规的引物设计中,均应包括两条引物,且位于待扩增片段的两端,引物方向均为5′→3′。扩增目的不同,选择的引物也会不同,但基本原则相同。
例1 (2014年·北京30题)(2)a基因是通过将T-DNA插入到A基因中获得的,用PCR法确定T-DNA插入位置时,应从图2中选择的引物组合是。(参考答案: II、 III)
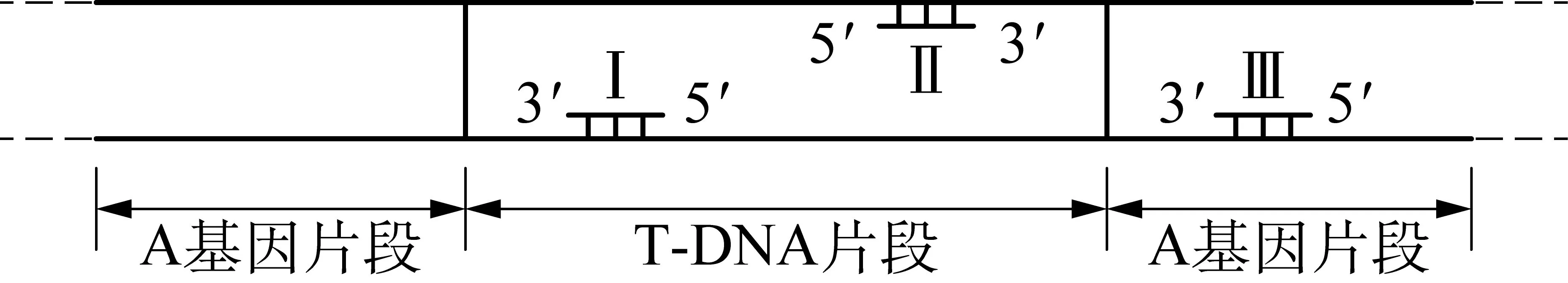
图2 链式DNA引物选择题型注: Ⅰ、 Ⅱ、 Ⅲ为引物
解析: 此题目中PCR是为了确认插入位置,故需要将T-DNA与A基因连接的位置扩增出来,引物应分别与上下两条链互补,图2给出了引物的方向(延伸方向为5′→3′),所以,只能选择II、 III。
例2 (2011年·北京31题)(6)根据T-DNA的已知序列,利用PCR技术可以扩增出被插入的基因片段。其过程是: 提取突变体植株的DNA,用限制酶处理后,再用将获得的DNA片段连接成环(图3),以此为模板,从图中A、 B、 C、 D四种单链DNA片断中选取作为引物进行扩增,得到的片断将用于获取该基因的全序列信息。(参考答案: DNA连接酶;B、 C)
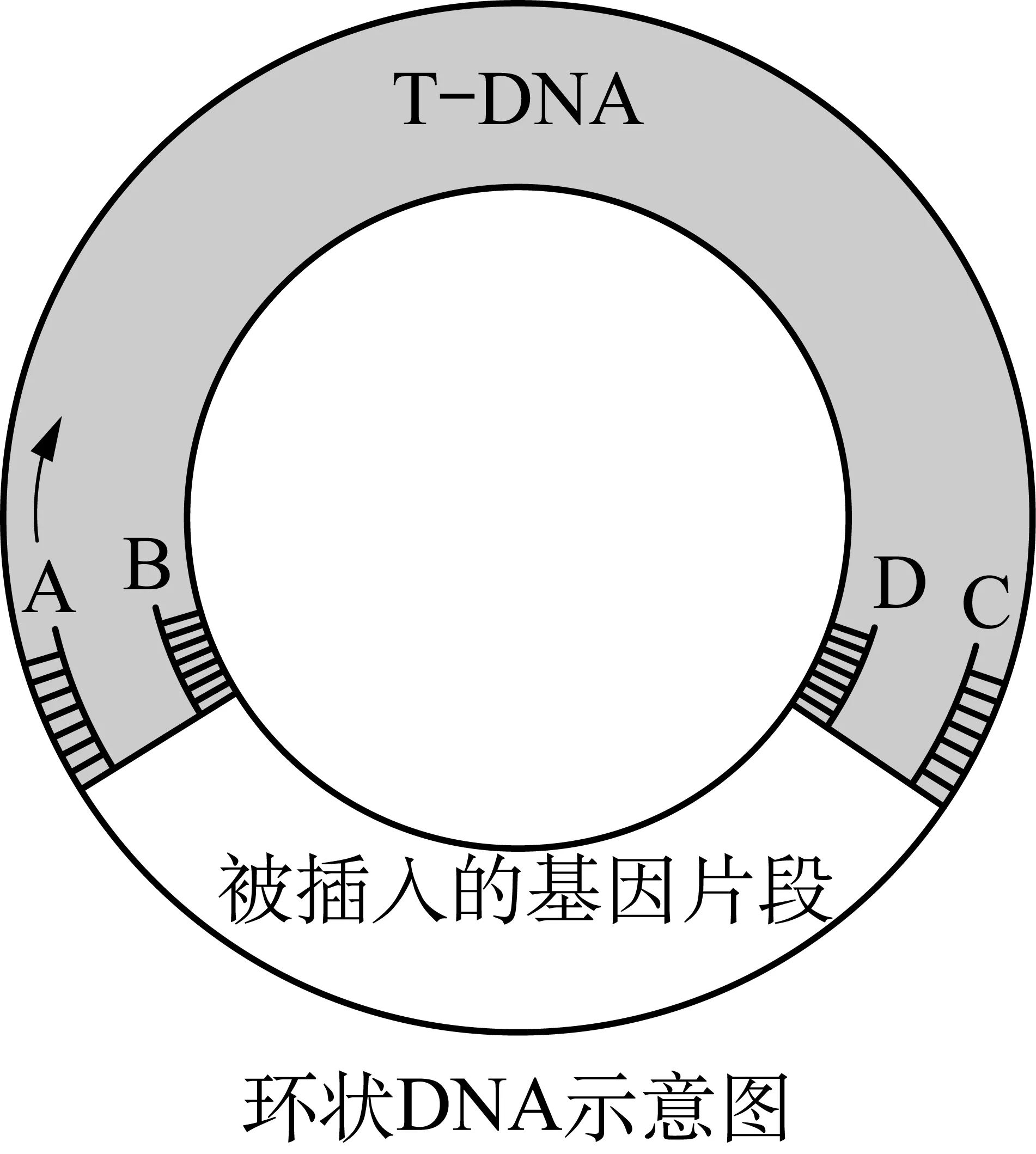
图3 环状DNA引物选择题型注: A、 B、 C、 D表示能与相应T-DNA链互补、长度相等的单链DNA片段;箭头指示DNA合成方向
解析: 此题通过PCR可以获取未知基因的全部序列信息,因此,需要将“被插入的基因片段”全部扩增出来,引物应位于其两端,且分别于两条链互补。图3给出了A的方向,根据DNA的结构特点可知,C与A方向相同,B、 D的方向与A相反,选用A、 D能够扩增出T-DNA序列,选用B、 C才能扩增出包含“被插入的基因片段”的序列信息,故选择B、 C。
例3 (北京·西城2017.1,26题)(2)②利用PCR技术从cDNA文库中获取AtNX1基因(基因序列未知),需根据AtNX1蛋白氨基酸序列设计引物,引物的序列可以有多种,原因是。在PCR体系中需加入其中的引物(填“全部”或“任意1种”或“其中2种”)。(参考答案: 全部)
解析: 此题的难度较例1、2有所增加,考查了引物与模板碱基互补配对的特点,由于密码子具有简并性(一种氨基酸对应多种密码子),并不能确定哪一种引物可以和未知序列碱基互补配对,因此,需要将全部引物加入,配对成功的引物可以完成PCR扩增过程,其他引物因无法配对不形成干扰。
2 基因定点突变的引物设计
例4 (北京·海淀2017.11,34题)(2)萝卜的蛋白A具有广泛的抗植物病菌作用,而且对人体没有影响。检测发现,转入的蛋白A基因在大肠杆菌细胞中表达效率很低,研究者推测不同生物对密码子具有不同的偏好,因而设计了与蛋白A基因结合的两对引物(引物B和C中都替换了一个碱基),并按图4方式依次进行4次PCR扩增,以得到新的蛋白A基因。①这是一种定点的技术。②图4所示的4次PCR应该分别如何选择图中所示的引物?请填写以下表格(若选用该引物划“√”,若不选用该引物则划“×”)。(参考答案: (2)① 基因突变 ② 如下表所示)
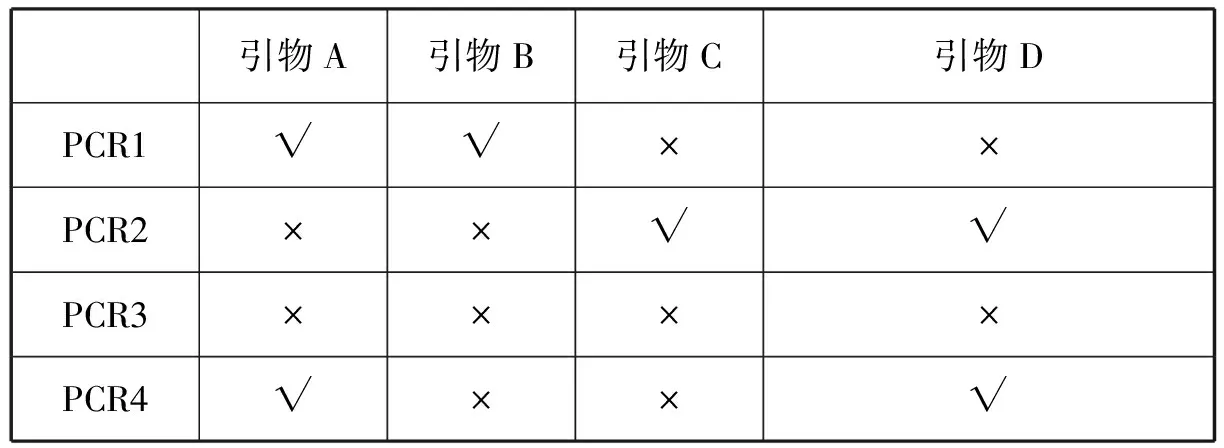
引物A引物B引物C引物DPCR1√√××PCR2××√√PCR3××××PCR4√××√
解析: 此题的引物设计与以往都不同,B和C中都替换了一个碱基,从图4分析可知,这两个引物位于基因的相同位置,替换的碱基应该能够互补配对,如此便可以实现将基因A的特定位置替换为新的碱基对。因此,属于一种基因突变的技术。后续的PCR过程就是为了完成基因的定点突变,根据引物设计原则,学生可以从PCR1、 PCR2的图示中选出引物A&B、 C&D。
PCR1、 PCR2的产物混合后退火,由图4可见,引物B、C所在位置的序列可以碱基互补配对结合在一起,从而使蛋白A基因的两条链以局部配对的方式结合,并彼此充当引物(不需要额外添加引物),完成5′→3′方向的延伸过程。因此,PCR3的结果是在替换了一个碱基的位置发生基因的定点突变。PCR4过程是为了获取更多新的蛋白A基因,选取的引物应位于其两端,根据扩增方向为5′→3′的原则,选择的引物应该是A和D。
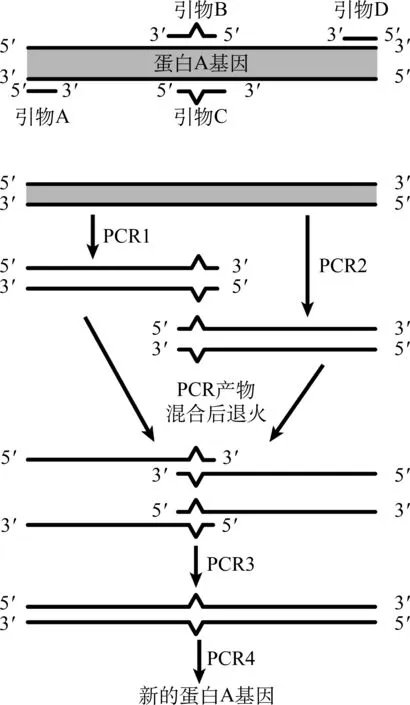
图4 基因定点突变原理示意图 注: 图中“”为碱基序列变化点
此题进行PCR扩增的目标是实现某一个特定位置碱基对的替换,因此,需要根据目的基因的序列进行分段PCR,最后再将两段拼接起来,是一种比较传统和实用的体外基因突变方法。随着科技的发展,CRISPR-Cas9定点突变技术应用更为广泛,其原理见例5。
例5 (北京·海淀2016.5)敲除基因时,sgRNA与红眼基因B的部分序列互补配对,细胞内的Cas9酶在配对区域定点剪切,引起红眼基因B发生碱基对,导致基因突变。该过程中,sgRNA和Cas9酶共同剪切DNA的作用类似于酶的作用,敲除后需要酶对DNA上的切口进行修复。(参考答案: 缺失,限制,DNA连接)
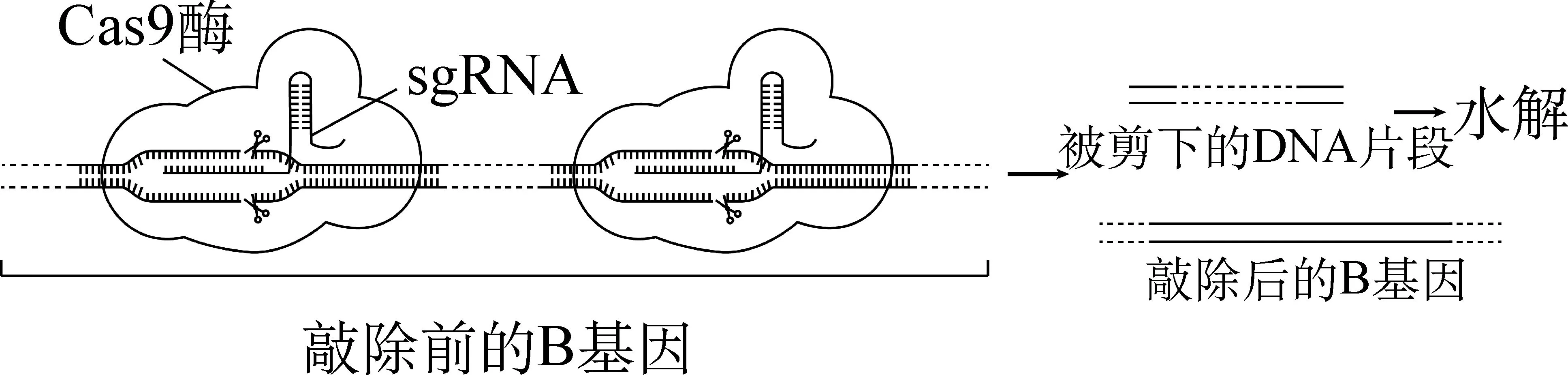
图5 CRISPR-Cas9技术原理图
解析: 此题中,基因突变是通过sgRNA(small guide RNA,向导序列)实现的,sgRNA是人工设计的能够与特定基因碱基互补配对的一小段RNA,当其与目的基因结合后,Cas9酶就可以对目的基因进行剪切,从而实现基因突变。
3 搭桥PCR与酶切位点的添加
例6 (北京·海淀2017.5,30题)(3)为研究基因A与四膜虫的耐药性是否有关,科研人员提取耐药个体的DNA,用图6所示的引物组合,分别扩增A基因的A1片段、A3片段。
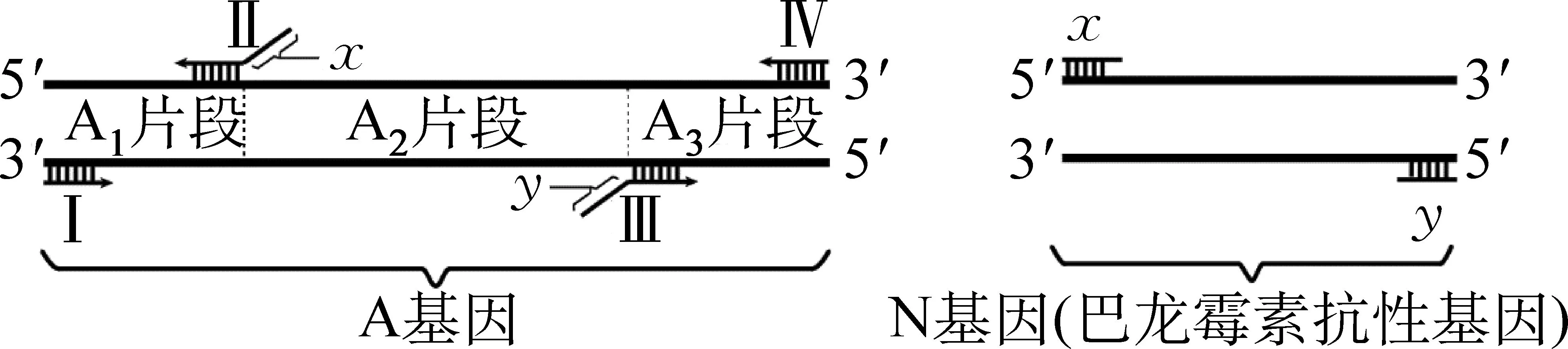
图6 添加特定序列的PCR引物选择注: 引物Ⅱ、 Ⅲ上的x、 y片段分别与N基因两端互补配对
① 据图6分析,用引物Ⅰ、 Ⅱ组合扩增后,得到的绝大部分DNA片段是下图中的。
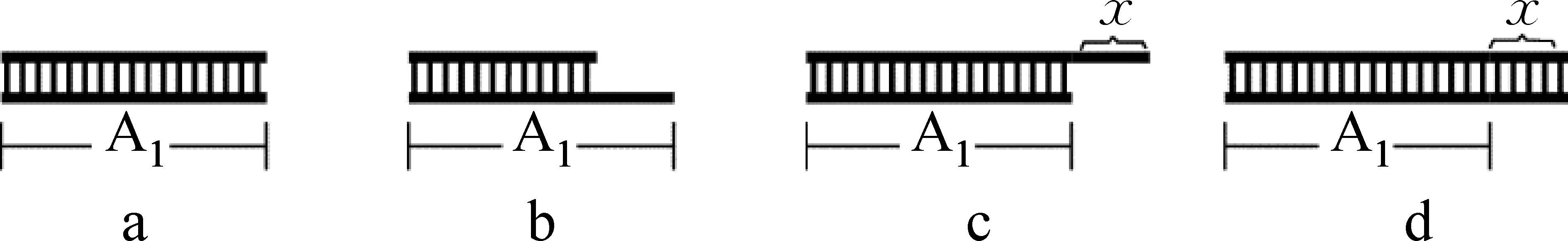
② 将大量N基因片段与扩增得到的A1片段、A3片段置于PCR反应体系中进行扩增,得到的绝大多数扩增产物是。(参考答案: (3)①d ②A1-N-A3)
解析: 此题的难点在于对PCR过程的分析,引物与模板链结合后,在Taq酶的作用下开始延伸,延伸的终点为模板链的终点(图7),第二轮扩增后会出现“A1-x”序列,之后以2n扩增,同时,以“A1-A2-A3”为模板进行的扩增在两轮后都会产生“A1+x”片段,最终反应体系中“A1-x”数量最多。同理,用III和IV扩增出的应为“y-A3”。
在N基因片段与扩增得到的A1片段、A3片段的PCR反应体系中,通过引物中x、 y片段局部配对的方式,两条链可以结合并分别充当彼此的引物,不再需要额外添加引物,其原理与例4的PCR3过程(图4)相同。最终,通过搭桥PCR的方式实现“A1-N-A3”的拼接。
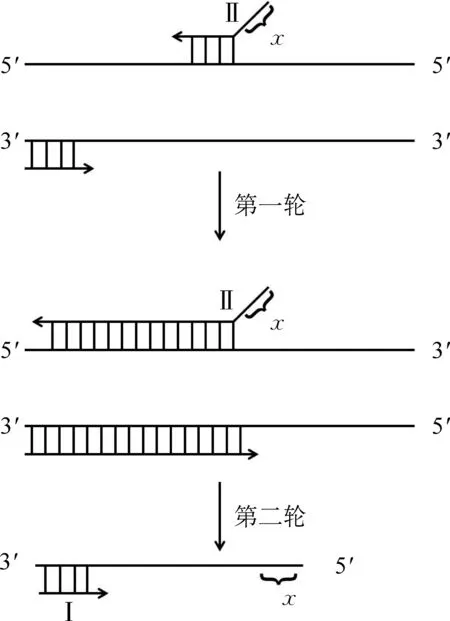
图7 添加特定序列的PCR原理
4 简并引物与突变位点确认
例7 原创试题(节选): 采用电击转化法将已知序列的外源DNA片段随机插入野生型衣藻基因组中可获得衣藻的鞭毛突变体,为进一步确定突变体衣藻的突变基因,设计以下实验(图8)。若要确定外源DNA片段插入X基因的位置,需要选用引物及,分别扩增出对应序列,再通过测序和序列比对确定外源DNA片段插入X基因的a和b两个位置。(参考答案: 1、3;2、4)
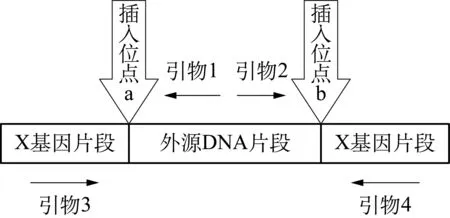
图8 确认突变位点的PCR引物选择
解析: 此题需要找出外源DNA在A基因中的插入位点a、 b,因此,需要分别扩增出包含a、 b两个位点的序列,按照图示应选择1、3扩增出左侧片段,选择2、4扩增出右侧片段,测序后即可得知插入位点的序列。
此项PCR技术中的X基因序列是未知的,因此,并不能设计出与其碱基互补配对的引物3、4。在实际操作中,通常会通过查询衣藻的全基因组序列找出其较为保守的一些序列,从而设计出一些能够与多个位点结合的简并引物(degenerated primers),在PCR过程中,同时加入简并引物3、4和1、2(与已知序列结合),通过调整退火温度,促使3、4引物与多个位点发生特异或非特异结合,其中有一部分即为包含a或者b的产物。之后,通过琼脂糖凝胶电泳技术,使不同长度的PCR产物分离开,选择含量较高的(条带较宽)的位置切下胶块进行DNA回收,测序后通过与外源DNA序列比对得出a、 b所在具体位点。
