内蒙古布氏田鼠地理种群遗传多样性
2016-12-09魏磊赵素华胡奕王承民何宏轩
魏磊, 赵素华, 胡奕, 王承民, 何宏轩*
(1. 亳州学院生物与化学工程系,安徽亳州 236800; 2. 宿州职业技术学院,安徽宿州234101;3.中国科学院动物研究所动物生态和保护生物学院重点实验室,国家野生动物疫病研究中心,北京100101)
内蒙古布氏田鼠地理种群遗传多样性
魏磊1, 3, 赵素华2, 胡奕3, 王承民3, 何宏轩3*
(1. 亳州学院生物与化学工程系,安徽亳州 236800; 2. 宿州职业技术学院,安徽宿州234101;3.中国科学院动物研究所动物生态和保护生物学院重点实验室,国家野生动物疫病研究中心,北京100101)
以MHCⅡ类基因第二外显子为分子标记,利用限制性内切酶分析法和序列分析法对8个布氏田鼠Lasiopodomysbrandtii地理种群进行遗传多样性分析。结果显示,8个布氏田鼠地理种群的MHCⅡ类基因第二外显子酶切共检测到6个等位基因,定义21种单倍型,其中有3个单倍型为不同区域种群共享,经卡方检验,6个酶切多态性位点上基因型频率不符合Hardy-Weinberg平衡;序列分析显示,在261 bp的核苷酸序列中,有57个变异位点,单倍型多样性为0.746 5~0.873 3、核苷酸多样性为0.006 06~0.016 55。谱系分析得到3个稳定的分支:锡林浩特、二连浩特、东乌珠穆沁旗和西乌珠穆沁旗4个种群形成一个单元分歧的进化分支(A区域),新巴尔虎左旗、陈巴尔虎旗和新巴尔虎右旗3个种群成一个单元分歧的进化分支(B区域),而位于浑善达克沙漠南部的正镶白旗种群形成一个独立单元分歧的进化分支(C区域),分别与采集的地理种群相吻合。AMOVA分析结果表明,区域类群间的遗传变异占总变异比率的64.08%,区域内种群间占7.78%,种群内占28.14%。除正镶白旗种群外,布氏田鼠种群具有较丰富的遗传多样性,遗传变异主要发生在区域类群之间和种群内。
布氏田鼠;MHCⅡ类基因;遗传多样性
布氏田鼠Lasiopodomysbrandtii隶属于啮齿目Rodentia仓鼠科Cricetidae田鼠亚科Microtinae,是半干旱型草原的主要啮齿类动物,分布在我国内蒙古中、东部,蒙古利亚共和国南部、东方艾玛格省东部以及俄罗斯(Shenbrot & Krasnov,2005)。在我国主要有2个间断的分布区,分属于内蒙古的呼伦贝尔草原和锡林郭勒的典型草原地区。近年来人类活动和气候变化破坏了草原生态系统,导致大面积荒漠的出现,形成了动物迁徙和重新分布的地理阻隔,使布氏田鼠种群呈点状、片段化、间断性分布(Zhongetal.,1985;Shi,1988;王万杰,任伯平, 2007)。进化生物学和群体遗传学理论表明,小的和孤立的种群将受到扩散的限制并导致遗传多样性降低,甚至因随机漂移和近亲繁殖的综合影响而丧失遗传变异(Frankham,2005)。Wang等(2011)基于微卫星标记对内蒙古东乌珠穆沁旗、阿巴嘎旗、新巴尔虎左旗和正镶白旗4个布氏田鼠地理种群遗传多样性分析显示,不同种群间等位基因平均数、有效等位基因平均数、信息指数、有效等位基因数和平均期望杂合度之间无显著差异,各种群内平均观测杂合度小于平均期望杂合度,4个种群28个位点中有14个位点背离Hardy-Weinberg平衡。结果表明布氏田鼠遗传变异主要发生在种群内,其中正镶白旗种群存在近交现象,遗传多样性最低。
分子标记技术为研究种内不同种群形成现有地理分布格局的历史原因和演化过程提供了有效的途径(Aviseetal., 1998)。主要组织相容性复合体(major histocompatibility complex, MHC)是脊椎动物体内与免疫应答调节密切相关的一个基因家族,由紧密连锁的高度多态性基因座位组成。MHC基因作为分子标记与微卫星标记相比显示了在选择平衡下的高度遗传多样性,其变异可反映基因组水平的变异,而多数受选择作用影响的分子标记则因为缺少变异性而无法阐述种群间的差异和适应性;微卫星位点因其主要受非选择性机制的影响而不适于阐述种群间的不同适应性(Quelleretal.,1993;Kimetal.,1999)。因此,通过分析MHC的遗传变异可以提供物种的遗传多样性水平、进化历史和种群动态及种群遗传结构等信息。
本文以MHCⅡ类基因第二外显子为分子标记,对内蒙古8个布氏田鼠地理种群进行遗传多样性分析,以期得到布氏田鼠种群遗传多样性的分子证据,为进一步开展布氏田鼠的分子生物学研究提供分子数据。
1 材料与方法
1.1 样品采集
样品来自于正镶白旗(115°16.331′E,42°25.611′N;n=62)、陈巴尔虎旗(119°44.735′E,49°32.652′N;n=52)、新巴尔虎左旗(118°03.776′E,49°18.152′N;n=56)、新巴尔虎右旗(118°22.573′E,48°21.625′N;n=50)、西乌珠穆沁旗(117°57.324′E,44°5.211′N;n=53)、二连浩特(112°23.564′E,43°53.245′N;n=65)、东乌珠穆沁旗(116°10.846′E,44°59.454′N;n=54)、锡林浩特(115°13.673′E,44°50.351′N;n=68)地区8个种群,共460只布氏田鼠,各取样点间的距离不小于200 km。样品采集信息见图1。
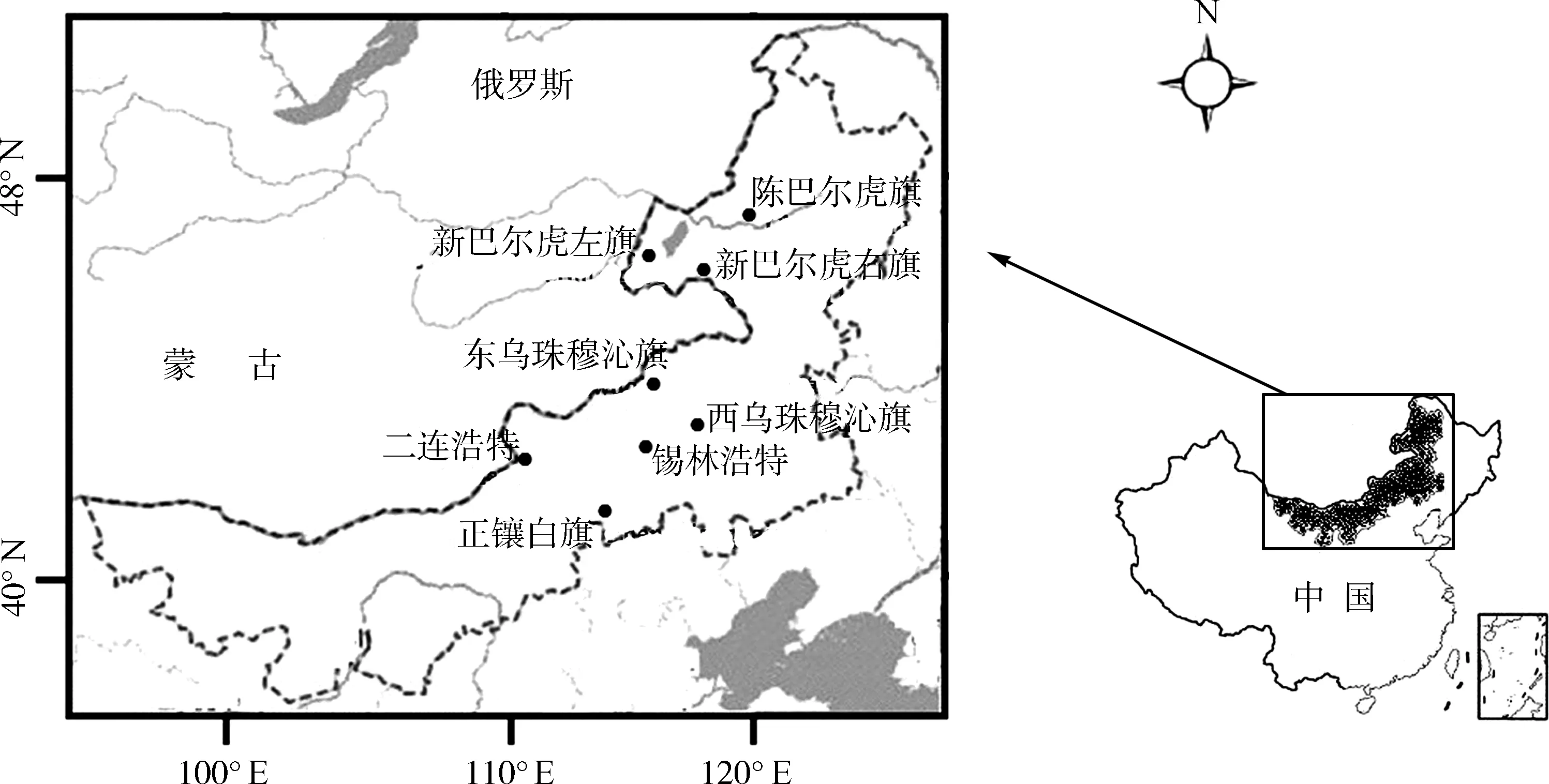
图1 布氏田鼠采集地点的分布
1.2 基因组DNA提取与PCR扩增
用DNA试剂盒提取基因组。以GenBank中登录号为AJ003232、AJ634270、AF084934、U88933、M73960、BC132163、K0012的序列作参考,设计1对引物:上游引物MF:5’-GGAATACGGTACATCTGAG-3’,下游引物MR:5’-CAACTCTCCGCCGCACAAGG-3’;PCR反应在PTC-200型DNA扩增仪上进行,PCR反应体系总体积为30 μL,其中1.0 mmol·L-1Tris-HCl(pH 8.3),1.5 mmol·L-1MgCl2,50 mmol·L-1KCl,150 μmol·L-1dNTPs,引物各0.25mol,1 U Taq DNA聚合酶,30 ng DNA。反应程序为:94 ℃变性45 s,54 ℃复性50 s,72 ℃延伸50 s,共30个循环。循环前95 ℃预变性5 min,循环后继续72 ℃延伸10 min。PCR产物用1.5%琼脂糖凝胶电泳检测并送生工生物工程(上海)股份有限公司进行正反测序。
1.3 限制性内切酶分析
限制性内切酶RsaⅠ购自宝生物工程(大连)有限公司。酶切体系和条件按说明书进行。酶切产物用含溴化乙锭的8%琼脂糖凝胶电泳后拍照,观察结果。
1.4 数据分析
用SPSS 20.0对酶切结果进行基因型统计。用Clustal X(Thompaonetal.,1997)进行序列比对。利用MEGA 6.0(Tamuraetal.,2013)中的非加权平均算术法(UPGMA)对各种群进行聚类分析,利用Bootstrap Test检验所得聚类结果可靠性。用Arlequin (version 3.0)(Excoffieretal.,2007)对基因序列进行分析,统计多态位点数(number of polymorphic sites,S),并以Pairwise difference分子距离计算单倍型多样性(haplotype diversity,H)、核苷酸平均差异数(mean number of nucleotide differences,π)和核苷酸多样性(nucleotide diversity,πn),种群结构的地理格局通过该软件的分子变异分析AMOVA模块来估测。
2 结果
2.1 MHCⅡ类基因第二外显子酶切单倍型及等位基因型频率
8个布氏田鼠地理种群MHCⅡ类基因第二外显子RsaⅠ酶切共检测到6个等位基因,13个酶切多态位点,定义了21种单倍型(表1),3个单倍型由8个种群共享;经卡方检验,6个酶切多态性位点上基因型频率不符合 Hardy-Weinberg平衡。正镶白旗种群纯合子基因型频率较高(0.064 5~0.112 9),杂合子基因型频率较低(0.016 2~0.048 4),其他种群纯合子基因型频率与杂合子基因型频率基本一致。

表1 布氏田鼠MHCⅡ类基因第二外显子酶切基因型
2.2 MHCⅡ类基因第二外显子序列分析
8个地理种群共460只布氏田鼠的MHCⅡ类基因第二外显子序列长度均为261 bp。选取纯合单倍型aa作为对比序列,经DnaSP分析,整个核苷酸序列无碱基空缺,共发现57个变异位点,突变位点百分率为21.84%(图2)。
2.3 聚类分析
用UPGMA法对8个布氏田鼠地理种群进行聚类分析显示,位于锡林郭勒高原东南浑善达克沙地南部的正镶白旗种群首先分化出来独立成一支,显示了这个区域与其他2个区域种群的隔离;位于锡林郭勒高原中部区域的二连浩特、锡林浩特、东乌珠穆沁旗和西乌珠穆沁旗种群形成一个单元分歧的进化分支;位于呼伦贝尔高原西南区域的新巴尔虎左旗、新巴尔虎右旗与陈巴尔虎旗种群形成一个单元分歧的进化分支。把这3个进化单元划分为A、B、C 3个区域(图3)。

图2 布氏田鼠MHCⅡ类基因第二外显子序列分析
Fig.2 Analysis of MHC class Ⅱ exon 2 ofLasiopodomysbrandtii
方框内为Rsa Ⅰ识别序列,向上箭头表示酶切位点,向下箭头表示碱基突变位点。
Boxes represent recognized sequence of Rsa Ⅰ; upward arrows represent restriction enzyme sites for Rsa Ⅰ; downward arrows represent mutation site.
2.4 布氏田鼠种群遗传多样性
根据地理分布及谱系结果对布氏田鼠进行种群遗传多样性分析。遗传多样性信息统计结果显示,B区域种群多态位点数、单倍型多样性和核苷酸多样性最高,A区域种群次之,C区域种群最低(表2),且核苷酸差异分析表明,C区域种群与其他区域种群之间的遗传差异明显。AMOVA分析结果表明,区域类群间的遗传变异占总变异比率的64.08%,区域内种群间占7.78%,种群内占28.14%。除正镶白旗种群外,布氏田鼠种群具有较丰富的遗传多样性。遗传变异主要发生在区域类群之间和种群内。
3 讨论
尽管濒危物种的遗传多样性是生物多样性研究中的热点问题(黄磊,王义权,2004;常青等,2005;黎燕琼等,2011),但生态学家及生物学家同样关注其他野生动物如啮齿类动物种群遗传多样性的研究。Bromham等(1996)和Li等(1996)对家鼠种群遗传多样性进行了研究,结果表明该物种的单倍型多样性和核苷酸多样性都较高。Hirota等(2004)研究结果揭示大林姬鼠Apodemusspeciosus隔离种群的核苷酸多样性较高,而单倍型多样性较低。蔡振媛等(2007)基于线粒体D-loop序列分析探讨了高原鼢鼠Myospalaxbaileyi8个种群的遗传多样性,研究结果揭示该物种的单倍型多样性较高而核苷酸多样性较低。谢建云等(2007)对东方田鼠Microtusfortis4个种群线粒体D-loop区遗传多态性分析显示,该区遗传变异丰富,呈广泛的种类多态性。梁君(2007)对蒙新区子午沙鼠Merionesmeridianus和长爪沙鼠M.unguiculatus的研究结果显示,2个物种的单倍型多样性和核苷酸多样性分别为0.991、0.047和0.976、0.007。Chen等(2010)基于线粒体控制区(D-loop)研究了分布于横断山脉白腹鼠Niviventerexcelsior种群的遗传多样性,结果显示该物种有较高的单倍型多样性和核苷酸多样性,表明在更新世冰期,横断山脉复杂的地形结构为白腹鼠种群提供了避难所从而使其保持了高水平的遗传多样性。Fan等(2012)基于线粒体细胞色素b基因研究了分布于青藏高原东南部的中华姬鼠Apodemusdraco种群的遗传多样性,结果表明该物种的单倍型多样性和核苷酸多样性较高,种群具有丰富的遗传多样性。黄翔和周立志(2012)基于8个多态性的微卫星位点,对子午沙鼠14个局域种群的160只个体进行了种群遗传多样性和遗传结构的分析,结果表明该物种具有高的遗传多样性。

表2 布氏田鼠遗传多样性

图3 布氏田鼠种群的UPGMA聚类分析
支点处的数字是置信度, 即2 000次bootstrap抽样检验的重复百分比; ER. 二连浩特, XL. 锡林浩特, XW. 西乌珠穆沁旗, DW. 东乌珠穆沁旗, XZ. 新巴尔虎左旗, XY. 新巴尔虎右旗, CB. 陈巴尔虎旗, ZB. 正镶白旗; 下同。
The numbers at nodes are the percent occurrence in 2 000 bootstrap replicates; ER. Erlianhaote, XL. Xilinhaote, XW. Xiwuqi, DW. Dongwuqi, XZ. Xinbaerhuzuoqi, XY. Xinbaerhuyouqi, CB. Chenbaerhuqi, ZB. Zhengxiangbaiqi; the same below.
遗传多样性是生物进化的内在源泉,其水平能反映出物种进化潜力和种群遗传适应潜力。遗传多样性常用单倍型多样性、核苷酸多样性来度量(Tajima,1983)。单倍型多样性、核苷酸多样性值越大,群体的遗传多样性越高。本研究结果显示,8个种群460只布氏田鼠MHCⅡ类基因第二外显子酶切基因型频率分析显示,除正镶白旗种群纯合子基因型频率较高,杂合子基因型频率较低外,其他种群纯合子基因型频率与杂合子基因型频率基本一致。卡方检验显示,13个酶切位点中6个位点偏离Hardy-Weinberg平衡,其中4个位点出现在正镶白旗种群,揭示正镶白旗种群纯合子所占的比例较高,杂合子所占的比例较低。序列分析表明,B区域种群序列多态性较高,A区域种群次之、C区域种群最低。8个种群460只布氏田鼠MHCⅡ类基因第二外显子酶切结果共检出21种单倍型,B区域的3个种群单倍型多样性最高,A区域的4个种群次之,C区域种群最低。本研究中B区域种群的单倍型多样性较A区域、C区域高,核苷酸多样性也比A区域、C区域高,符合上述规律。布氏田鼠与子午沙鼠和长爪沙鼠的单倍型多样性和核苷酸多样性类似,呈现出单倍型多样性高、核苷酸多样性低的特点。究其原因:首先,单个碱基的突变可以影响单倍型多样性,但对核苷酸多样性的影响却较小,与单倍型多样性相比,核苷酸多样性的提高需要更长时间的积累。因此,环境反复变化造成的种群缩小和快速增长被认为是造成这种遗传多样性特征的主要原因之一(缑敬轩,2004;曹华猛,2005)。布氏田鼠长期受特定的地理环境条件胁迫和栖息地片段化的双重作用,与其他北方啮齿类一样,种群大小受反馈调节的影响,数量在短时间内经历了涨落波动(Elton,1942;Zhongetal.,1985),种群数量极端的波动即种群增长瓶颈效应能消除过去时间积累的核苷酸多样化变异,但又能快速导致少数碱基位点的快速进化而引起种群遗传结构的变化(Wright,1943)。其次,由于布氏田鼠营地下生活,环境相对稳定以及有限的迁徙扩散能力限制了基因流,任何短时间内的变异都能积累起来,从而使单倍型的多样性高,但提高核苷酸多样性却需要长时间的积累。
国内外许多学者研究过不同地理种群遗传多样性水平的差异并探讨了差异产生的原因。Paetkau等(1998)和周乐等(2007)研究表明,种群的遗传多样性水平与其种群的大小有显著的相关性。Wisely等(2002)对黑足鼬Mustelanigripes的研究表明,大种群的遗传多样性水平明显高于被隔离的小种群。这些研究结果揭示种群的遗传多样性水平与其种群的大小有显著的相关性,大种群的遗传多样性水平明显高于被隔离的小种群。本研究结果表明布氏田鼠种群总的遗传多样性水平较高,但就区域种群而言,A区域、B区域的种群均显示出较高的单倍型多样性和核苷酸多样性,C区域的正镶白旗种群较低。出现这种情况可能有以下原因:首先,布氏田鼠作为内蒙古草原的主要啮齿类动物,分布范围从内蒙古东部的典型草原、干草原到中部的荒漠草原,分布区中地理环境复杂多样,表明布氏田鼠对高寒环境有良好的适应能力,环境条件的差异对其遗传多样性水平影响较小。其次,A区域种群分布在呼伦贝尔高原,B区域种群分布在锡林郭勒高原,区域内没有大的山脉、河流的阻隔,地理屏障作用不明显,且区域内种群相对较大,种群之间的基因交流也相对较多。再次,C区域种群位于浑善达克沙地南部,因沙漠形成了它与其他地理种群之间基因交流的自然屏障而成为隔离封闭种群,且种群都相对较小,这可能是C区域种群遗传多样性较低的重要原因。
致谢:感谢中国科学院植物研究所内蒙古锡林郭勒草原生态系统国家野外观测研究站的全体工作人员,中国科学院动物研究所农业虫害鼠害国家重点实验室的张强博士和郑州大学生物系谢东明在样品采集过程中给予的帮助。
蔡振媛, 张同作, 慈海鑫, 等. 2007. 高原鼢鼠线粒体谱系地理学和遗传多样性[J]. 兽类学报, 27(2): 130-137.
曹华猛. 2005. 白头叶猴(Trachypithecusleucocephalus)线粒体DNA遗传多样性的研究[D]. 桂林: 广西师范大学.
常青, 曹发华, 朱立峰, 等. 2005. 长江下游地区夜鹭种群微卫星变异及遗传多样性[J]. 动物学报, 51(4): 657-663.
缑敬轩. 2004. 川金丝猴(Rhinopithecusroxellanae)种群遗传结构和分子系统地理学初步研究[D].西安: 西北大学.
黄磊, 王义权. 2004. 扬子鳄种群的微卫星DNA 多态及其遗传多样性保护对策分析[J]. 遗传学报, 31(2): 143-150.
黄翔, 周立志. 2012. 蒙新区子午沙鼠种群的遗传多样性和遗传结构[J]. 兽类学报, 32(3): 179-187.
黎燕琼, 郑绍伟, 龚固堂, 等. 2011. 生物多样性研究进展[J]. 四川林业科技, 32(4): 12-19.
梁君. 2007.子午沙鼠和长爪沙鼠的分子系统地理学研究[D]. 合肥: 安徽大学.
王万杰, 任伯平. 2007. 北魏以来的浑善达克沙地和科尔沁沙地[J]. 太原师范学院学报(自然科学版), 6(1): 107-110.
谢建云, 冯洁, 邵伟娟, 等. 2007. 东方田鼠四个种群线粒体D-loop区遗传多态性分析[J]. 兽类学报, 27(4): 407-412.
周乐, 杨生妹, 于智勇, 等. 2007. 高原鼠兔四个地理种群的遗传多样性与遗传分化[J]. 兽类学报, 27(3): 221-228.
Avise JC, Walker D, Johns GC. 1998. Speciation durations and Pleistocene effects on vertebrate phylogeography[J]. Proceeding of the Royal Society, 265: 1702-1712.
Bromham L, Rambaut A, Harvey PH. 1996. Determinants of rate variation in mammalian DNA sequence evolution[J]. Journal of Molecular Evolution, 43: 610-621.
Chen WC, Liu SY, Liu Y,etal. 2010. Phylogeography of the large white-bellied ratNiviventerexcelsiorsuggests the influence of Pleistocene Glaciations in the Hengduan Mountains[J]. Zoologial Science, 27: 487-493.
Elton C. 1942. Voles, mice and lemmings: problems in population dynamics[M]. Oxford: Clarendon Press.
Excoffier L, Laval G, Schneider S,etal. 2007. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 1: 47-50.
Fan ZX, Liu SY, Liu Y,etal. 2012. Phylogeography of the south China field mouse (Apodemusdraco) on the southeastern Tibetan Plateau reveals high genetic diversity and glacial refugia[J]. PLoS ONE, 7(5): e38184. DOI:10.1371/journal.pone.0038184.
Frankham R. 2005. Genetics and extinction[J]. Biological Conservation, 126: 131-140.
Hirota T, Hirohata T, Mashima H,etal. 2004. Population structure of the large Japanese fieldmouse,Apodemusspeciosus(Rodentia: Muridae), in suburban landscape, based on mitochondrial D-loop sequences[J]. Molecular Ecology, 13: 3275-3282.
Kim TJ, Parker KM, Hedrick PW. 1999. Major histocompatibility complex differentiation in Sacramento River Chinook salmon[J]. Genetics, 151: 1115-1122.
Li WH, Ellsworth DL, Krushkal J,etal. 1996. Rates of nucleotide substitution in primates and rodents and the generation-time effect hypothesis[J]. Molecular Phylogenetics and Evolution, 5: 182-187.
Paetkau D, Waits LP, Clarkson PL,etal. 1998. Variation in genetic diversity across the range of North American brown bears[J]. Conservation Biology, 12(2): 418-429.
Queller DC, Strassmann JE, Hughes CR. 1993. Microsatellites and kinship[J]. Trends in Ecology and Evolution, 8: 285-288.
Shenbrot GI, Krasnov BR. 2005. An atlas of the geographic distribution of the Arvicoline rodents of the world (Rodentia, Muridae: Arvicolinae)[M]. Moscow: Pensoft Press.
Shi DZ. 1988. A preliminary study on Brandt’s voles’ distribution region in China and its relation to vegetation and water-temperature condition[J]. Acta Theriologica Sinica, 8: 299-306.
Tajima F. 1983. Evolutionary relationship of DNA sequences infinite populations[J]. Genetics, 105: 437-460.
Tamura K, Stecher G, Peterson D,etal. 2013. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 30: 2725-2729.
Thompaon JD, Gibson TJ, Plewniak F,etal. 1997. The CLUSTAL X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 25(24): 4876-4882.
Wang D, Guo YW, Shi DZ. 2011. Genetic structure of Brandt’s vole (Lasiopodomysbrandtii) populations in Inner Mongolia, China, based on microsatellite analysis[J]. Conservation Genetics, 12: 659-667.
Wisely SM, Buskirk SW, Fleming MA,etal. 2002. Genetic diversity and fitness in black-footed ferrets before and during a bottleneck[J]. The American Genetic Association, 93(4): 231-237.
Wright S. 1943. Isolation by distance[J]. Genetics, 28: 114-138.
Zhong WQ, Zhou QQ, Sun CL. 1985. The basic characteristics of the rodent pests on the pasture in Inner Mongolia and the ecological strategies of controlling[J]. Acta Theriologica Sinica, 5: 241-249.
Genetic Diversity ofLasiopodomysbrandtiiPopulations in Inner Mongolia
WEI Lei1, 3, ZHAO Suhua2, HU Yi3, WANG Chengmin3, HE Hongxuan3*
(1. Bozhou University, Bozhou, Anhui Province 236800, China; 2. Suzhou Vocational Technology College, Suzhou,Anhui Province 234101, China; 3. National Research Center for Wildlife Born Diseases, Key Laboratory of Animal Ecology and Conservation Biology, Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China)
The genetic diversity of 8 geo-populations ofLasiopodomysbrandtiiwere analyzed using PCR-RFLP and sequence analysis based on MHC class Ⅱ exon 2. The results showed that 6 alleles, 21 haplotypes and 13 restriction enzyme polymorphic loci were detected, and 3 haplotypes were shared by all the populations. The Chi-square test results suggested that the 8 populations ofL.brandtiiin the present study did not follow with the Hardy-Weinberg equilibrium at six restriction enzyme polymorphic loci. Sequence analysis showed that there were 57 bases mutations. Haplotype diversity (H) and nucleotide diversity (πn) were 0.746 5~0.873 3 and 0.006 06~0.016 55, respectively. Phylogenetic analyses suggested that the sampled individuals were clustered into three clades: populations of Xilinhaote, Erlianhaote, Dongwuqi, and Xiwuqi were clustered as Zone A; populations of Xinbaerhuzuoqi, Chenbaerhuqi and Xinbaerhuyouqi were clustered as Zone B; while the Zone C was comprised of the sole population from Zhengxiangbaiqi. And this clustering pattern was consistent with their geographical distributions. The result of AMOVA analysis showed that regional genetic variation was accounted for 64.08% of the total, intraregional population variation was accounted for 7.78%, and individual variation was accounted for 28.14%. There was a rich genetic diversity among the populations ofL.brandtiiexcept Zhengxiangbaiqi population. The majority of genetic variation occurred among groups and within populations.
Lasiopodomysbrandtii; MHC class Ⅱ gene; genetic diversity
2016-06-06 接受日期:2016-08-22
安徽省高校学科(专业)拔尖人才学术资助重点项目(gxbjZD2016108); 安徽省教育厅自然科学基金重点项目(KJ2012Z406); 国家重点基础研究发展规划项目(9732007BC109103); 国家自然科学基金项目(31101806)
魏磊(1969—), 男, 教授, 博士, 研究方向: 功能基因、分子系统地理学, E-mail:weileisu@163.com
*通信作者Corresponding author, E-mail:hehx@ioz.ac.cn
10.11984/j.issn.1000-7083.20160157
Q959.837
A
1000-7083(2016)06-0818-07
