藏羚羊基因组微卫星分析与初步验证
2016-12-09都玉蓉谭春敏徐永涛周智红马建滨郭松长
都玉蓉, 谭春敏, 徐永涛, 周智红, 马建滨, 2*, 郭松长
(1. 青海师范大学生命与地理科学学院,西宁810008; 2.青海省青藏高原药用动植物资源重点实验室,西宁810008; 3. 青海省三江源民族中学,西宁810001;4. 中国科学院高原生物适应与进化重点实验室,西宁810007)
藏羚羊基因组微卫星分析与初步验证
都玉蓉1, 2, 谭春敏3, 徐永涛1, 周智红1, 马建滨1, 2*, 郭松长4*
(1. 青海师范大学生命与地理科学学院,西宁810008; 2.青海省青藏高原药用动植物资源重点实验室,西宁810008; 3. 青海省三江源民族中学,西宁810001;4. 中国科学院高原生物适应与进化重点实验室,西宁810007)
为明确藏羚羊Pantholopshodgsoni核DNA中微卫星分布情况和特征,利用MISA工具对藏羚羊基因组进行微卫星扫描。在全长2 696.89 Mb的藏羚羊基因组中,搜索到723 135个微卫星座位,其中完全型微卫星有675 809个。6种重复类型中,单核苷酸重复最多,有471 142个,占69.72%;其次是二核苷酸、三核苷酸,分别为88 832个和86 658个,占13.14%和12.82%;六核苷酸最少,仅215个,约占0.03%。以藏羚羊基因组DNA为模板对微卫星座位进行验证,在100个微卫星座位中筛选到8个具有多态性的微卫星座位,多态比例约为8%。本研究将为研究藏羚羊微卫星标记、群体遗传多样性、藏羚羊保护生物学提供基础。
藏羚羊;基因组;微卫星;多态性
微卫星又称为简单重复序列(simple sequences repeats),是以1~6个核苷酸为基本重复单位的串联重复序列,其长度在100 bp以内,它们广泛存在于各类真核生物的基因组中,原核生物基因组中也含有少量的微卫星(罗文永等,2003)。微卫星因分布广泛、变异度高和易于检测等特征而被应用于分子生态、群体遗传学等研究领域(张于光等,2003)。
目前,开发微卫星标记的方法主要分为以下3类:(1)从公共数据库或相关文献中查找微卫星座位;(2)近缘物种间引物交叉扩增;(3)从基因组DNA中筛选微卫星座位(程晓凤等,2011)。近年来,随着基因组测序技术的发展,越来越多的物种进行了大规模测序并对物种的基因组进行了微卫星分布特征分析(马秋月等,2013),从而为在全基因组层面上分析基因组中微卫星的分布、丰度等特性提供了可能。研究微卫星在物种基因组中的丰度及其特征还有助于了解物种基因组的结构和进化(张学勇,李大勇,2000)。
藏羚羊Pantholopshodgsoni是国家Ⅰ级保护动物,对藏羚羊特异性标记资源以及遗传多样性的研究已有报道。贺培建(2004)、Ruan等(2005)、周惠等(2006)以及Du等(2010)利用mtDNA D-loop的遗传变异对藏羚羊的遗传多样性、遗传分化和种群动态等进行了分析。陈笑(2005)和Zhou等(2007)采用跨物种引物分别对藏羚羊的40个、25个微卫星座位进行分析,筛选出10对和9对可稳定扩增、多态性较高的引物。由于藏羚羊全基因组序列已测序完成(Geetal.,2013),并于2013年在线公布,此数据库成为开发微卫星标记的有利资源,但藏羚羊特异性的标记资源及基因组序列信息还比较匮乏,藏羚羊基因组水平上微卫星特征分析研究还未见报道。因此,本研究主要通过查找藏羚羊基因组开发出的微卫星标记,对藏羚羊基因组所含不同类型的微卫星特征和组成情况进行分析,并初步验证微卫星标记;为藏羚羊遗传多样性研究奠定一定的基础。
1 材料与方法
1.1 藏羚羊微卫星查找和引物设计
微卫星检出设置为:单核苷酸≥10次,二核苷酸≥8次,三核苷酸、四核苷酸、五核苷酸、六核苷酸≥5次。下载藏羚羊基因组数据,利用MISA工具(http://pgrc.ipk-gatersleben.de/misa/)从藏羚羊全基因组数据库中筛选微卫星座位。微卫星引物设计由Primer 3联合Perl脚本完成。
1.2 藏羚羊基因组微卫星的分布及出现频率
根据Weber(1990)的划分标准,不同核苷酸重复的微卫星排列方式可以划分为3种类型:完全型、不完全型和复合型。本研究仅对藏羚羊基因组中1~6碱基重复的完全型微卫星进行统计分析。根据微卫星重复的序列特征,统计分析不同核苷酸重复类型所占比例,找出不同类型微卫星中的优势重复单元;并对1~6核苷酸重复类型中不同的微卫星的长度变异情况进行分析。
1.3 藏羚羊基因组微卫星标记初步验证
1.3.1 基因组DNA的提取 藏羚羊基因组DNA采用苯酚-氯仿-异戊醇方法提取,1%琼脂糖凝胶电泳检测,4 ℃、-20 ℃保存备用。
1.3.2 微卫星引物验证 去掉单核苷酸重复类型和不能设计引物的微卫星座位,得到46 286个微卫星座位及其引物序列,随机挑选并合成100对引物(可发邮件向作者索取)进行后续筛选与初步分析。
经温度梯度PCR筛选出有扩增产物的引物对后,混合4个藏羚羊的基因组DNA做模板进行PCR扩增,以聚丙烯酰胺凝胶电泳检测产物是否具有多态。如该引物检测到该座位具有多态,则以2013年采集于可可西里国家级自然保护区的24个藏羚羊个体的基因组DNA进行扩增和多态检测。微卫星多态检测委托上海生工生物工程有限公司采用ABI 3730测序仪进行。
扩增条件:95 ℃预变性5 min,95 ℃变性30 s,X ℃(各引物对相应的退火温度见表3)退火30 s,72 ℃延伸30 s,30个循环,最后72 ℃延伸10 min,4 ℃保存。扩增体系为20 μL,含ddH2O 14.4 μL,10×PCR buffer 2.0 μL,dNTPs 1.6 μL,上、下游引物各0.4 μL,模板DNA 1.0 μL(50 ng·μL-1),Taq酶1 U。
2 结果与分析
2.1 藏羚羊基因组微卫星丰度分析
在全长约为2 696.89 Mb的藏羚羊基因组中共有723 135个微卫星,平均每隔3 729个碱基就有1个。其中,完全型微卫星675 809个,占93.46%。在675 809个完全型微卫星中,单核苷酸471 142个,占69.72%;二核苷酸88 832个,占13.14%;三核苷酸86 658个,占12.82%;四核苷酸13 561个,占2.01%;五核苷酸15 401个,占2.28%;六核苷酸215个,占0.03%(图1)。

图1 微卫星座位类型(核苷酸碱基数目)分布
2.2 藏羚羊基因组微卫星中优势重复单元碱基的组成
根据碱基互补配对原则,每个微卫星和其理论上对等的序列都合并为一类。因此,单核苷酸重复有2种重复单元;二核苷酸重复有4种重复单元;三核苷酸重复有10种重复单元;四核苷酸重复有33种重复单元;五核苷酸重复有102种重复单元;六核苷酸重复有350种重复单元。
单核苷酸中A/T是主要的重复单元,共450 879个(95.70%),而G/C重复单元只有20 263个。
二核苷酸重复类型中,AC/GT重复单元最常见,共61 045个(68.72%);其次是AT/TA重复单元,共21 935个(24.69%);再次是AG/CT,共5 788个(6.52%);GC/CG数量最少,仅出现64次。
三核苷酸重复以AGC/GTC和ACG/CTG为主,分别有31 379个(36.21%)和31 145个(35.94%);其他重复单元类型及频次分别为:AAC/GTT,8 974个(10.36%);AAT/ATT,6 726个(7.76%);ACC/GTG,3 278个(3.78%);AGG/CTC,1 712个(1.98%);AAG/CTT,1 132个(1.31%);
ACT/ATG,991个(1.14%);ATC/GTA,927个(1.07%);CCG/CGG,394个(0.45%)。
四核苷酸中A/T丰富的重复单元(AAAT/ATTT,AATT/ATTA,AAAG/CTTT,AAAC/GTTT,AACT/ATTG,AAGT/ATTC,AATC/GTTA,AATG/CTTA,ACAT/GTAT,AGAT/ATCT)共有10 836个,占79.91%;其中AAAT/ATTT 30.94%,AAAC/GTTT 22.54%,AAAG/CTTT 7.26%,AACT/ATTG及AATC/GTTA最少,分别占0.48%和0.45%。而G/C丰富的重复单元(CCGG/CGGC,CCCG/CGGG,CCCT/AGGG,CCCA/TGGG,CCAG/CGGT,CCTG/CGGA,CCGA/TGGC,CCGT/AGGC,CACG/CGTG,CTCG/CGAG)共551个,占4.06%;其中CCCT/AGGG 1.69%,CACG/CGTG 0.87%,CCGG/CGGC没有出现。
五核苷酸中A/T重复单元(AAAAN)n1 027个、(AAANN)n14 113个,占98.31%(N代表除A以外的任何碱基,下同);而G/C重复单元所占比例少。
六核苷酸中A/T丰富的重复单元(AAAAAN)n71个、(AAAANN)n28个,也构成了很大一部分六核苷酸重复(46.05%)。
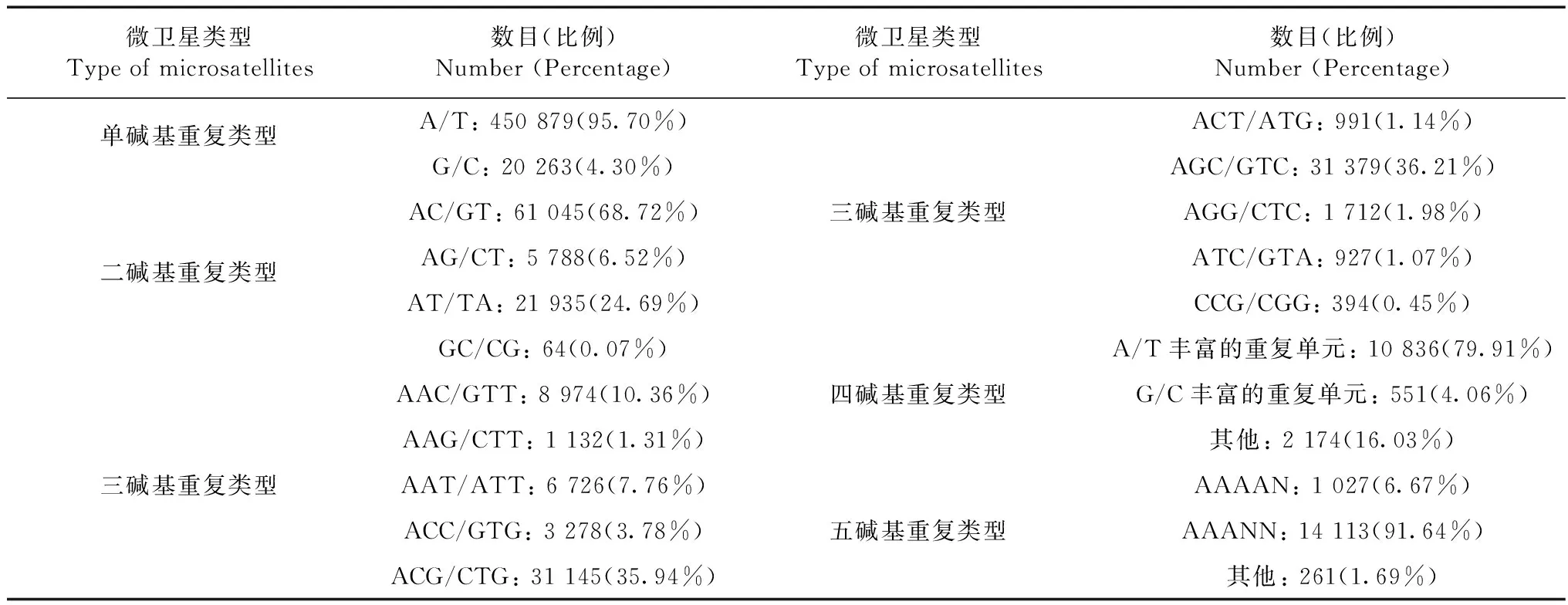
表1 不同碱基重复微卫星在藏羚羊基因组中的分布
2.3 藏羚羊基因组微卫星长度分布分析
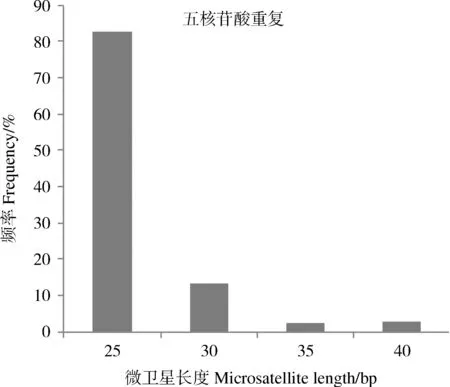
对不同重复微卫星类型中的重复长度进行统计分析,发现重复次数>20的微卫星相对较少,在单核苷酸中只有8 596个,占1.82%;二核苷酸中4 315个,占4.86%;三核苷酸中215个,占0.25%;四核苷酸中41个,占0.30%;五核苷酸中87个,占0.56%;六核苷酸中3个,占1.40%(表2)。以微卫星核心重复序列的长度为横坐标,分布频率为纵坐标作图,可知每种重复微卫星类型都随着重复次数的递增,频率呈递减趋势,即:微卫星长度越长,其丰度越低(图2)。
2.4 藏羚羊基因组微卫星标记初步验证
100对引物中,有70对引物可获得清晰的条带,占引物总数的70%。混合4个藏羚羊的基因组DNA为模板并进行扩增,发现8个微卫星座位具有多态。各座位对应的引物如表3所示。
2.5 微卫星座位多态检测
24个藏羚羊样本在座位P6、P63、P67、P73、P75、P78、P90、P96上的等位基因数(NA)分别为7、11、9、9、14、14、12和9个。除座位P73和P67观测杂合度(Ho)较低外,其余6个微卫星座位的Ho均大于0.7,具有较高的杂合度;而各座位的期望杂合度(He)均较高;且多态信息含量(PIC)也均大于0.5(表4)。以上表明所筛选的8个微卫星座位具有较高的遗传变异。


3 讨论
分析结果显示藏羚羊基因组中的优势重复序列类型是单核苷酸重复序列,其次是二核苷酸和三核苷酸重复序列,之后是五核苷酸、四核苷酸重复序列,六核苷酸重复序列最少,这与牛Bostaurus、山羊Caprahircu、绵羊Ovisaries(王月月等,2015)和牦牛Bosmutus、水牛Bubalusbubalis(戚文华等,2015)各重复类型微卫星在分布数的顺序上完全一致,与猪Susscrofa、犬Canislupusfamiliaris和马Equuscaballus(王月月等,2015)以及大熊猫Ailuropodamelanoleuca、北极熊Ursusmaritimus(李午佼等,2014)的微卫星分布顺序略有不同。采用MISA工具并采用相同微卫星检出设置,对上述10个物种的基因组微卫星验证分析得到了类似的结果,即藏羚羊1~6碱基重复微卫星分布顺序与牛、牦牛、水牛、山羊、绵羊等牛科物种完全一致,且其分布频率也与这5个物种更接近;而藏羚羊1~6碱基重复微卫星的分布顺序和频率与其他5个物种均存在较大差异(表5)。在分类上,藏羚羊与牛、牦牛、水牛、山羊和绵羊均属于牛科。因此,上述结果表明物种间微卫星分布存在一定的保守性,亲缘关系越近,其分布规律越相似。

表4 藏羚羊种群多态信息

表5 11个物种微卫星类型分布与频率
注: 括号内数字为该型微卫星占物种微卫星总数的百分比。
Note: The number in the bracket is the percentage of the repeat unit in all repeat types.
在单核苷酸重复类型中A/T(450 879)占多数,而C/G(20 263)的分布相对较少。在目前已知全基因组的物种中,如人Homosapiens、果蝇Drosophilamelanogaster、秀丽隐杆线虫Caenorhabditiselegans、酵母Saccharomycescerevisiae和拟南芥Arabidopsisthaliana的基因组单碱基微卫星中也发现了相同的现象(Tóthetal.,2000)。
在二核苷酸重复类型中,比例较高的是AC/GT(68.72%),其次是AT/TA(24.69%)、AG/CT(6.52%),GC/CG最少。人和果蝇中,二碱基的重复由高到低依次是AC、AT和AG,拟南芥中最多的是AT,其次是AG,而在酵母中AT占有绝对优势(Tóthetal.,2000)。对于CG 含量稀少的现象,Schorderet和Gartlar(1992)进行了解释:根本原因是由于基因组DNA中的CpG的甲基化,使得胞嘧啶C很容易经过脱氨基作用转变成胸腺嘧啶T。同时基因组DNA 中CpG的甲基化会成为突变的热点,而且少量的GC又是维持DNA 热力学稳定性所必需的,这可能是导致GC含量偏少的原因。
在三核苷酸重复类型中,微卫星重复的10种类型可全部被发现。AGC/GTC数量最多(31 379),其次是ACG/CTG(31 145),AAC/GTT(8 974),AAT/ATT(6 726),CCG/CGG最少(394)。而河豚Takifugurubripes基因组微卫星中以AAT最多,其次是AGG和ATC,GCC最少(崔建洲等,2006);人类基因组微卫星的重复拷贝类别中AAT和AAC最多,接下来是AAG和AGG,ACG最少(Subramanianetal.,2003);果蝇中AGC最多,拟南芥和秀丽隐杆线虫含有较多的AAG重复,而酵母中含有更多的AAT、AAG、ATG 和AGC 重复(Kattietal.,2001)。
研究结果显示,不同重复类型核苷酸中,随着微卫星核心单元重复次数增加,其出现频率呈递减的趋势,且核心单元重复次数>20的微卫星仅占1.96%,与牦牛、水牛基因组微卫星各核心重复单元的拷贝数分布区间高度一致(戚文华等,2015)。
本研究从46 286个藏羚羊微卫星中随机挑选出100对引物进行合成并进行温度梯度扩增,电泳结果显示约70%的引物可以得到有效扩增。进一步对上述可稳定扩增的微卫星座位进行筛选、测序,发现有8个微卫星座位具有多态,占总座位数的8%。据报道,通过构建微卫星文库,研究人员在中国鲎Tachypleustridentatus、卤虫Artemia中分别获得了7.5%(宁晔凤等,2015)和9.52%(刘海珍等,2016)的多态引物,这表明通过基因组数据与通过微卫星文库构建法筛选多态引物具有类似的效率。因此,随着测序技术的发展,利用日益丰富的物种基因组数据开发目标物种的多态微卫星座位具有简便、高效、可靠的特点。
陈笑. 2005. 藏羚羊遗传多样性的微卫星分析[D]. 杭州: 浙江大学.
程晓凤, 黄福江, 刘明典, 等. 2011. 454测序技术开发微卫星标记的研究进展[J]. 生物技术通报, 8: 82-90.
崔建洲, 申雪艳, 杨官品, 等. 2006. 红鳍东方鲀基因组微卫星特征分析[J]. 中国海洋大学学报(自然科学版), 36(2): 249-254.
贺培建.2004.藏羚羊遗传多样性研究[D]. 杭州: 浙江大学.
李午佼, 李玉芝, 杜联明, 等. 2014. 大熊猫和北极熊基因组微卫星分布特征比较分析[J]. 四川动物, 33(6): 874-878.
刘海珍, 邹小艳, 郭松长, 等. 2016. 卤虫(AC)n和(AG)n微卫星文库构建[J]. 四川动物, 35(3): 356-360.
罗文永, 胡骏, 李晓方. 2003. 微卫星序列及其应用[J]. 遗传, 25(5): 615-619.
马秋月, 戴晓港, 陈赢男, 等. 2013. 枣基因组的微卫星特征[J]. 林业科学, 49(12): 81-87.
宁晔凤, 黎中宝, 戴刚, 等. 2015. 中国鲎基因组微卫星富集文库的构建及分析[J]. 海洋科学, 39(4): 15-20.
戚文华, 蒋雪梅, 杜联明, 等. 2015. 牦牛和水牛全基因组微卫星分布规律及其比较分析[J]. 基因组学与应用生物学, 34(7): 1406-1412.
王月月, 刘雪雪, 董坤哲, 等. 2015. 7种家养动物全基因组微卫星分布的差异研究[J]. 中国畜牧兽医, 42(9): 2418-2426.
张学勇, 李大勇. 2000. 小麦及其近亲基因组中的DNA重复序列研究进展[J]. 中国农业科学, 33(5): 14-24.
张于光, 李迪强, 饶立群, 等. 2003. 东北虎微卫星DNA遗传标记的筛选及在亲子鉴定中的应用[J]. 动物学报, 49(1): 118-123.
周慧, 李迪强, 张于光, 等. 2006. 藏羚羊mtDNA D-loop区遗传多样性研究[J]. 遗传, 28(3): 299-305.
Du YR, Guo SC, Wang ZF,etal. 2010. Demographic history of the Tibetan antelopePantholopshodgsoni(chiru)[J]. Journal of Systematics and Evolution, 48: 490-496.
Ge RL, Cai QL, Shen YY,etal. 2013. Draft genome sequence of the Tibetan antelope[J]. Nature Communications, 2013: 1-7.
Katti VM, Ranjekar KP, Gupta SV. 2001. Differential distribution of simple sequence repeats in eukaryotic genome sequences[J]. Molecular Biology and Evolution, 18(7): 1161-1167.
Ruan XD, He PJ, Zhang JL,etal. 2005. Evolutionary history and current population relationships of the chiru (Pantholopshodgsoni) inferred from mtDNA variation[J]. Journal of Mammalogy, 86: 881-886.
Schorderet DF, Gartlar SM. 1992. Analysis of CpG suppression in methylated and nonmethylated species[J]. Proceedings of the National Academy of Sciences, 89(3): 957-961.
Subramanian S, Madgula VM, George R,etal. 2003. Triplet repeats in human genome: distribution and their association with genes and other genomic regions[J]. Bioinformatics, 19(5): 549-552.
Tóth G, Gáspári Z, Jurka J. 2000. Microsatellites in different eukaryotic genomes: survey and analysis[J]. Genome Research, 10(7): 967-981.
Weber JL. 1990. Informativeness of human (dC-dA)n·(dG-dT)npolymorphisms[J]. Genomics, 7(4): 524-530.
Zhou H, Li DQ, Zhang YG,etal. 2007.Genetic diversity of microsatellite DNA loci of Tibetan antelope (chiru,Pantholopshodgsonii) in Hoh Xil National Nature Reserve, Qinghai, China[J]. Journal of Genetics and Genomics, 34(7): 600-607.
Genome-wide Characterization and Identification of Microsatellites inPantholopshodgsoni
DUYurong1, 2,TANChunmin3,XUYongtao1,ZHOUZhihong1,MAJianbin1, 2*,GUOSongchang4*
(1. School of Life and Geography Science, Qinghai Normal University, Xining 810008, China; 2. Key Laboratory of Medicinal Plant and Animal Resources in Qinghai-Tibetan Plateau, Qinghai Province, Xining 810008, China; 3. Sanjiangyuan Nationalities High School of Qinghai Province, Xining 810001, China; 4. Key Laboratory of Evolution and Adaptation of Plateau Biota, Northwest Institute of Plateau Biology, Chinese Academy of Sciences, Xining 810007, China)
To investigate the distribution and feature of simple sequence repeats or microsatellites (1~6 bp repeats), we screened the entire genome of Tibetan antelope (Pantholopshodgsoni)(2 696.89 Mb) by using MISA. A total of 723 135 microsatellite loci including 675 809 perfect microsatellites were detected and characterized within the whole genome. Among these microsatellites, the most abundant repeat type was mononucleotide (69.72%), followed by di- (13.14%), tri- (12.82%), penta- (2.28%), tetra- (2.01%), hexa- (0.03%) nucleotide repeat types, and the motifs AC/GT (9.09%) and ACG/CTG (4.64%) appeared in high frequency. An approximate evaluation was conducted by randomly amplifying 100 microsatellite loci, and 8 loci (8%) were proved to be polymorphic. This study provide abundant microsatellite markers for analysis of genetic variation, construction of genetic map, as well as the conservation biology investigation inP.hodgsoni.
Pantholopshodgsoni; genome; microsatellite; polymorphism
2016-08-03 接受日期:2016-10-08
青海省应用基础研究计划项目(2013-Z-751; 2013-Z-750)
都玉蓉(1968—), 女, 教授, 研究方向:动物分子生态学, E-mail:xndyr@163.com
*通信作者Corresponding author, E-mail:mjb_117@163.com; guo@nwipb.cas.cn
10.11984/j.issn.1000-7083.20160213
Q959.8
A
1000-7083(2016)06-0845-07
