陕西省部分地区猪流行性腹泻病毒S、M和N基因的遗传变异分析
2016-12-09庞文静何亚鹏付明哲许信刚郭抗抗
庞文静,何亚鹏,付明哲,许信刚,郭抗抗,张 琪
(西北农林科技大学动物医学院,陕西杨凌 712100)
陕西省部分地区猪流行性腹泻病毒S、M和N基因的遗传变异分析
庞文静,何亚鹏,付明哲,许信刚,郭抗抗*,张 琪*
(西北农林科技大学动物医学院,陕西杨凌 712100)
为了解陕西省部分地区猪流行性腹泻病毒(PEDV)的遗传和变异情况,采集陕西省部分地区规模化猪场的5份疑似PEDV感染的猪小肠内容物,进行PEDV S、M和N基因的RT-PCR扩增,并对扩增产物进行序列测定和遗传变异分析。结果表明,5份病料均能扩增出PEDV S、M和N基因,5株病毒分别命名为SX-SL、SX-BJ、SX-YL、SX-WN和SX-HZ株。序列分析表明,5株毒株之间的S、M和N基因核苷酸序列的同源性分别为96.7%~99.8%、98.4%~100%和97.2%~99.9%;氨基酸序列的同源性分别为97.4%~99.9%、98.2%~100%和98.2%~100%。该5株病毒与中国疫苗株CV777的S、M和N基因核苷酸序列的同源性分别为93.9%~99.8%、98.1%~100%和95.3%~99.9%,氨基酸序列的同源性为93.6%~99.9%、96.2%~100%和98.2%~100%。遗传进化分析结果显示,5个陕西分离株的S基因与中国疫苗株CV777亲缘关系较远,与近年来中国株、日本株以及韩国株亲缘关系较近。SX-SL株、SX-BJ株和SX-YL株的M和N基因与中国疫苗株CV777亲缘关系较近,且与中国株CHGD-01亲缘关系密切。SX-WN株和SX-HZ株的M和N基因与中国疫苗株CV777亲缘关系较远。该5株病毒的S基因以及SX-WN 株和SX-HZ 株的M基因和N基因变异程度较大,而SX-SL株、SX-BJ株和SX-YL株三个流行株均与中国株CHGD-01亲缘关系密切,并且与近年在陕西省流行的PEDV也不完全相同。
猪流行性腹泻病毒;S基因;M基因;N基因;序列分析
猪流行性腹泻(Porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)引起的一种高度接触性肠道传染病,各年龄段的猪都可能感染发病,主要特征是体温升高、食欲下降、呕吐、腹泻以及脱水,肠道黏膜出血以及糜烂发炎,其中哺乳仔猪较其他生长期猪发病率高,病死率可达100%[1-2]。该病在流行特点、临床特征和病理变化等方面与猪传染性胃肠炎极其相似,临床诊断时很难将它们区分开[3-4]。PEDV属于冠状病毒科冠状病毒属成员,基因组为单链正股的RNA,具有感染性,基因组全长约28 kb,有6个ORF,编码的蛋白从5′-3′依次为replicase基因编码的复制酶多聚蛋白(replicases 1a和1b,PP1ab)、S基因编码的纤突蛋白、ORF3基因编码的ORF3蛋白、E基因编码的小膜蛋白、M基因编码的膜糖蛋白、N基因编码的核衣壳蛋白,其中4种主要结构蛋白分别为S蛋白、E蛋白、M蛋白、和N蛋白,ORF3基因是PEDV基因组中唯一的辅助基因[5-6]。研究报告显示,在2015年以前,全球已经有30多个国家和地区发现并确认有PEDV的存在[7-9]。2010年下半年,PED在中国广泛流行,导致猪的大批死亡,病死率可达100%[10-11]。最近几年,PEDV的变异毒株在许多国家相继出现,大大增加了PED防治的难度,这也是该病不断大规模流行的最根本原因[12-13]。S蛋白具有识别靶细胞并且使病毒和细胞膜融合的作用,其在免疫反应中起重要作用,S基因易变异,因此分析当地PEDV毒株的流行和遗传变异情况,S基因具有重要价值[14-16]。PEDV的主要跨膜糖蛋白为M蛋白,该蛋白在病毒的装配和出芽过程中发挥着重要的作用,并且其保守性强,是作为检测PEDV的重要靶基因[17]。核衣壳蛋白即N蛋白,具有较强的保守性,并在病毒基因组的转录复制中起到一定的作用,能够诱发机体产生有效的免疫应答[18]。因此,研究PEDV的S、M和N基因变异情况,具有重要意义。近年来陕西省不断有猪流行性腹泻的暴发和流行,本研究自2015年-2016年采集陕西省部分地区规模化养猪场5份疑似猪流行性腹泻的肠内容物,进行PEDV的S、M和N基因扩增及测序,并进行流行毒株的遗传和变异分析,以了解陕西省PEDV毒株基因的遗传变异特点,为陕西省PED的防控提供理论依据。
1 材料与方法
1.1 材料
1.1.1 病料 采集陕西省部分地区规模化猪场发生严重腹泻的病死仔猪小肠内容物5份。以病料的采集地点,将样品分别命名为SX-HZ(汉中)、SX-SL(商洛)、SX-WN(渭南)、SX-BJ(宝鸡)、SX-YL(榆林)。
1.1.2 主要试剂 RNA反转录试剂盒(HiScript®Q RT Super Mix for qPCR)为诺唯赞生物技术有限公司产品;Trizol Reagent为天根生化科技(北京)有限公司产品;2×TaqMaster Mix和DL 2 000 DNA Marker为北京康为世纪生物科技有限公司产品。
1.2 方法
1.2.1 引物设计与合成 根据GenBank中已发表的PEDV的S、M和N基因,利用Primer 5.0分别设计扩增M基因、N基因和S基因的特异性引物(表1)。由于S基因较长,将S基因分成3个相互重叠的基因片段,分别命名为S1、S2和S3三段扩增。引物由Invitrogen生命技术有限公司(上海)合成,将各引物浓度稀释至20 μmol/L,置-20℃保存备用。

表1 扩增PEDV S、M和N基因的PCR引物
1.2.2 病毒RNA的提取 将病料研磨匀浆后,-20℃/37℃反复冻融3次,转移至离心管中,8 000 r/min离心5 min,取200 μL上清,按照Trizol Reagent说明书方法提取病毒RNA,提取的RNA 置-80℃保存备用。
1.2.3 RT-PCR扩增 以提取的病毒基因组RNA为模板,按照Hi Script®Q RT Super Mix for qPCR试剂盒说明书将病毒RNA反转录为cDNA。RT反应参数为RNA与随机引物在56℃水浴5 min,冰浴2 min后加2×RT Mix Hi Script®Enzymeb Mix,25℃ 5 min,50℃ 45 min,85℃ 5 min。再以cDNA为模板进行PCR扩增S、M和N基因。PCR反应参数为94℃ 5 min;94℃ 45 s,55℃ 60 s,72℃ 60 s,35个循环;72℃延伸10 min。
1.2.4 序列测定 将获得的S、M和N基因的PCR产物进行核酸凝胶电泳,经回收纯化PCR产物,送北京擎科生物科技有限公司进行序列测定。
1.2.5 序列分析 使用DNA Star软件中的MegAlign功能将S1、S2和S3基因拼接成完整的S基因。登录NCBI GenBank,利用其BLAST功能对所测得的S、M和N基因的核苷酸和氨基酸与GenBank已收录的10株国内外PEDV毒株进行比对分析,并构建遗传进化树。10株PEDV参考毒株分别为中国株PEDV-LY(KM609210.1)、YN1-PEDV(KT021227.1)、CH-GDZQ(KM242131.1)、CHGD-01(JX261936.1)和CV777(AF353511.1);日本株AOM-3-JPN(LC063833.1)和FKO-1-JPN(LC063811.1);美国株USA-Minnesota236(KR265794.1)和PC177(KR078300.1);韩国株 KUIDL-PED(KJ588063.1)。
2 结果
2.1 PEDV S、M和N基因的PCR扩增结果
分别以反转录后的5株PEDV毒株(SX-HZ、SX-SL、3SX-WN、SX-BJ 和SX-YL)的cDNA为模板,经PCR扩增S(S1、S2、S3)、M和N基因后得到了相应的目的片段,各扩增片段的大小与预期片段大小一致。S1、S2和S3基因片段大小分别为1 405、1426、1 394 bp(图1),M基因片段大小为681 bp(图2),N基因片段大小为1 326 bp(图3)。
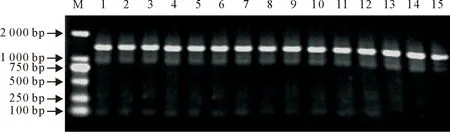
M.DNA标准 DL 2 000;1~5.S1基因;6~10.S2基因;11~15.S3基因

M.DNA 标准 DL 2 000;1~5.M基因PCR扩增产物
M.DNA Marker DL 2 000;1-5.PCR products of M gene
图2 PFDV M基因的扩增
Fig.2 The amplification of PFDV M gene

M.DNA 标准DL 2 000; 1~5.N基因PCR扩增产物
2.2 PEDV S基因的序列同源性分析
用生物分析软件对5株PEDV的S基因进行分析。结果表明,SX-WN(渭南)株和SX-HZ(汉中)株基因组含4161个核苷酸,编码1 386个氨基酸,而SX-YL(榆林)株、SX-SL(商洛)株和SX-BJ(宝鸡)株是由4 158个核苷酸组成,编码1 385个氨基酸。对5株PEDV的S基因进行核苷酸和推导的氨基酸序列进行分析比较。5个毒株间的核苷酸序列同源性为96.6%~99.8%,氨基酸序列同源性为97.3%~99.9%;5个毒株与疫苗株CV777之间核苷酸和氨基酸序列的同源性分别为93.9%~99.8%和93.6%~99.9%;与中国分离株PEDV-LY(KM609210.1)、YN1-PED(KT021227.1)、CH-GDZQ (KM242131.1)和CHGD-01(JX261936.1)的核苷酸和氨基酸的同源性分别为96.3%~99.8%和96.5%~99.9%;与日本分离株AOM-3-JPN(LC063833.1)和FKO-1-JPN(LC063811.1)的序列同源性分别为96.7%~99.8%和97.3%~99.9%;与美国分离株USA-Minnesota236(KR265794.1)和PC177(AF353511.1)的同源性分别为96.6%~99.8%和97.2%~99.9%;与韩国分离株KUIDL-PED(KJ588063.1)的同源性分别为96.7%~99.8%和96.6%~99.8%。
2.3 PEDV S基因的遗传进化树分析
从PEDV S基因的进化树结果可以看出(图4),有9株PEDV国内外流行毒株(CV777除外)与该5株PEDV毒株属于同一个进化分支,并且SX-YL、SX-SL和SX-BJ与中国分离株CHGD-01(JX261936.1)亲缘关系极其密切,而SX-WN和SX-HZ株与中国分离株PEDV-LY(KM609210.1)关系密切,与中国株CH-GDZQ(KM242131.1)和YN1-PEDV(KT021227.1),日本株AOM-3-JPN(LC063833.1)和FKO-1-JPN( LC063811.1),美国株USA-Minnesota236(KR265794.1)和PC177(KR078300.1)以及韩国株KUIDL-PED(KJ588063.1)亲缘关系较密切。此外,5株PEDV与中国现用疫苗株CV777(AF353511.1)均不在同一个进化分支上,亲缘关系远。
2.4 PEDV M基因的序列同源性分析
分析陕西省5株PEDV毒株的M基因发现,其M基因均由681个核苷酸组成,编码227个氨基酸,用DNA Star软件中的Meg Align功能对5个毒株的M基因进行核苷酸和推导的氨基酸序列比较显示,毒株之间的核苷酸序列同源性为98.4%~100%,氨基酸序列同源性为98.2%~100%;该5个毒株与疫苗株CV777之间核苷酸和氨基酸序列同源性分别为98.1%~100%和98.2%~100%,与中国分离株PEDV-LY(KM609210.1)、YN1-PED(KT021227.1)、CH-GDZQ(KM242131.1)和CHGD-01(JX261936.1)的序列同源性分别为98.1%~100%和98.2%~100%,与日本株AOM-3-JPN(LC063833.1)和FKO-1-JPN(LC063811.1)的序列同源性分别为98.4%~100%和98.2%~100%,与美国株USA-Minnesota236 (KR265794.1)和PC177(AF353511.1)的序列同源性分别为98.4%~100%和98.2%~100%,与韩国株KUIDL-PED(KJ588063.1)的序列同源性分别为98.4%~100%和98.2%~100%。
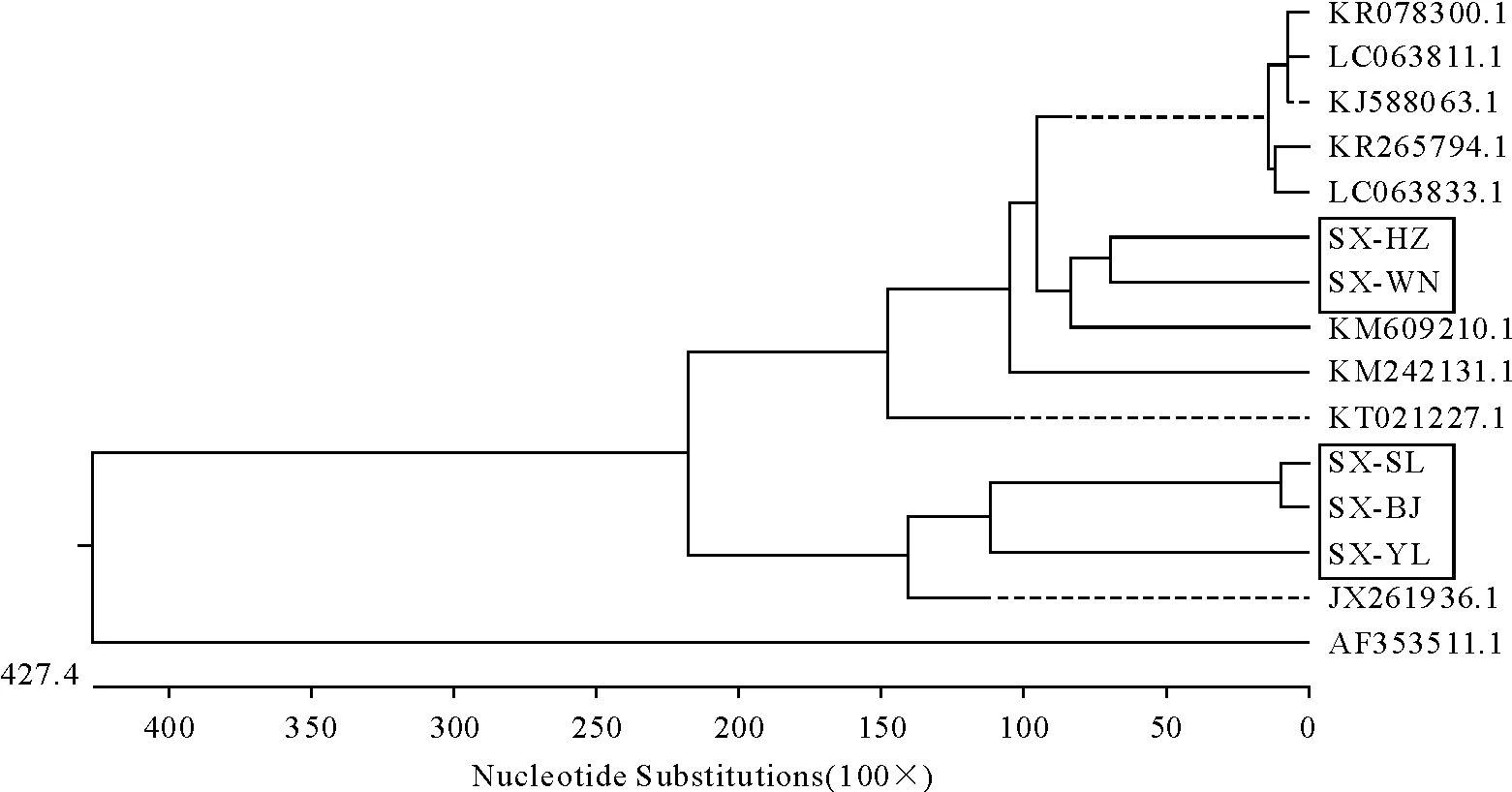
图4 基于PEDV S基因核苷酸序列构建的进化树
2.5 PEDV M基因遗传进化树分析
基于PEDV M基因的遗传进化树可以看出(图5),PEDV分成2个分支,SX-WN和SX-HZ株与中国分离株PEDV-LY(KM609210.1)、YN1-PED(KT021227.1)和CH-GDZQ(KM242131.1),日本株AOM-3-JPN(LC063833.1)和FKO-1-JPN(LC063811.1),美国株USA-Minnesota236(KR265794.1)和PC177(AF353511.1)以及韩国株KUIDL-PED(KJ588063.1)同属于一个进化分支,亲缘关系极其密切。而SX-SL、SX-BJ 和SX-YL株与中国株CHGD-01(JX261936.1)和中国疫苗株CV777(AF353511.1)同属一个进化分支并且亲缘关密切。
2.6 PEDV N基因序列同源性分析
该5株病毒N基因均由1 326个核苷酸组成,编码442个氨基酸。毒株之间的核苷酸序列同源性在97.2%~99.9%,氨基酸序列同源性在98.2%~100%;该5个毒株与疫苗株CV777之间核苷酸和氨基酸序列的序列同源性分别为95.3%~99.9%和96.2%~100%,与中国株PEDV-LY(KM609210.1)、YN1-PED(KT021227.1)、CH-GDZQ-2014(KM242131.1)和CHGD-01(JX261936.1)同源性分别为96.0%~100%和96.6%~100%;与日本株AOM-3-JPN-2014(LC063833.1)和FKO-1-JPN-2014 LC063811.1)的同源性分别为97.2%~99.9%和98.2%~100%;与美国株USA-Minnesota236(KR265794.1)和PC177(KR078300.1)的同源性分别为97.2%~99.9%和98.2%~100%,与韩国株KUIDL-PED的同源性分别为97.2%~99.9%和98.2%~100%。
2.7 PEDV N基因遗传进化树分析
由PEDV N基因的遗传进化树可以看出(图6),陕西省分离株SX-WN和SX-HZ与中国株PEDV-LY(KM609210.1)和CH-GDZQ-2014(KM242131.1)关系极其密切,与日本株AOM-3-JPN(LC063833.1)和FKO-1-JPN(LC063811.1),美国株USA-Minnesota236(KR265794.1)和PC177(KR078300.1)以及KUIDL-PED(KJ588063.1)同属于一个进化分支,亲缘关系较为密切,而与中国疫苗株(CV777)亲缘关系较远。SX-SL、SX-BJ和SX-YL与中国株YN1-PEDV(KT021227.1)、CHGD-01(JX261936.1)和中国疫苗株CV777(AF353511.1)同属于一个进化分支,与中国株CHGD-01(JX261936.1)亲缘关系尤为密切。
3 讨论
近年来,PED的疫情在我国各个省份相继流行,陕西也不例外,大多数规模化养殖场对PEDV都进行了正常免疫,但许多猪场还是暴发了PED,给养猪业带来巨大的经济损失。在环境、营养和管理都越来越规模化的养殖条件下,PED能频繁的流行跟PEDV自身的变异是否存有一定的联系。本试验对陕西省部分地区的PEDV毒株进行S、M和N基因的遗传变异分析,为PEDV的防控提供理论依据。

图5 基于PEDV M基因核苷酸序列构建的进化树
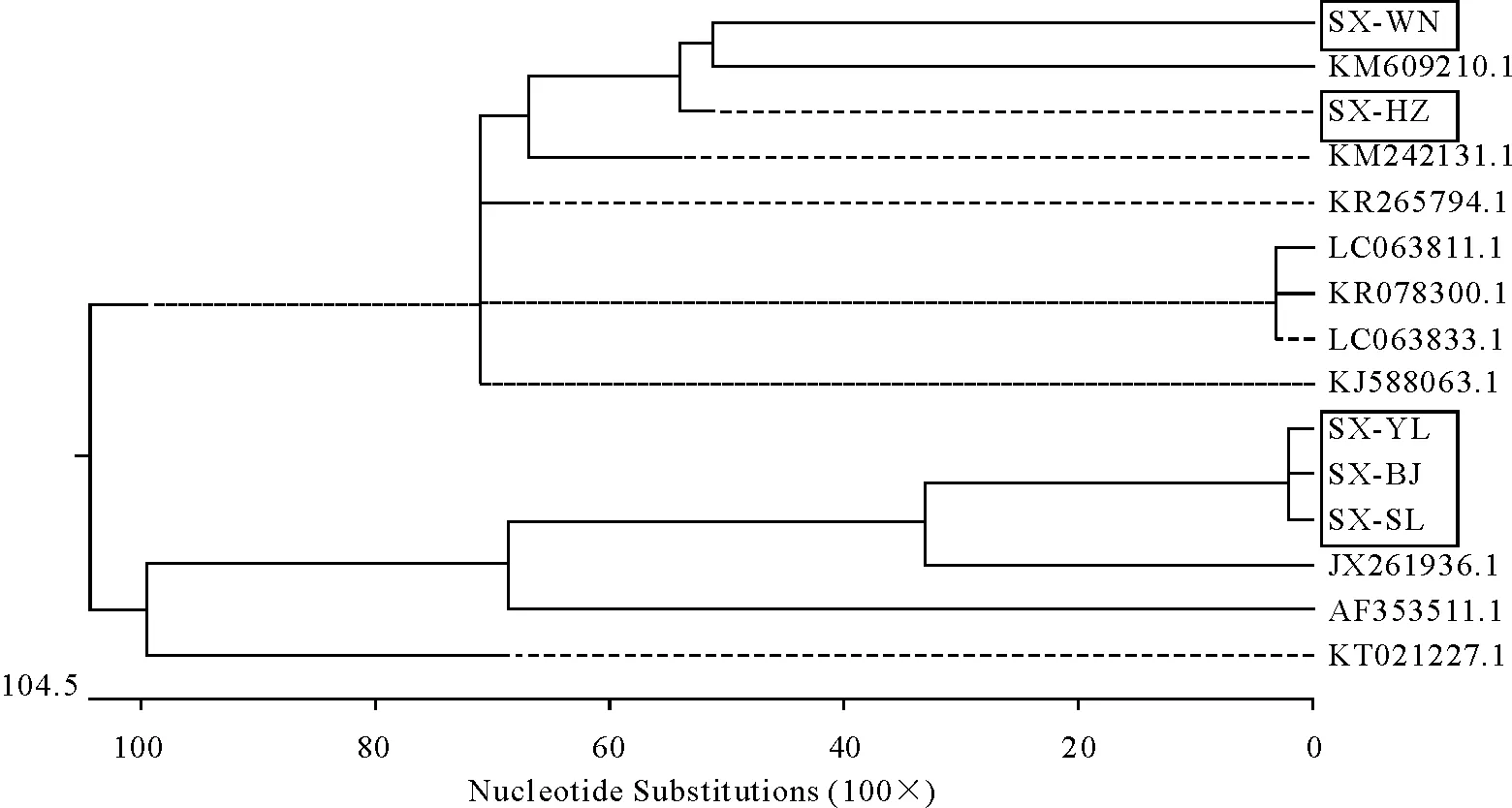
图6 基于PEDV N基因核苷酸序列构建的进化树
将陕西省部分地区具有代表性的5株PEDV进行S、M以及N基因测序分析,该5个毒株间的S、M和N基因之间的核苷酸同源性分别为96.7%~99.8%、98.4%~100%和97.2%~99.9%,氨基酸同源性分别为97.4%~99.9%、98.2%~100%和98.2%~100%。而与中国疫苗株CV777进行比较,5个分离株的S、M和N基因与中国疫苗株CV777之间核苷酸同源性分别为93.9%~99.8%、98.1%~100%和95.3%~99.9%,氨基酸同源性分别为93.6%~99.9%、96.2%~100%和98.2%~100%,表明5株分离株的S基因与疫苗株CV777存在相对较大的差异。
5株PEDV的S基因与中国疫苗株CV777亲缘关系较远,与其他9株国内外PEDV毒株亲缘关系较近,说明S基因的遗传变异大。由M和N基因的进化分析显示,M和N基因均分为2个进化分支,且分离株中SX-SL株、SX-BJ 株和SX-YL株同属于一个进化分支,与中国疫苗株CV777和中国株CHGD-01(JX261936.1)亲缘关系密切,而分离株SX-WN和SX-HZ同属于一个进化分支,与中国疫苗株CV777亲缘关系远,说明SX-WN株和SX-HZ株的M基因和N基因遗传变异较大,SX-SL株、SX-BJ 株和SX-YL株的M基因和N基因遗传变异小。针对S、M和N基因的这些变异情况,推测这可能是现用疫苗免疫失败而致使陕西省PEDV流行的原因。另一方面,测得的陕西省该5株PEDV流行株中SX-SL株、SX-BJ 株和SX-YL株可能是由中国株CHGD-01(JX261936.1)进化而来,而分离株SX-WN和SX-HZ与韩国株、日本株以及其他地区中国的PEDV流行毒株的亲缘关系较近,这提示PEDV流行株的变异可能与周边国家畜产品的贸易往来有一定的联系,可能源于韩国和日本等周边国家,其内在原因和机理需进一步的研究。同时说明最近在陕西广泛流行的PEDV也并不完全相同。
[1] Pan Y,Tian X,Wei L,et al.Isolation and characterization of a variant porcine epidemic diarrhea virus in China.[J].Virol J,2012,9(1):195.
[2] 卓秀萍,朱 玲,乔小改,等.人工感染猪流行性腹泻病毒的哺乳仔猪的病理学观察[J].中国兽医科学,2015,45(2):202-207.
[3] Shibata I,Tsuda T,Mori M,et al.Isolation of porcine epidemic diarrhea virus in porcine cell cultures and experimental infection of pigs of different ages [J].Vet Microbiol,2000,72(3-4):173-182.
[4] Vlasova A N,Marthaler D,Wang Q,et al.Distinct characteristics and complex evolution of PEDV strains,North America,May 2013-February 2014[J].Emerg Infect Dis,2014,20(10):1620-1628.
[5] 高君恺,刘浩飞,杨 倩.猪流行性腹泻病毒的研究进展[J].南京农业大学学报,2014,37(1):1-5.
[6] Brian D A,Baric R S.Coronavirus genome structure and replication [J].Current Topics Microbiol Immunol,2005,287:1-30.
[7] Stevenson G W,Hoang H,Schwartz K J,et al.Emergence of porcine epidemic diarrhea virus in the United States:clinical signs,lesions,and viral genomic sequences.[J].J Vet Diagn Invest,2013,25(5):649-654.
[8] 翁善钢.2014年国际猪病疫情回顾—猪流行性腹泻在全球的流行与传播[J].肉类工业,2015(2):49-50.
[9] Sozzi E,Luppi A,Lelli D,et al.Comparison of enzyme-linked immunosorbent assay and RT-PCR for the detection of porcine epidemic diarrhoea virus[J].Res Vet Sci,2010,88(1):166-168.
[10] 孙振钊,赵伟男,王靖飞.猪腹泻病例中PEDV、TGEV及PRV的感染调查[J].动物医学进展,2014,35(5):132-135.
[11] 牛俊超,黄复深,胡佩佩.猪流行性腹泻研究进展[J].动物医学进展,2014,35(10):107-110.
[12] 杨丽梅,马 力,徐倩倩,等.我国猪病毒性腹泻的诊断与流行病学调查研究概况[J].动物医学进展,2014,35(2):115-119.
[13] 常铁城,陈建飞,冯 力,等.2014年部分地区猪流行性腹泻病毒流行病学调查[J].中国预防兽医学报,2016,38(4):335-338.
[14] 谢红玲,崔尚金,崔玉东.猪流行性腹泻及病毒研究进展[J].畜禽业,2008,225(1):10-13.
[15] Oh J,Lee K W,Choi H W,et al.Immunogenicity and protective efficacy of recombinant S1 domain of the porcine epidemic diarrhea virus spike protein.[J].Arch Virol,2014,159(11):2977-2987.
[16] 陈弟诗,任玉鹏,张 斌,等.猪流行性腹泻病毒S基因研究进展[J].动物医学进展,2014,35(7):77-81.
[17] Gao Y,Kou Q,Ge X,et al.Phylogenetic analysis of porcine epidemic diarrhea virus field strains prevailing recently in China[J].Arch Virol,2013,158(3):711-715.
[18] Zhao P D,Bai J,Jiang P,et al.Development of a multiplex TaqMan probe-based real-time PCR for discrimination of variant and classical porcine epidemic diarrhea virus[J].J Virol Meth,2014,206(1):150-155.
Genetic Variation Analysis of S,M and N Genes of Porcine Epidemic Diarrhea Virus in Partial Regions of Shaanxi Province
PANG Wen-jing,HE Ya-peng,FU Ming-zhe,XU Xin-gang,GUO Kang-kang,ZHANG Qi
(CollegeofVeterinaryMedicine,NorthwestA&FUniversity,Yangling,Shaanxi,712100,China)
In order to understand the variation of PEDV in Shaanxi province,five suspected samples of porcine epidemic diarrhea virus (PEDV) infection of intestinal contents were collected from five large-scale pig farms in partial regions of Shaanxi province and S,M and N genes were amplified using RT-PCR.The analysis results showed that the PEDV S,M and N genes of 5 samples could be amplified and the 5 virus strains were named SX-SL,SX-BJ,SX-YL,SX-WN and SX- HZ respectively.The nucleotide sequence of S,M and N genes shared 96.7%-99.8%,98.4%-100% and 98.4%-99.8% identity within them.The amino acids sequence of S,M and N gene shared 97.4%-99.9%,98.2%-100% and 98.2%-100% identity within them.The nucleotide sequence of S M and N genes shared 93.9%-99.8%,98.1%-100% and 95.3%-99.9% and the amino acid sequence of S,M and N genes,shared 93.6%-99.9%,96.2%-100% and 98.2%-100% identity with CV777 strain.Phylogenetic analysis indicated that S gene of the 5 PEDV strains had a farther relationship with CV777 strain and had a closer relationship with the China,South Korea and Japan strains.SX-SL,SX-BJ and SX-YL strains had a closer relationship with CV777 strain and had a closer relationship with the CHGD-01 strain.But the M and N genes of the SX-WN and SX-HZ strains had a farther relationship with CV777 strain.S gene of the 5 PEDV strains and M and N genes of SX-WN and SX-HZ strains varied greatly,and these strains might origin from China,South Korea and Japan strains.SX-SL,SX-BJ and SX-YL strains had a closer relationship with the CHGD-01 strain and these strains might origin from the CHGD-01strain.The epidemic strains of PEDV in Shaanxi province were different.
PEDV; S gene; M gene; N gene; sequence analysis
2016-05-16
陕西省农业科技创新与攻关项目(2016NY-095);西北农林科技大学基本科研业务费项目(Z109021427)
庞文静(1991-),女,江苏连云港人,硕士,主要从事分子病原学与免疫学研究。*通讯作者
S852.659.6
A
1007-5038(2016)11-0025-06
