布鲁氏菌Omp10蛋白抗原表位的生物信息学分析
2016-10-22朱玥洁胡金伟张峰波甫拉提热西提张春桃丁剑冰
杨 妍, 朱玥洁, 胡金伟, 张峰波, 甫拉提·热西提, 张春桃, 贾 斌, 庞 盼, 丁剑冰
(新疆医科大学第一附属医院1输血科, 2生殖助孕中心, 3检验科, 乌鲁木齐 830054; 4新疆医科大学基础医学院免疫学教研室, 乌鲁木齐 830011)
·基础医学研究·
布鲁氏菌Omp10蛋白抗原表位的生物信息学分析
杨妍1, 朱玥洁2, 胡金伟3, 张峰波3, 甫拉提·热西提4, 张春桃4, 贾斌3, 庞盼3, 丁剑冰4
(新疆医科大学第一附属医院1输血科,2生殖助孕中心,3检验科, 乌鲁木齐830054;4新疆医科大学基础医学院免疫学教研室, 乌鲁木齐830011)
目的用生物信息学技术预测布鲁氏菌Omp10蛋白的抗原表位,寻找布鲁氏菌Omp10的优势表位。方法利用IEDB在线软件、DNAStar软件、ProPred MHC Class-Ⅱ Binding Peptide Prediction Server软件和SYFPEITHI软件,通过分子结构、亲水性、柔韧性、表面可及性以及抗原指数等在线分析Omp10蛋白的二级结构、B细胞抗原表位及T细胞抗原表位。结果布鲁氏菌Omp10蛋白B细胞抗原表位有4个,为26-34、65-75、97-102和115-120氨基酸残基。Omp10蛋白T细胞抗原表位有6个,为64-79、72-87、104-119、6-21、9-24和78-93氨基酸残基。结论通过综合分析布鲁氏菌Omp10蛋白有4个B细胞抗原表位和6个T细胞抗原表位,为进一步筛选具有一定抗原保护性和免疫原性的优势表位和多价疫苗构建奠定了基础。
布鲁氏菌; Omp10蛋白; 抗原表位
布鲁氏菌病(Brucellosis)是公认的一种常见的人兽共患疾病,是由布鲁氏菌通过皮肤黏膜、呼吸道、消化道传播感染宿主动物,通过接触或食入感染动物的分泌物、体液等来建立感染的疾病[1-2]。引起的临床症状有波状热[3-5]、睾丸炎[6-7]、脊柱炎、关节炎[8-9]、心内膜炎等等,可引起人类全身性感染,且不受年龄和性别的影响,临床症状多变,难以控制[10-11]。该病分布广泛,目前已有170多个国家有该病的发生和流行。近年来,该病有逐年增多的趋势,没有有效的治疗方法,使得患者和社会承受了巨大的经济负担[12]。随着分子生物学技术的飞速发展,大量研究表明布鲁氏菌的外膜蛋白有很强的免疫原性,可能与布鲁氏菌在巨噬细胞内存活有关[13]。为了减少疾病的痛苦与社会的损失,有必要开发安全有效的疫苗。
Omp10是布鲁氏菌重要的免疫原性蛋白,分子量大小为10 ku,属于菌体外膜脂蛋白,存在于已知所有的种型中。Omp10蛋白具有良好的抗原性与免疫保护性,不仅可刺激体液免疫而且对细胞免疫也有刺激作用。在目前疫苗研制过程中,Omp10蛋白被认为是一种较为理想的疫苗分子,越来越引起人们的关注,但是人们对Omp10蛋白的抗原特异性及表位研究较少,故本研究对布鲁氏菌Omp10蛋白的抗原表位进行了生物信息学结构分析和预测。
1 材料与方法
1.1材料Omp10蛋白氨基酸序列:从GenBank中获得的氨基酸序列:MESMDMKRFRIVAPLAL-
MSLALAACETTGPGSGNAPIIAHTPAGIEGSW-
VDPNGIASSFNGGIFETRTTDTNEKLAEGNYL-
YLSPQLVEINMRSIVRGTTSKVNCALVSPTQL-
NCTSSAGSRFSLTRRNAG,共131个氨基酸。运用IEDB在线分析软件和DNAStar软件进行B细胞表位的分析预测。运用ProPred MHC Class-Ⅱ Binding Peptide Prediction Server软件和SYFPEITHI软件在线分析进行T细胞表位预测。
1.2方法利用Gamier-Robson和Chou-Fasman方法预测Omp10蛋白的二级结构。利用Emini法预测特定的氨基酸残基组成的区域位于蛋白分子表面的可能性,反映了蛋白抗原内外各层残基的分布情况;利用Hydropathy-Kyte-Doolitte法来预测蛋白分子的疏水区和亲水区;利用Karplus-Schulz法来预测蛋白分子多肽链骨架区的柔韧性;利用Antigenicity-Jameson-Wolf法来预测潜在的蛋白抗原决定簇。
2 结果
2.1Omp10 蛋白B细胞抗原表位的预测分析
2.1.1IEDB在线软件预测分析
2.1.1.1线性表位根据得分由高到低筛选了5段优势区段,分别为26-40、42-58、64-76、100-102、112-120(图1)。
2.1.1.2β转角得分最高的为28-34,其次为29-35、30-36、27-33、47-53、48-54、56-62、114-120、31-37、26-32、115-121(图2)。高分的位置主要分布在27-37氨基酸区域。
2.1.1.3表面可及性得分最高的是69-74,其次为65-70、67-72、66-71、68-73、70-75、5-10、97-102、124-129、126-131(图3)。高分的位置主要集中在65-75氨基酸区域。
2.1.1.4亲水性得分最高的是68-74,其次是67-73、65-71、66-72、26-32、28-34、116-122、27-33、29-35、114-120(图4)。高分的位置集中在65-74氨基酸区域。
2.1.1.5柔韧性得分由高到低依次为28-34、29-35、27-33、26-32、97-103、30-36、96-102、98-104、68-74、69-75(图5)。高分位置主要分布在26-35氨基酸区域。
2.1.1.6抗原指数得分高的前10位是103-109、11-17、102-108、82-88、19-25、105-111、81-87、104-110、20-26、80-86氨基酸区域(图6)。
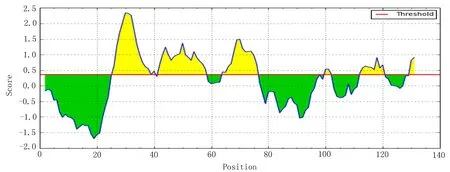
图1 线性表位预测
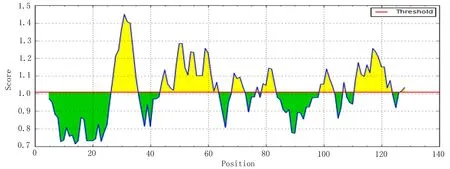
图2 β转角预测
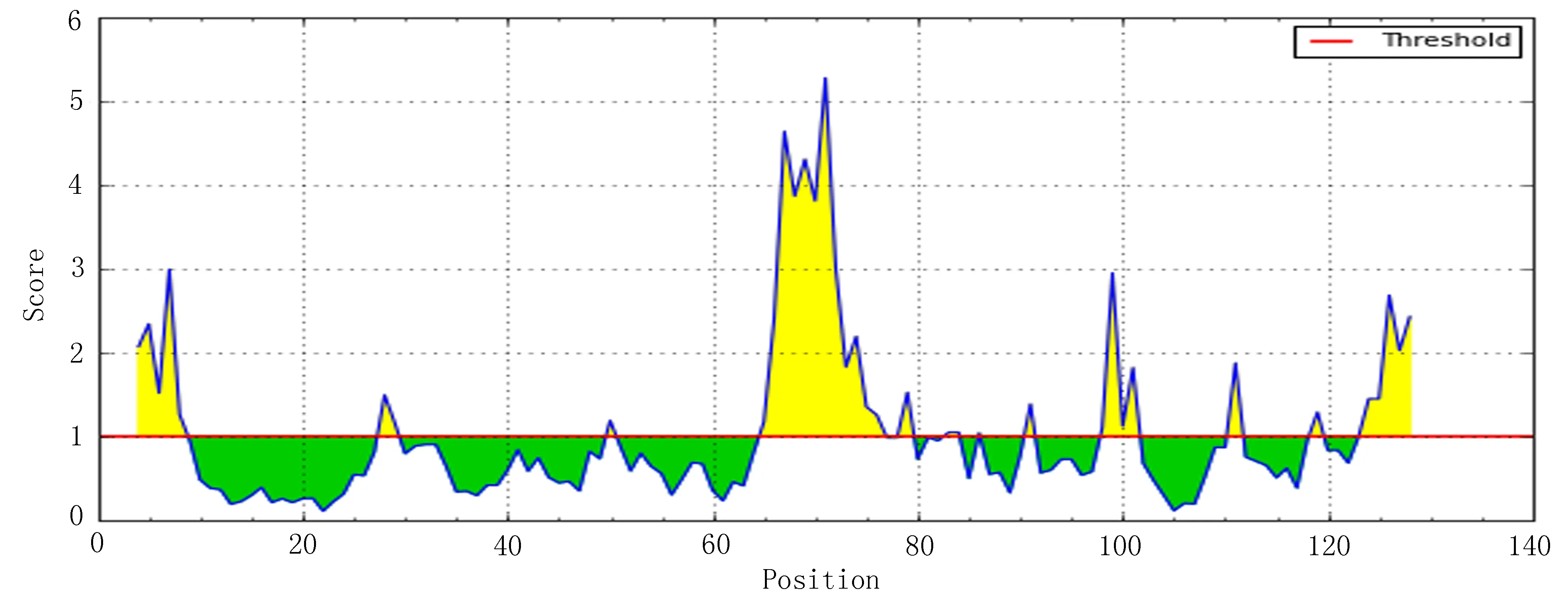
图3 表面可及性预测
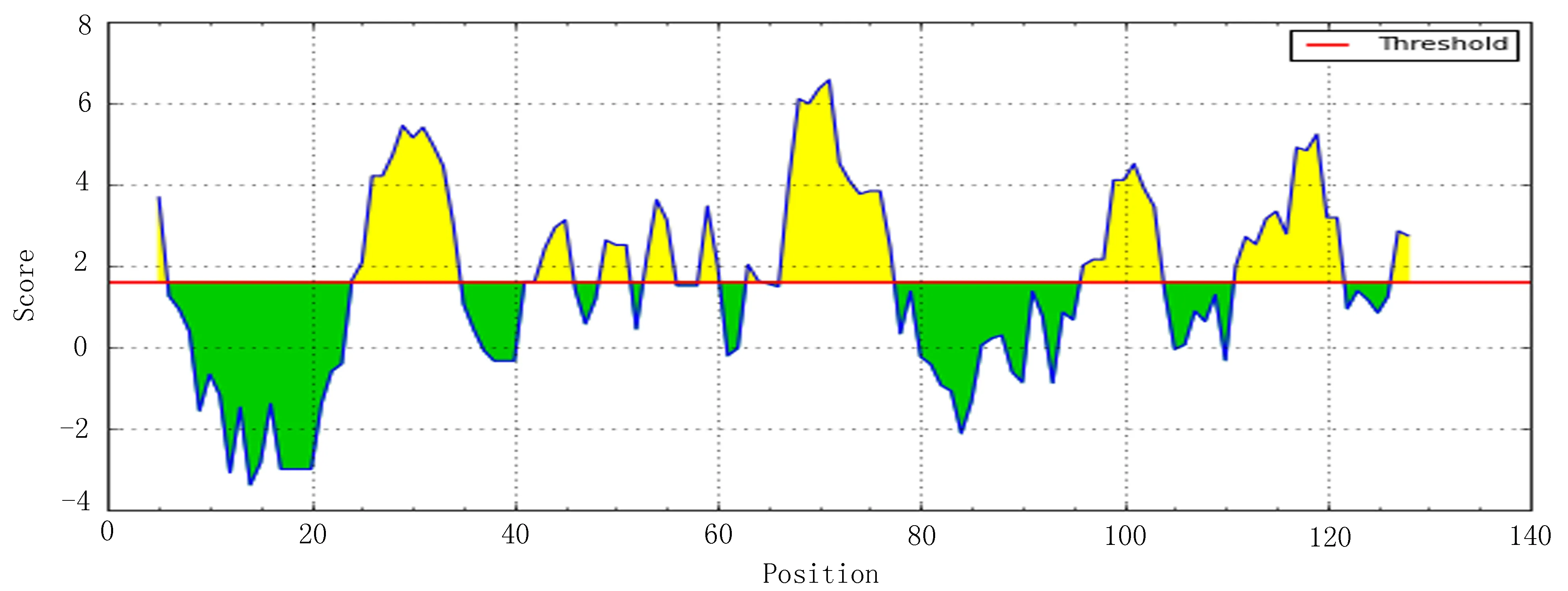
图4 亲水性预测
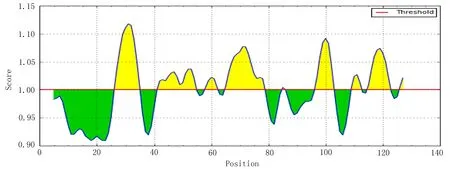
图5 柔韧性预测
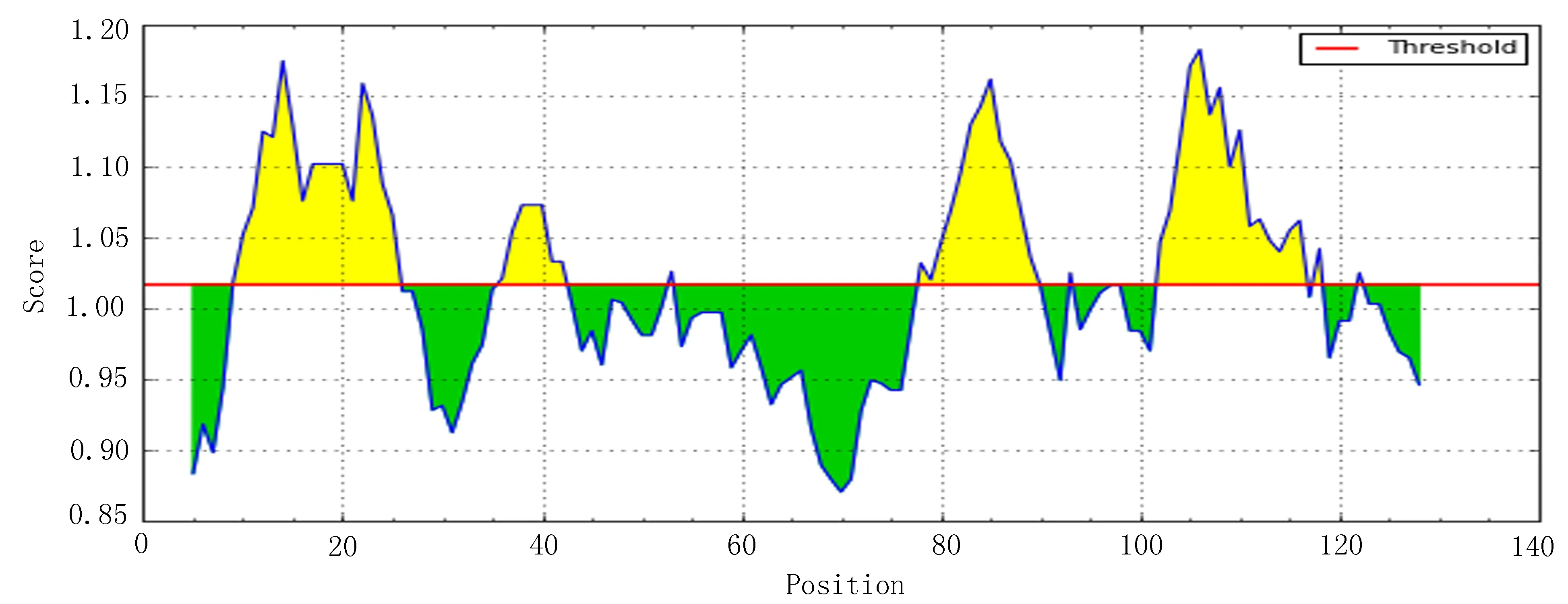
图6 抗原指数预测
2.1.2DNAStar软件预测分析综合Gamier-Robson方法和Chou-Fasman方法分析,Omp10蛋白的α-螺旋优势表位区段为:1-29、71-79;β转角的优势抗原表位区段为:24-28、34-41、46-50、55-60、63-68、79-97、101-116、121-126。在α-螺旋和β转角之间分布着13段无规则卷曲,分别为:27-33、41-45、46-49、50-54、59-63、68-73、76-79、83-87、90-94、97-102、107-111、113-122、123-130。Emini法分析显示,表面可及性较好的区段,分别为:1-8、64-75、123-127。Hydropathy-Kyte-Doolitte法分析表明,Omp10蛋白有5段亲水性较好的区域,分别为:1-10、26-34、47-55、61-78、109-130。Karplus-Schulz法分析显示,Omp10蛋白有8段柔韧性较好的区段,分别为:27-35、41-54、56-61、64-78、96-102、107-111、113-121、124-126。Antigenicity-Jameson-Wolf法分析显示,Omp10蛋白抗原指数较高的区域有:1-11、26-34、41-55、59-76、94-102、115-130(图7)。
综上,根据两款软件的分析结果,显示26-34、65-75、97-102、115-120氨基酸残基区域可能为潜在B细胞抗原表位。
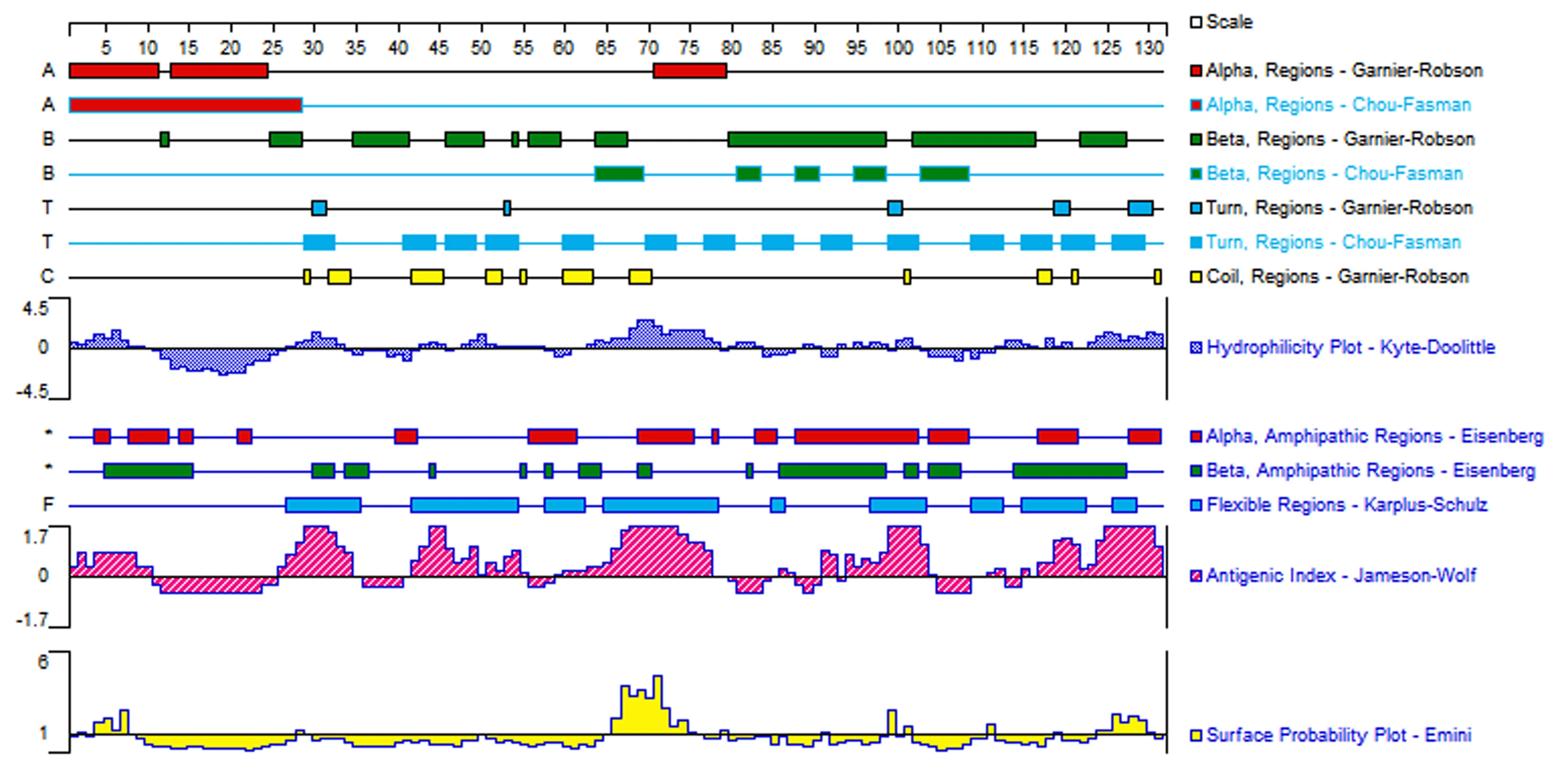
图7 DNAStar分析Omp10蛋白的各项参数(二级结构、表面可及性、亲水性、柔韧性及抗原指数分析)
2.2Omp10蛋白T细胞抗原表位的预测分析为了提高预测的准确性,本研究选取等位基因HLA-DRB1*0301,用ProPred MHC Class-ⅡBinding Peptide Prediction Server软件在线分析得出9-17、88-103段出现了优势表位(图8)。运用SYFPEITHI软件在线分析,得分最高的区段为64-79,其次为72-87、104-119、6-21、9-24、78-93、71-86、89-104、45-60、52-67(图9)。根据这两款软件的分析结果,显示64-79、72-87、104-119、6-21、9-24、78-93氨基酸残基区域为潜在T细胞抗原表位。
3 讨论
布鲁氏菌属胞内寄生菌,可在机体内长期存活。急性期患者如得不到及时有效的治疗,往往会转为慢性感染,机体很难再将该病菌彻底清除,然而长期服用抗菌药物很容易产生耐药性。因此,预防布鲁氏菌病最有效的途径是对易感动物和人群进行疫苗接种。减毒活疫苗是目前公认的预防措施,具有良好的免疫效果,但却难以广泛开展,原因是普遍存在毒性较大、无法区分人工免疫与自然感染等问题,而表位疫苗可以解决这些问题,所以研究者们将关注的重点逐渐转移到表位疫苗上,如马秀敏等[14]通过对AgB1抗原表位预测分析为研制包虫病表位疫苗提供了理论依据。李江英等[15]为研制抗结核疫苗对Rv1410c结核分枝杆菌抗原表位进行分析预测。通过对抗原表位的预测,可为筛选优势的表位疫苗奠定基础。表位也被称为抗原决定簇,是决定抗原的特异性的化学基团,是刺激机体产生抗体并能够被其识别的部位。表位可分为B细胞表位和T细胞表位。B细胞表位主要位于抗原分子表面,能与未经抗原呈递细胞加工的抗原发生反应,诱导体液免疫应答产生特异性抗体;T细胞表位是短线性肽,从MHC分子抗原呈递给T细胞受体与细胞免疫反应相关联[16]。线性表位由肽链上顺序连续的氨基酸组成。完整蛋白的抗体可以与模拟这种表位的短线性肽结合,由于结合力弱且并不含完整天然蛋白的构象,所以只能代表复杂表位的一部分。线性表位多见于T细胞表位和部分B细胞表位。抗原表位可以通过生物信息学分析预测,本研究对Omp10蛋白的抗原表位进行预测,单参数预测的正确率不是很高,所以通常要综合考虑多种参数[17]。

图8 ProPred MHC Class-ⅡBinding Peptide Prediction Server软件在线分析优势表位
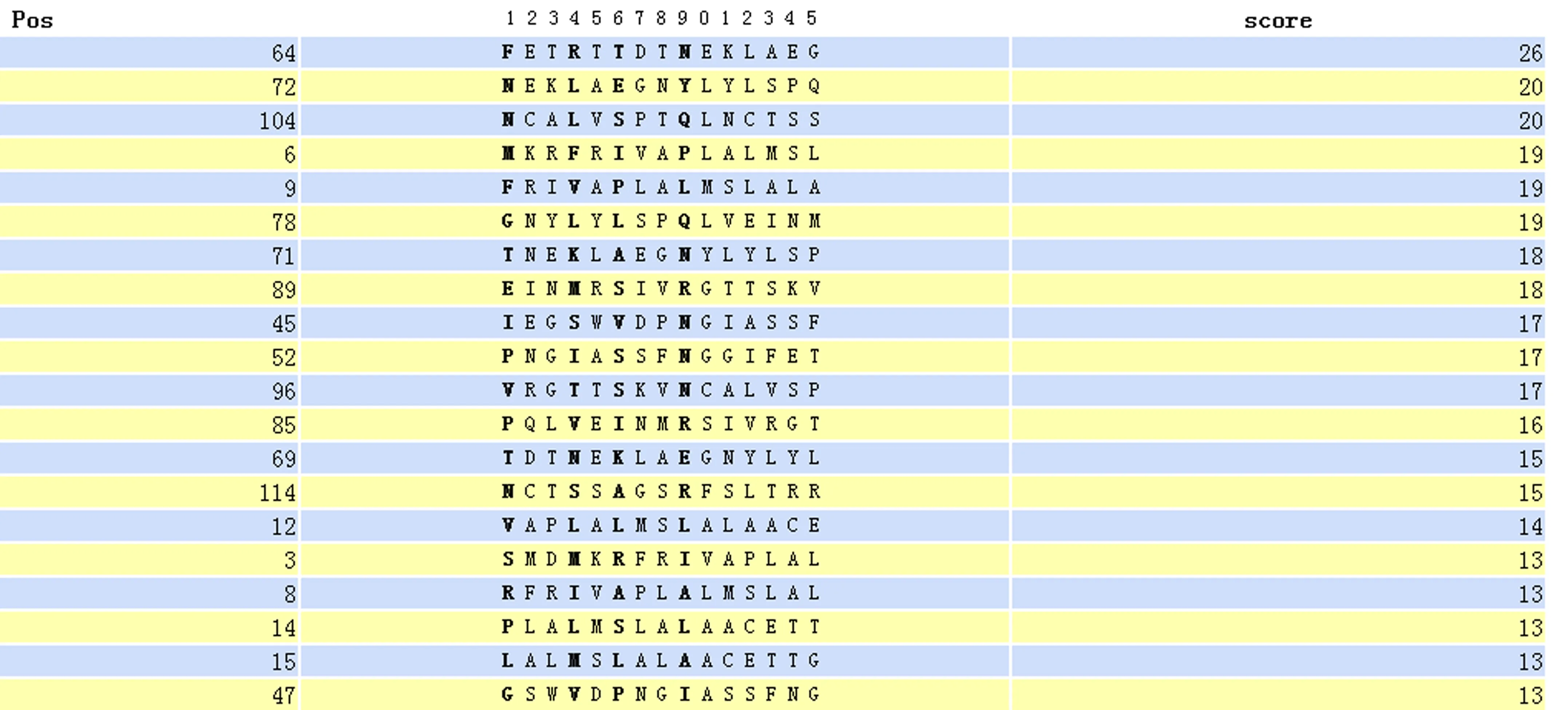
图9 SYFPEITHI软件在线分析得分区段
在蛋白质的二级结构中,α-螺旋和β-折叠的氢键键能高,形成稳定的刚性结构,在蛋白质内部起骨架作用且不易变形,不易与抗体结合。相反,转角和卷曲区域的氢键键能低,易形成盘旋、扭曲等松散的柔性结构出现在蛋白质抗原表面,利于与抗体结合,易形成抗原表位。预测Omp10蛋白抗原表位的多个参数包括表面可及性、亲水性、柔韧性及抗原指数。表面可及性是指蛋白抗原内、外残基的分布情况,其反映了氨基酸残基被溶剂分子接触的可能性,表面可及性越大越易形成表位。蛋白亲水部位与抗原表位有着密切的联系,亲水性残基主要位于蛋白质表面,而疏水性残基主要包埋在蛋白质的内部。蛋白质的多肽骨架具有折叠和弯曲的能力,也就是蛋白质的柔性区域。柔韧性高的氨基酸残基即灵活性强的位点容易形成抗原表位。抗原指数分析表明了抗原的免疫原性区域,潜在的占主导地位的表位很有可能位于高抗原指数区域。结合蛋白的多个参数各自的阈值,综合其峰值可预测其表位[18],并提高准确率。本研究通过IEDB在线预测分析软件预测的B细胞抗原表位位于26-35、65-75、97-102、114-120、126-129共5个氨基酸残基区域。DNAStar软件分析预测的Omp10蛋白B细胞抗原表位可能位于26-34、47-55、59-61、64-75、96-102、109-111、115-121、123-126共8个氨基酸残基区域。由于IEDB在线预测分析软件和DNAStar软件在分析抗原指数参数时不尽相同,可能与不同软件的分析方法不一致有关系,所以结合两款软件的分析结果,显示26-34、65-75、97-102、115-120氨基酸残基区域可能为潜在B细胞抗原表位。根据这两款软件的分析结果,显示64-79、72-87、104-119、6-21、9-24、78-93氨基酸残基区域为潜在T细胞抗原表位。64-79、72-87、104-119、6-21、9-24、78-93氨基酸残基区域为潜在T细胞抗原表位。
表位疫苗的研究和开发是一项困难而有针对性的工作,疫苗制备的关键步骤是获得所需信息相关的抗原表位,表位疫苗是根据抗原表位的氨基酸序列产生的。随着生物信息学技术的发展,表位预测技术有了显著的提高,多种方法多参数预测大大提高了抗原表位的预测准确度。在目前研究中,Omp10的这些表位可以作为新的疫苗候选,为有效疫苗的开发提供新的途径,为预防布鲁氏菌病感染奠定一定的实验基础。
[1]Franco MP, Mulder M, Gilman RH,et al. Human brucellosis[J].Lancet Infect Dis,2007,7(12):775-786.
[2]Arimi SM,Koroti E,Kang′ethe EK,et al. Risk of infection with Brucella abortus and Escherichia coli O157:H7associated with marketing of unpasteurized milk in Kenya[J].Acta Trop,2005,96(1):1-8.
[3]Whatmore AM. Current understanding of the genetic diversity of Brucella, an expanding genus of zoonotic pathogens[J].Infect Genet Evol,2009,9(6):1168-1184.
[4]Forland F, De Carvalho Gomes H, Nokleby H, et al. Applicability of evidence-based practice in public health: risk assessment on Q fever under an ongoing outbreak[J]. Euro Surveill,2012,17(3): 20060.
[5]Dean AS, Bonfoh B, Kulo AE, et al. Epidemiology of brucellosis and q Fever in linked human and animal populations in northern togo[J]. PLoS One,2013,8(8): e71501.
[6]Purwar S, Metgud SC, Gokale SK. Exceptionally high titres in atypical presentation of occult epididymo-orchitis due to brucellosis[J].J Med Microbiol,2012,61(Pt 3): 443-445.
[7]Erdem H, Elaldi N, Ak O, et al. Genitourinary brucellosis: results of a multicentric study[J].Clin Microbiol Infect,2014,20(11): 847-853.
[8]Hasanoglu I, Guven T, Maras Y, et al. Brucellosis as an aetiology of septic arthritis[J].Trop Doct,2014,44(1): 48-49.
[9]Baldi PC, Giambartolomei GH. Pathogenesis and pathobiology of zoonotic brucellosis in humans[J]. Rev Off Int Epizoot,2013,32(1): 117-125.
[10]Herrick JA, Lederman RJ, Sullivan B, et al. Brucella arteritis: clinical manifestations, treatment, and prognosis[J].Lancet Infect Dis,2014,14(6): 520-526.
[11]Akhvlediani T, Clark DV, Chubabria G, et al. The changing pattern of human brucellosis: clinical manifestations, epidemiology, and treatment outcomes over three decades in Georgia[J].BMC Infect Dis,2010,10: 346.
[12]Ducrotoy MJ, Bertu WJ, Ocholi RA, et al. Brucellosis as an emerging threat in developing economies: lessons from Nigeria[J].PLoS Negl Trop Dis,2014,8(7): e3008.
[13]董炳梅,王金良,唐娜,等.布鲁氏菌的致病机制与细胞免疫机制研究进展[J].中国人兽共患病学报,2012,28(6):635-639,643.
[14]马秀敏,胡晓安,阿尔孜古丽·吐尔逊,等.细粒棘球绦虫AgB1抗原表位的生物信息学预测[J].科技导报,2013,31(27):27-30.
[15]李江英,白雪娟,梁艳,等.用DNAStar软件预测Rv1410c结核分枝杆菌蛋白抗原表位[J].细胞与分子免疫学杂志,2015,31(4):474-477.
[16]Liu XL, Zhao H, Cao WY, et al. Bioinformatic prediction of the antigenic epitopes of recombinant ferritin of Echinococcus granulosus[J].Mol Med Rep,2016,13(1):888-894.
[17]Li YH, Liu XF, Zhu YJ, et al. Bioinformatic prediction of epitopes in the Emy162 antigen of Echinococcus multilocularis[J].Exp Ther Med,2013,6(2): 335-340.
[18]Zhang FB, Lu XB, Guo N, et al. The prediction of T- and B-combined epitope of Ag85B antigen of Mycobacterium tuberculosis[J].Int J Clin Exp Med,2016,9(2):1408-1421.
(本文编辑王艳)
Bioinformatics analysis of Brucella′s Omp10 antigen epitope
YANG Yan1, ZHU Yuejie2, HU Jinwei3, ZHANG Fengbo3, Pulati Rexiti4, ZHANG Chuntao4, JIA Bin3,PANG Pan3, DING Jianbing4
(1DepartmentofBloodTransfusion,2DepartmentofReproductiveFertilityCenter,3DepartmentofClinicalLaboratory,theFirstAffiliatedHospitalofXinjiangMedicalUniversity,Urumqi830054,China;4DepartmentofImmunology,SchoolofPreclinicalMedicine,XinjiangMedicalUniversity,Urumqi830011,China)
ObjectiveTo predicte the dominant epitopes of brucella Omp10 protein by bioinformatics analysis. MethodsThe secondary structure, B cell antigen epitope and T cell antigen epitope of OMP10 protein were infered from the molecular structure, hydrophily, flexibility, the surface accessibility, antigen index via the IEDB software, DNAStar software, ProPred MHC Class-Ⅱ Binding Peptide Prediction Server software and SYFPEITHI software online. ResultsOmp10 protein contains four B cell antigen epitopes(consist of 26-34, 65-75, 97-102 and 115-120 amino acid residues, respectively) and six T cell antigen epitopes(composed by 64-79, 72-87, 104-119, 6-21, 9-24 and 78-93 amino acid residues, separately). ConclusionThe results will provide a key clue for screening dominate epitope and constructing multivalent vaccines for the prevention of brucellosis.
Brucella; Omp10 protein; epitope
国家自然科学基金(81560322); 新疆维吾尔自治区自然科学基金(2015211C100); 新疆医科大学科学创新基金(XYDCX201507)
杨妍(1983-),女,硕士,主管技师,研究方向:布鲁氏菌感染疾病的研究。
作者简介:朱玥洁(1987-),女,在读硕士,研究方向:生殖免疫。
丁剑冰,女,教授,博士生导师,研究方向:感染免疫和生殖免疫研究,E-mail: djbing002@sina.com。
S852.61
A
1009-5551(2016)10-1261-06
10.3969/j.issn.1009-5551.2016.10.013
2016-02-18]
