夏眠期仿刺参肠组织实时荧光定量PCR分析中内参基因的筛选❋
2016-08-13张莉恒常亚青包振民
张莉恒, 王 师, 常亚青, 包振民, 丁 君❋❋
(1.大连海洋大学农业部北方海水增养殖重点实验室,辽宁 大连 116023;2.中囯海洋大学海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003)
夏眠期仿刺参肠组织实时荧光定量PCR分析中内参基因的筛选❋
张莉恒1, 王师2, 常亚青1, 包振民2, 丁君1❋❋
(1.大连海洋大学农业部北方海水增养殖重点实验室,辽宁 大连 116023;2.中囯海洋大学海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003)
摘要:在仿刺参(Apostichopus japonicus)正常时期、夏眠初期和中期,应用实时荧光定量PCR技术,检测CA043、dpolm、ro60、grb2、hiat1、nlrc4、farp1、cyc和gapdh 9个候选内参基因在肠组织中的表达稳定性。利用Delta Ct、GeNorm、NormFinder和BestKeeper 4种程序进行统计分析。发现上述条件中,9个候选内参基因在上述条件中的表达稳定性存在一定差异,不同的软件处理分析,得到的基因稳定性排序不完全一致。综合4种程序的方法,筛选出基因grb2表达最稳定,可用作内参基因,其次为基因ro60及dpolm,而表达最不稳定的是CA043,不适宜作为内参基因。本研究为仿刺参中基因表达定量分析奠定了基础,同时为仿刺参中内参基因的选择提供一定参考依据。
关键词:仿刺参;夏眠;内参基因;荧光定量PCR
引用格式:张莉恒, 王师, 常亚青, 等. 夏眠期仿刺参肠组织实时荧光定量PCR分析中内参基因的筛选[J].中国海洋大学学报(自然科学版), 2016, 46(7): 35-43.
ZHANG Li-Heng, WANG Shi, CHANG Ya-Qing, et al. Selection of reference genes for quantitative real-time PCR of Apostichopus japonicus intestine during different periods of aestivation[J].Periodical of Ocean University of China, 2016, 46(7): 35-43.
仿刺参(Apostichopusjaponicus)为中国现有海参中最主要的经济种类,目前已有学者从遗传育种学、发育学和免疫学等多个领域对仿刺参进行了研究,随着分子生物学的发展,对仿刺参的功能基因等分子生物学领域的研究已成为近年来研究热点。关于了解基因表达方式、揭示基因功能及阐明基因在生物体发育过程中的调控机制等研究中,基因表达分析具有至关重要的作用。在基因表达分析方法中,由于实时荧光定量PCR(Quantitative Real-time PCR)具有较高的敏感性和自动化程度、较好的重复性、较快的检测速度等优点,已被广泛应用[1-7]。利用该方法时,需要采用合适的内参基因对可能存在的差异进行校正及标准化。理想的内参基因是指在各种类型细胞、组织及不同处理条件下均表达恒定,但大量研究表明,大多常用内参基因只在特定细胞或组织、特定条件下表达较稳定,而在不同类型细胞、不同发育阶段及不同处理条件下表达具有差异[8-11],从而使实时荧光定量PCR实验的准确性受到内参基因选择的影响。因此,通过比较不同生理条件下、不同类型细胞及不同生长阶段等情况下基因的表达水平,选出合适的内参基因使数据得到标准化具有重要意义。近年来,内参基因的选择已得到研究者的高度重视。常见的内参基因有GAPDH基因、β-actin基因等管家基因[12-15]。目前,国内内参基因相关研究多见于高等动植物[16],水产动物相关研究较少,主要有杨桂梅等[17]利用抑制差减技术检测出gapdh和β-肌动蛋白基因在牙鲆(Paralichthysolivaceus)发育过程中表达水平上可能存在差异;此外,杨爱馥等[18]对仿刺参cytb和β-actin基因表达稳定性进行了比较;封利颖等[19]利用实时荧光定量技术对12种候选内参基因在虾夷扇贝不同组织及不同发育阶段的表达稳定性进行了分析;国外学者分析研究了斑点叉尾鮰(Ictaluruspunctatus)[20]、大西洋鲑(Salmosalar)[21]等的内参基因稳定性。关于仿刺参夏眠特定条件下内参基因表达稳定性的研究尚未见报道。
本研究首次通过对仿刺参转录组数据分析,选择了9个候选内参基因,利用Delta Ct、GeNorm、NormFinder及BestKeeper 4种方法比较各候选内参基因在仿刺参正常时期、夏眠初期及夏眠中期肠组织中的表达稳定性,选择出不同生理条件下稳定表达的内参基因,旨在探究仿刺参基因表达定量分析中内参基因的选择问题,为海参功能基因表达中内参基因的选择提供了参考。
1 材料与方法
1.1 试验材料
1.1.1 试验动物试验所用仿刺参取自大连黑石礁海域,分为3个时间点进行取样,即仿刺参正常时期(2013-07-01)、夏眠初期(2013-09-11)和夏眠中期(2013-10-09)。每个时期取3头,取其肠道组织,收集到1.5mL离心管内,投入液氮中迅速冷冻,存于-80℃冰箱保存备用。
1.1.2 主要试剂与仪器DEPC水;Trizol;氯仿;3mol/L NaAc(pH=5.2);75%乙醇;异丙醇;Nuclease-free水;1×TAE;琼脂糖;10mmol/L dNTP;Oligo(dT)18 Primer;Recombinant RNase Inhibitor;Reverse Transcriptase M-MLV(RNase H-);SYBR Green Master(Rox);移液枪(Eppendorf);高速匀浆机(IKEA);PCR仪(Bio-RAD);纯水系统(Milliore);高速冷冻离心机(Eppendorf); Nanoview(GE);琼脂糖电泳仪;凝胶成像系统(SIM);制冰机;涡旋振荡仪;超净无菌工作台;Qubit核酸浓度测定仪;高压灭菌锅;LightCycler实时荧光定量PCR仪480Ⅱ。
1.2 试验方法
1.2.1 总RNA提取仿刺参不同夏眠时期的9个个体肠组织的RNA均用TRIzol Reagent(Invitrogen,CA,USA)提取,且所有操作均在低温且无RNA酶的条件下进行。通过1.5%琼脂糖凝胶电泳确定RNA的完整性,使用Nanoview(GE)检测RNA的纯度和浓度,选用A260/A280值为1.8~2.2的RNA进行后续实验。结束检测后将RNA放置-80℃冰箱冻存。
(2)缺乏针对工业控制网络安全防御的相关技术手段。由于工业控制网络对于运行的连续性与响应时间等方面的要求较高,有别于信息网络的要求,研究针对工业控制网络的安全防御理论及技术非常必要。
1.2.2 cDNA合成取2μgRNA,向其中加入1μL Oligo dT,并加入DEPC水补至体系为12.5μL,整个过程要求在冰上进行,然后将其置于预热至65℃的PCR仪中孵育5min,迅速取出置于冰上。向上述反应物中加入4μL的5×Reaction Buffer、0.5μL的20URNasein、2μL的10mmol/L dNTP及1μL的200U M-MVL Reverse Transcriptase,混匀后置于42℃ 90min,70℃ 10min孵育以灭活反转录酶。
1.2.3 实时荧光定量PCRQPCR反应是20μL体系、2μL的稀释20倍的cDNA、4μL的2μmol/L引物S、4μL的2μmol/L引物A及10μL的SYBR Green Mastermix。将上述反应液混匀离心后,置于LightCycler实时荧光定量PCR仪480Ⅱ系统中,每个基因设置3个生物重复和3个技术重复。按照以下条件进行PCR扩增:50℃ 2min;95℃ 10min;95℃ 15s。各引物最佳退火温度下反应1min,40次循环;95℃ 15s,60℃ 1min,0.5℃/10s降至60℃,60℃1min。PCR仪检测荧光信号强度的时间设定在扩增反应的退火过程中。为了排除引物二聚体及非特异性扩增产物对基因表达定量的影响,在PCR反应结束后进行了熔解曲线分析,温度从95℃以0.5℃/10s速率缓慢降至60℃,连续采集样品的荧光信号以获取熔解曲线。
1.2.4 候选内参基因选择及引物设计所有候选内参基因序列由本实验室通过仿刺参转录组Illumina测序技术(Hiseq2000)测定而得。测序获得仿刺参转录组数据,将序列与Swissprot蛋白数据库比对,得到基因注释,同时比较分析各基因的表达水平,最后共选出10个候选内参基因用来分析基因表达稳定性,包括CA043(Uncharacterized protein C1orf43 homolog)、dpolm(dna-directed dna/rna polymerase mu)、ro60(60 kda ss-a ro ribonucleoprotein)、grb2(growth factor receptor-bound protein 2)、hiat1(hippocampus abundant transcript 1 protein)、nlrc4(nlr family card domain-containing protein 4)、farp1(FERM, RhoGEF and pleckstrin domain-containing protein 1)、cyc(cytochrome c)、gapdh(glyceraldehyde-3-phosphate dehydrogenase)。利用Primer5.0软件设计引物,见表1。引物由上海生工生物技术有限公司合成。
1.2.5 普通PCR检测扩增产物为了检测cDNA的质量、扩增产物是否准确及其是否具有特异性,在进行实时荧光定量PCR之前,先通过普通PCR验证。普通PCR反应的10μL体系包括1μL的cDNA、1μL的Primer-F、1μL的Primer-R、0.8μL的10mmol/L dNTP、1μL的10×PCR buffer、0.1μL的rTaq酶及5.1μL的ddpO。将上述体系反应物置于程序:95℃预变性2min后,按照95℃,30s;60℃,30s;72℃,15s的反应程序进行30个循环,终延伸72℃,5min后置于4℃保存。通过1%琼脂糖凝胶电泳检测扩增产物。
1.2.6 数据处理和分析数据处理与分析采用Excel 2003和4种常用的内参稳定性评价软件,包括Delta Ct、geNorm (http://medgen.ugent.be/~jvdesomp/genorm/)[22],NormFinder[23]和BestKeeper[24]软件,根据各软件说明对候选内参基因在仿刺参正常时期、夏眠初期及夏眠中期肠组织中的表达稳定性进行分析。具体操作如下:设定不同样本中某一内参基因Ct值最小者表达量为1,在Excel 2003中标准化处理其他样品

表1 9个候选内参基因引物序列及相关信息
Note: ①Unigene; ②Annotation; ③Primer sequence(5’-3’); ④Length of amplified product
此内参基因的表达量,即此内参基因想其他样本中的相对表达量为2-ΔCt(ΔCt=各样本Ct值-最小Ct值),将数据结果按格式要求导入GeNorm软件,该软件能够计算各候选内参基因的平均表达稳定值M以及分析内参基因标准化因子的配对差异,从而筛选出表达较稳定的内参基因以及所需内参基因的最适数目。NormFinder软件与geNorm软件类似,而BestKeeper软件则输入原始数据即可。
2 结果与分析
2.1 仿刺参正常时期、夏眠初期及夏眠中期肠组织总RNA提取
图1 总RNA
2.2 候选内参基因引物特异性分析
普通PCR扩增后,1%琼脂糖凝胶电泳检测扩增产物。结果如图2所示,在100~200bp之间,基因CA043、dpolm、ro60、grb2、hiat1、nlrc4、farp1、cyc和gapdh扩增片段条带清晰,片段长度与预期片段大小一致,且条带单一、不含杂带与引物二聚体;同时对各候选内参基因引物进行qRT-PCR分析,结果发现各候选内参基因的熔解曲线均为显著的单一峰(见图3)。综合分析,说明9个候选内参基因引物均具有较高特异性,可用于后续的qRT-PCR分析;同时发现每个样品扩增曲线具有较好的重复性,这说明qRT-PCR的分析结果准确可信。

(1:CA043;2:dpolm;3:ro60;4:grb2;5:hiat1;6:nlrc4;7:farp1;8:cyc;9:gapdh;M为DNA标记M is DNA Marker.)
图29 个候选内参基因PCR 扩增产物
Fig.2PCR products of 9 candidate reference genes
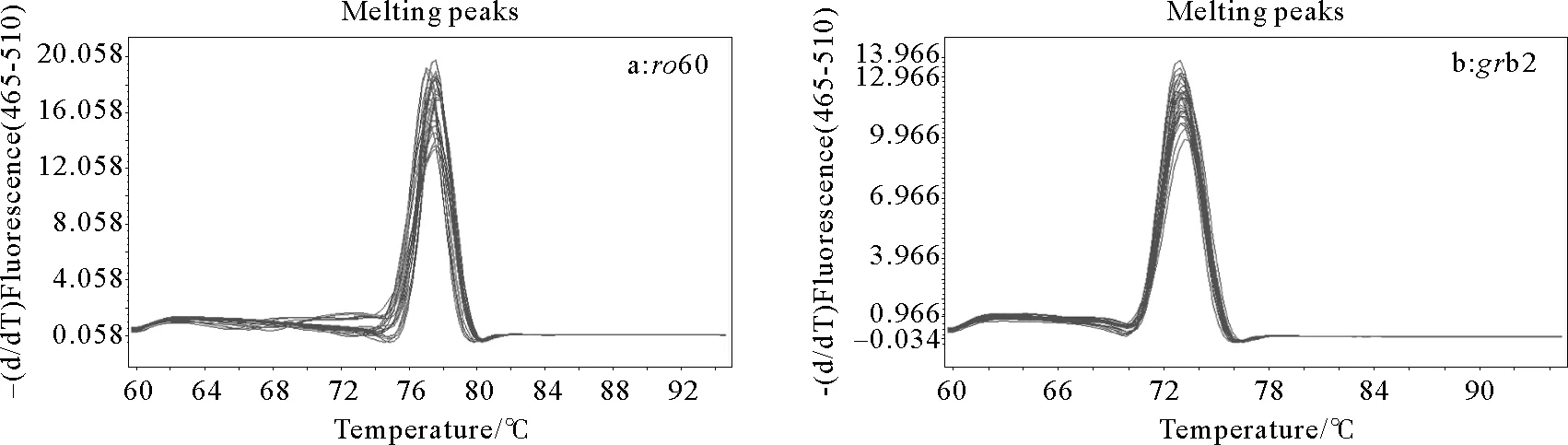
图3 9对候选基因引物的熔解曲线
2.3 内参基因稳定性评价分析
2.3.1 geNorm软件评价分析结果仿刺参正常时期、夏眠初期及夏眠中期肠组织中9个候选内参基因的表达稳定性见图4。geNorm软件是依据表达稳定度平均值(M)的大小对内参基因进行评价,M值越小,说明内参基因表达越稳定;反之,则表明内参基因表达稳定性差。经过geNorm软件的评价分析发现,在仿刺参正常时期、夏眠初期及夏眠中期肠组织中9个候选内参基因的表达稳定性的平均值M从大到小依次为CA043>gapdh>farp1>cyc>nlrc4>hiat1>dpolm>ro60=grb2(见图4a),由此可见,稳定性最好的基因为ro60和grb2,其平均表达稳定性M值均为0;而基因CA043与gapdh稳定性最差,其平均表达稳定性M值分别为1.59和1.42。除基因CA043外,其他8个候选内参基因M值均小于1.5,在此条件下可作为内参基因。此外,geNorm软件通过分析标准化因子配对差异值,来确定所需内参基因的最适数目,当标准化因子配对差异(pairwise variations)值低于0.15,即Vn(n+1)<0.15时,说明有n个基因可以作为实时荧光定量分析的内参基因,不需要选择使用数量大于等于n+1个基因作为内参基因。如图4b所示,本研究中标准化因子配对差异值均大于0.15,但是以V3/4最小,即在此之后增加更多的内参基因也不能降低标准化因子配对差异值(Vn/n+1),所以理想的内参基因数目应选为3个,建议选用基因grb2、ro60和dpolm作为内参基因。

(其中a为选内参基因表达稳定度的平均值,b为内参基因标准化因子的配对差异分析。a is Average expression stability M, b is Pairwise Variations.)
图4GeNorm软件分析候选基因在仿刺参不同夏眠时期肠组织的表达稳定性
Fig.4Average expression stability values of the candidate reference genes in intestine of
japonicus during different aestivation analyzed by GeNorm
2.3.2 Delta Ct方法评价分析结果根据实时荧光定量PCR结果,利用Delta Ct方法对仿刺参正常时期、夏眠初期及夏眠中期肠组织中9个候选内参基因的表达稳定性进行分析,结果见图5。由图5可知9个候选内参基因平均稳定性值从大到小排序为CA043>gapdh>cyc>farp1>nlrc4>hiat1>dpolm>ro60=grb2,基因grb2和ro60稳定性较高,平均表达稳定性值均为0.205,之后依次为基因dpolm、hiat1、nlrc4、farp1、cyc、gapdh,基因CA043表达稳定性最差,其平均表达稳定性值为0.44。
2.3.3 NormFinder 软件评价分析结果NormFinder软件分析内参基因的原理与GeNorm软件相似,都是根据基因平均表达稳定值的大小评估,稳定值越小,基因表达的稳定性越高。但是该软件不能预测内参基因组合的数目。经NormFinder软件评价分析发现,NormFinder分析结果与GeNorm分析结果基本一致(见图6)。基因dpolm平均表达稳定值最小,为0.04,可作为候选内参基因;其次为基因grb2与ro60,平均稳定值均为0.072;同样基因CA043平均稳定值最高,为0.408。
2.3.4 BestKeeper软件评价分析结果BestKeeper软件与GeNorm、NormFinder软件原理不同,该软件是根据标准差(SD)与调节系数标准差SD[±X-fold]对基因表达稳定性进行评判,其值越小则表示基因表达越稳定。经过BestKeeper软件评估分析,仿刺参正常时期、夏眠初期及夏眠中期肠组织中9个候选内参基因的表达稳定性见图7与表2。由表2和图7可知,与geNorm和NormFinder软件分析结果不同,BestKeeper软件分析结果以基因CA043表达最为稳定,SD为0.76;同时基因grb2、ro60及dpolm表达稳定性仍排列在前四名,其SD值分别为1.01、1.17、1.20;而表达最不稳定的内参基因为farp1,SD值为1.40。
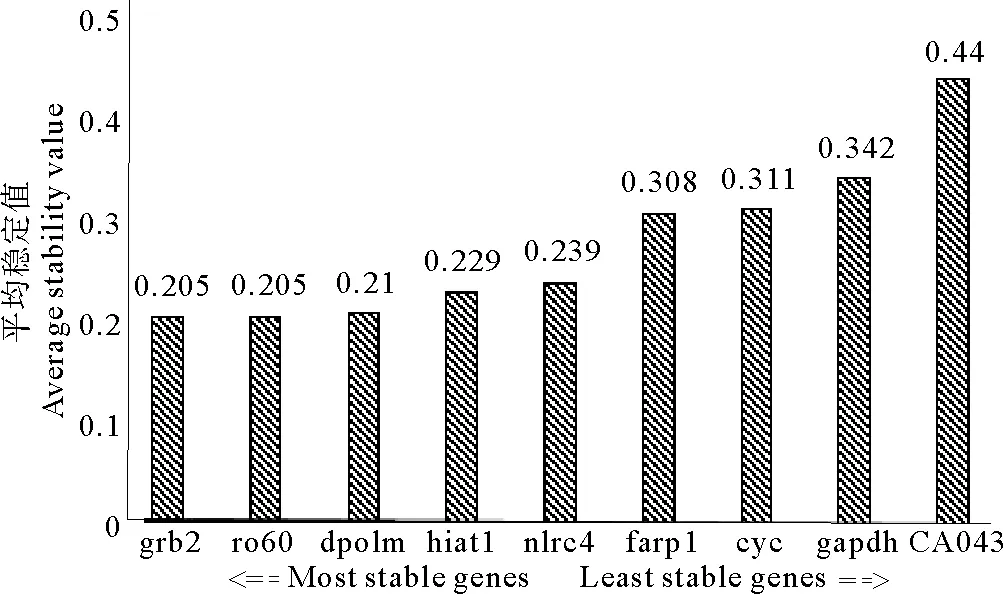
图5 Delta Ct法分析候选基因在仿刺参不同夏眠时期肠组织的表达稳定性
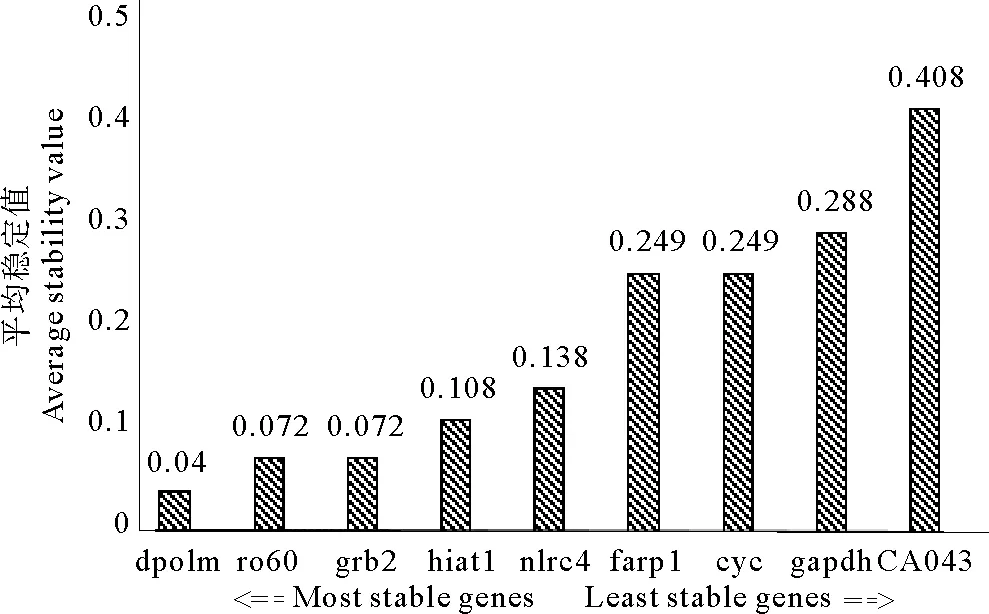
图6 利用NormFinder评价内参候选基因仿刺参不同夏眠时期肠组织的表达稳定性
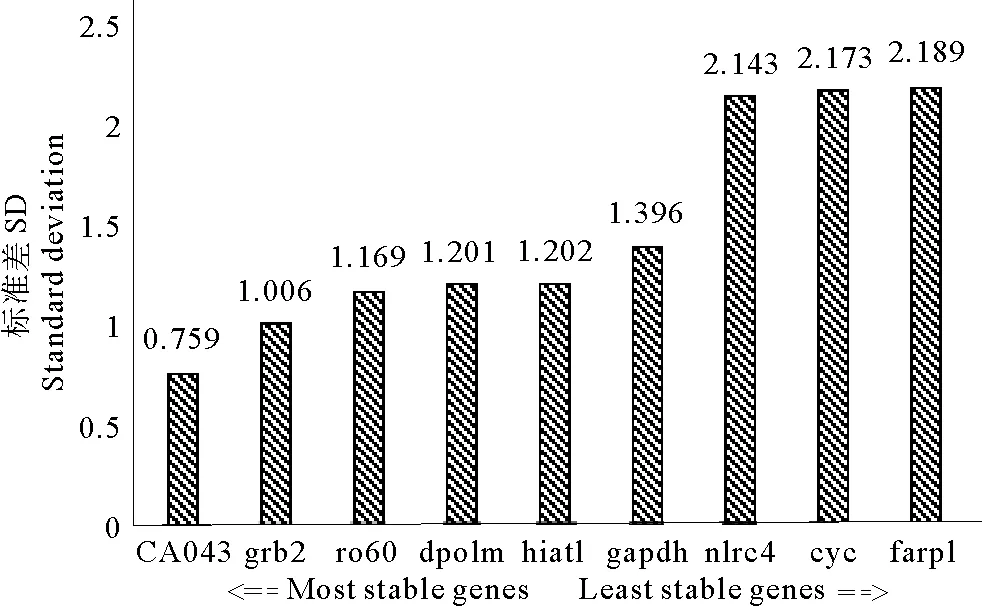
图7 利用BestKeeper评价内参候选基因在仿刺参不同夏眠时期肠组织的表达稳定性

参数Parameters基因GenesCA043dpolmro60grb2hiat1nlrc4farp1cycgapdhGeoMeans[CP]22.9827.4128.2126.7426.2727.2327.3124.8226.99ARMean[CP]23.0027.4428.2426.7626.3127.3327.4324.9527.03Min[CP]22.0425.5726.5525.0324.4624.8024.7822.0824.17Max[CP]24.9729.3031.4928.8528.5031.7633.5030.2929.72SD[±CP]0.761.201.171.011.202.142.192.171.40CV[%CP]3.304.384.143.764.577.847.988.715.16Min[X-fold]-1.92-3.59-3.16-3.26-3.51-5.37-5.78-6.70-7.05Max[X-fold]3.973.729.724.344.6923.0773.0944.336.67SD[±X-fold]1.672.302.252.012.304.424.564.512.63
本研究利用Delta Ct、geNorm、Normfinder和BestKeeper 4种软件综合分析筛选出仿刺参正常时期、夏眠初期及夏眠中期肠组织中表达相对稳定的内参基因。表3显示,基因grb2表达最稳定,其次为ro60基因及dpolm基因,而基因CA043表达稳定性较差。因此,可选基因grb2、ro60及dpolm作为内参基因。
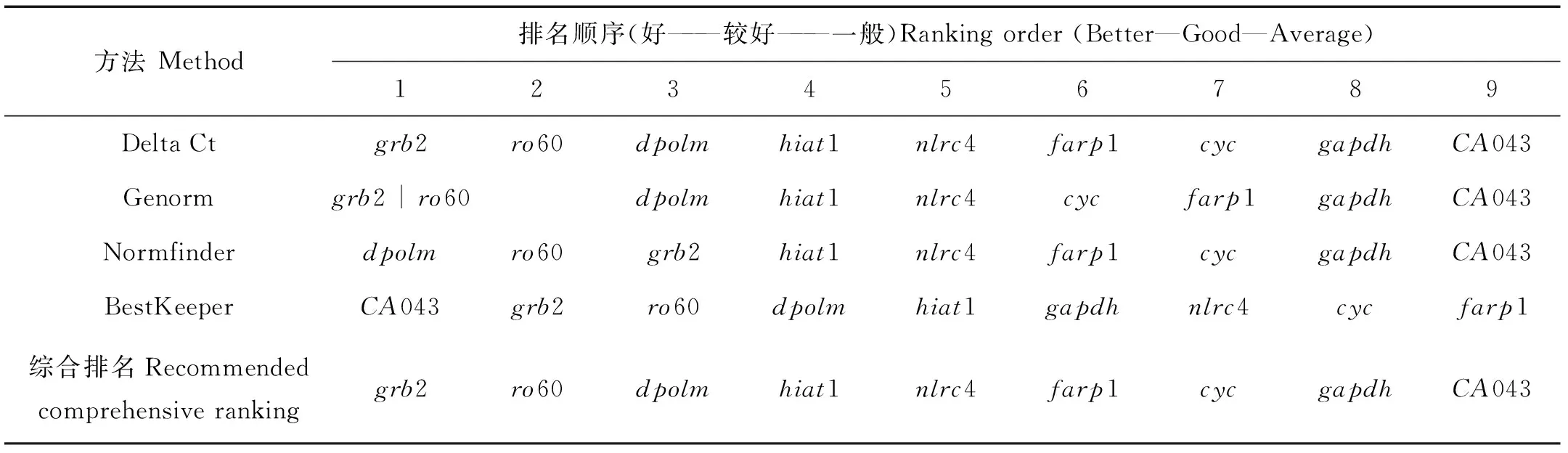
表3 9个候选内参基因稳定度排序
3 讨论
夏眠是仿刺参特有的重要生理习性,由学者Mitsukuri于日本首次发现[25]。夏眠期间仿刺参会产生一系列的生理生态变化,主要表现为活动降低、停止摄食、体质量减轻、消化道退化萎缩、代谢降低等。这一生理习性在一定程度上影响了仿刺参的质量及产量。因此,研究夏眠过程中仿刺参的代谢生理调控机制,以期望打破夏眠这一生理习性,对能够更好的实现仿刺参的经济价值具有重要意义。另外,由于夏眠是仿刺参的特殊生理习性,能够准确的取到各夏眠时期仿刺参样品具有一定的难度。目前,关于夏眠期间仿刺参内参基因选择的研究尚未见报道,本文首次比较了9个候选基因在仿刺参正常时期、夏眠前期及夏眠中期肠组织中的表达稳定性,以期望得到表达稳定的内参基因,为以后关于仿刺参夏眠过程中代谢调控机制的研究提供参考。
对于实时荧光定量PCR研究而言,选择合适的内参基因是其能够准确分析基因表达稳定性的前提。通常理想的内参基因是指在不同类型细胞或组织器官、不同发育阶段及各种处理条件下均稳定表达[26]。但实际上几乎不存在能同时满足这些条件的基因,目前所研究的基因在不同类型的细胞、组织器官及实验条件下表达均存在差异[27-29]。因此,研究者可以特定验条件对内参基因的稳定性进行分析。本研究即对仿刺参夏眠这一特殊生理条件下的肠道组织进行内参基因的稳定性的分析。
之前大量学者多以文献中报道过的常用管家基因作为候选内参基因来进行基因表达稳定性分析。而本研究首次通过由本实验室对仿刺参转录组进行Illumina测序,获得仿刺参转录组数据,将序列与Swissprot蛋白数据库比对,得到基因注释,同时比较分析各基因的表达水平,从而获得CA043、dpolm、ro60、grb2、hiat1、nlrc4、farp1、cyc和gapdh共9个候选内参基因用来分析基因表达稳定性。其中基因grb2,即生长因子受体结合蛋白2,具有促进细胞增殖、细胞生长及细胞分化等功能,在多个信号通路中担任着“接力棒”的角色,目前已成为研究最为透彻的接头蛋白。董迎辉等[30]扩出泥蚶grb2基因的全长cDNA序列,并研究分析了其组织表达、生物信息学及发育阶段表达特征;周维霞等[31]研究发现基因grb2的表达与胰腺癌的生物学行为密切相关。此外,grb2基因在哺乳动物中已被证实是调控生长以及胚胎发育相关信号通路的重要基因,基于夏眠期间仿刺参的生理生态变化的表现,这一结论与本研究得到的grb2基因表达最稳定的结果一致。Eli Eisenberg等[32]认为CA043基因可用作内参基因;ro60参与了RNA聚合酶III启动子的转录的生物过程;基因polm在人、牛、小鼠等中是保守的;Neil Romberg等[33]研究表明nlrc4的突变可导致小肠结肠炎;BinQuan Zhuang等[34]研究发现基因farp1能促进脊柱运动神经元树突的生长;基因cyc即为细胞色素C,具有增强细胞氧化与诱导细胞凋亡作用;基因gapdh为甘油醛-3-磷酸脱氢酶,该酶是糖酵解反应中的一个酶。封利颖等[19]利用实时荧光定量技术对12种候选内参基因在虾夷扇贝不同组织及不同发育阶段的表达稳定性进行了分析,并得到基因cyc和gapdh可作为内参基因,本研究结果与其不同。为了能够更充分了解各基因的相关信息,上述基因有待于进一步深入研究。
目前常用于评价分析内参基因稳定性的生物学软件有GeNorm、NormFinder及BestKeeper 3种,如果单独选择其中一种生物学软件分析候选内参基因的表达稳定性以筛选最佳内参基因,得到的结果并不理想[35]。因此本研究利用基于Excel 2003平台的Delta Ct、GeNorm、NormFinder及BestKeeper 4种软件对仿刺参正常时期、夏眠初期及夏眠中期条件下肠道组织中的9个候选内参基因的表达稳定性进行了分析。结果表明,经过Delta Ct、GeNorm及NormFinder软件分析的结果基本一致,基因grb2、ro60及dpolm表达稳定性排序均为前3,GeNorm与NormFinder软件的区别是前者能够确定合适内参基因组合的数目,而后者只能得到表达最稳定的基因。与上述3种方法分析结果不同,BestKeeper软件分析结果为基因CA043最为稳定,其他基因稳定性排序也与另外3种分析方法差异,这可能是由于BestKeeper软件的原理是直接将原始数据Ct值输入到程序中,这样计算出的数据会产生虚假的变化[36-37]。综合分析,合适的内参基因组合数目为3个,分别为基因grb2、ro60及dpolm;并筛选出了在仿刺参夏眠期条件下的表达最稳定的内参基因为grb2。
4 结语
本研究首次尝试研究仿刺参在夏眠这一特殊生理条件下评价分析候选内参基因表达稳定性。为使实验结果更具说服力,利用Delta Ct、GeNorm、NormFinder和BestKeeper 4种生物学软件,综合系统地比较仿刺参正常时期、夏眠初期及夏眠中期肠道组织的Real-time PCR数据,筛选出了适合研究仿刺参夏眠期条件下的基因表达的内参基因组合数目是3个,其中表达最稳定的内参基因为grb2。研究结果为仿刺参夏眠相关基因表达分析中的内参基因的选择提供了实验依据,同时也对仿刺参不同组织及不同实验条件处理下内参基因的筛选提供了参考资料。
参考文献:
[1]Bustin S A, Benes V, Nolan T, et al. Quantitative real-time RT-PCR-a perspective[J]. J Mol Endocrinol, 2005, 34(3): 597-601.
[2]Kubista M, Andrade J M, Bengtsson M, et al. The real-time poly-merase chain reaction[J]. Mol Aspects Med, 2006, 27(2-3): 95-125.
[3]VanGuilder H D, Vrana K E, Freenman W M. Twenty-five years quantitative PCR for gene expression analysis[J]. Biotechniques, 2008, 44(5): 619-626.
[4]岳志芹, 汪俊, 梁成珠, 等. 实时定量RT-PCR方法检测太平洋牡蛎中的诺沃克样病毒[J]. 海洋科学, 2008, 32(8): 1-4.
Zhiqin Yue, Jun Wang, Chengzhu Liang, et al. Detection of norwalk-like virus in oyster by real-time quantitative RT-PCR [J]. Marine Sciences, 2008, 32(8): 1-4.
[5]吴绍强, 李海艳, 林祥梅, 等. 贝类派琴虫实时荧光定量PCR检测方法的建立和应用[J]. 渔业科学进展, 2009, 30(5): 58-63.
Shaoqiang Wu, Haiyan Li, Xiangmei Lin, et al. Establishment and application of real-time PCR method for detection ofPerkinsussp. in bivalves[J]. Progress in Fishery Sciences, 2009, 30(5): 58-63.
[6]Wang H, Song L S, Li C H, et al. Cloning and characterization of a novel C-type lectin from Zhikong scallopChlamysfarreri[J]. Molecular Immunology, 2007, 44(5): 722-731.
[7]钱曦, 华雪铭, 黄旭雄, 等. 异育银鲫c型溶菌酶全长cDNA序列的生物信息学分析及其组织表达分析[J]. 中国生物化学与分子生物学报, 2008, 24(4): 337-347.
Xi Qian, Xueming Hua, Xuqiong Huang, et al. Molecular cloning of full-length c-type lysozyme fromCarassiusauratusgibelioand its tissue-specific expression and bioinformatic analysis[J]. Chinese Journal of Biochemistry and Molecular Biology, 2008, 24(4): 337-347.
[8]Bustin S A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays[J]. J Mol Endocrinol, 2000, 25(2): 169-193.
[9]Thellin O, Zoriz W, Lakaye B, et al. Housekeeping genes as internal standards: Use and limits[J]. J Bitech, 1999, 75(2-3): 291-295.
[10]Radoni A, Thulke S, Mackay I M, et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochemical and Biophysical Research Communications, 2004, 313: 856-862.
[11]Bas J W Dekkers, Leo Willems, George W Bassel, et al. Identification of reference genes for RT-qPCR expression analysis in arabidopsis and tomato seeds[J]. Plant Cell Physiol, 2012, 53(1): 28-37.
[12]Pugnale P, Latorre P, Rossi C, et al. Real-time multi-plex PCR assay to quantify hepatitis C virus RNA in peripheral blood mononuclear cells[J]. J Virol Methods, 2006, 133(2): 195-204.
[13]Regis S, Grossi S, Lualdls, et al. Diagnosis of pelizae-us merzbacher disease: detection of proteoLipid protein gene copy number by real-time PCR[J]. Neurogenetics, 2005, 6(2): 73-78.
[14]Nguyen van N, Taglineger K, Helps C R, et al. Measurement of cytokine mRNA expression in intestinal biopsies of cats with inflammatory enteropathy using quantitative real-time RT-PCR[J]. Vet Immunol Immunopathol, 2006, 113(3/4): 404-414.
[15]Tsui N B, Loym. Molecular analysis of circulating RNA in plasma[J]. Methods Mol BIOL, 2006, 336: 123-134.
[16]袁伟, 万建红, 杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J]. 植物学报, 2012, 47(4): 427-436.
Wei Yuan, Jianhong Wan, Yuejian Yang. Characterization and selection of reference genes for real-time quantitative RT-PCR of plant[J]. Chinese Bulletin of Botany, 2012, 47(4): 427-436.
[17]杨桂梅, 鲍宝龙, 任大明. 3-磷酸甘油醛脱氢酶、β-肌动蛋白和18S rRNA作为相对定量的内标在牙鲆发育阶段的稳定性比较[J]. 上海水产大学学报, 2005, 14(1): 84-88.
Guimei Yang, Baolong Bao, Daming Ren. Comparison between ribosomal 18S rRNA, GAPDH and β-actin genes as internal standard for quantitative comparison of mRNA levels in development of flounder,Paralichthysolivaceus[J]. Journal of Shanghai Fisheries University, 2005, 14(1): 84-88.
[18]杨爱馥, 周遵春, 董颖, 等. 仿刺参cytb和β-actin基因表达稳定性比较[J]. 中囯农业科技导报, 2010, 12(1): 78-84.
Aifu Yang, Zunchun Zhou, Ying Dong, et al. Stability comparison of cytb and β-actin gene expression in Sea cucumber(Apostichopusjaponicus)[J]. Journal of Agricultural Science and Techology, 2010, 12(1): 78-84.
[19]Liying Feng, Qian Yu, Xue Li, et al. Identification of reference genes for qRT-PCR analysis in Yesso scallopPatinopectenyessoensis[J]. PLOS ONE, 2013, 8(9): 1-10.
[20]Small B C, Murdock C A, Biodeau-Bourgeois A L, et al. Stability of reference genes for real-timr PCR analyses in channel catfish(Ictaluruspunctatus)tissues under varying phyisiological conditions[J]. Compara-tive Biochemistry and Physiology Part B, 2008, 151(3): 296-304.
[21]Ingerslev H C, Pettersen E F, Jakobsen R A, et al. Expreesion profling and validation of reference gene candidates in immune revevant tissues and cells from Alantic salmon (Salmosalar)[J]. Molecular Immunology, 2006, 43(8): 1194-1201.
[22]Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J]. Genome Biology, 2002, 3(7): 1-12.
[23]Andersen C L, Jensen J, Orntoft T F. Normalization ofReal-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identifygenes suited for normalization, applied to bladderandcoloncancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250.
[24]Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excelbased tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515.
[25]Mitsukuri K. Notes on the habits and life history ofStichopusjaponicusSelenka[J]. Annot Zool Japan, 1903, 5: 1-21.
[26]Hu R B, Fan C M, Fu Y F. Reference gene selection in plant real-time quantitative reverse transcription PCR (qRT-PCR)[J]. J Agric Sci Technol, 2009, 11(6): 30-36.
[27]Lee P D, Sladek R, Greenwood C M T, et al. Control genes and variability: Absence of ubiquitous reference transcripts in diverse mammalian expression studies[J]. Genome Res, 2002, 12: 292-297.
[28]Tang R Y, Dodd A, Lai D, et al. Validation of zebrafish (Daniorerio) reference genes for quantitative Real-time RT-PCR normalization[J]. Acta Biochimica Et Biophysica Sinica, 2007, 39(5): 384-390.
[29]Long X Y, Wang J R, Ouellet T, et al. Genome-wide identification and evaluation of novel internal control genes for Q-PCR based transcript normalization in wheat[J]. Plant Molecular Biology, 2010, 74(3): 307-311.
[30]董迎辉, 项翔, 姚韩韩, 等. 泥蚶(Tegillarcagranosa)生长因子受体结合蛋白2(GRB2)基因的克隆与表达分析[J]. 海洋与湖沼, 2013, 44(4): 937-943.
Yinghui Dong, Xiang Xiang, Hanhan Yao, et al. Cloning and expression of growth factor receptor-bound 2 inTegillarcagranosa[J]. Chinese Journal of Oceanology and Limnology, 2013, 44(4): 937-943.
[31]周维霞, 蒋晓红, 杨勇, 等. 人类真核延伸因子1A2与生长因子受体结合蛋白2在胰腺癌组织中的表达及临床病理意义[J]. 世界华人消化杂志, 2014, 22(14): 2049-2054.
Weixia Zhou, Xiohong Jiang, Yong Yang, et al. Expression and clinical pathological significance ofeukaryotic elongation factor 1A2 and growth factor receptor-bound 2 in pancreatic carcinoma[J]. World Chinese Journal of Digestology, 2014, 22(14): 2049-2054.
[32]E li Eisenberg, Erez Y. Human housekeeping genes, revisited[J]. Trends in Genetics, 2013, 29(10): 569-574.
[33]Neil Romberg, Khatoun A Moussawi, Carol Nelson-Williams, et al. Mutation of NLRC4 causes a syndrome of enterocolitis and autoinflammation[J]. Nature Genetics, 2014, 46: 1135-1139.
[34]BinQuan Zhuang, YouRong Sophie Su, Shanthini Sockanathan. FARP1 promotes the dendritic growth of spinal motor neuron subtypes through transmembrane semaphorin6A and plexinA4 signaling[J]. Neuron, 2008, 61(6): 359-372.
[35]Mascia T, Santovito E, Gallitelli D, et al. Evaluation of reference genes for quantitative reverse-transcription polymerase chain reaction normalization in infected tomato plants[J]. Molecular Plant Pathology, 2010, 11(6): 805-816.
[36]Khanlou K M, Van Bockstaele E. A critique of widely used normalization software tools and an alternative method to identify reliable reference genes in red clover (TrifoliumpratenseL. ) [J]. Planta, 2012, 236(5): 1381-1393.
[37]Livak K J, Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and he 2-ΔΔCt method[J]. Methods, 2001, 25(4): 402-408.
责任编辑朱宝象
基金项目:❋国家高技术研究发展计划项目(2012AA10A412);辽宁省农业攻关及成果产业化项目(2015203003)资助
收稿日期:2015-03-05;
修订日期:2016-05-05
作者简介:张莉恒(1990-),女,硕士生,主要从事水产养殖研究。E-mail:shiqudeyoushang@126.com ❋❋通讯作者:E-mail: dingjun1119@dlou.edu.cn
中图法分类号:S917
文献标志码:A
文章编号:1672-5174(2016)07-035-09
DOI:10.16441/j.cnki.hdxb.20150051
Selection of Reference Genes for Quantitative Real-Time PCR ofApostichopusaponicusIntestine During Different Periods of Aestivation
ZHANG Li-Heng1, WANG Shi2, CHANG Ya-Qing1, BAO Zhen-Min2, DING Jun1
(1.The Key Laboratory of Mariculture & Stock Enhancement in North China′s Sea, Ministry of Agriculture, Dalian Ocean University, Dalian 116023, China; 2.The Key Laboratory of Marine Genetics and Breeding (MGB), Ministry of Education, College of Marine Life Sciences, Ocean University of China, Qingdao 266003, China)
Abstract:Apostichopus japonicus is the most important economic species in China. Its special physiological habit is aestivation. There have been many studies on breeding, development, immunology and others, molecular biological research has become important. Domestic reference gene-related research is more in higher plants and animals than in aquatic animal. The study on the stability of reference gene in aestivation has not been reported. From transcriptome data analysis, we selected nine candidate refe-rence genes. In normal times, and at beginning of aestivation and in the mid of aestivation, With quantitative real-time PCR, we assessed the expression stability of nine candidate reference genes (CA043, dpolm, ro60, grb2, hiat1, nlrc4, farp1, cyc and gapdh) in intestinal tissues of A. japonicus. The stability of nine candidate reference genes analyzed by the 4 procedures (Delta Ct, GeNorm, NormFinder and BestKeeper). In conclusion, through the different software, the stability of candidate reference genes were not entirely consistent. The most stable gene was grb2, which can be used as the reference. The stability of ro60 and dpolm was the second; and the least stable was CA043. This study was the first to evaluate and analyze the expression stability of candidate reference genes in aestivation, laid a foundation for the further quantitative analysis of gene expression in A. japonicus.
Key words:Apostichopus japonicus; aestivation; reference gene; quantitative real-time PCR
Supported by the National High Technology Researchand PevelopmEnt Program (2012AA10A412); Industrialization Project of Agricultural Research and Achievements in Liaoning Province (2015203003)
