基于转录组的疏花软紫草低拷贝核基因引物开发
2016-06-04义何承忠巨苗苗田西南林业大学云南省云南省高校林木遗传改良与繁育重点实验室云南昆明650224国家林业局西南地区生物多样性保育重点实验室云南昆明650224中国科学院昆明植物研究所中国科学院东亚植物多样性与生物地理学重点实验室云南昆明650201
付 义何承忠巨苗苗田 斌,3(1.西南林业大学云南省云南省高校林木遗传改良与繁育重点实验室,云南昆明650224;2.国家林业局西南地区生物多样性保育重点实验室,云南昆明650224;3.中国科学院昆明植物研究所中国科学院东亚植物多样性与生物地理学重点实验室,云南昆明650201)
基于转录组的疏花软紫草低拷贝核基因引物开发
付 义1,2何承忠1,2巨苗苗1,2田 斌1,2,3
(1.西南林业大学云南省云南省高校林木遗传改良与繁育重点实验室,云南昆明650224;
2.国家林业局西南地区生物多样性保育重点实验室,云南昆明650224;
3.中国科学院昆明植物研究所中国科学院东亚植物多样性与生物地理学重点实验室,云南昆明650201)
摘要:基于高通量的转录组测序,为大量并快速的开发分子标记提供契机。本研究对紫草科植物疏花软紫草进行转录组测序,共得到92 042 086条reads,从疏花软紫草158 446个Unigene中搜索到332个低拷贝核基因。为进行多态性验证,从中随机挑选30个设计引物,并对3个疏花软紫草种群共9个个体进行PCR扩增,结果发现其中16对引物能够扩增出单一稳定的目的条带,从中随机挑选7个片段进行群体测序并计算DNA多态性。结果表明,7对引物的多态性位点平均值为6.57,单倍型平均值为5.14,单倍型多态性(HD)在0.389~0.944之间,核苷酸多态性(Pi)为0.001 48~0.017 23。通过转录组测序技术开发的低拷贝核基因有较高的多态性,且可以有效地运用于群体遗传学和谱系地理学研究中。另外,这些低拷贝核基因能够被运用于软紫草属植物系统发育重建以及物种形成等研究工作中。
关键词:疏花软紫草;转录组;低拷贝核基因;引物
长久以来,植物系统进化研究主要依赖于细胞质DNA,但是由于自然杂交现象在植物中的普遍存在,和细胞质DNA单亲遗传的限制,其包含的遗传信息并不能很好的反应物种的进化历史。然而,用基于双亲遗传的核基因能很好地解决物种之间和物种内不同种群间的亲缘关系。如李建华利用低拷贝核基因对桦木科铁木属和鹅耳枥属进行研究,结果表明铁木属为单系鹅耳枥属为并系[1]。利用低拷贝核基因在金丝桃科植物系统发育重建方面的研究,也取得了很好的效果[2]。刘勉等人利用低拷贝核基因重建了菊科紫菀亚科族间的系统发育关系,研究结果显示低拷贝核基因可以更好地解决菊科科以下分类阶元的系统发育关系[3]。随着高通量测序技术的快速发展,利用其进行转录组测序分析能更全面快速地了解真核生物复杂的转录组,且对于无参考基因组的的物种也能通过转录组测序分析,从而获得该物种的单一基因序列集(Unigene)数据[4]。基于这些数据,可以进行多种遗传多样性分析以及分子标记开发。目前,转录组测序分析在分子标记的开发中应用比较广泛。如黄海燕等人基于杜仲(Eucommia ulmoides)转录组序列进行了SSR分子标记的开发,结果发现,设计SSR引物时,扩增片段长度控制在100~250 bp,多态性会比较高,扩增效果也会较好[5]。张振等完成了红松(Pinus koraiensis)转录组SSR分析,印证了利用红松转录组数据开发SSR标记的可行性[6]。近年来的研究发现,基于转录组测序技术也能开发植物的低拷贝核基因引物[7]。然而对非模式植物的研究报道较少。
疏花软紫草(Arnebia szechenyi)是紫草科(Boraginaceae)软紫草属(Arnebia)的多年生草本植物。该植物茎高20~30 cm,有疏分枝,密生灰白色短柔毛。花黄色,喉部有黑色的蜜导,雄蕊着生花冠筒中部(长柱花)或喉部(短柱花),花药长约1.6 mm;子房4裂,花柱丝状,稍伸出喉部(长柱花)或仅达花冠筒中部,先端浅2裂。小坚果三角状卵形,长约2.7 mm,有疣状突起和短伏毛,花果期6—9月。在我国主要分布于内蒙古西部、宁夏、甘肃西北部及青海东部和南部的山坡向阳处[8]。由于该植物为二型花柱,花较大,并且花喉部有明显的蜜导[9],因此是繁殖生态学研究的合适材料;此外,其分布在西北干旱地区,对其种群遗传结构的研究能很好地解释我国北方地区荒漠化的形成时间和机理。然而目前能用于该物种的分子标记较少。因此,本研究拟基于高通量转录组测序技术对疏花软紫草的低拷贝核基因引物进行筛选,以期为疏花软紫草乃至软紫草属植物在植物系统进化以及繁殖机理等方面的研究提供参考。
1 材料与方法
1.1研究材料
用于提取总RNA的疏花软紫草采自青海省循化孟达乡(N 35°50.664′,E 102°35.729′)。在采摘分子材料的时候尽可能采摘新鲜没有病虫害的幼嫩叶片,然后立即置于液氮中,之后转置于-80℃低温保存。用于低拷贝核基因引物验证的疏花软紫草样本分别采自甘肃省张掖北山、甘肃省皋兰什川十字沟、宁夏中卫沙坡头等3个地方(表1),样本采集原则也是主要采集新鲜没有病虫害的幼嫩叶片,然后置于硅胶中干燥,用于提取总DNA进行低拷贝核基因验证。

表1 疏花软紫草样本的地理分布位置Table 1 Geographic location of sample population of Arnebia szechenyi
1.2研究方法
1.2.1样本DNA提取
在每个地点采集的疏花软紫草种群中各挑选3个个体,然后用上海生工生物工程股份有限公司的Ezup柱式植物基因组DNA抽提试剂盒提取3个种群共9个个体的总DNA,DNA的质量和浓度用1%琼脂糖凝胶电泳及Nanodrop 2000检测,质量和浓度均达到要求的DNA置于-80℃冰箱中备用。
1.2.2样本RNA的提取及转录组测序
样品采集后,用CTAB法提取疏花软紫草叶片的总RNA,并用1%琼脂糖凝胶电泳及Nanodrop 2000检测其浓度和质量。在此基础上,采用OligodT磁珠试剂盒分离纯化出mRNA。纯化后,加入缓冲液将mRNA打断,并以其为模板,以6碱基随机序列为引物合成单链cDNA。之后,加入dNTPs、缓冲液和DNA聚合酶合成双链cDNA,并利用AMPure XP beads将其纯化。最后,将已纯化的双链cDNA进行末端修复、加多聚腺嘌呤尾,连接测序接头,片段大小选择及PCR富集等过程后构建cDNA文库,并采用高通量测序平台Illumina HiSeqTM2500对其进行测序(武汉未来组生物科技有限公司)。
1.2.3数据过滤及拼接
获得转录组测序的原始数据后,利用自行编写的perl脚本过滤原始数据。整个过程包含4个步骤:去除含有测序接头的序列;去除N%(N为无法确定碱基信息的碱基)大于10%的序列;去除低质量序列以及去除冗余重复的序列。数据过滤后,采用Trinity[10]软件将所有过滤序列进行拼接,并用CDHIT[11]软件(http:/ / www.bioinformatics.org/ cd-hit/)对拼接后的序列进行聚类以及冗余序列的去除。经过拼接和聚类以后,得到的转录本称为Unigene。
1.2.4低拷贝核基因筛选
以被子植物数据库为参照,将疏花软紫草转录本序列用Hamstr[12]算法寻找同源基因,同源比对的e值设定为10-5。将比对结果进行筛选,结果与被子植物数据库存在同源关系,但与自身其他序列不存在同源关系的基因为筛选出的疏花软紫草低拷贝核基因。
1.2.5低拷贝核基因的验证
从筛选的疏花软紫草低拷贝核基因中随机挑选出30个基因进行引物设计及试验验证。用Primer3[13]分别对30个基因设计PCR引物,引物设计原则为:长度为18~22 bp,PCR产物大小为300~500 bp。为了验证这些引物的有效性和多态性,随后,用所设计的30对引物分别对表1中的3个种群共9个个体在25 μL PCR体系下扩增,扩增条件为:94℃预变性5min;94℃变性30s,56℃退火45s,72℃延伸1 min 30 s,30个循环;用1%琼脂糖凝胶电泳检测。排除非特异性扩增的情况,电泳结果中,同一泳道中只出现1条扩增条带,且扩增条带的大小位于300~500 bp之间的基因为低拷贝核基因。
2 结果与分析
2.1转录组测序及拼接
基于高通量测序技术对疏忽软紫草进行转录组测序,共得到92 042 086条序列,总长度为9 112 166 514 bp,经过去除包含测序接头的序列,去除N的比例大于10%的序列,去除低质量序列和去除冗余序列的4个数据过滤过程后,得到80 406 986条序列,为原始序列的87.36%,过滤后碱基总长度为7960291614 bp。采用Trinity软件对过滤数据进行拼接和用CD-HIT软件对拼接后的序列进行聚类分析以后,最终得到了疏花软紫草的转录组数据(表2)。从表2中可看出,通过拼接并聚类后,得到了158 446个contig,总长度为183 270 167 bp,contig长度分布在201~15 803 bp之间,平均长度为1 157 bp,N50(按照长度将拼接转录本从大到小排序,累加转录本的长度为总长50%的拼接转录本长度)为1 987 bp,N90为454 bp。
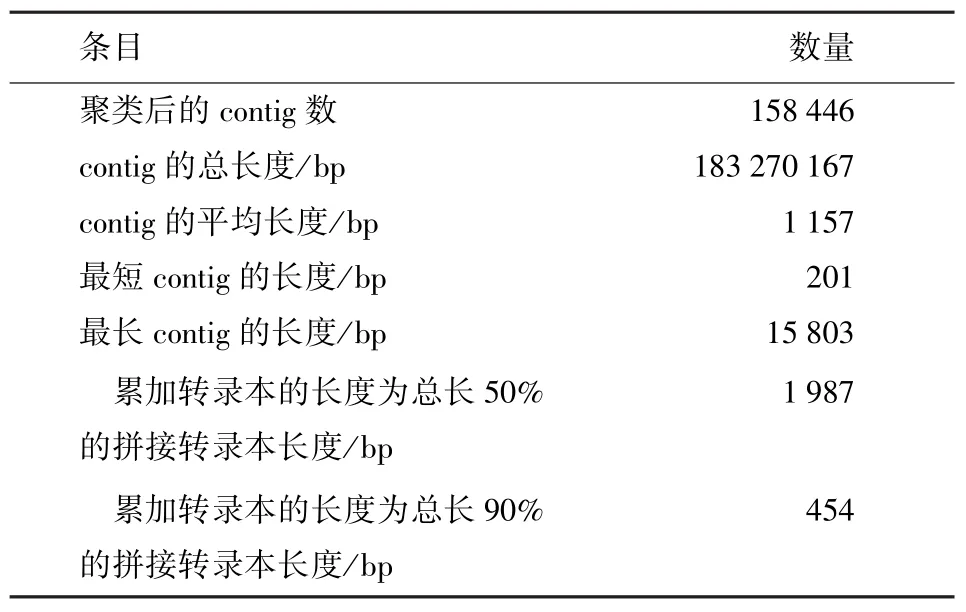
表2 转录组基本数据Table 2 Basic date of the transcriptome
2.2低拷贝核基因引物筛选和验证
以被子植物数据库为参照,将疏花软紫草转录本序列用Hamstr算法寻找同源基因,共找到了与自身其他序列不存在同源关系的基因332个,即332个低拷贝核基因。随机挑选30个基因设计引物。为了验证引物的有效性和多态性,随后,用所设计的30对引物分别对ZY、DT013、DT015共3个种群9个个体进行扩增,用1%琼脂糖凝胶电泳检测PCR扩增产物,结果显示,有16对引物能扩增出稳定且单一的目的条带,且PCR产物片段大小均与预测结果相吻合。随后,又对这16对引物所对应的基因进行注释,引物编号、序列以及注释结果见表3。然后从这16对引物中随机挑选7对引物计算DNA多态性,多态性数据见表4。由表4可知,7对引物的多态性位点平均值为6.57,单倍型平均值为5.14,单倍型多态性(HD)在0.389~0.944之间,核苷酸多态性(Pi)在0.00 148~0.01 723之间。

表3 低拷贝引物序列信息Table 3 The primer sequence information of low-copy nuclear genes

表4 基于7对低拷贝核基因引物的DNA多态性数据Table 4 DNA polymorphism data based on 7 pairs of low-copy nuclear gene primers
3 结论与讨论
本研究通过高通量的转录组测序技术对疏花软紫草的低拷贝核基因进行了初步筛选,得到了16对可以有效扩增的引物。并对其中的7对引物进行了多态性验证。结果表明,7对引物扩增产物的多态性位点平均值为6.57,单倍型平均值为5.14,单倍型多态性(HD)在0.389~0.944之间,对其做方差分析,发现不同引物对HD具有极显著差异(P= 0.000<0.01)。核苷酸多态性(Pi)在0.001 48~0.017 23之间,对其做方差分析,发现不同引物对Pi也存在极显著差异(P=0.008<0.01)。微卫星标记是当下科研工作者应用比较广泛的一种分子标记,其具有多态性高、重复性好、易于分析等显著特点[14]。但是近年来大量关于微卫星标记的研究显示,微卫星标记虽然能较好地反应物种内的遗传多样性,但是在物种间的通用性还是存在一定的局限性。熊敏等人对濒危植物华木莲(Sinomanglietia glauca)的基因组微卫星以及通用性的研究显示,华木莲微卫星引物在近缘种中扩增效率较低,最低达到了28.5%[15]。此外,SSR分子标记不能很好地反应物种的自然选择历史。因此,如果要更高效的对种间的进化历史、遗传结构等进行研究,有必要开发在多态性更高,并且在物种间通用性比较高的分子标记。研究表明,通过转录组测序技术开发的低拷贝核基因有较高的多态性,并可以有效地运用于群体遗传学和谱系地理学研究中。另外,这些低拷贝核基因能够被运用于软紫草属植物系统发育重建以及物种形成等研究工作中。
[参考文献]
[1]Li J H.Sequences of low-copy nuclear gene support the monophyly of Ostrya and paraphyly of Carpinus(Betulaceae)[J].Journal of Systematics and Evolution,2008,46(3):333-340.
[2]Andrea S M,Isabel S,Thomas M,et al.Utility of lowcopy nuclear markers in phylogenetic reconstruction of Hypericum L.(Hypericaceae)[J].Plant Systematics and Evolution,2014,300(6):1503-1514.
[3]刘勉,张彩飞,黄建勋,等.利用低拷贝核基因重建菊科紫菀亚科族间系统发育关系[J].植物学报,2015,50(5):549-564.
[4]岳桂东,高强,罗龙海,等.高通量测序技术在动植物研究领域中的应用[J].中国科学(生命科学),2012,42(2):107-124.
[5]黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J].林业科学,2013,49 (5):176-181.
[6]张振,张含国,莫迟,等.红松转录组SSR分析及EST-SSR标记开发[J].林业科学,2015,51(8):114-120.
[7]Zeng L P,Zhang Q,Sun R R,et al.Resolution of deep angiospermphylogeny using conserved nuclear genes and estimates of early divergencetimes[J].Nature Communications,2014,5:4956.
[8]中国科学院中国植物志编辑委员会.中国植物志:第64卷[M].北京:科学出版社,1989.
[9]Zhang C,Wang L L,Lan D,et al.Pollination ecology of Arnebia szechenyi(Boraginaceae),a Chinese endemic perennial characterized by distyly and heteromorphic self-incompatibility[J].Annales Botanici Fennici,2014,51(5):297-304.
[10]Grabherr M G,Haas B J,Yassour M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nature Biotechnology,2011,29(7):644-652.
[11]Fu L M,Niu B F,Zhu Z W,et al.CD-HIT:Accelerated for clustering the next-generation sequencing data [J].Bioinformatics,2012,28(23):3150-3152.
[12]Ebersberger I,Strauss S,von Haeseler A.HaMStR:Profile hidden markov model based search for orthologs in ESTs[J].BMC evolutionary biology,2009,9(9):157-165.
[13]Koressaar T,Remm M.Enhancements and modifications of primer design program Primer 3[J].Bioinformatics,2007,23(10):1289-1291.
[14]邹喻苹,葛颂,王晓东.系统与进化植物学中的分子标记生命科学专论[M].北京:科学出版社,2001.
[15]熊敏,王静,张志荣,等.濒危植物华木莲核基因组微卫星引物开发研究[J].植物分类与资源学报,2011,33(5):535-539.
(责任编辑张坤)
第1作者:付义(1991—),男,硕士生。研究方向:植物学/植物生物技术。Email:2367772079@qq.com。
The Development of Low-copy Nuclear Gene Primers Based on Transcriptome for Arnebia szechenyi
Fu Yi1,2,He Chengzhong1,2,Ju Miaomiao1,2,Tian Bin1,2,3
(1.Key Laboratory for Forest Genetic and Tree Improvement&Propagation in Universities of Yunnan Province,Southwest Forestry University,Kunming Yunnan 650224,China;2.Key Laboratory of Biodiversity Conservation in Southwest China,State Forestry Administration,Kunming Yunnan 650224,China;3.Key Laboratory for Plant Diversity and Biogeography of East Asia,Kunming Institute of Botany,Chinese Academy of Sciences,Kunming Yunnan 650201,China)
Abstract:Transcriptome sequencing based on high throughput has provided an opportunity for rapidly developing more molecular markers.92 042 086 reads were obtained in this research based on transcriptome sequence of Arnebia szechenyi,then acquiring 332 low-copy genes by searching 158 446 unigene of A.szechenyi.In order to verify DNA polymorphism,this research randomly selected 30 low-copy genes as designing primers and 9 individuals in three natural populations of A.szechenyi were detected by PCR amplification.It was found that 16 pairs of 30 low-copy gene primers could amplify steadily single target band.After that,7 fragments randomly selected from 16 primers were sequenced and used to calculate DNA polymorphism,the results showed that mean value of polymorphic sites based on 7 primers is 6.57,hyplotype is 5.14,hyplotype diversity(HD)was from 0.389 to 0.944,nucleotide diversity(Pi)was from 0.00148 to 0.01723.Our findings indicated that low-copy nuclear genes developed in this study based on transcriptome sequencing have relatively high polymorphism,and it could be validly used for the study of populations genetic and phylogeography.Furthermore,book=87,ebook=90these low-copy nuclear genes could be applied to the fields of phylogeny reconstruction and speciation of Arnebia.
Key words:Arnebia szechenyi,transcriptome,low-copy nuclear gene,primer
通信作者:田斌(1983—),男,副教授。研究方向:林木遗传育种及植物种群遗传学。Email:tianbinlzu@163.com。
基金项目:云南省高校林木遗传改良与繁育重点实验室研究生开放基金(YNGB201507)资助;西南地区生物多样性保育国家林业局重点实验室开放基金资助。
收稿日期:2015-12-15
doi:10.11929/ j.issn.2095-1914.2016.03.015
中图分类号:S718.46
文献标志码:A
文章编号:2095-1914(2016)03-0086-05
