蒺藜苜蓿全基因组中U-box基因家族的筛选与特征分析
2016-01-27郑兴卫邵麟惠李聪
郑兴卫,邵麟惠,李聪
(中国农业科学院北京畜牧兽医研究所,北京100193)
蒺藜苜蓿全基因组中U-box基因家族的筛选与特征分析
郑兴卫,邵麟惠,李聪*
(中国农业科学院北京畜牧兽医研究所,北京100193)
摘要:植物基因组中广泛存在U-box基因,其编码蛋白大部分作为泛素系统中决定底物特异性识别的E3泛素连接酶,广泛地调控植物生长生殖发育以及响应逆境胁迫等过程。本文利用蒺藜苜蓿基因组数据库,通过生物信息学手段,鉴定蒺藜苜蓿U-box家族基因;采用MEGA6软件进行系统进化树分析;通过GSDS在线工具和Mapinspect软件进行基因结构及染色体定位分析;利用已有的蒺藜苜蓿芯片数据进行组织表达和胁迫响应表达分析。结果表明,蒺藜苜蓿基因组中含有41个U-box基因,不均匀地分布于蒺藜苜蓿的8条染色体上。根据结构域组成和系统进化分析将这些U-box蛋白分成6类。基因表达模式分析发现,该家族成员的表达具有一定的组织特异性,并能响应盐、干旱和氮素胁迫。这些研究结果为蒺藜苜蓿U-box基因家族的功能分析奠定了基础。
关键词:蒺藜苜蓿;U-box基因家族;进化;表达模式
DOI:10.11686/cyxb2015102http://cyxb.lzu.edu.cn
郑兴卫,邵麟惠,李聪. 蒺藜苜蓿全基因组中U-box基因家族的筛选与特征分析. 草业学报, 2015, 24(8): 130-141.
Zheng X W, Shao L H, Li C. Genome-wide screening and characterization of the U-box gene family inMedicagotruncatula. Acta Prataculturae Sinica, 2015, 24(8): 130-141.
收稿日期:2015-02-25;改回日期:2015-04-09
基金项目:国家自然科学基金(31372362)项目和“十二五”国家科技支撑计划(2011BAD17B01)资助。
作者简介:郑兴卫(1987-),女,河北定州人,在读博士。E-mail:smilezxw@126.com
通讯作者*Corresponding author. E-mail:licong0520@sina.com
Genome-wide screening and characterization of the U-box gene family inMedicagotruncatula
ZHENG Xing-Wei, SHAO Lin-Hui, LI Cong*
InstituteofAnimalSciences,ChineseAcademyofAgriculturalSciences,Beijing100193,China
Abstract:The U-box domain genes typically exist in multiple variants within plant genomes, and most of them belong to the ubiquitin/proteasome system as E3ubiquitin ligase. They play key roles in the regulation of growth, in development, in reproduction as well as in response to environmental stress. In this study, bioinformatics methods were applied to identify U-box family genes of M. truncatula through whole genome screening. A phylogenetic tree was created using the MEGA6 program. Gene structure and chromosome location were defined by GSDS and Mapinspect. A total of 41 U-box genes distributed unevenly on 8 chromosomes were identified from M. truncatula. The U-box proteins were classified into six major classes based on their motif compositions and phylogeny relationships. The expression profiles of M. truncatula U-box genes showed characteristics of tissue specificity and response to salt, drought and nitrogen stress. These results will be useful for future functional analyses of the U-box family genes in M. truncatula.
Key words:Medicago truncatula; U-box gene family; phylogenetic analysis; expression patterns
U-box基因家族是一类具有U-box结构域的基因家族,其编码蛋白大部分是泛素系统中决定底物识别特性的泛素连接酶E3[1],也有部分U-box蛋白质属于泛素链聚集因子E4[2]。作为E3的U-box蛋白在真核生物,特别是植物中广泛存在,在与细胞周期、信号转导、逆境反应、转录调节、DNA修复、细胞凋亡、形态发生、分泌途径等过程有关的蛋白质降解途径中发挥着重要作用[3-6]。
U-box基序最初是从具有泛素链聚集酶 E4活性的酵母蛋白质 UFD2 中鉴定出来的[7]。第一个被鉴定出具体结构的植物U-box蛋白为拟南芥(Arabidopsisthaliana)AtPUB14蛋白质[8]。近年来,随着越来越多的 U-box 蛋白质被鉴定出来,其结构与功能也逐渐引起了人们的关注。研究表明,U-box 结构域由大约70个氨基酸组成,该结构域在真菌、植物和动物等真核生物中高度保守。在目前已鉴定出的真核生物U-box蛋白中,植物U-box(plant U-box protein,PUB)蛋白的数目远远多于其他生物[3]。如酵母中有 2 个 U-box 基因,在人中有 21 个 U-box 基因[4,7,9],而经基因组序列分析预测,拟南芥基因组中存在64个U-box基因[10-11],水稻(Oryzasativa)中已鉴定出77个带有U-box结构的基因[12]。到目前为止,多种植物的PUB蛋白基因功能已经被验证:1)PUB蛋白参与植物的防御反应。编码拟南芥AtPUB17和与其同源的烟草(Nicotianabenthamiana)ACRE276蛋白的基因能正调控细胞凋亡和植物防御的过程[13]。AtPUB19通过负调节 ABA信号调控植物的干旱胁迫响应过程[14]。超表达拟南芥的 PUB22 和 PUB23能够增加其对干旱胁迫的敏感性[15]。辣椒(Capsicumannuum)中的U-Box 蛋白质CaPUB1,在拟南芥中超表达后植株的耐盐性增加[16]。CMPG1 在烟草与番茄(Solanumlycopersicum)中参与多个抗病基因介导的抗性反应[17]。2)一些植物U-box蛋白调控植株生长和生殖发育过程。水稻OsPUB15突变后,导致种子不能长出初生根,幼苗生长缓慢甚至死亡[18]。拟南芥AtPUB4基因突变后,影响绒毡层细胞降解途径,使花粉粒粘连导致雄性不育[19]。ACR1在芸薹属植物雌蕊中作用于其下游的S 受体激酶,促进了雌蕊中兼容性因子的泛素化降解,最终导致花粉自交不亲和[20]。并且拟南芥PUB13和同源基因水稻SPL11同时能够调控植物的细胞死亡、抗病和开花时间[21],证明植物在发育和先天免疫反应方面具有信号交叉。
蒺藜苜蓿是豆科苜蓿属一年生植物,由于其具有生育期短(80~100 d)、基因组较小(454~526 Mb)、二倍体(2n=16)、自花授粉、易于转化、再生时间较短等特点被作为豆科模式植物进行研究[22-23]。蒺藜苜蓿与大部分豆科植物具有相似的遗传性,从蒺藜苜蓿获得的信息对其他豆科植物具有重要的参考价值。因此,蒺藜苜蓿成为继拟南芥和水稻进行基因组序列测定之后第3个基因组完整测序的模式植物[24-25]。蒺藜苜蓿全基因组序列公布后仍在不断完善,目前,利用生物信息学方法分析基因家族的特征、进化关系成为热点。本研究利用生物信息学方法对蒺藜苜蓿全基因组中的U-box基因家族的种类、数目、基因结构、染色体定位、系统发生和基因表达模式等进行分析,旨在为豆科植物育种提供重要的理论依据。
1材料与方法
1.1 数据来源
试验于2014年8月至2015年1月进行。最新的蒺藜苜蓿(Medicagotruncatula)全基因组数据和蛋白质数据(Mt4.0)下载于JVCI蒺藜苜蓿测序网站(http://medicago.jcvi.org/medicago/)[26];拟南芥U-box基因家族蛋白序列和基因序列下载于拟南芥U-box基因家族网站(http://www.arabidopsis.org/browse/genefamily/pub.jsp)[10];水稻U-box基因家族蛋白序列下载于水稻全基因组数据库(http://rice.plantbiology.msu.edu/)[27];蒺藜苜蓿基因芯片表达数据下载自蒺藜苜蓿基因表达图谱数据库(MtGEA v3,http://mtgea.noble.org/v3/)[28]。
1.2 蒺藜苜蓿U-box 基因家族成员鉴定
首先利用蒺藜苜蓿全基因组序列构建本地BLAST数据库[29],以目前拟南芥已经鉴定出的所有U-box基因家族成员,共64个蛋白的基因序列对蒺藜苜蓿基因组数据库执行本地BLAST搜索,E value设为0.001;同时将Pfam数据库[30]中U-box保守结构域序列下载并提取出来(PF04565),利用Hmmer v3.1b1[31](http://hmmer.janelia.org/)构建隐马尔科夫模型(HMM),在Mt4.0v1蛋白数据库中搜索含有U-box结构域的候选序列。合并上述两部分结果,手工剔除无完整读码框的序列。所得结果利用Pfam和SMART[32](http://smart.embl-heidelberg.de/)在线工具进一步分析结构域,去除无典型U-box结构域的序列,最终得到蒺藜苜蓿的U-box基因。利用ExPASy Proteomics Server(http://www.expasy.org/proteomics)对所有蒺藜苜蓿U-box蛋白氨基酸序列进行分子量、等电点预测。
1.3 U-box家族系统发育树的构建及蛋白结构域分析
通过Clustal W[33]程序对蒺藜苜蓿U-box蛋白进行多序列联配比对分析,序列联配比对结果使用MEGA6[34]程序采用邻接法(Neighbor-Joining,NJ)生成基因的系统进化树,替换模式为“Poisson model”,缺口设置为“Pairwise deletion”,校验参数Bootstrap重复1000次。利用在线工具SMART分析U-box基因家族蛋白的结构域组成。
1.4 蒺藜苜蓿U-box家族基因结构及染色体定位分析
在得到每个基因信息的同时得到了基因的DNA序列,包括基因的cDNA序列,再用在线工具GSDS[35](http://gsds.cbi.pku.edu.cn/)得到基因结构图。同时获得了这些基因的染色体位置信息,利用Mapinspect工具标出每个U-box结构域基因在染色体组上的位置,从而得到每个U-box结构域基因在基因组中的分布情况。
1.5 蒺藜苜蓿U-box家族基因表达模式分析
根据41个蒺藜苜蓿U-box基因对应的CDS序列,在蒺藜苜蓿基因芯片平台BLAST搜索其对应的探针,获得探针的表达量信息。将39个不同组织器官、不同处理下的基因芯片数据通过CIMminer在线分析软件鉴定蒺藜苜蓿U-box家族表达差异基因,并作出热图(heat-map),最后对蒺藜苜蓿U-box基因的表达情况进行聚类分析。
2结果与分析
2.1 蒺藜苜蓿U-box基因家族信息
通过本地BLAST比对以及Hmmer搜索,去除不含U-box典型结构域的序列,最终从已发表的蒺藜苜蓿全基因组中鉴定出41个U-box 基因家族成员,如表1所示。通过Pfam和SMART工具进行蛋白质结构分析,41个蒺藜苜蓿U-box蛋白均含有约60~70个氨基酸的U-box保守结构域。通过ExPASy工具分析,蒺藜苜蓿中最长的U-box蛋白(登录号:Medtr1g090320.1)包含1490个氨基酸残基,分子量大小为41个蛋白中的最大值167.08 kD,最短的U-box蛋白(登录号:Medtr2g007630.1)包含259个氨基酸残基,分子量最小,为28.73 kD。等电点范围为5.28(Medtr7g078330.1)~9.24(Medtr2g007630.1)。
2.2 蒺藜苜蓿U-box家族系统进化及蛋白结构域分析
为研究蒺藜苜蓿U-box基因家族系统进化关系,对最终筛选出的41个蒺藜苜蓿U-box蛋白进行了系统进化树的构建。经过SMART蛋白结构域分析发现,除U-box结构域外,蒺藜苜蓿U-box蛋白还含有其他结构域。根据进化树及蛋白结构域(图1),并参考拟南芥和水稻的U-box基因家族分类结果(表2),本文将蒺藜苜蓿中41个U-box蛋白分为6种类型,分别包含18,12,5,3,2和1个蒺藜苜蓿U-box蛋白家族成员。ARM亚家族在植物中研究最多,并且在拟南芥和水稻中都被鉴定为最大的亚类,在蒺藜苜蓿中为第2大亚类。在拟南芥和水稻中根据N端保守位点甘氨酸、赖氨酸/精氨酸和亮氨酸定义的GKL+U-box亚家族,因为其结构域分析与U-box only亚类没有区别,因此在本文中归为U-box only亚类,U-box only结构的PUB蛋白在蒺藜苜蓿中的数量最多,共18个。Kinase亚家族有5个成员,其中3个成员含丝/苏氨酸蛋白激酶结构域(S_TKc domain),另外两个为丝/苏/酪氨酸蛋白激酶结构域(STYKc domain)。含有WD40重复结构和TPR结构的U-box基因在蒺藜苜蓿中分别有3和2个。MtPUB28是蒺藜苜蓿中唯一含有UFD2结构的U-box蛋白。
为了深入分析与其他物种同源基因的进化关系,对蒺藜苜蓿与拟南芥和水稻的U-box/ARM亚类进行了系统进化树的构建(图2)。结果显示,与拟南芥或水稻中的同源基因相比,大部分蒺藜苜蓿U-box/ARM基因与蒺藜苜蓿家族内部的同源基因的遗传距离更加接近。然而,MtPUB25、MtPUB32和MtPUB34在拟南芥中都存在与其遗传距离最近的直系同源蛋白。
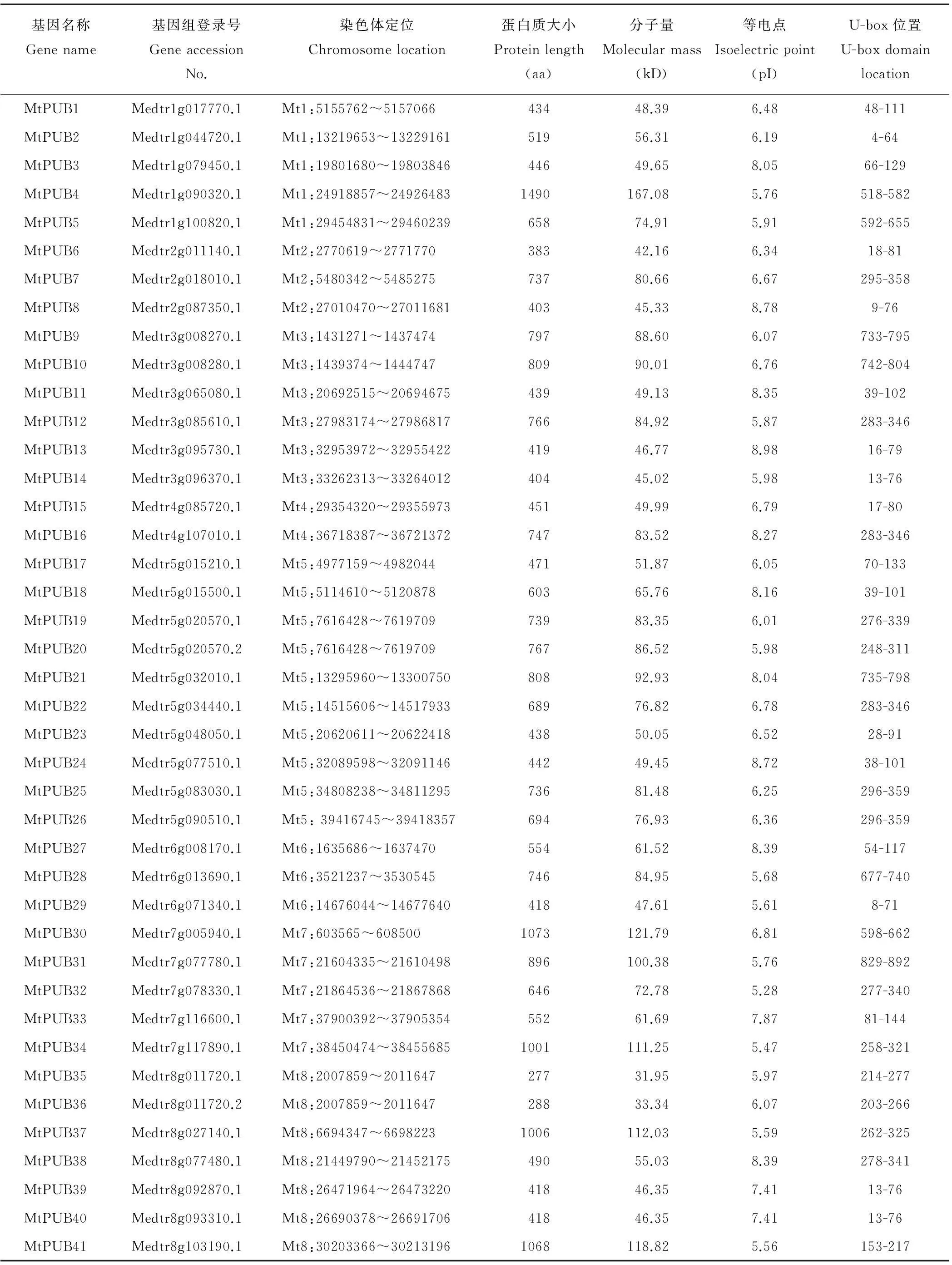
表1 蒺藜苜蓿基因组中的U-box基因
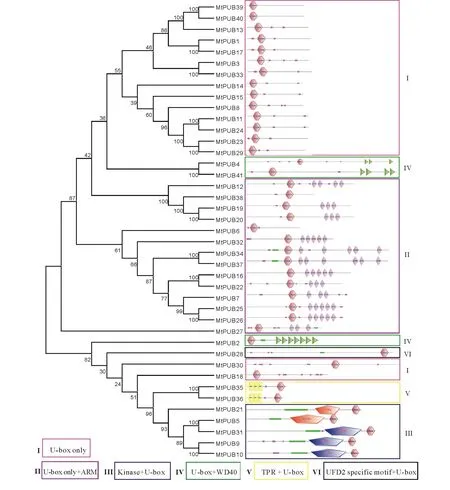
图1 蒺藜苜蓿U-box家族进化树及蛋白结构Fig.1 The neighbor joining phylogenetic tree and structure of U-box proteins in M. truncatula 使用U-box蛋白的氨基酸序列构建进化树,分支上数字代表校验参数Bootstrap重复1000次时该节点可信度The phylogenetic tree was constructed by using the amino acid sequences of the U-box proteins. The numbers on the branches mean the reliability percent of Bootstraps value based on 1000 replication.
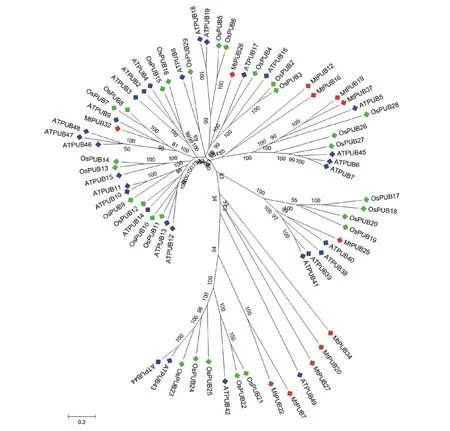
图2 蒺藜苜蓿与拟南芥、水稻U-box/ARM蛋白的系统发生关系Fig.2 Phylogenetic relationship of M. truncatula, Arabidopsis and O. sativa U-box/ARM proteins 红色表示蒺藜苜蓿U-box/ARM蛋白,蓝色表示拟南芥U-box/ARM蛋白,绿色表示水稻U-box/ARM蛋白。The red labels indicate U-box/ARM protein in M. truncatula, while blue and green ones indicate U-box/ARM in Arabidopsis and O. sativa, respectively.
2.3 蒺藜苜蓿U-box家族基因结构及染色体定位分析
对家族成员的基因结构分析显示(图3),蒺藜苜蓿U-box家族基因结构差异较大,内含子数目从0个到16个数目不等。进一步分析发现,蒺藜苜蓿U-box蛋白分类中的I类,即U-box only亚类,其基因结构较为简单,大多数不含内含子或仅含有1个内含子,仅有3个基因含有3个以上的内含子。II类(U-box/ARM亚类)中每个基因的内含子数目不超过4个。III类(Kinase/U-box亚类)中基因的内含子数目在6~9个之间,IV类(U-box/WD40亚类)的3个基因所含内含子数目最多,分别为16个、15个和13个。V类(TPR/U-box亚类)的2个基因均含有7个内含子。仅包含1个基因的VI类(UFD2/U-box亚类),Medtr6g013690.1基因含有15个内含子。
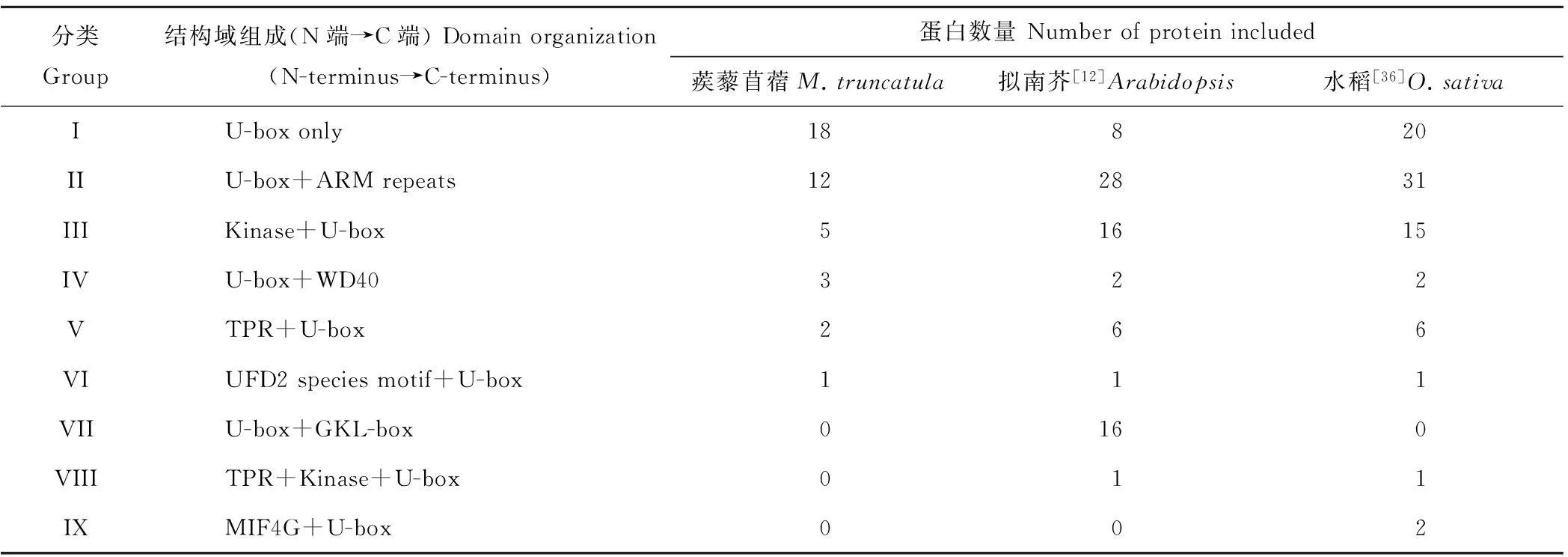
表2 蒺藜苜蓿、拟南芥和水稻U-box蛋白的结构域组成
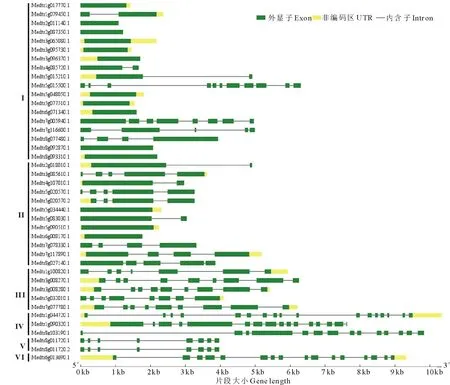
图3 41个蒺藜苜蓿U-box基因的结构分析Fig.3 Gene structure of 41 U-box gene in M. truncatla
通过提取蒺藜苜蓿U-box基因的染色体定位信息,得到41个U-box基因在蒺藜苜蓿8条染色体上的定位图(图4)。由图4可知,蒺藜苜蓿U-box基因在染色体上呈不均等分布。其中5号染色体分布最多,共含有10个U-box基因,而4号染色体上分布的U-box基因最少,仅有2个U-box成员。另外,U-box基因在染色体上的分布呈现区域性,从3号、5号、7号和8号染色体中可以看到,在染色体上的某一区域家族成员个数比较密集。
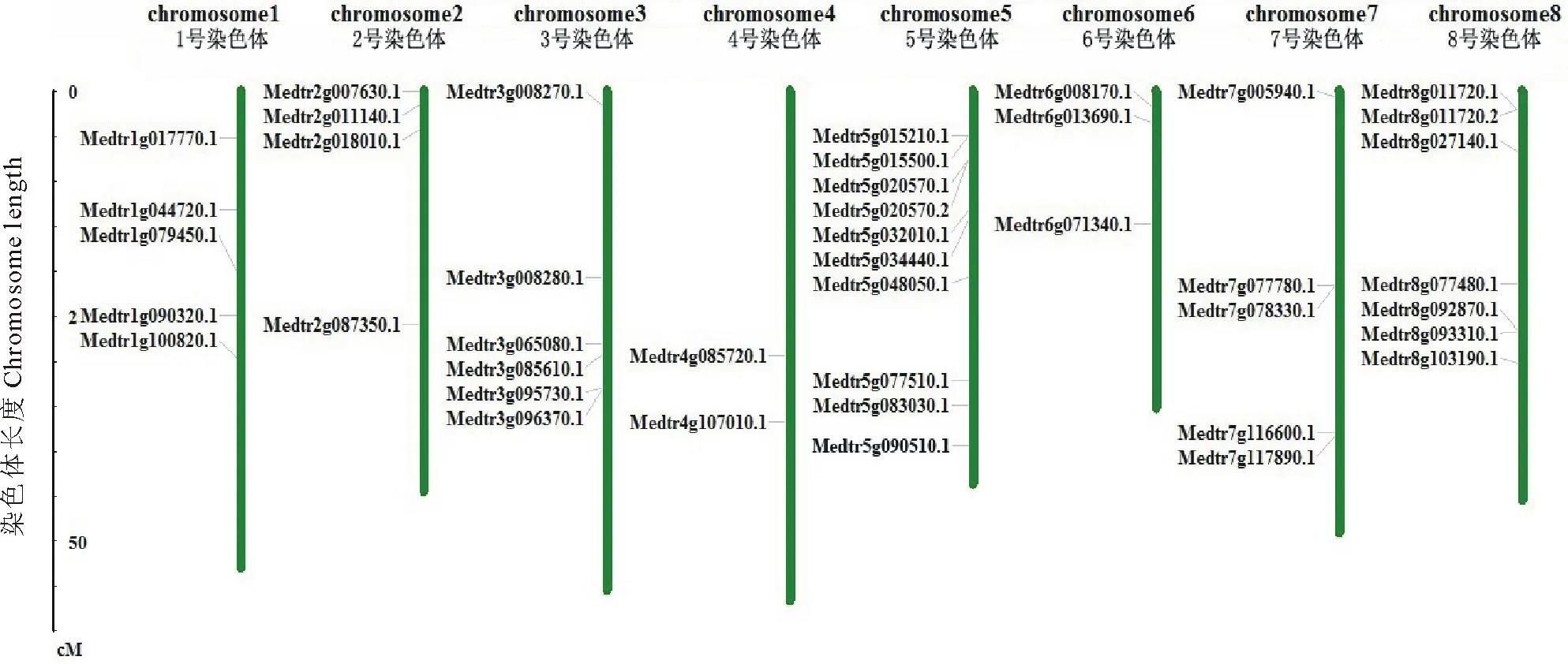
图4 蒺藜苜蓿U-box基因在染色体上的定位Fig.4 Chromosomal locations of U-box genes in M. truncatula
2.4 蒺藜苜蓿U-box家族基因表达模式分析
利用蒺藜苜蓿MtGEA数据库进行的组织表达分析结果(图5)表明,共有37个U-box基因在数据库中有表达数据。聚类图中用绿-黑-红三色代表基因表达的强度,红色越亮代表信号越强,绿色越亮代表信号越弱,黑色代表中间水平。结果显示,Medtr5g020570.1、Medtr5g020570.2在叶片中特异性表达,Medtr5g077510.1和Medtr3g008082.1主要在根中表达,Medtr1g017770.1与Medtr3g065080.1在营养器官中表达量高,Medtr1g079450.1和Medtr7g116600.1在种皮中特异性表达。Medtr1g044720.1普遍存在于蒺藜苜蓿叶、茎、花和种子等组织中,表达量较为丰富,而Medtr5g048050.1、Medtr5g083030.1、Medtr5g090510.1、Medtr6g071340.1和Medtr7g005940.1在各组织中表达量都很低。
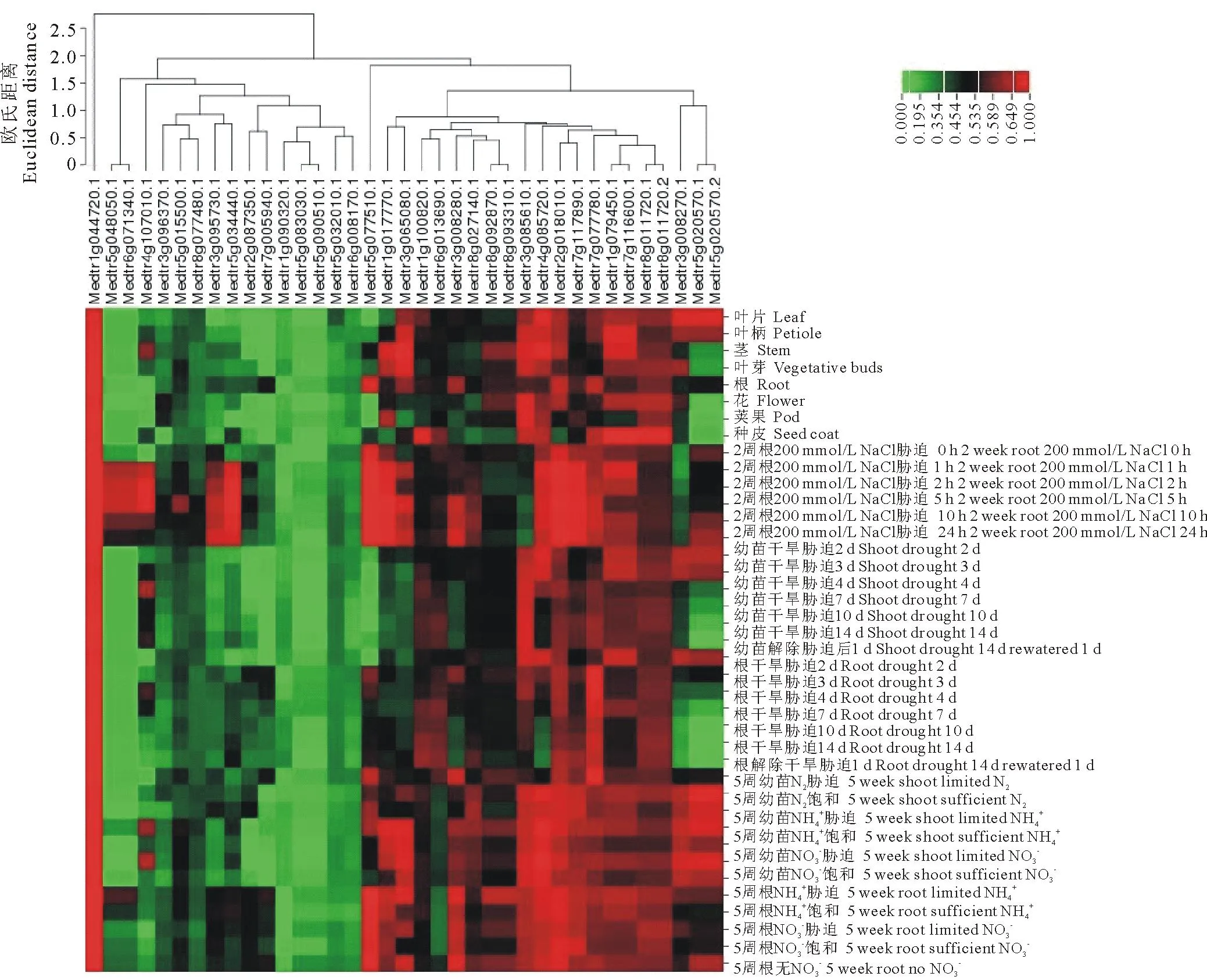
图5 蒺藜苜蓿U-box基因表达模式Fig.5 The expression profile of the M. truncatula U-box genes
同时,根据不同处理下的芯片数据,还分析了不同胁迫处理下蒺藜苜蓿U-box基因的表达模式,包括盐(200 mmol/L NaCl)、干旱、低氮(N2、NO3-、NH4+)处理。结果显示,盐胁迫下9个基因(Medtr1g017770.1、Medtr2g018010.1、Medtr3g095730.1、Medtr4g107010.1、Medtr5g034440.1、Medtr5g048050.1、Medtr5g077510.1、Medtr6g071340.1和Medtr7g117890.1)在根中表达量上调,表明这些基因可能会参与植株幼苗期对盐胁迫的响应。水分胁迫下,在幼苗和根中检测到的U-box基因表达量变化并不一致。干旱处理后在幼苗中表达量上调的基因有Medtr4g107010.1和Medtr6g013690.1两个,Medtr4g085720.1、Medtr5g020570.1、Medtr5g020570.2和Medtr7g116600.1四个基因则表现为干旱时表达量降低,胁迫解除后又恢复甚至超过正常值。而在干旱胁迫后的根中,Medtr1g044720.1基因表达量上调,表达量下降的有10个基因(Medtr3g008270.1、Medtr3g008280.1、Medtr3g095730.1、Medtr4g085720.1、Medtr5g020570.1、Medtr5g020570.2、 Medtr7g005940.1、Medtr7g077780.1、Medtr8g092870.1和Medtr8g093310.1)。N素(N2、NO3-、NH4+)胁迫下,Medtr4g085720.1和Medtr4g107010.1 在幼苗中响应胁迫,表达量都上调,而根中这两个基因表达量基本不受N素(NO3-、NH4+)胁迫的影响。Medtr7g005940.1在幼苗和根中对NH4+胁迫响应都比NO3-敏感。
3讨论
随着多种植物基因组测序的完成,利用全基因组数据对基因家族进行系统分析和预测基因功能已成为植物基因功能研究所关注的重要问题[37-39]。蒺藜苜蓿作为豆科的模式植物,其基因组测序完成后,WRKY[40-41]、LBD[42]、MADS-box[43]及NBS[44]等家族都进行了广泛的生物信息学分析。U-box蛋白在植物生殖发育及抗逆机制中发挥着至关重要的作用,是当前生物学研究的热点。本研究在蒺藜苜蓿基因组中鉴定出41个U-box基因,这与之前拟南芥(64个)[39]和水稻(77个)[36]等植物的报导存在一定差异,这说明U-box基因家族并不与基因组大小有直接的关系,这与Zeng等[12]在水稻U-box基因家族研究中的结论一致。
U-box功能域是一种被修饰的RING-finger结构,它不具有RING-finger type蛋白质中由半胱氨酸和组氨酸及两个锌离子构成的金属离子螯合残基,而是用盐键和氢键来稳定其结构[45]。除了N端的U-box结构域外,U-box蛋白中通常还存在一些与蛋白相互作用密切相关的二级结构域,主要用来介导U-box蛋白与底物蛋白的特异性识别。本研究中根据除U-box结构域外的其他结构域的种类,将蒺藜苜蓿中的41个U-box蛋白家族成员分为6个亚家族。U-box/ARM亚家族成员是目前植物U-box家族基因中功能研究最多的亚类。拟南芥和水稻均为研究最为深入的模式植物,突变体类型较蒺藜苜蓿更为丰富,芯片数据更加全面完整,U-box基因功能也得到了更加广泛的验证。研究表明,U-box/ARM蛋白通过多个ARM基序相互衔接形成独特的结构以提供蛋白-蛋白相互作用的场所,参与植物的自交不亲和、抗病、相应非生物胁迫、激素信号转导等多个过程。本文对蒺藜苜蓿与拟南芥、水稻的U-box/ARM亚家族进行了联合系统进化分析,发现与单子叶的水稻相比,蒺藜苜蓿U-box基因与同是双子叶的拟南芥更容易聚为一类,并且U-box基因家族在物种间进化上是较为保守的(图2),所以可以认为与拟南芥聚类在一起的蒺藜苜蓿U-box基因也能具有类似的功能。基于拟南芥中基因功能已报导或基因组中已注释的U-box/ARM基因的分析结果[46-47],可以对蒺藜苜蓿的U-box基因家族进行功能预测分析。在磷酸盐缺失条件下,拟南芥AtPUB9与其上游信号AtARK2共同调控植株侧根发育[48],与其对应的蒺藜苜蓿同源蛋白MtPUB32可能也具有类似功能。拟南芥AtPUB18和AtPUB19是同源基因,参与ABA介导的干旱胁迫响应过程,并且与AtPUB22和AtPUB23响应干旱胁迫的过程相互独立[49-52],蒺藜苜蓿中MtPUB26与AtPUB18和AtPUB19同源,可能也参与到类似的干旱响应过程。
本文通过对蒺藜苜蓿基因芯片信息分析发现,根据蒺藜苜蓿U-box家族基因在8个不同组织的表达显示,不同的U-box家族成员在表达上具有一定的组织特异性,暗示U-box家族成员间存在功能特异性。而陈浩等[53]通过选取水稻中4个U-box蛋白质进行Western blotting检测和EST数据比较分析,得出4个U-box蛋白质在水稻生长发育的不同时期或部位为组成型表达,且表达量接近。这可能与物种间存在差异有关,或者与不同研究中所涉及的转录谱数据和EST数据存在差异有关。多个U-box蛋白基因在盐、干旱和氮素胁迫条件下,表达量出现上调或下调的变化,暗示这些基因在调控逆境胁迫过程中能够发挥作用。
目前国内外对U-box基因功能的研究主要采用酵母双杂交筛选与其相互作用的下游蛋白[54],以及基于RNA沉默或基因敲除的反向遗传学办法[19]。通过研究蒺藜苜蓿U-box蛋白的结构与功能,寻找与其相互作用的靶蛋白,将有助于了解植物体内蛋白与蛋白的互作机制,从而揭示U-box基因如何调控蒺藜苜蓿的生长发育,以及如何参与对逆境胁迫的信号转导过程。
References:
[1]Shigetsugu H, Kei-ichi I N. U-box proteins as a new family of ubiquitin ligases. Biochemical and Biophysical Research Communications, 2003, 302(4): 635-645.
[2]Aravind L, Eugene V K. The U box is a modified RING finger-a common domain in ubiquitination. Current Biology, 2000, 10(4): 132-134.
[3]Patterson C. A new gun in town: the U box is a ubiquitin ligase domain. Science’s STKE, 2002, 2002(116): pe4.
[4]Hatakeyama S, Yada M, Matsumoto M,etal. U box proteins as a new family of ubiquitin-protein ligases. The Journal of Biological Chemistry, 2001, 276(35): 33111-33120.
[5]Hatakeyama S, Nakayama K I. U-box proteins as a new family of ubiquitin ligases. Biochemical and Biophysical Research Communications, 2003, 302(4): 635-645.
[6]Andersen P, Kragelund B B, Olsen A N,etal. Structure and biochemical function of a prototypicalArabidopsisU-box domain. The Journal of Biological Chemistry, 2004, 279(38): 40053-40061.
[7]VanDemark A P, Hill C P. Structural basis of ubiquitylation. Current Opinion Structural Biology, 2002, 12(6): 822-830.
[8]Koegl M, Hoppe T, Schlenker S,etal. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell, 1999, 96(5): 635-644.
[9]Ohi M D, Vander Kooi C W, Rosenberg J A,etal. Structural insights into the U-box, a domain associated with multi-ubiquitination. Nature Structural Biology, 2003, 10(4): 250-255.
[10]Azevedo C, Santos-Rosa M J, Shirasu K. The U-box protein family in plants. Trends in Plant Science, 2001, 6(8): 354-358.
[11]Wiborg J, O’Shea C, Skriver K. Biochemical function of typical and variantArabidopsisthalianaU-box E3 ubiquitin-protein ligases. Biochemistry Journal, 2008, 413(3): 447-457.
[12]Zeng L R, Park C H, Venu R C,etal. Classification, expression pattern, and E3 ligase activity assay of rice U-box-containing proteins. Molecular Plant, 2008, 1(5): 800-815.
[13]Yang C W, GonzAilez-Lamothe, Rocio,etal. The E3 ubiquitin ligase activity ofArabidopsisPLANT U-BOX17 and its functional tobacco homolog ACRE276 are required for cell death and defense. The Plant Cell, 2006, 18(4): 1084-1098.
[14]Samuel M A, Mudgil Y, Salt J N,etal. Interactions between the S-domain receptor kinases and AtPUB-ARM E3 ubiquitin ligases suggest a conserved signaling pathway inArabidopsis. Plant Physiology, 2008, 147(4): 2084-2095.
[15]Cho S K, Ryu M Y, Song C,etal.ArabidopsisPUB22 and PUB23 are homologous U-Box E3 ubiquitin ligases that play combinatory roles in response to drought stress. Plant Cell, 2008, 20(7): 1899-1914.
[16]Cho S K, Chung H S, Ryu M Y,etal. Heterologous expression and molecular and cellular characterization of CaPUB1 encoding a hot pepper U-Box E3 ubiquitin ligase homolog. Plant Physiology, 2006, 142(4): 1664-1682.
[17]Gonzalez-Lamothe R, Tsitsigiannis D I, Ludwig A A,etal. The U-box protein CMPG1 is required for efficient activation of defense mechanisms triggered by multiple resistance genes in tobacco and tomato. Plant Cell, 2006, 18(4): 1067-1083.
[18]Park J J, Yi J, Yoon J,etal. OsPUB15, an E3 ubiquitin ligase, functions to reduce cellular oxidative stress during seedling establishment. The Plant Journal, 2011, 65(2): 194-205.
[19]Wang H, Lu Y, Jiang T,etal. TheArabidopsisU-box/ARM 1 repeat E3 ligase AtPUB4 influences growth and degeneration of tapetal cells, and its mutation leads to conditional male sterility. The Plant Journal, 2013, 74(3): 511-523.
[20]Stone S L, Anderson E M, Mullen R T,etal. ARC1 is an E3 ubiquitin ligase and promotes the ubiquitination of proteins during the rejection of self-incompatibleBrassicapollen. Plant Cell, 2003, 15(4): 885-898.
[21]Liu J, Li W, Ning Y,etal. The U-box E3 ligase SPL11/PUB13 is a convergence point of defense and flowering signaling in plants. Plant Physiology, 2012, 160(1): 28-37.
[22]Wang Y, Liu W X, Ma L C,etal. Research progresses inMedicagotruncatulagenetic transformation. Pratacultural Science, 2014, 31(3): 504-511.
[23]Wei Z W, Gai J Y. Model legume:Medicagotruncatula. Acta Prataculturae Sinica, 2008, 17(1): 114-120.
[24]Gary S, Marc L, Laurent B,etal. Genetics and functional genomics of legume nodulation. Current Opinion in Plant Biology, 2006, 9(2): 110-121.
[25]Endre G, Kereszt A, Kevei Z,etal. A receptor kinase gene regulating symbiotic nodule development. Nature, 2002, 417: 962-966.
[26]Tang H, Krishnakumar V, Bidwell S,etal. An improved genome release (version Mt4.0) for the model legumeMedicagotruncatula. BMC Genomics, 2014, 15: 312.
[27]Kawahara Y, de la Bastide M, Hamilton J P,etal. Improvement of theOryzasativaNipponbare reference genome using next generation sequence and optical map data. Rice, 2013, 6: 4.
[28]He J, Benedito V A, Wang M,etal. TheMedicagotruncatulagene expression atlas web server. BMC Bioinformatics, 2009, 10: 441.
[29]Braun R C, Pedretti K T, Casavant T L,etal. Parallelization of local BLAST service on workstation cluster. Future Generation Computer System, 2001, 17(6): 745-754.
[30]Finn R D, Mistry J, Schuster-Bockler B,etal. Pfam: clans, web tools and services. Nucleic Acid Research, 2006, 34(S1): 247-251.
[31]Finn R D, Clements J, Eddy S R.HMMER web server: interactive sequence similarity searching. Nucleic Acid Research, 2011, 39(S2): 29-37.
[32]Letunic I, Doerks T, Bork P. SMART 6: recent updates and new developments. Nucleic Acid Research, 2009, 37(S1): 229-232.
[33]Larkin M A, Blackshields G, Brown N P,etal. Clustal W and Clustal X version 2.0.Bioinformatics, 2007, 23(21): 2947-2948.
[34]Tamura K, Stecher G, Peterson D,etal. MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30: 2725-2729.
[35]Hu B, Jin J, Guo A,etal. GSDS 2.0: an upgraded gene feature visualization server. Bioinformatics, 2014, doi: 10.1093/bioinformatics/btu817.
[36]Cao Y H. The Characteristics of Rice U-Box Gene Family with its Transcription and Expression Pattern Analysis[D]. Baoding: Agricultural University of Hebei Province, 2012.
[37]Jiang G G, Song L L, Guo D L,etal. Genome-wide association mapping of aluminum tolerance inMedicagotruncatula. Acta Prataculturae Sinica, 2013, 22(4): 170-178.
[38]Kang J M, Zhang T J, Yang Q C,etal. Research progress in the quantitative trait loci (QTL) and genomic selection of alfalfa. Acta Prataculturae Sinica, 2014, 23(6): 304-312.
[39]Lu Y Q, Guan N, Li C. Advanced in the study on U-box proteins ofArabidopsisthaliana. Molecular Plant Breeding, 2008, 6(5): 941-948.
[40]Jiang T, Lin Y X, Lin X,etal. Genome-wide analysis of the WRKY transcription factor family inMedicagotruncatula. Acta Prataculturae Sinica, 2011, 20(3): 211-218.
[41]Song H, Nan Z B. Genome-wide identification and analysis of WRKY transcription factors inMedicagotruncatula. Hereditas, 2014, 36(2): 152-168.
[42]Jia X T, Liu W X, Xie W G,etal. Genome-wide analysis of the LBD transcription factor family inMedicagotruncatula. Acta Botanica Boreali-Occidentalia Sinica, 2014, 34(10): 2176-2187.
[43]Zhang J, Song L L, Guo D L,etal. Genome-wide identification and investigation of the MADS-box family inMedicagotruncatula. Acta Prataculturae Sinica, 2014, 23(6): 233-241.
[44]Zhao Y, Jiang H Y, Wang J M,etal. Analysis of genome-wide disease resistance genes and their expansion inMedicagotruncatula. Journal of Agricultural Biotechnology, 2009, 17(6): 1062-1069.
[45]Donna Y, Daphne R G. The diversity of plant U-box E3 ubiquitin ligases: from upstream activators to downstream target substrates. Journal of Experimental Botany, 2009, 60(4): 1109-1121.
[46]Yang J, Lan X G, Li Y H. U-box/ARM proteins in plants. Plant Physiology Communications, 2008, 44(6): 1216-1222.
[47]Vogelmann K, Subert C, Danzberger N,etal. Plasma membrane-association of SAUL1-type plant U-box armadillo repeat proteins is conserved in land plants. Frontiers in Plant Science, 2014, 5: 37.
[48]Drechsel G, Bergler J, Wippel K,etal. C-terminal armadillo repeats are essential and sufficient for association of the plant U-box armadillo E3 ubiquitin ligase SAUL1 with the plasma membrane. Journal of Experimental Botany, 2011, 62(2): 775-785.
[49]Srijani D, Subramanian S, Gayathri W,etal. The S-domain receptor kinase AtARK2 and the U- box/ARM-repeat-containing E3 ubiquitin ligase 9 module mediates lateral root development under phosphate starvation inArabidopsis. Plant Physiology, 2014, 165(4): 1647-1656.
[50]Yashwanti M, Shiu S H, Sophia L S,etal. A large complement of the predictedArabidopsisARM repeat protein are members of the U-box E3 ubiquitin ligase family. Plant Physiology, 2004, 134(1): 59-66.
[51]Jakob W, Charlotte O S, Karen S. Biochemical function of typical and variantArabidopsisthalianaU-box E3 ubiquitin-protein ligase. Biochemical Journal, 2008, 413: 447-457.
[52]Seo D H, Ryu M Y, Jammes F,etal. Roles of fourArabidopsisU-Box E3 ubiquitin ligases in negative regulation of abscisic acid-mediated drought stress responses. Plant Physiology, 2012, 160(1): 556-568.
[53]Chen H, Li L Y, Bai H,etal. Expression analysis of rice U-Box proteins at different developmental stages. Progress in Biochemistry and Biophysics, 2009, 36(9): 1208-1214.
[54]Mbengue M, Camut S, de Carvalho-Niebel F,etal. TheMedicagotruncatulaE3 ubiquitin ligase PUB1 interacts with the LYK3 symbiotic receptor and negatively regulates infection and nodulation. The Plant Cell, 2010, 22(10): 3474-3488. doi: 10.1105/tpc.110.075861.
参考文献:
[22]王宇, 刘文献, 马利超, 等. 蒺藜苜蓿遗传转化体系研究进展. 草业科学, 2014, 31(3): 504-511.
[23]魏臻武, 盖钧镒. 豆科模式植物——蒺藜苜蓿. 草业学报, 2008, 17(1): 114-120.
[36]曹英豪. 水稻U-box基因家族的特征及转录表达模式分析[D]. 保定: 河北农业大学, 2010.
[37]姜格格, 宋丽莉, 郭东林, 等. 蒺藜苜蓿耐酸铝性状的全基因组关联分析. 草业学报, 2013, 22(4): 170-178.
[38]康俊梅, 张铁军, 杨青川, 等. 紫花苜蓿QTL与全基因组选择研究进展及其应用. 草业学报, 2014, 23(6): 304-312.
[39]鲁玉清, 关宁, 李聪. 拟南芥U-box蛋白的研究进展. 分子植物育种, 2008, 6(5): 941-948.
[40]江腾, 林勇祥, 林雪, 等. 苜蓿全基因组WRKY转录因子基因的分析. 草业学报, 2011, 20(3): 211-218.
[41]宋辉, 南志标. 蒺藜苜蓿全基因组中WRKY转录因子的鉴定与分析. 遗传, 2014, 36(2): 152-168.
[42]贾喜涛, 刘文献, 谢文刚, 等. 蒺藜苜蓿LBD转录因子家族全基因组分析. 西北植物学报, 2014, 34(10): 2176-2187.
[43]张军, 宋丽莉, 郭东林, 等. MADS-box基因家族在蒺藜苜蓿的全基因组分析. 草业学报, 2014, 23(6): 233-241.
[44]赵阳, 江海洋, 汪结明, 等. 蒺藜苜蓿全基因组抗病基因和基因扩张分析. 农业生物技术学报, 2009, 17(6): 1062-1069.
[46]杨佳, 蓝兴国, 李玉花. 植物U-box/ARM蛋白. 植物生理学通讯, 2008, 44(6): 1216-1222.
[53]陈浩, 李莉云, 白辉, 等. 水稻U-Box蛋白质在不同发育时期的表达分析. 生物化学与生物物理进展, 2009, 36(9): 1208-1214.
