羟基苯甲腈类抑制剂与Cdc25B磷酸酯酶相互作用的结合位点残基观察
2015-12-02王喆赵宏涛
王喆,赵宏涛
(天津医科大学第二医院,天津 300211)
Cdc25磷酸酯酶属于双特异性蛋白磷酸酶,在细胞周期中发挥重要作用,可将细胞周期蛋白依赖性激酶(CDKs)去磷酸化,从而使其激活[1]。Cdc25磷酸酯酶包括Cdc25A、Cdc25B和Cdc25C三种亚型[2],其中Cdc25B可以与CDK2/Cyclin A复合物结合,水解CDK2中ATP结合位点loop上两残基的磷酸根,进而将其激活[3]。Cdc25B在多种肿瘤细胞如结肠癌、胰腺癌、卵巢癌等过度表达,对肿瘤细胞的发展起促进作用,下调Cdc25B表达会引起肿瘤细胞周期阻滞,抑制肿瘤细胞增殖。因此,Cdc25B已经成为治疗癌症的潜在药物作用靶点[4,5]。传统的设计思路是针对Cdc25B的催化活性位点设计抑制剂,但该位点空间体积较小,导致小分子抑制剂设计难度增加。最近研究[6]报道了新型羟基苯甲腈类Cdc25B抑制剂,此类抑制剂并不与Cdc25B催化活性位点结合,而是作用于Cdc25B与CDK2/Cyclin A复合物的结合位点,从而抑制Cdc25B酶活性。目前对Cdc25B中专门负责识别羟基苯甲腈类抑制剂残基的认识还十分有限,本研究利用分子动力学模拟的方法确定了羟基苯甲腈类抑制剂与Cdc25B磷酸酯酶相互作用的结合位点残基。
1 材料与方法
1.1 Cdc25B蛋白及其结构预处理 从RCSB蛋白数据库下载人Cdc25B/A8H(PDB编号:4WH7)和Cdc25B/3M8(PDB编号:4WH9)复合物晶体结构,去除晶体结构中的硫酸根离子和甘油分子,只保留小分子抑制剂周围0.4 nm范围内的结构水分子。
1.2 A8H、3M8与Cdc25B磷酸酯酶相互作用的分子动力学模拟参数设置 分子动力学模拟所用软件为 Gromacs 4.5.5,蛋白采用 Amber FF99SB 力场[7],小分子抑制剂采用GAFF(General Amber Force Field)[8],通过量子化学半经验AM1方法对小分子抑制剂进行结构优化,并利用AM1-BCC方法拟合出小分子抑制剂原子的局部电荷。在进行分子动力学模拟前,先将两个蛋白/抑制剂复合物晶体结构置于TIP3P水盒子中,水盒子边缘到最近的溶质原子的距离为1.0 nm。此外,模拟使用周期边界条件,利用PME(Particle Mesh Ewald)方法校正长程静电作用,使用LINCS算法限制连有氢原子化学键的键长,分子动力学模拟所用范德华作用阈值为1.0 nm,模拟温度设定为300 K,恒温模拟算法为Velocity Rescaling[9]。分子动力学模拟积分步长为1.0 fs,每隔10 ps保存1次轨迹。在进行分子动力学平衡前,对两个蛋白/抑制剂复合物结构进行最速下降法优化,直到体系原子所受力最大值不超过1 000 kJ/(mol·nm2)。为平衡溶剂水分子,在恒温恒容条件下,给两个蛋白/抑制剂复合物重原子加1 000 kJ/(mol·nm2)的限制力进行100 ps分子动力学模拟。然后去掉该限制力,在恒温恒容条件下进行1 ns分子动力学平衡过程以及10 ns正式分子动力学模拟。
1.3 Cdc25B/羟基苯甲腈类抑制剂复合物的稳定性观察 为研究Cdc25B/抑制剂复合物在分子动力学模拟过程中的稳定性,本研究以10 ns正式分子动力学模拟的起始结构为参照,计算了Cdc25B/A8H和Cdc25B/3M8复合物蛋白骨架Cα原子均方根偏差(RMSD)随时间变化的曲线。各体系RMSD数值在5 ns后基本在0.15~0.2 nm范围内波动,上述各体系在5 ns后达到分子动力学模拟的平衡状态,分子动力学模拟产生的后5 ns轨迹可以用于进一步的氢键作用和相互作用能分析。
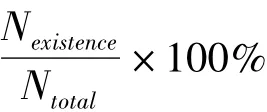
1.5 A8H、3M8与CDC25B结合位点残基相互作用能计算方法 根据Amber力场计算公式[12],计算小分子抑制剂(A8H和3M8)与CDC25B活性位点残基的静电相互作用能和范德华相互作用能,A8H、3M8与残基之间的总相互作用能为静电相互作用能和范德华相互作用能之和,相互作用能计算所使用的轨迹与氢键作用分析相同。
2 结果
2.1 A8H、3M8与Cdc25B结合位点残基的氢键作用 A8H与Cdc25B结合位点各残基Y382、D397、K399、R485、R488、R492 形成氢键的概率分别为 0、2.00%、0.40%、0.60%、0、0,3M8 与 Cdc25B 结合位点各残基 Y382、D397、K399、R485、R488、R492 形成氢键的概率分别为 11.38%、12.97%、0.80%、4.19%、94.41%、97.21%。3M8 的磺酸根可以与R488、R492侧链的胍基以及Y382侧链的酚羟基形成氢键作用,3M8的酚羟基与D397侧链的羧基形成氢键作用,其中3M8与Y382、D397之间的氢键作用是不能从晶体结构看到的。
2.2 A8H、3M8与Cdc25B结合位点残基的相互作用能 D397、L398、C484、R485、R488、M505 与 A8H形成了较强相互作用,Y382、D397、L398、C484、R485、R488、R492、M505 与 A8H 形成了较强相互作用,详见图1。
3 讨论
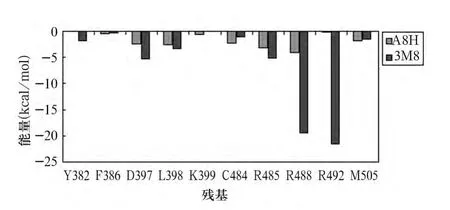
图1 A8H、3M8与Cdc25B结合位点残基相互作用能
确定生物大分子中参与配体分子识别的残基,是进行基于结构药物设计的基础。虽然Lund等[6]通过X射线晶体结构,发现了Cdc25B中对R488和R492的识别作用,但是Cdc25B对抑制剂的识别是一个动态的过程,其他一些极性残基也在这两个残基附近,在动态的分子识别过程中,它们也可能与小分子抑制剂形成强极性相互作用,从而参与该类抑制剂的识别。除了极性作用,非极性作用也是分子识别的关键。由于羟基苯甲腈类抑制剂具有像苯环这样的芳香基团,推测Cdc25B中某些疏水残基在该类抑制剂的识别过程中同样发挥重要作用。单纯观察蛋白/抑制剂复合物晶体结构,很难发现哪些疏水残基参与了抑制剂的识别。相比静态蛋白晶体结构,分子动力学方法可以通过计算,模拟出Cdc25B与羟基苯甲腈类抑制剂之间的动态相互作用情况,并在此基础上,通过分析氢键作用概率和相互作用能,确定参与抑制剂识别的残基。
氢键作用是蛋白对小分子进行识别的主要方式之一,在氢键作用分析中,本研究计算了小分子抑制剂与Cdc25B结合位点残基氢键作用概率,那些与A8H或3M8形成稳定氢键作用的残基被认为参与了Cdc25B对此类小分子抑制剂的生物识别。本研究结果表明,Y382、D397、R488和R492可以通过氢键作用方式,帮助Cdc25B识别羟基苯甲腈类小分子抑制剂。由于氢键作用分析只是在一定程度反映了小分子抑制剂与Cdc25B结合位点残基之间静电作用强度的差异,不能反映非极性的范德华相互作用大小。为进一步研究Cdc25B结合位点残基对该类小分子抑制剂的识别机制,就需要了解Cdc25B结合位点残基与这两个小分子抑制剂之间的相互作用强度。考虑到这两个抑制剂都占据了Cdc25B相同的区域,两个小分子抑制剂A8H和3M8与这些残基的相互作用强度无法通过分析晶体结构来判断。为进一步研究Cdc25B结合位点残基与小分子抑制剂的相互作用,就必须计算Cdc25B结合位点残基与这两种小分子抑制剂的相互作用能。本研究结果表明,Y382、D397、L398、C484、R485、R488、R492、M505参与了Cdc25B对羟基苯甲腈类抑制剂的识别作用。
综上,本研究利用分子动力学模拟的方法,通过分析Cdc25B与羟基苯甲腈类抑制剂之间氢键作用概率和相互作用能,发现 R488和 R492参与了Cdc25B对羟基苯甲腈类抑制剂的识别,这与既往研究[6]结果相吻合。在此基础上,本研究还发现Y382、D397、L398、C484、R485 和 M505 同样参与了Cdc25B对羟基苯甲腈类抑制剂的识别。因此,研究人员在设计Cdc25B抑制剂时,需要注意调控小分子化合物与这些残基之间的相互作用。
[1]Sarkis M,Tran DN,Kolb S,et al.Design and synthesis of novel bis-thiazolone derivatives as micromolar CDC25 phosphatase inhibitors:effect of dimerisation on phosphatase inhibition[J].Bioorg Med Chem Lett,2012,22(24):7345-7350.
[2]Sibille E,Bana E,Chaouni W,et al.Development of a matrix-assisted laser desorption/ionization-mass spectrometry screening test to evidence reversible and irreversible inhibitors of CDC25 phosphatases[J].Anal Biochem,2012,430(1):83-91.
[3]Song Y,Lin X,Kang D,et al.Discovery and characterization of novel imidazopyridine derivative CHEQ-2 as a potent CDC25 inhibitor andpromising anticancer drug candidate[J].Eur J Med Chem,2014,(82):293-307.
[4]Contour-Galcera MO,Sidhu A,Prévost G,et al.What's new on CDC25 phosphatase inhibitors[J].Pharmacol Ther,2007,115(1):1-12.
[5]Brault L,Bagrel D.Activity of novel Cdc25 inhibitors and preliminary evaluation of their potentiation of chemotherapeutic drugs in human breast cancer cells[J].Life Sci,2008,82(5-6):315-323.
[6]Lund G,Dudkin S,Borkin D,et al.Inhibition of CDC25B phosphatase through disruption of protein-protein interaction[J].ACS Chem Biol,2015,10(2):390-394.
[7]Lindorff-Larsen K,Piana S,Palmo K,et al.Improved side-chain torsion potentials for the Amber ff99SB protein force field[J].Proteins,2010,78(8):1950-1958.
[8]Wang J,Wolf RM,Caldwell JW,et al.Development and testing of a general amber force field[J].J Comput Chem,2004,25(9):1157-1174.
[9]Bussi G,Donadio D,Parrinello M.Canonical sampling through velocity rescaling[J].J Chem Phys,2007,126(1):014101.
[10]Cosentino U,Pitea D,Moro G,et al.Conformational behaviour determines the low-relaxivity state of a conditional MRI contrast agent[J].Phys Chem Chem Phys,2009,11(20):3943-3950.
[11]Wang Q,Gao J,Liu Y,et al.Molecular dynamics simulation of the interaction between protein tyrosine phosphatase 1B and aryl diketoacidderivatives[J].J Mol Graph Model,2012,(38):186-193.
[12]Steinbrecher T,Koslowski T,Case DA.Direct simulation of electron transfer reactions in DNA radical cations[J].J Phys Chem B,2008,112(51):16935-16944.
