基于海甘蓝RNA-Seq 序列开发EST-SSR 分子标记
2014-12-23戚维聪程计华黄邦全
戚维聪, 程计华, 黄邦全, 李 坦, 林 峰
(1.江苏省农业科学院农业生物技术研究所,江苏 南京210014;2.荷兰瓦赫宁根大学植物育种系,荷兰 瓦赫宁根6708PB;3.中国科学院武汉植物园,湖北 武汉430074;4.湖北大学生命科学院,湖北 武汉430062)
海甘蓝(Crambe abyssinica)是异源六倍体(2n=6x=90),原产于地中海地区,属于十字花科(Brassicaceae)两节荠属(Crambegenus)[1-2]。生产芥酸是海甘蓝经济价值的主要体现,海甘蓝的芥酸含量达到60%。芥酸(顺-13-二十二碳一烯酸)是一种长碳链不饱和脂肪酸,在化工中应用广泛,其衍生物芥酸酰胺是塑料生产的必须添加剂,主要作用是润滑,防止粘连。除此之外芥酸还可以被用在制药、生产船舶表面涂料和尼龙等多个方面[1,3]。全球对芥酸年需求量大且增长迅速,从1990 年到2010 年,总消费量从1.8 ×107t 增长至3.5 ×107t,大约翻了一番。近年来由于大面积推广种植双低油菜,高芥酸资源出现短缺。为了满足工业上对芥酸这一重要化工原料的需求,美国、荷兰等国相继培育、释放、种植了一系列高芥酸含量的海甘蓝新品种。海甘蓝中度耐盐,适合在盐碱等边际性土地上生长。中国拥有近2.2 ×106hm2海洋滩涂,利用这些边际性土地种植高芥酸海甘蓝将产生显著经济效益[4-5]。
海甘蓝作为一个“新”的经济作物,其遗传研究较为薄弱。目前,NCBI 数据库只搜集了37 条海甘蓝EST 序列,基于海甘蓝遗传信息开发的分子标记尚未见报道。二代测序技术的推广和应用为快速有效地发展“新”经济作物的分子标记提供了契机[6-7]。通过对转录组RNA-Seq 数据的从头拼接(De novo)和组装,海量测序短片段被连接成一类较长的重叠群(Contigs),这些重叠群包含了特定发育时期的部分转录本信息[8]。转录本含有大量的简单重复序列(Simple sequence repeat,SSR),由于简单重复序列变异度较高,这些位点已成为开发功能分子标记的理想靶位点[8-9]。利用RNA-Seq 组装的重叠群序列开发EST-SSR 分子标记的技术已在蓖麻、草坪草、芝麻,红薯和咖啡等植物中广泛应用[10-12]。但是由于海甘蓝研究中尚未获得足够数量的EST 和RNA-Seq 数据信息,开发EST-SSR 分子标记仍是空白。
海甘蓝种子发育阶段是合成芥酸的主要时期,关键基因在这段时间内相继表达。基于此,我们将对发育时期种子进行高通量深度测序,利用从头拼接软件获得重叠群,对这些重叠群序列进行SSR 位点扫描,并对海甘蓝基因组DNA 进行PCR 扩增,以期为揭示海甘蓝遗传背景的多样性以及遗传连锁图谱的构建提供第一代SSR 分子标记。
1 材料与方法
1.1 供试材料及全基因组测序
海甘蓝品种为C.abyssinicacv.Galatic。去壳种子置于铺有3 层润湿滤纸的培养皿中,发芽后移入温室土壤栽培,待植物开花后挂牌标记。取开花后20 d 的种子提取mRNA(10 粒为一组),试剂盒为iScriptTMcDNA Synthesis Kit (Bio-rad,USA)。DNA提取材料种植条件如前所述,取0.5 g 新鲜叶片提取基因组DNA。
1.2 组装拼接
采用Illumina HiSeq 2000 测序仪进行测序。进一步利用SolexaQA[13]软件包对原始测序数据进行质量(Q20,Phred-Score≥20 即1%的错误率)和测序长度(L40,长度≥40 bp)过滤。随后,利用Trinity转录组组装软件[14]按默认参数对清理后的序列进行转录组从头de novo组装。
1.3 SSR 全基因组挖掘及引物设计
对Trinity 程序组装获得的转录组重叠群序列,用MISA 程序(http://pgrc.ipk-gatersleben.de/misa/misa.html)扫描转录组SSR 位点(http://pgrc.ipkgatersleben.de/misa/),满足单碱基重复超过10 次或双碱基重复超过8 次或三碱基重复超过5 次或四碱基重复超过4 次或五碱基重复超过2 次或六碱基重复超过3 次被定义为SSR 位点。通过Perl 语言抽取SSR 位点侧翼序列后利用Primer 3(http://biotools.umassmed.edu/bioapps/primer3_www.cgi)进行PCR 特异引物设计,退火温度为(58 ±3)℃。
1.4 海甘蓝SSR 位点在白菜基因组的定位及注释
为了进一步揭示海甘蓝EST-SSRs 相关的基因组信息及变异效应,利用所开发的SSRs 分子标记保守侧翼序列,结合白菜基因组序列信息(http://www.ncbi.nlm.nih.gov/nuccore/AENI00000000),通过Blast[14]同源比对,去除冗余以及进行全基因组物理定位。SSR 位点多态性比较则主要通过电子ePCR 软件(http://www.ncbi.nlm.nih.gov/projects/e-pcr/)完成,利用本地数据库预测白菜基因组中PCR 扩增产物的大小。
1.5 PCR 扩增和电泳
PCR 反应总体系为25.0 μl,包括10 ×Buffer(含Mg2+)2.5 μl,dNTP 2.5 μl,Taq酶0.1 μl,引物2.0 μl,模板DNA 2.0 μl,ddH2O 15.9 μl,上覆20.0 μl矿物油。PCR 反应程序为:94 ℃预变性3 min;94℃变性30 s,58 ℃退火1 min,72 ℃延伸80 s,共进行38 个循环。用2%琼脂糖胶加压100 V 进行电泳,紫外透射仪上观察、拍照。
2 结果与分析
2.1 组装拼接
利用Illumina Paired-End 100 bp×2 双端测序,经Q20 质量过滤和L40 长度清理,获得了86 224 256 条平均长度约93 bp,共计7 999 663 632个碱基的重叠群。数据进一步利用Trinity 组装程序进行从头de novo组装,共获得长度等于200 个碱基的重叠群序列234 622条,重叠群平均长度为956 bp,其中N50 为1 428 bp,最长的重叠群为16 475 bp。由于组装后的重叠群存在选择性剪切产生的冗余,利用Blastn (1e-50)去除高相似度的序列后得到186 778条重叠群,这些重叠群序列用于后续的SSR 挖掘。
2.2 EST-SSR 位点扫描
MISA 软件扫描重叠群后共发现在38 601条序列中含有47 073个SSR 位点,其中有6 816条重叠群含有1 个以上的SSR 位点,平均每4 500 bp 含有1 个SSR位点。除了单碱基重复10 次的SSR 位点外(45%),三碱基重复SSR 位点占了总数的29%,双碱基重复占10%,五碱基重复占7%,六碱基重复占5%,四碱基重复占2%。三碱基重复中AAG/CTT 基序存在于4 993条序列中,丰度最高(表1、表2、图1)。

表1 海甘蓝EST-SSR 搜索结果Table 1 Summary of EST-SSR searching resulting results
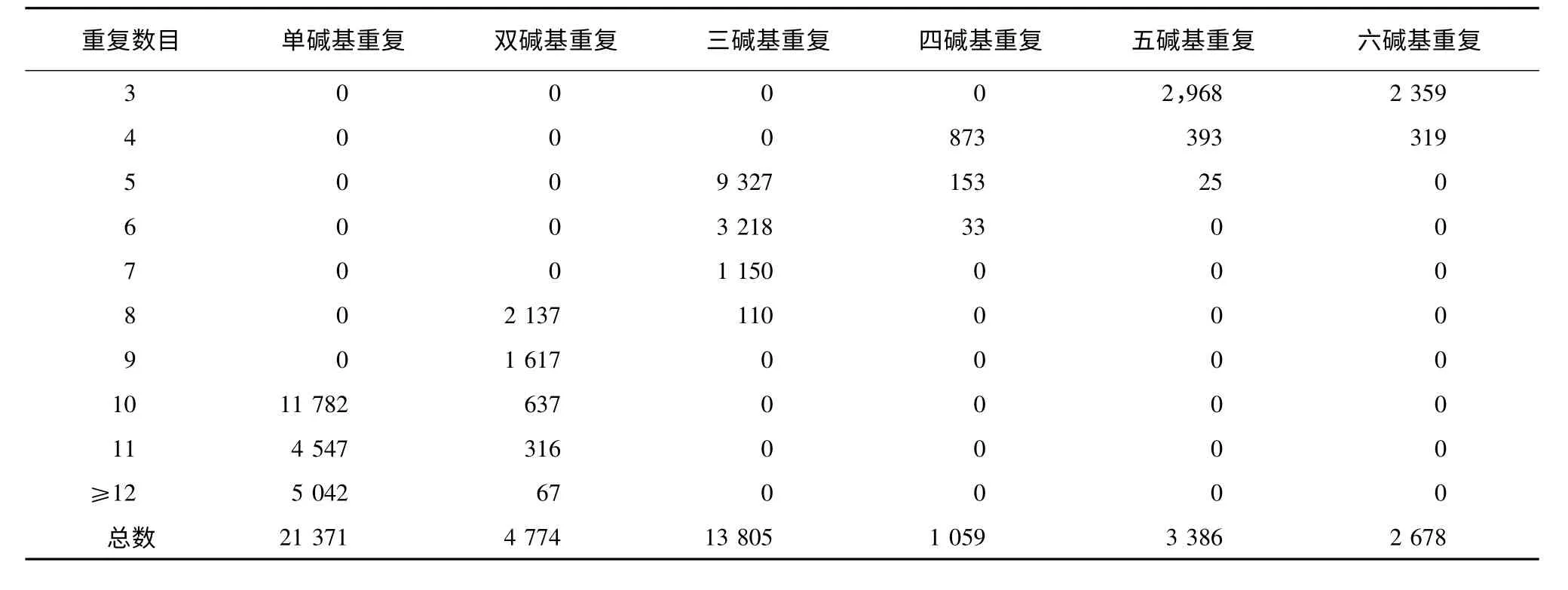
表2 海甘蓝EST-SSR 位点长度信息Table 2 Length distribution of EST-SSR based on the number of repeat units
2.3 EST-SSR 引物设计及过滤
利用SSR 位点侧翼保守序列,共设计了16 355对SSR 引物。PCR 产物扩增范围为150 ~400 bp。利用Blastn 对电子PCR 产物进行了冗余性评估,发现只有6 639条引物可以专化扩增出唯一特异性条带。利用已测序白菜基因组信息对6 639对引物进行筛选,在5'端允许3 个碱基错配,3'端不允许错配,PCR 产物长度大于200 bp,小于500 bp 的条件下,获得了1 206条物种间可通用的EST-SSR 引物。根据白菜基因组每10 kb DNA 区间内只允许存在一个分子标记的条件下,共获得了688 个分子标记。图2 列出了这些分子标记在白菜基因组的物理位置。除个别位置外,ESTSSR 分子标记的位置在染色体上基本呈均匀分布。
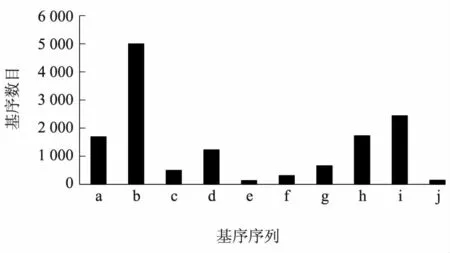
图1 三核苷酸不同基序排列分布Fig.1 Frequency distribution of trinucleotide EST-derived SSRs based on motif sequences
2.4 EST-SSR 分子标记验证
基于白菜基因组信息,在每条染色体臂两端各挑选了一个SSR 位点,共计20 对EST-SSR 引物在海甘蓝中进行引物验证(表2)。通过琼脂糖凝胶电泳发现,在检测的20 对SSR 引物中,扩增成功率达100%。20 对PCR 产物片段大小均与预测结果完全吻合(图3)。
3 讨论
通过对已公开EST 序列进行SSR 位点扫描开发分子标记的方法已被广泛地运用。海甘蓝作为一个“新”作物,其EST 信息在NCBI 数据库中几乎为空白。我们通过二代测序技术,首次获得了大量海甘蓝EST 序列。其中约有3%左右的序列含有SRR位点,这些位点侧翼序列为设计引物提供了模板。虽然通过以上的方法可以快速高效设计EST-SSR引物,但是在研究过程中发现,二代测序拼接结果中含有大量冗余(Redundancy)序列,基于这些冗余序列开发的一系列EST-SSR 引物只能被认定为一个位点的分子标记。导致大量冗余序列的主要原因是RNA 选择性剪切和测序过程中的污染。因此,在设计海甘蓝EST-SSR 引物前,我们首先对Trinity 拼接结果进行了过滤,通过序列之间相互对比,去除了30%高相似度的重叠群。其次对余下的序列进行了功能注释,删除明显来自人类和细菌的污染序列。最后,利用海甘蓝近缘物种白菜基因组序列,淘汰了物理位置在10 kb 之内的分子标记。通过以上3 种过滤,本研究最大程度地获得了688 个全基因组较为分散的SSR 分子标记。
由于EST 主要编码功能基因,受选择压力大,序列保守性高,导致EST-SSR 所揭示的多态性相对基因组SSR 低[13,15-17]。研究表明,在EST 的3'区存在较高的三核苷酸变异率。如控制人类亨丁顿舞蹈症的HTT基因是在第四对染色体,HTT基因重复排序数目CAG 太多时会造成亨丁顿舞蹈症,重复数28 次以下没有亨丁顿舞蹈症表现,而40 以上有亨丁顿舞蹈症,基于CAG 重复序列开发的SSR 标记在人类不同个体间具有较高的多态性[9]。玉米Prolamin 蛋白结合因子(PBF)在3'区也存在一个高变异率的ACC 三核苷酸序列,基于该重复序列开发的umc1065 EST-SRR 分子标记在玉米不同自交系间揭示极高的多态性[18]。尽管根据EST 设计的SSR 较根据基因组设计的多态性偏低,但由于与功能基因紧密相连,可被开发成功能性分子标记,显著提高分子标记对目的功能基因的选择效率。
EST-SSR 引物较根据基因组信息设计的SSR 引物扩增成功效率偏低,原因主要一方面引物结合位点落在外显子与外显子的连接区,该区段在基因组上被内含子隔开,导致引物无特定结合区域;另一方面,PCR 扩增区域若含有较长的内含子,引物在基因组DNA 为模板时由于区段太长,无法获得有效的扩增。由于缺乏海甘蓝基因组信息,无法仅通过生物信息学手段预测内含子的位置和大小。为了最大程度解决EST-SSR 引物扩增效率偏低的问题,我们利用海甘蓝的近缘种白菜的基因组测序信息来过滤外显子连接区和大内含子区,最终获得了近688 个高质量的引物信息。这些白菜和海甘蓝通用分子标记(Transferable EST-SSR markers)可以被用于这两个物种间的比较基因组研究,利用白菜的遗传信息加速海甘蓝关键基因的鉴定和克隆。
注:本研究获得的EST-SSR 引物及对应的具体信息可向作者索取。
[1] KANEKO Y,BANG S,MATSUZAWA Y.Wild crop relatives:genomic and breeding resources[M].Berlin:Springer,2011:247-258.
[2] WARWICK S,GUGEL R.Genetic variation in theCrambe abyssinica-C.hispanica-C.glabratacomplex[J].Genetic Resources and Crop Evolution,2003,50(3):291-305.

图2 海甘蓝EST-SSR 分子标记在白菜基因组物理分布Fig.2 The physical localization of EST-SSR on Brassica napa genome

图3 20 对SSR 引物基因组扩增带型Fig.3 PCR banding patterns of Crambe abyssinice cv.Galatic by 20 SSR primer pairs
[3] ABADA Y S K,NGUYEN H P,SCHREIBER R,et al.Assessment of motor function,sensory motor gating and recognition memory in a novel BACHD transgenic rat model for Huntington disease[J].PLoS ONE,2013,8(7):e68584.
[4] METTERNICHT G,ZINCK J.Remote sensing of soil salinity:potentials and constraints [J].Remote Sensing of Environment,2003,85(1):1-20.
[5] RHOADES J,MANTEGHI N,SHOUSE P,et al.Soil electrical conductivity and soil salinity:new formulations and calibrations[J].Soil Science Society of America Journal,1989,53(2):433-439.
[6] VARSHNEY R K,NAYAK S N,MAY G D,et al.Next-generation sequencing technologies and their implications for crop genetics and breeding[J].Trends in Biotechnology,2009,27(9):522-530.
[7] DAVEY J W,HOHENLOHE P A,ETTER P D,et al.Genomewide genetic marker discovery and genotyping using next-generation sequencing[J].Nature Reviews Genetics,2011,12(7):499-510.
[8] RAHMAN M,SUN Z,MCVETTY P B.High throughput genomespecific and gene-specific molecular markers for erucic acid genes inBrassica napus(L.)for marker-assisted selection in plant breeding[J].Theoretical and Applied Genetics,2008,117(6):895-904.
[9] RIESS O,NOERREMOELLE A,SOERENSEN S A,et al.Improved PCR conditions for the stretch of (CAG)n repeats causing Huntington's disease[J].Human Molecular Genetics,1993,2(6):637.
[10] AGGARWAL R K,HENDRE P S,VARSHNEY R K,et al.Identification,characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species[J].Theoretical and Applied Genetics,2007,114(2):359-372.
[11] FEINGOLD S,LLOYD J,NORERO N,et al.Mapping and characterization of new EST-derived microsatellites for potato (Solanum tuberosumL.)[J].Theoretical and Applied Genetics,2005,111(3):456-466.
[12] ZHANG T,GE M,YE X,et al.Construction of a linkage map for quantitative trait loci associated with economically important traits in creeping bentgrass (Agrostis stoloniferaL.)[J].Euphytica,2012,188(3):347-360.
[13] GORBACH D M,HU Z L,DU Z Q,et al.Mining ESTs to determine the usefulness of SNPs across shrimp species[J].Animal Biotechnology,2010,21(2):100-103.
[14] ALTSCHUL S F,MADDEN T L,SCHÄFFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[15] MUCHERO W,DIOP N N,BHAT P R,et al.A consensus genetic map of cowpea[Vigna unguiculata(L.)Walp.]and synteny based on EST-derived SNPs[J].Proceedings of the National Academy of Sciences,2009,106(43):18159-18164.
[16] SEHGAL D,RAJARAM V,ARMSTEAD I P,et al.Integration of gene-based markers in a pearl millet genetic map for identification of candidate genes underlying drought tolerance quantitative trait loci[J].BMC Plant Biology,2012,12(1):9.
[17] XUE S,ZHANG Z,LIN F,et al.A high-density intervarietal map of the wheat genome enriched with markers derived from expressed sequence tags [J].Theoretical and Applied Genetics,2008,117(2):181-189.
[18] VICENTE-CARBAJOSA J,MOOSE S P,PARSONS R L,et al.A maize zinc-finger protein binds the prolamin box in zein gene promoters and interacts with the basic leucine zipper transcriptional activator Opaque2[J].Proceedings of the National Academy of Sciences,1997,94(14):7685-7690.
