多重PCR鉴定三种颚口线虫方法的建立
2014-04-13李树清李雯雯张鸿满陈韶红陈志飞王巧全张永年黄维义
李树清,李雯雯,张鸿满,陈韶红,李 健,陈志飞,王巧全,张永年,黄维义
(1. 上海出入境检验检疫局,上海 200135;2. 中山大学附属第一医院动物实验中心,广州 510080;3. 广西疾病预防控制中心,南宁 530021; 4. 中国疫病预防控制中心寄生虫病预防控制所,上海 200025;5. 广西大学动物科学技术学院,南宁 530005)
多重PCR鉴定三种颚口线虫方法的建立
李树清1,李雯雯2,张鸿满3,陈韶红4,李 健5,陈志飞1,王巧全1,张永年4,黄维义5
(1. 上海出入境检验检疫局,上海 200135;2. 中山大学附属第一医院动物实验中心,广州 510080;3. 广西疾病预防控制中心,南宁 530021; 4. 中国疫病预防控制中心寄生虫病预防控制所,上海 200025;5. 广西大学动物科学技术学院,南宁 530005)
为了建立快速、灵敏、可靠的鉴别颚口线虫虫种的方法,根据GenBank中发表的棘颚口线虫、日本颚口线虫和杜氏颚口线虫ITS-2序列,设计了3对特异引物,建立了这3种颚口线虫的单一PCR和多重PCR检测方法,并分别对单一PCR和多重PCR方法的特异性和敏感性进行了鉴定。结果显示单一PCR和多重PCR均能特异扩增出棘颚口线虫、日本颚口线虫和杜氏颚口线虫,其片段大小分别为282、358、183 bp,单一PCR对棘颚口线虫、日本颚口线虫和杜氏颚口线虫虫体DNA最小检出量分别为0.2、0.01、0.01 ng/μL。对宫脂线虫、异尖线虫、棘口吸虫以及迭宫绦虫均不能进行扩增。用建立的多重PCR方法对菲律宾、印度尼西亚、黑龙江颚口线虫等12条颚口线虫DNA模板进行扩增,经鉴定菲律宾与印度尼西亚的颚口线虫为棘颚口线虫,黑龙江的颚口线虫为日本颚口线虫。鉴定结果与原虫体样本的形态鉴定和测序分析结果一致。研究显示建立的多重PCR方法具有很强的特异性和较高的敏感性,可用于棘颚口线虫、日本颚口线虫和杜氏颚口线虫虫种的鉴定和流行病学调查。
棘颚口线虫;日本颚口线虫;杜氏颚口线虫;多重PCR;虫种鉴定
颚口线虫病是因人食入生的或未熟的含有颚口属(Gnathostoma)幼虫的淡水鱼引起的人畜共患寄生虫病。颚口线虫属有13个有效种,目前已报道的致病种有棘颚口线虫(G.spinigerum)、刚刺颚口线虫(G.hispidum)、杜氏颚口线虫(G.doloresi)、日本颚口线虫(G.nipponicum)、马来颚口线虫(G.malaysiae)和双核颚口线虫(G.binucleatum),对食品中检出的颚口线虫幼虫虫种的鉴别对该病的诊断与防治有重要意义[1,2]。颚口线虫种类传统的鉴定方法是以形态特征为鉴别指标,但颚口线虫的生活史复杂,不同发育阶段形态变化较大,有些虫种的第三期幼虫形态相似,传统形态方法很难鉴别,因而不能作为虫种鉴别的精确指标,且传统方法也费时费力。
多重PCR(multiplex PCR),又称多重引物PCR或复合PCR,它是在同一PCR反应体系里加上2对以上引物,同时扩增出多个核酸片段的PCR反应,该技术可以高效、系统、经济、简便的应用于同时检测或鉴定病原微生物,及某些遗传病和癌基因的分型鉴定。寄生虫种类鉴别研究的目的基因多以核糖体DNA(rDNA)为主。真核生物rDNA的内转录间隔区(internal transcribed spacer,ITS)包括ITS-1和ITS-2,进化较快,具有种间特异性和种内保守性,是进行种间分类的极好材料,可设计特异性的引物进行扩增,并且可以通过比较ITS序列的差异来鉴定形态学上难以区别的近缘种[3,4]。根据ITS序列用多重PCR方法来鉴别颚口线虫虫种,目前国内外尚无相关报道。棘颚口线虫、杜氏颚口线虫、日本颚口线虫是亚洲最常见的3种颚口线虫致病种,本研究根据ITS-2序列,分别设计这3种颚口线虫的特异引物,进行多重PCR扩增,旨在建立一种快速、灵敏、可靠的鉴别颚口线虫种类的方法。
1 材料与方法
1.1 虫种G.spinigerum、G.doloresi由广西疫病预防控制中心张鸿满主任医师惠赠,G.nipponicum由本室从泥鳅中分离,3种标准虫种已经过形态学和分子生物学鉴定。其ITS2基因序列GenBank登录号分别为:JN408326、JN408329和JN408315,CO1基因序列登录号分别为JN408310、JN408299和JN408298。虫体保存于70%乙醇中。
1.2 对照虫种宫脂线虫(Hysterothylacium)、异尖线虫(Anisakis)由本室保存;棘口吸虫(Echinostoma)、迭宫绦虫(Spirometra)分别由广西大学黄维义教授与广西疫病预防控制中心张鸿满主任医师惠赠。
1.3 待鉴定样品待鉴定样品为2010年~2011年分离自菲律宾、印度尼西亚进口黄鳝与黑龙江泥鳅的颚口线虫。2条菲律宾与8条印度尼西亚分离的颚口线虫经形态学与分子生物学鉴定为G.spinigerum(ITS2登录号:JN408316~JN408325,CO1登录号为JN408300~JN408309);2条黑龙江分离的颚口线虫经形态学与分子生物学鉴定为G.nipponicum(ITS2登录号为JN408313和JN408314,CO1登录号为JN408296和JN408297)。
1.4 主要试剂Taq聚合酶、PCR试剂、DNAMarker(DL500)购自宝生物工程有限公司;DNA提取试剂盒购自QIAGEN公司。
1.5 仪器及设备梯度PCR扩增仪(美国AppliedBiosystems公司)、电泳仪(美国Labnet公司)、G:BOX凝胶成像系统(英国Syngene公司)。
1.6 引物设计与合成从GenBank中获得G.spinigerum(AB181155)、G.doloresi(AB181156)、G.nipponicum(AB181157)ITS2序列,应用DNAStar7.0分子生物学软件分析后设计引物,由上海生工生物工程技术服务有限公司合成(表1)。

表1 多重PCR引物序列Table 1 Primers used in multiplex PCR
1.7 虫体DNA提取从70%乙醇保存液中取出单个虫体,置于1.5 mLEppendorf管中,用蒸馏水反复浸泡清洗3次,每隔2h换水1次,按照QIAGEN公司DNA提取试剂盒(DNeasyBlood&Tissuekit)使用说明书提取颚口线虫基因组DNA,将提取的基因组DNA置于-20℃保存备用。
1.8 PCR反应条件的优化25μL的反应体系:25mmol/LMg2+2.5μL、10×PCRbuffer2.5μL、2.5mmol/LdNTP1μL、5U/μLTaq聚合酶0.1μL、适当浓度的上游和下游引物、DNA模板1μL。以抽提的DNA为模板,对PCR各循环参数、退火温度和各引物浓度等进行优化,以确定最佳的PCR模式。
1.9 单一PCR扩增将上述提取的3种颚口线虫DNA分别用各自的引物进行PCR扩增。25μL反应体系:10×buffer2.5μL、Mg2+2.5μL(25mmo1)、dNTP1μL(2.5mmol)、上下游引物各0.5μL(10μmo1)、Taq聚合酶0.1μL(5U/μL)、模板DNA1μL,加灭菌ddH2O至25μL。反应过程:95℃预变性3min;95℃变性30s,55℃退火30s,72℃延伸30s,30个循环,72℃延伸5min。
1.10 多重PCR扩增25μL反应体系:10×buffer2.5μL、Mg2+2.5μL(25mmo1)、dNTP1μL(2.5mmol)、G.doloresi与G.nipponicum上下游引物各0.3μL(10μmo1/L)、G.pinigerum上下游引物各0.4μL(10μmo1/L)。Taq聚合酶0.1μL(5U/μL)、模板DNA各1μL,加灭菌ddH20至25μL。操作程序和反应参数同上。
1.11 PCR扩增的特异性单一PCR:用G.spinigerum、G.doloresi或G.nipponicum的单一引物,分别对G.spinigerum、G.doloresi、G.nipponicum、Hysterothylacium、Anisakis、Echinostoma与Spirometra的DNA模板,在同等条件下进行PCR反应,以鉴定其特异性;多重PCR:同时用3对引物,分别对G.spinigerum、G.doloresi和G.nipponicum及其不同组合、Hysterothylacium、Anisakis、Echinostoma与Spirometra的DNA模板,在同等条件下进行PCR反应,以鉴定其特异性。
1.12 PCR扩增的敏感性以10倍梯度稀释法分别将G.spinigerum、G.doloresi、G.nipponicum3种颚口线虫的DNA模板稀释成5个浓度梯度,按以上条件进行PCR扩增,测定单一PCR和多重PCR反应的敏感性。
1.13 对检疫样品的检测将各地检出的颚口线虫虫体(均经过形态学和分子生物学鉴定),提取DNA模板,按上述建立的多重PCR方法进行扩增。
2 结果
2.1 颚口线虫DNA模板的制备结果将3种颚口线虫所提取的DNA经分光光度计测定,OD260/OD280在1.8~2.0范围内,纯度符合PCR要求。
2.2 PCR反应条件优化结果单一引物扩增相应虫种DNA模板时,退火温度在55℃~61℃ 时条带无明显差异变化;3对引物混合扩增3个虫种的混合DNA模板时,51℃和53℃退火温度G.doloresi条带稍暗,当退火温度在55℃~61℃时各条带最亮,且无非特异性条带(图1)。最后选用55℃作为多重PCR退火温度,多重PCR引物最佳混合比例:G.doloresi、G.nipponicum上下游引物各0.3μL(10 μmo1/L),G.spinigerum上下游引物各0.4μL(10 μmo1/L)。
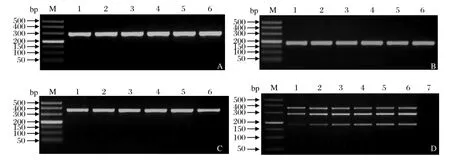
图1 棘颚口(A)、杜氏颚口(B)、日本颚口(C)单一PCR和多重PCR(D)退火温度的确定Fig.1 Agarose gel electrophoresis of single PCR for G. spinigerum (A), G. doloresi (B), G. nipponicum(C)and multiplex PCR(D)amplif cation products
2.3 单一PCR扩增结果及特异性单一PCR扩增后,产物经3.0% 琼脂糖凝胶电泳检测,可见G.spinigerum、G.doloresi、G.nipponicum分别扩增出1条282、183和358 bp的条带,与各自的预期相符;3种引物对Hysterothylacium、Anisakis、Echinostoma与Spirometra均未扩增出任何条带(图2)。G.spinigerum、G.doloresi和G.nipponicum单一PCR特异性扩增产物经测序分析后,结果显示与该3种颚口线虫的ITS2目的基因序列相吻合。
2.4 多重PCR扩增结果及其特异性同时用3对引物分别对G. spinigerum、G. doloresi和G. nipponicum的单一DNA模板进行PCR扩增,均扩增出1条目的条带(图3:泳道5~7);对G.spinigerum与G. doloresi、G.spinigerum与G.nipponicum和G.doloresi与G.nipponicum3种不同组合的DNA模板进行PCR扩增,分别扩增出2条目的条带(图3:泳道2~4),而对Hysterothylacium、Anisakis、Echinostoma、SpirometraDNA 均未扩增出相应条带(图3:泳道8~11);对G.spinigerum、G.doloresi和G.nipponicum3种颚口线虫的混合DNA 模板,扩增出3条目的条带(图3:泳道1)。
2.5 敏感性试验结果经不同稀释浓度的DNA模板按上述方法进行PCR扩增,G.doloresi、G.nipponicum、G.spinigerumDNA模板原浓度分别133、125、204 ng/μL。单一PCR对G.doloresi、G.nipponicum和G.spinigerum的最小检出量分别为0.01、0.01、0.2 ng/μL。混合模板多重PCR最小检出量与单一PCR相同(图4)。
2.6 临床虫种的鉴定用建立的多重PCR方法对2条菲律宾、8条印度尼西亚分离到的颚口线虫的DNA模板进行扩增,电泳条带与G.spinigerum阳性对照的条带位置一致,为282 bp;对2条黑龙江分离到的颚口线虫DNA模板进行扩增,电泳条带与G.nipponicum阳性对照条带位置一致,为358 bp(图5)。多重PCR鉴定结果与原样品形态鉴定和分子鉴定结果完全相符。

图2 单一引物PCR特异性Fig.2 Specif city of single PCR

图3 3对引物多重PCR特异性Fig.3 Specif city of multiplex PCR

图4 PCR敏感性试验Fig.4 PCR sensitivity assay

图5 多重PCR方法鉴定样品结果Fig.5 Sample test assay using multiplex PCR method
3 讨论
颚口线虫病流行病学调查和病例报告见于世界各地。国外主要流行于泰国、日本、印度、越南、孟买、柬埔寨、墨西哥等地[5-10]。我国已报道50多例,分布于16个省(市),其中以上海市、福建省及广东省较为多见,虫种以棘颚口线虫为主,刚刺颚口线虫和杜氏颚口线虫病例偶见[11]。FAO(联合国粮农组织)已将寄生虫列为水产品中生物危害因子之首[12],其中包括颚口线虫。随着人们对食品安全的重视,水产品颚口线虫的检疫也需要有快速灵敏的虫种鉴定方法作为辅助工具,本研究选择多重PCR方法直观且简便快速,不需要太多复杂的步骤,便于检疫工作者操作。
在建立多重PCR的检测方法时,引物的设计至关重要,引物的长度、G+C含量、Tm值应尽量一致,且各扩增片段的大小要适宜,既要避免电泳不能分辨,又不能相差过大。在颚口线虫属中,各虫种之间ITS-2基因序列有65%~89%的同源性,差异较大,并且ITS2序列较长,适合用于特异引物的设计,G.spinigerum、G.doloresi和G.nipponicum3种颚口线虫的ITS2同源性为75%~83%,差异较大易于特异引物的设计。本文研究设计的3对引物,GC 含量相近,Tm值相近,这就保证了在相同的退火温度下,G.spinigerum、G.doloresi和G.nipponicum都能得到很好的扩增。其扩增片段分别为282、183、358 bp,这使得在检测时能在同一个电泳胶上进行,为颚口线虫多重PCR方法的成功奠定了基础。
传统上颚口线虫种类的鉴定主要依靠虫体形态学特征,但不同种间幼虫形态学特征区别较小,而同一虫种在不同的中间宿主其大小也有差异,导致区分困难,鉴定费时费力。目前有对颚口线虫CO1与ITS2设计通用引物分析鉴定虫种的报道,但未见设计特异引物进行虫种鉴定的报道。本研究建立的鉴定方法显示,无论是单一PCR还是多重PCR,均能特异性扩增出G.spinigerum、G.doloresi和G.nipponicum条带,单一PCR与多重PCR敏感性一致,且敏感性能够满足对所提虫体DNA浓度的要求,表明建立的方法具有很强的特异性和较高的敏感性。该方法仅通过条带的位置就可以判定所属的虫种,不需要进行测序等繁琐费时的步骤。将该方法运用于样品的鉴定,鉴定结果与虫体形态学与序列分析结果一致[13,14],表明该检测方法的可靠性,证明了本研究建立的多重PCR方法能同时鉴定G.spinigerum、G.doloresi和G.nipponicum3个种,可用于这3种颚口线虫的诊断和种类调查。
Sugiyama等[15]对卫氏并殖吸虫进行了ITS2特异引物研究,目前球虫与锥虫方面也有不少多重PCR检测技术的研究,而在颚口线虫方面本研究是首次对G.spinigerum、G.doloresi、G.nipponicum3种颚口线虫设计特异引物并建立多重PCR鉴定虫种的方法,国内外目前尚未有相关的文献报道。由于条件的限制,本实验室只有这3种颚口线虫,缺乏颚口线虫其他虫种的特异性对比,但本研究结果为今后建立更完善的颚口线虫多重PCR虫种鉴定方法奠定了基石,为检验检疫工作者提供了G.spinigerum、G.doloresi、G.nipponicum3种颚口线虫简单、快速、灵敏的鉴定方法。
[1] Ligon B L. Gnathostomiasis: A review of a previously localized zoonosis now crossing numerous geographical boundaries[J]. Semin Pediatr infect Dis, 2005, 16(2): 137-143.
[2] Nomura Y, Nagakura K, Kagei N,et al.Gnathostomiasis possibly caused by Gnathostoma malaysiae[J]. Tokai J Exp Clin Med, 2000, 25(1):1-6.
[3] Almeyda-Artigas R J, Bargues M D, Mas-Coma S. ITS-2 rDNA sequencing of Gnathostoma species(nematoda) and elucidation of the species causing human Gnathostomasis in the Amercas[J]. J Parasitol, 2000, 86(3): 537-544.
[4] Hashimoto K, Watanabe T, Liu C X,et al.Mitochondrial DNA and nuclear DNA indicate that the Japanese Fasciola species is F. gigantica[J]. Parasitol Res, 1997, 83 (3), 220-225.
[5] Herman J S, Wall E C, Van-tulleken C,et al.Gnathostomiasis acquired by british tourists in botswana[J]. Emerq Infect Dis, 2009, 15(4): 594-597.
[6] Strady C, Dekumyoy P, Clement-riqolet M,et al.Longterm follow-up of imported gnathostomiasis shows frequent treatment failure[J]. Am J Trop Med Hyg, 2009 , 80(1): 33-35.
[7] Hennies F, Jappe U, Kapaun A,et al.Gnathostomiasis import from Laos[J]. J Dtsch Dermatol Ges, 2006, 4(5): 414-416.
[8] Akahane H, Sano M, Kobayashi M. Three cases of human gnathostomiasis caused by Gnathostoma hispidum. With particular reference to the identification of parasitic larvae[J]. Southeast Aaian J Trop Med Pubic Health, 1998, 29(3): 611.
[9] Herman J S, Chiodini P L. Gnathostoma, another emerging imported disease[J]. Clin Microbiol Rev, 2009, 22(3): 484-492.
[10] Lore V, Gluckman S J. Eosinophilic meningitis due to Gnathostoma spinigerum[J]. J Infect, 2002, 45(2): 117-120.
[11] 张鸿满, 黎学铭, 欧阳颐, 等. 广西首次发现颚口线虫病[J]. 应用预防医学, 2008, 14(5): 275-276.
[12] Huss H H, Ababouch L, Gram L. Assessment and management of seafood safety and quality[M]. Rome: FAO, 2003: 60-69.
[13] 李树清, 李雯雯, 陈志飞, 等. 进口黄鳝颚口线虫检疫及虫种鉴定[J]. 中国寄生虫学与寄生虫病杂志, 2011, 29(5): 81-85.
[14] 李雯雯, 李树清, 张子群, 等. 黑龙江与广州颚口线虫幼虫分离株的形态学观察及其分子鉴定[J]. 中国预防兽医学报, 2012, (2): 104-107.
[15] Sugiyama H, Morishima Y, Kameoka Y,et al.Polymerase chain reaction (PCR)-based molecular discrimination between Paragonimus westermani and P. miyazakii at the metacercarial stage[J]. Mol Cell Probe, 2002, 16(3): 231-236.
DEVELOPMENT OF A MULTIPLEX PCR ASSAY TO IDENTIFY GNATHOSTOMA SPINIGERUM, GNATHOSTOA NIPPONICUM AND GNATHOSTOMA DOLORESI
LI Shu-qing1, LI Wen-wen2, ZHANG Hong-man3,CHEN Shao-hong4, LI Jian5, CHEN Zhi-fei1, WANG Qiao-quan1, ZHANG Yong-nian4, HUANG Wei-yi5
(1. Shanghai Entry-Exit Inspection and Quarantine Bureau, Shanghai 200135, China; 2. Department of Animal Lab Center, First Affiliated Hospital, Sun Yat-Sen University, Guangzhou 510080, China; 3. Guangxi Center for Disease Control and Prevention, Nanning 530021, China; 4. Chinese Center for Disease Control and Prevention, National Institute of Parasitic Diseases, Shanghai 200025, China; 5. College of Animal Science and Technology, Guangxi University, Nanning 530005, China)
Three pairs of primers specifc forG.spinigerum(JN408326),G.nipponicum(JN408315) andG.doloresi(JN408329) were designed based on their ITS-2 sequences. Accordingly, single and multiplex PCR methods were developed to identify Gnathostoma species. The amplif ed products in both single and multiplex PCR methods were 282 bp forG.spinigerum,358 bp forG.nipponicumand 183 bp forG.doloresi.The DNA detection limits were 0.2 ng forG.spinigerum,0.01 ng forG.nipponicumand also 0.01 ng forG.doloresi.No PCR products were amplified from genomes ofHysterothylacium, Anisakis, EchinostomaandSpirometra erinaceieuropaei.In addition, 12 species ofGnathostomaisolated from Philippines, Indonesia and Heilongjiang were identif ed in multiplex PCR and the results were in agreement with morphology and sequence analysis. The isolates from Philippines and Indonesia wereG.spinigerumand isolates from Heilongjiang wereG. nipponicum.These data suggested that multiplex PCR methods could be used for identifyingG. spinigerum, G. nipponicumandG. doloresi.
G.spinigerum; G.nipponicum ; G.doloresi;multiplex PCR; identif cation
S852.731
A
1674-6422(2014)06-0038-08
2014-07-11
上海市科委技术标准专项(11dz0503100 );国家质检总局科技专项(2010IK013)
李树清,女,研究员,主要从事进出境动物检疫工作
李雯雯,E-mail:liwenwen.1027@163.com
