通过改进Red重组方法快速构建肠炎沙门菌毒力岛SPI-1缺失株
2014-03-08吕雪莲于申业倪宏波田秋丰刘思国
吕雪莲,于申业,倪宏波,衣 菲,2,田秋丰,2,刘思国
(1.黑龙江八一农垦大学 动物科技学院,黑龙江 大庆 163319;2.中国农业科学院哈尔滨兽医研究所 兽医生物技术国家重点实验室/动物细菌病研究室,黑龙江 哈尔滨 150001)
肠炎沙门菌(Salmonella enterica)是引起人类食源性疾病的主要革兰阴性菌之一,并可以导致禽类、家畜等哺乳动物产生不同的疾病。沙门菌的致病力与其毒力基因密切相关,SPI-1 和SPI-2 是沙门菌的两个重要毒力岛,其中SPI-1 主要编码Ⅲ型分泌系统(T3SS)通过宿主的细胞膜转运肠炎血清蛋白,此外,研究表明SPI-1 能够侵染细胞诱导巨噬细胞产生细胞毒性[1-2]。SPI-1 编码序列约为40 kb,具有一定的稳定性,在自然分离株中极少存在SPI-1 部分或全部缺失的情况[3]。Red 重组是革兰阴性菌最常用的反向遗传操作系统,常规的方法是利用λ 噬菌体Red 重组酶将导入细胞的线性DNA 片段与染色体(或载体)的特定靶序列进行同源重组[4]。本研究室前期基于I-SceI 酶切原理对Red 同源重组系统进行改进,提高了重组效率、缩短了实验周期。本实验利用改进的系统对S.enterica 毒力岛SPI-1 进行一步法敲除,为其生物学特性的研究提供了依据。
1 材料和方法
1.1 菌株及质粒 S.enterica SM6、E.coil DH5α、Red 重组系统工具质粒pKD3、pCP20 及pET-28a 均由中国农业科学院哈尔滨兽医研究所细菌室保存;温敏复制型质粒pKD46a(基于pKD46 质粒骨架,经L-阿拉伯糖诱导后可产生Exo、Gam 和Bet 3 种同源重组酶以及I-SceI 酶)由中国农业科学院哈尔滨兽医研究所细菌室构建。
1.2 主要试剂 L-阿拉伯糖购自Sigma 公司;PCR试剂购自TaKaRa 公司;限制性内切酶、T4 连接酶购自MBI 公司;DNA 胶回收试剂盒和质粒提取试剂盒购自天根生化科技(北京)有限公司。
1.3 引物设计及合成 根据GenBank 中登录的沙门菌毒力岛SPI-1 基因序列(FJ496648.1)设计引物S1/S2 扩增上游同源臂、S3/S4 扩增下游同源臂。氯霉素抗性基因片段的扩增引物为C1/C2,质粒pKD46a 的鉴定引物为P1/P2,毒力岛SPI-1 敲除后的鉴定引物为D1/D2(表1),引物由博仕生物技术有限公司合成。
1.4 S.enterica感受态细胞的制备 将S.enterica 在LB 液体培养基中过夜培养,按1∶100 转接到100 mL LB 培养基,培养至OD600nm约为0.4 时,冰浴预冷10 min~30 min。先后以预冷的无菌水和10 %甘油各离心洗涤2 次,将菌体悬浮于600 μL 10 %甘油中,分装后-80 ℃冻存。

表1 PCR 引物序列Table 1 Primers used in this study
1.5 打靶质粒的构建 以质粒pKD3 为模板,扩增氯霉素抗性片段;以SM6 为模板,扩增毒力岛SPI-1 上游和下游同源臂,PCR 产物纯化经相应酶切后胶回收。氯霉素抗性片段与载体pET-28a 连接,转化到E.coil DH5α 中,PCR 与测序鉴定正确后,分别在氯霉素抗性片段的上下游连接SPI-1 上下游同源臂,重组质粒命名为pET-SPI-1FCmR。
1.6 电转化以及Red重组系统的诱导 取质粒pKD46a 和pET-SPI-1FCmR 各1.5 μL 加入S.enterica感受态细胞中混匀,冰浴10 min,进行电转化。转化菌涂布于含氨苄青霉素(100 μg/mL)、卡那霉素(50 μg/mL)和氯霉素(30 μg/mL)的LB 平板上,30 ℃培养24 h。挑取单菌落进行PCR 鉴定,获得共转化重组菌。
将菌液转接于含有氨苄和氯霉素的LB 培养基中,30 ℃220 r/min 培养至OD600nm约为0.3,加入L-阿拉伯糖至终浓度30 mmol/L,30 ℃180 r/min 诱导1 h,30 ℃静置培养7 h~11 h,涂布含有氨苄青霉素和氯霉素的LB 平板,30 ℃静置培养12 h 后挑取单克隆进行PCR 鉴定。阳性菌株命名为SM6ΔSPI-1::cat。
1.7 氯霉素抗性基因的敲除 按1.4 方法制备SM6ΔSPI-1::cat 感受态细胞,将pCP20 质粒转入其中,涂布于含氨苄青霉素的LB 平板,30 ℃静置培养24 h,挑菌鉴定正确后,转接到无抗性的LB 培养基中,42 ℃220 r/min 诱导15 h~16 h 使FLP 重组酶表达。生长的菌落分别点到含氨苄青霉素的平板和氯霉素的平板上,两个平板对应的位置均不长菌,利用鉴定引物D1/D2 进行PCR 鉴定,正确后的菌株命名为SM6ΔSPI-1。
1.8 体外遗传稳定性的检测 挑取单个菌落接种于LB 培养基中,37 ℃220 r/min 培养至对数期,按1∶100 转接到新鲜的LB 培养基中,连续传代,取第5、10、15、20、25 及30 代菌液利用鉴定引物进行PCR 鉴定,检测缺失菌株的遗传稳定性。
1.9 生长速率的比较 为对比缺失株SM6ΔSPI-1与野生株SM6 的生长状态有无差异,分别挑取二者的单菌落接种于LB 培养基中,37 ℃220 r/min 过夜培养,将其按1∶100 比例分别转接到5 mL LB 培养基中,调整初使菌液浓度(OD600nm约为0.05),37 ℃220 r/min 培养,每隔1 h 取200 μL 菌液测定OD600nm值,直至细菌生长处于稳定期,重复试验3 次,绘制SM6ΔSPI-1 和SM6 的生长曲线。
1.10 统计分析 所有数据采用SPSS17.0 软件进行统计分析。
2 结果
2.1 缺失株SM6ΔSPI-1的构建与鉴定
2.1.1 打靶质粒pET-SPI-1FCmR 的构建 将氯霉素抗性片段、SPI-1 上游和下游同源臂分别连入pET-28a 载体构建重组质粒pET-SPI-1FCmR,并以T7/T7ter 为引物鉴定重组质粒,结果显示,PCR 产物约2 500 bp(图1),测序结果与预期2 478 bp 一致。

图1 重组质粒pET-SPI-1FCmR 的PCR 鉴定Fig.1 PCR product identified recombinant plasmid of pET-SPI-1FCmR
2.1.2 质粒的共转化 将pET-SPI-1FCmR 和质粒pKD46a 共转入S.enterica 感受态细胞中,以引物P1/P2 进行PCR 鉴定,结果显示扩增产物约500 bp,与预期相符(图2)。
2.1.3 氯霉素抗性基因的敲除与PCR 鉴定 将pCP20 质粒转入阳性菌株中,热诱导消除氯霉素抗性基因,构建缺失菌SM6ΔSPI-1,以SM6ΔSPI-1 和SM6ΔSPI-1::cat 为模板,利用引物D1/D2 和T7/T7ter进行PCR 鉴定,结果显示扩增产物分别约为1 500 bp和2 500 bp,大小与预期相符,表明氯霉素基因已经从SM6ΔSPI-1::cat 中敲除,野生株SM6 毒力岛SPI-1 基因序列约为40 kb,片段过长,因此PCR 扩增结果为阴性(图3)。

图2 pKD46a 转化到Salmonella 的PCR 鉴定Fig.2 Identification of the plasmid pKD46a in transformed Salmonella by PCR

图3 缺失株SM6ΔSPI-1 的PCR 鉴定Fig.3 Identification of the SM6ΔSPI-1 by PCR
2.2 S.enterica毒力岛SPI-1缺失株的体外遗传稳定性 对S.enterica 缺失株进行体外连续传代,取第5、10、15、20、25、30 代单菌落利用引物D1/D2进行PCR 鉴定,结果显示未发生回复突变,表明缺失菌株能够在体外稳定遗传(图4)。
2.3 SM6与SM6ΔSPI-1生长速率的比较 在相同条件下培养SM6 和SM6ΔSPI-1,并且每间隔1 h测一次OD600nm值,绘制生长速率曲线。结果显示SM6ΔSPI-1 在体外正常生长并且SM6ΔSPI-1 生长速率高于SM6,SM6ΔSPI-1 与SM6 在6 h~9 h 生长速率相比差异显著(p<0.05)(图5)。
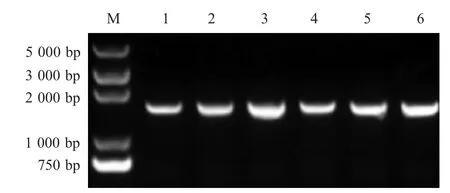
图4 SM6ΔSPI-1 遗传稳定性PCR 鉴定结果Fig.4 Stability test of the SM6ΔSPI-1 by PCR
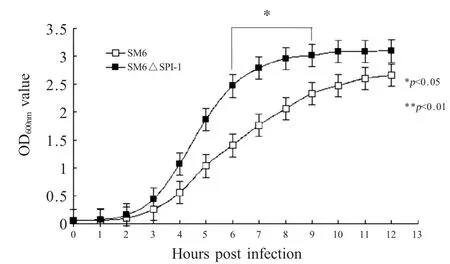
图5 缺失菌株与野生菌株生长速率的比较Fig.5 Growth rate of mutant strain SM6ΔSPI-1 and the wild stain SM6
3 讨论
基因敲除是研究微生物蛋白功能重要的方法。细菌基因敲除有多种策略,包括非复制型质粒载体敲除法、不稳定型质粒载体敲除法、温度敏感型质粒载体敲除法、线形转化敲除法和结合转化敲除法[5]。同源重组是目前最常用的细菌基因突变方法,Red重组技术应用于沙门菌染色体DNA、大肠杆菌染色体DNA、细菌人工染色体基因组DNA 中,Red 重组技术也可以精确修饰不同大小的DNA 分子[6-9]。
由于毒力岛SPI-1 编码的碱基长达40 kb,应用的常规Red 重组方法难以将S.enterica 毒力岛SPI-1敲除。本实验室前期基于I-SceI 酶切原理对pKD46质粒进行改造,构建pKD46a。本研究利用这种改进的Red 重组系统,通过一步法构建了SM6ΔSPI-1,不需要再将毒力岛基因进行逐段敲除。改进的Red系统与常规方法相比具有快速、准确、高效的优势。
另外,SM6ΔSPI-1 遗传稳定性试验显示,SM6ΔSPI-1 具有良好的遗传稳定性。SM6ΔSPI-1 的生长速率较SM6 快,表明毒力岛SPI-1 的缺失可能减少SM6 生长负担,推测SPI-1 中某些组分的合成导致沙门菌生长变缓。本实验还在敲除毒力岛SPI-1过程中检测了几代菌液制成感受态细胞,结果显示传10 代后的菌液更利于质粒pCP20 的导入。表明菌液里的pKD46a 质粒影响质粒pCP20 的转入,传代次数增多使质粒pKD46a 自然丢失。总之,S.enterica 毒力岛SPI-1 的敲除为其后续功能研究奠定了基础,为制备基因工程减毒疫苗提供一种快捷的缺失方法。
[1]Kaniga K,Trollinger D,Galan J E.Identification of two targets of the type III protein secretion system encoded by the inv and spa loci of Salmonella typhimurium that have homology to the Shigella IpaD and IpaA proteins[J].J Bacteriol,1995,177(24):7078-7085.
[2]Chen L M,Kaniga K,Galan J E.Salmonella spp.are cytotoxic for cultured macrophages[J].Mol Microbiol,1996,21(5):1101-1115.
[3]Ginocchio C C,Rahn K,Clarke R C,et al.Naturally occurring deletions in the centisome 63 pathogenicity island of environmental isolates of salmonella spp[J].J Infect Immun,1997,65(4):1267-1272.
[4]Datsenko K A,Wanner B L.One-step inactivation of chromosomal genes in Escherichia coli K12 using PCR products[J].Proc Natl Acad Sci USA,2000,97(12):6640-6645.
[5]谢承佳,何冰芳,李霜.基因敲除技术及其在微生物代谢工程方面的应用[J].生物加工过程,2007,(3):10-14.
[6]孟霞,孟宪臣,朱春红,等.肠炎沙门菌hfp 缺失株的构建和对flic 基因的调控分析[J].中国预防兽医学报,2013,35(9):720-724.
[7]崔文禹,李山虎,姜飞,等.应用Red 重组工程技术建立asd 基因缺失的大肠杆菌DH10B 菌株[J].生物技术通讯,2006,17(4):493-495.
[8]Muyrers J P,Zhang You-ming,Testa G,et al.Rapid modification of bacterial artificial chromosomes by ET recombination[J].Nucleic Acids Res,1999,27(6):1555-1557.
[9]杨建岭,顾淑萍,陈臣,等.用Red/ET 重组酶构建基因打靶载体[J].生物工程学报,2006,22(6):919-924.
