忽地笑加兰他敏生物合成代谢途径中COMT酶基因克隆及序列分析
2013-11-26桂柳姿崔培梧潘清平唐金凤鲁耀邦
桂柳姿,崔培梧,潘清平,唐金凤,鲁耀邦
(1.湖南中医药大学,湖南 长沙410208;2.湖南省中药现代化研究重点实验室,湖南 长沙410208;3.广东医学院,广东;4.中药药性与药效三级科研实验室,湖南 长沙410208)
忽地笑(Lycoris aurea),又名黄花石蒜,是石蒜科(Amaryllidaceae)石蒜属(Lycoris Herb.)多年生草本植物,其在生长繁殖过程中会产生多种生物碱,其中加兰他敏(Galanthamine)是一种苄基乙胺类生物碱[1],属乙酰胆碱酯酶抑制剂[2],为治疗阿尔茨海默氏病(Alzheimer’s Disease,AD)的首选药物之一。由于化学合成加兰他敏成本高昂,目前药用加兰他敏主要从石蒜科植物的鳞茎中提取分离得到[3]。在本课题组的前期研究中,比较了黄花石蒜花蕊和花葶组织中总RNA 的提取方法[4],以及使用以RNA 差异显示法筛选黄花石蒜中加兰他敏合成相关基因[5]。
目前认为加兰他敏在植物体内的生物合成途径主要分为六个步骤,首先是原儿茶醛与酪胺(tyramine)合成降孤挺花啶(norbelladine),然后降孤挺花啶在儿茶酚氧位甲基转移酶 (catechol-O-methytransferase,COMT)的催化下接受一个甲基基团转变为4-氧-甲基降孤挺花啶,而4-氧-甲基降孤挺花啶是加兰他敏、文殊兰胺(crinine)、石蒜碱(lycorine)等石蒜科生物碱的共同前体[3]。COMT 是一种S-腺苷-L-甲硫胺酸(SAM)依赖的甲基转移酶,目前在烟草(Nicotiana tabacum)[6]、罂粟(Papaver somniferum)、唐松草(Thalictrum tuberosum)[7]和玉米(Zea mays)[8]等多种植物中被克隆。而本实验首次从忽地笑克隆COMT 酶基因并对其编码的蛋白质进行了生物信息学分析。
1 材料与方法
1.1 材料
1.1.1 实验材料 忽地笑花瓣于2012年7月10日采自湖南衡山,经湖南中医药大学中药鉴定学教研室潘清平教授鉴定后于-80 ℃保存。
1.1.2 主要试剂 RNAprep pure 植物总RNA 提取试剂盒(TIANGEN,北京),SuperScriptTMIII Firststrand Synthesis System (Invitrogen,美国)、3’Full RACE Core set Ver.2.0、5’ Full RACE kit、pMD19-T Vector、Primer STARRHS DNA Polymerase、LA Taq、A-Tailing 试剂盒、Eoli.DH5α 感受态细胞(大连宝生物公司),QIAquick Gel Extraction Kit (QIAGEN,德国),BIOWEST AGAROSE(Genetech,美国),100 bp DNA ladder Marker(北京鼎国生物技术有限公司)。
1.1.3 仪器 5332 PCR 仪、Biophotometer 6131 核酸蛋白分析仪(Eppendorf,德国),HE-120 Gen 多功能水平电泳槽、Tanon Eps300 型恒压恒流电泳仪、GZS-1000B 凝胶图像处理系统 (上海天能科技有限公司),恒温震荡培养箱(太仓市华美生化仪器厂),洁净工作台(上海博迅实业有限公司医疗设备厂),MV-100 微型真空离心浓缩仪 (日本TOMY 公司),TGL-18M 型台式高速冷冻离心机(湖南赛特湘仪高速离心机有限公司),超纯水系统(北京帕思特科技有限公司),THA-3560C 高压灭菌锅(造鑫企业有限公司)。
1.2 方法
1.2.1 忽地笑花瓣总RNA 提取 从-80 ℃取出忽地笑花瓣,用植物总RNA 提取试剂盒提取其总RNA,具体操作按照说明书进行。提取后,用1.0%琼脂糖凝胶电泳检测总RNA 的完整性,并用核酸蛋白仪检测其浓度,剩余量存于-80 ℃中。
1.2.2 忽地笑花瓣cDNA 第一链的合成 以总RNA 为模板,参照逆转录试剂盒说明书合成cDNA第一链,合成后存于-20 ℃。
1.2.3 忽地笑COMT 基因部分序列的克隆 参照NCBI 中已发表的高等植物的COMT 氨基酸序列同源区,利用Primer Premier5 软件设计1 对简并引物LaCOMT-R 和LaCOMT-F(表1)进行PCR 扩增。PCR 扩增条件:94 ℃预变性5 min;94 ℃ 30 s,53 ℃1 min,72 ℃1 min,35 个循环数;72 ℃后延伸10 min;4 ℃保存。扩增产物用1.5%琼脂糖凝胶电泳检测,回收片段,连接转化,蓝白筛选,再取白色菌落送交至长沙博尚公司测序。
1.2.4 3’RACE 和5’RACE 扩增根据已获得的COMT 基因的部分序列,利用Primer Premier5 软件及Oligo 6.0 软件设计3’RACE 和5’RACE 的特异性引物3R1、3R2、5R1 和5R2(表1),其分别与试剂盒提供的3’RACE Out Primer、3’RACE Inner Primer、5’ RACE Out Primer 和 5’RACE Inner Primer 进行PCR 扩增(表1)。3’ RACE Out PCR 扩增条件:94 ℃预变性5 min;94 ℃30 s,53 ℃30 s,72 ℃1 min,35 个循环数;72 ℃后延伸10 min;4 ℃保存。3’RACE Inner PCR 扩增条件:94 ℃预变性5 min;94 ℃30 s,53 ℃50 s,72 ℃1 min,30个循环数;72 ℃后延伸10 min;4 ℃保存。5’RACE Out PCR 扩增条件:94 ℃预变性3 min;94 ℃30 s,55 ℃50 s,72 ℃1 min,25 个循环数;72 ℃后延伸10 min;4 ℃保存。5’RACE Outer PCR 扩增条件:98 ℃预变性3 min;98 ℃10 s,55.2 ℃15 s,72 ℃90 s,20 个循环数;72 ℃后延伸10 min;4 ℃保存。5’RACE Inner PCR 扩增条件:94 ℃预变性3 min;94 ℃30 s,57 ℃30 s,72 ℃90 s,30 个循环数;72 ℃后延伸10 min;4 ℃保存。扩增产物用1.5%琼脂糖凝胶电泳检测,回收片段,连接转化,蓝白筛选,再取白色菌落送交至上海生工测序。
1.2.5 忽地笑COMT 基因全长的获得 使用序列拼接软件ContigExpress 对测序获得的3’RACE 与5’RACE 片段进行拼接,得到COMT 基因的全长序列。

表1 忽地笑COMT 基因PCR 扩增引物
1.2.6 忽地笑COMT 基因序列的生物信息学分析
(1) 核苷酸性质分析 使用NCBI 提供的在线ORF Finder 寻找基因的编码框;并使用BLAST搜索克隆的LaCOMT 氨基酸序列的同源序列,分析其的同源性。
(2) 蛋白质分析 用ProtParam 软件(http://www.expasy.ch/tools/protparam.html)在线分析La-COMT 基因编码蛋白质的分子量,等电点和不稳定系数等;用ProtScale 软件(http://www.expasy.ch/tools/protscale.html)在线分析蛋白质的亲/疏水性;用SignalP 软件(http://www.cbs.dtu.dk/services/SignalP/)在线检测蛋白质信号肽;用TMHMM 软件(http://www.cbs.dtu.dk/services/TMHMM) 在线检测蛋白质跨膜结构;二级结构使用SOPMA 软件(http://npsapbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/nps a_sopma.html) 在线分析;三级结构使用SWISSMODEL 软件(http://swissmodel.expasy.org/)在线自动建模。
(3) 基于COMT 氨基酸序列的物种进化分析使用DNAMAN 软件将NCBI 中不同物种的13 种与LaCOMT 氨基酸序列具有一定同源性的序列进行比对,并用Observed Divergency 的距离模式将其集合在一起绘制系统发育树。
2 结果与分析
2.1 目的基因的克隆
用简并引物LaCOMT-R 和LaCOMT-F 对忽地笑cDNA 第一链进行PCR 扩增,得到1 段492 bp碱基序列(图1A)。根据这条序列为3’ RACE 与5’RACE 的巢式PCR 设计特异性引物,最后得到La-COMT 基因的3’端片段约600 bp(图1B)和5’端片段约850 bp (图1C)。通过序列拼接软件ContigExpress 对3’ 端片段与5’ 端片段进行拼接,得到La-COMT 基因的全长序列约1300 bp,再通过NCBI 网站上的ORF finder 在线软件分析LaCOMT 基因的开放读码框,结果显示LaCOMT 基因序列有12 bp 的polyA 结构和1101 bp 编码的366 aa 的开放阅读框。
2.2 儿茶酚氧位甲基转移酶基因系统进化分析
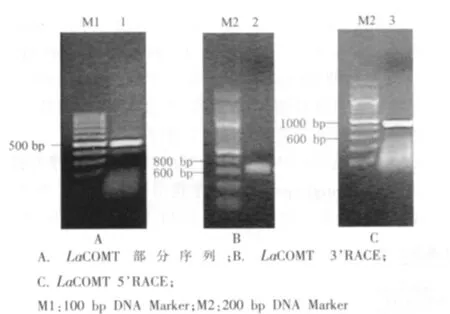
图1 LaCOMT 的PCR 扩增
在NCBI 网站上通过BLAST 比对出LaCOMT 基因编码的氨基酸序列,发现其不仅与其他植物的氧位甲基转移酶(COMT)具有一定的同源性,而且与其他植物的咖啡酸氧位甲基转移酶(CAOMT)也具有较高的同源性。使用DNAMAN 软件对其进行系统发育树的构建,发现忽地笑COMT 与荷兰鸢尾(Iris x hollandica)CAOMT 的同源性最高,与罂粟(Papaver somniferum)、唐 松 草(Thalictrum tuberosum)和烟草(Nicotiana tabacum)COMT 的同源性次之,而玉米(Zea mays)COMT 与蜀黍(Sorghum bicolor)、苇状羊茅 (Festuca arundinacea) 和黑麦草(Lolium perenne)CAOMT 聚为一类 (图2)。由于COMT 与CAOMT 均属于依赖S-腺苷-L-甲硫胺酸(SAM) 的氧位甲基转移酶,通过上述分析可推断COMT 与CAOMT 在进化过程中具有一定的保守性,其进化来源也可能具有一定的同源性。
2.3 LaCOMT 编码蛋白序列与其他植物COMT 同源性比对分析
通过上述的系统发育进化分析后,再进一步分析LaCOMT 与其他植物COMT 同源性,发现其与忽地笑、唐松草、烟草和玉米的同源性高达74%,且具有依赖SAM 的甲基转移酶蛋白的三个保守区:(V/I/L)VDVGGGXG、VPX (A/P/G)DAXXMKWI 和 AL PXXGKVIXXEXILP(图3)。

图2 基于忽地笑儿茶酚氧位甲基转移酶基因的不同物种的基因进化树

图3 LaCOMT 氨基酸序列与其他植物儿茶酚氧位甲基转移酶比对结果
2.4 忽地笑儿茶酚氧位甲基转移酶蛋白特性分析
2.4.1 LaCOMT 蛋 白 理 化 性 质 分 析 根 据Prot-Param 在线软件预测表明,LaCOMT 蛋白分子式为C1824H2861N467O527S23,分子量为40501.9;等电点pI 为5.95;带负电荷氨基酸残基(Asp+Glu)为42,带正电荷氨基酸残基(Arg+Lys)为36;稳定系数为38.13,该值在40 以下,说明LaCOMT 蛋白单体是稳定的;脂肪系数为90.63;总平均亲水性为-0.037,同时结合ProtScale 在线软件预测LaCOMT 蛋白的亲/疏水性氨基酸分布图(图4)表明,亲水性氨基酸和疏水性氨基酸较均匀的分布于整个氨基酸序列中,故此蛋白单体没有明显的疏水性;估计体外半衰期为30h。含量较高的氨基酸依次为Leu (9.6% )、Ala(7.9%)、Lys (7.7%)、Ile (7.4%)、Gly (7.1%)、Ser(6.8%)、Asp(5.7%)、Glu(5.7%)、Val(5.7%)和Pro(5.5%)。
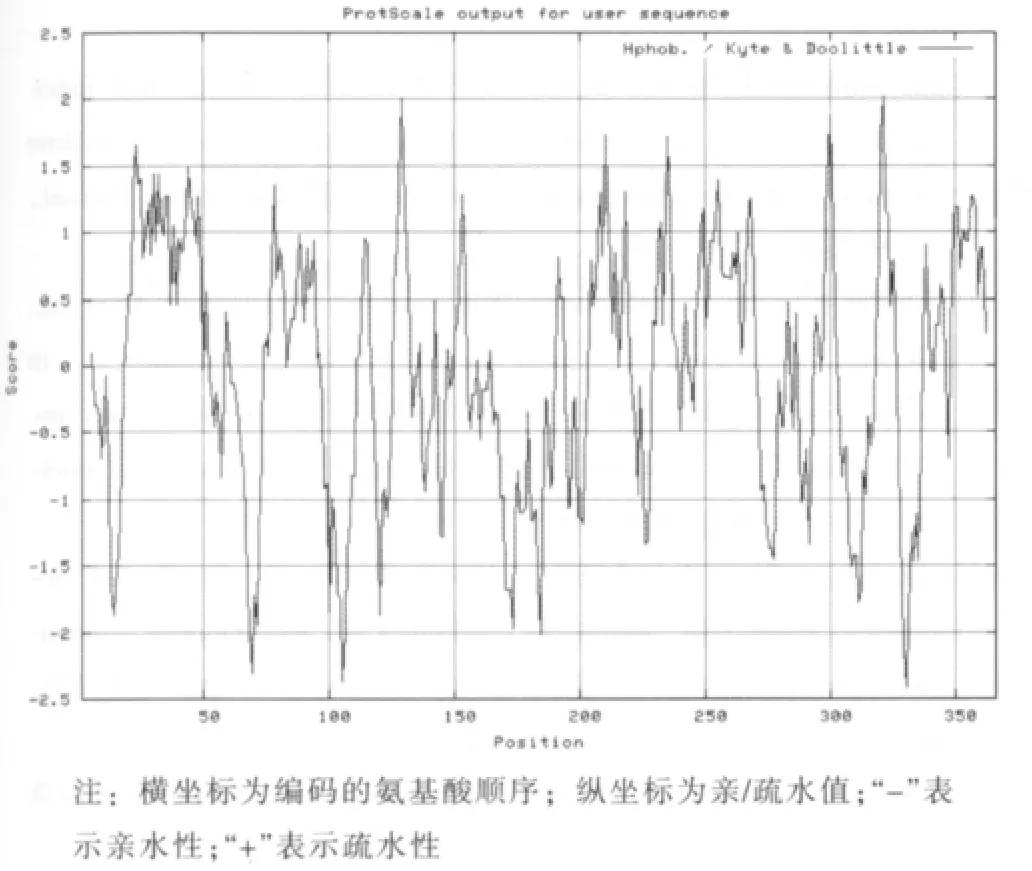
图4 LaCOMT 蛋白疏水性分析图
2.4.2 LaCOMT 蛋白氨基酸序列信号肽及跨膜结构域的预测和分析 用SignalP 在线软件对LaCOMT蛋白的N 端的前70 bp 氨基酸片段进行的信号肽检测表明,其C 值、S 值和Y 值的计算结果均为NO,且平均值少于0.5,预测出此蛋白无信号肽(图5),推断LaCOMT 蛋白为非分泌蛋白。用TMHMM在线软件分析蛋白的跨膜结构域,预测出此蛋白无跨膜区,推断LaCOMT 酶发挥功能的部位不结合于生物膜上。

图5 LaCOMT 蛋白信号肽分析图
2.4.3 LaCOMT 蛋白二级结构预测 使用SOPMA在线软件预测LaCOMT 蛋白二级结构,结果表明,有168 个氨基酸可形成α 螺旋,54 个氨基酸可形成延伸带,23 个氨基酸可用于形成β 转角,其余121 个氨基酸可形成无规卷曲。
2.4.4 LaCOMT 蛋白三级结构预测 使用SWISSMODEL 预测LaCOMT 蛋白三级结构,采用自动模式建模,预测出LaCOMT 基因编码的蛋白对应1kyzE 模型(图6A)的E 值为4.05e-141,序列的一致性为67.61%,1kyzE 是植物中咖啡酸氧位甲基转移酶的模板,LaCOMT 亦具有甲基转移酶的功能,说明以1kyzE 模型为样板预测的LaCOMT 单体三级结构图是可靠的(图6B)。
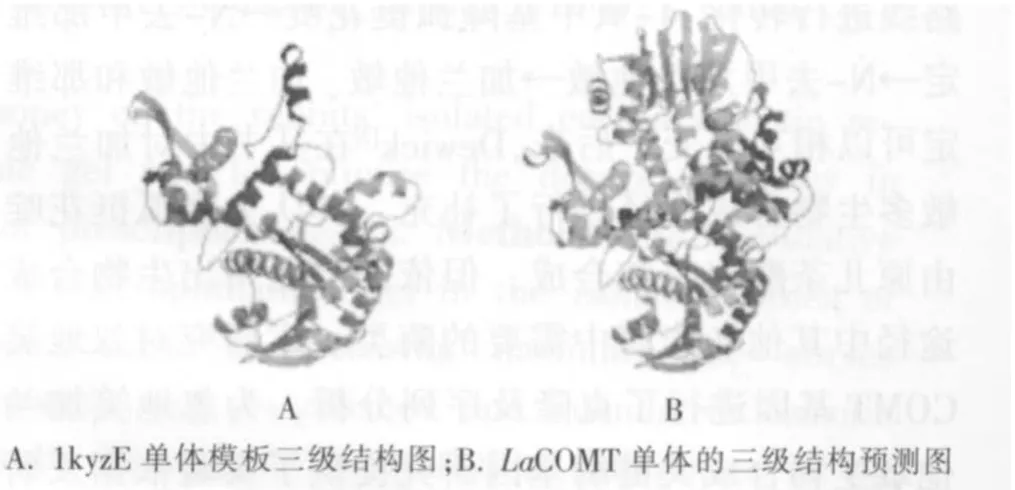
图6 忽地笑儿茶酚氧位甲基转移酶的三级结构预测图
3 讨论
本实验获得忽地笑儿茶酚氧位甲基转移酶(LaCOMT)基因序列后,通过分析其开放阅读区编码的氨基酸序列发现其具有依赖S-腺苷甲硫胺酸(SAM)的甲基转移酶蛋白的三个保守区[9]:(V/I/L)VDVGGGXG、VPX (A/P/G)DAXXMKWI 和AL PXXGKVIXXEXILP,SAM 的作用是在甲基转移酶的作用下为底物提供甲基,故此保守序列可初步说明此次实验所克隆得到的序列为COMT 基因序列。同时对COMT 进行同源性比对发现LaCOMT 与来源于烟草、唐松草、玉米和罂粟的COMT 的同源性高达74%,这进一步说明此次实验所得的序列为COMT 序列。本实验克隆的LaCOMT 基因读码框共1101bp,共编码366 个氨基酸,与梁丽建等[10]研究的石蒜COMT(LrCOMT)基因编码的氨基酸数目一致,但氨基酸序列有所不同。
LaCOMT 基因编码的氨基酸序列的理化性质和二级结构分析结果表明该基因编码的蛋白质稳定,为非分泌性蛋白,其酶促作用的发生不结合生物膜。COMT 进行系统发育树构建以及LaCOMT 蛋白三级结构建模结果表明COMT 与咖啡酸氧位甲基转移酶(CAOMT)具有较高的同源性,各自作用于不同底物,分别为儿茶酚胺类化合物和5-羟基松伯醇[11],并且CAOMT 也是依赖SAM 的一种甲基转移酶,可初步推断COMT 与CAOMT 在进化可能具有同一祖先且COMT 在进化上具有相当的保守性。
Eichhom 等[12]以Leucojum Amaestivum 为 材 料通过放射性和重同位素标记生物合成的可能前体,研究加兰他敏的生物合成途径,推测出加兰他敏的生物合成的可能途径主要有五个步骤,首先确定了降孤挺花啶在儿茶酚氧位甲基转移酶的作用下获得一个甲基转变为4-氧甲基降孤挺花啶,再按照以下路线进行转化:4-氧甲基降孤挺花啶→N-去甲那维定→N-去甲加兰他敏→加兰他敏。加兰他敏和那维定可以相互转变。后来,Dewick[3]在其书中对加兰他敏多生物合成途径进行了补充,其认为降孤挺花啶由原儿茶醛与酪胺合成,但依旧没能指出生物合成途径中其他各步骤中需要的酶类。本研究对忽地笑COMT 基因进行了克隆及序列分析,为忽地笑加兰他敏生物合成关键酶基因研究提供了实验依据及研究思路。但忽地笑加兰他敏生物合成途径的其它酶促反应机制有待进一步研究。
[1]郑 颖,刘 鑫,丁立生.苄基苯乙胺类生物碱的植物化学研究进展[J].天然产物研究与开发,2009(21):171-176.
[2]Ping Jia,Rong Sheng,Jing Zhang,et al..Design,synthesis and evaluation of galanthamine derivatives as acetylcholinesterase inhibitors [J].European Journal of Medicinal Chemistry,2009,44(2):772-784.
[3]Paul M,Dewick.Medicinal nature products: a biosynthetic approach (2nd ediction)[M].Antony Rowe Lid,Chippenham,Wilts,2002:345-346.
[4]唐金凤,鲁耀邦,桂柳姿.黄花石蒜花蕊和花葶组织中总RNA 提取方法的比较[J].湖南中医药大学学报,2012,32(1):31-34.
[5]唐金凤,鲁耀邦,桂柳姿.mRNA 差异显示法筛选黄花石蒜中加兰他敏合成相关基因[J].湖南中医药大学学报,2012,32(7):34-36.
[6]Jaeck E,Martz F,Stiefel V,Fritig B.Legrand M.Expression of class I O-methyltransferase in healthy and TMV-infected tobacco[J].Molecular Plant-Microbe Interactions.1996,9(8): 681-688.
[7]Susanne Frick,Toni M,Kutchan.Molecular cloning and functional expression of O-methyltransferases common to isoquinoline alkaloid and phenylpropanoid biosynthesis [J].The Plant Journal,1999,17(4): 329-339.
[8]Guillet-Claude C,Birolleau-Touchard C,Manicacci D,et al..Genetic diversity associated with variation in silage corn digestibility for three O-methyltransferase genes involved in lignin biosynthesis[J].Theoretical and Applied Genetics,2004,110(1): 126-135.
[9]Chandrashekhar P,Joshi,Vincent L,Chiang.Conserved sequence motifs in plant S -adenosyl -L -methionine -dependent methyltransferases [J].Plant Molecular Biology,1998(37):663-674.
[10]梁丽建,江玉梅,夏 冰,等.石蒜儿茶酚氧位甲基转移酶基因克隆与原核表达[J].西北植物学报,2012,32(1):23-28.
[11]田 铃,嵇保中,刘曙文,等.甲基转移酶的功能和分类[J].生命的化学,2007,27(5):425-427.
[12]Jorg Eichhom,Takeshi Takada,Yasuyuki Kita,et al..Biosynthesis Of the amaryllidaceae alkaloid galanthamine[J].Phytochemistry,1998,49(4):1 037-1 047.
