细胞酶联反应法 (cell-ELA) 测定特异结合U87-EGFRvⅢ细胞的适配子的亲和力
2013-09-03谭燕梁惠玉伍锡栋高宇博张兴梅
谭燕,梁惠玉,伍锡栋,高宇博,张兴梅
1 南方医科大学基础医学院 神经生物学教研室,广东 广州 5105152 南方医科大学第一临床医学院,广东 广州 510515
细胞酶联反应法 (cell-ELA) 测定特异结合U87-EGFRvⅢ细胞的适配子的亲和力
谭燕1,梁惠玉1,伍锡栋1,高宇博2,张兴梅1
1 南方医科大学基础医学院 神经生物学教研室,广东 广州 510515
2 南方医科大学第一临床医学院,广东 广州 510515
谭燕, 梁惠玉, 伍锡栋, 等. 细胞酶联反应法 (cell-ELA) 测定特异结合 U87-EGFRvⅢ细胞的适配子的亲和力. 生物工程学报, 2013, 29(5): 664−671.
Tan Y, Liang HY, Wu XD, et al. Cell-ELA-based determination of binding affinity of DNA aptamer against U87-EGFRvIII cell. Chin J Biotech, 2013, 29(5): 664−671.
通过细胞-指数级富集的配基系统进化 (Cell based systematic evolution of ligands by exponential enrichment,cell-SELEX) 方法从随机文库中筛选得到与稳定过表达人表皮生长因子受体Ⅲ型突变体(Epidermal growth factor receptor variant Ⅲ,EGFRvⅢ) 的胶质瘤细胞U87细胞株 (U87-EGFRvⅢ) 特异结合的DNA适配子。以U87-EGFRvⅢ细胞作为检测对象,筛选到的适配子A15作为检测分子,建立一种细胞酶联反应 (Cell enzyme-linked assay,cell-ELA) 方法测定适配子的亲和力,并用EGFR抗体作为对照来比较此种DNA适配子的亲和力。结果显示,所测定的 DNA适配子 A15的解离平衡常数 (Equilibrium dissociation constants,Kd) 小于100 nmol/L,对U87-EGFRvⅢ细胞的结合能力与抗体相似。Cell-ELA法可较方便地用于cell-SELEX中适配子亲和力的测定。
细胞酶联反应法,细胞-指数级富集的配基系统进化,DNA适配子,解离平衡常数
适配子是一类与靶分子特异结合的能形成稳定空间结构的寡核苷酸分子。人工筛选得到的适配子有许多优于抗体的特点[1-4]:1) 通过各种SELEX技术获得,方法简单可行,并能在体外大量合成;2) 与靶分子具有较高的亲和力,解离平衡常数Kd值在pmol/L~nmol/L范围内;3) 免疫原性低;4) 易合成、保存和修饰,因此可应用于疾病的诊断和治疗。经过 20余年的研究,SELEX技术得到了改进和发展。近年来,cell-SELEX技术发展迅速,用活细胞或者活细胞上的某些生物分子作为靶标,在生理状态下筛选到的适配子更有意义,这将为肿瘤细胞表面特异生物标志物的筛选提供平台。
在 cell-SELEX筛选中需要用合适的方法来鉴定适配子是否富集;富集得到的适配子也需要合适的方法来测定其与细胞的亲和力。本研究利用经典ELISA的方法和生物素-链亲和素系统,建立并进行 cell-ELA实验来定量鉴定适配子与靶细胞的亲和力,为 cell-SELEX筛选得到的适配子的亲和力检测提供了一种简便方法。
1 材料与方法
1.1 材料
HEK293、U87以及稳定过表达 EGFRvⅢ的 U87-EGFRvⅢ细胞株由本实验室保存,SH-SY5Y细胞株从ATCC (美国模式培养物集存库) 购买。PCR用的DNATaq酶购自TaKaRa公司;DNA文库、测序及探针标记引物由Invitrogen公司合成;辣根过氧化物酶标记的链亲和素 (1∶1 000) 购自Sigma公司,抗EGFR蛋白抗体 (兔多克隆抗体) 购自Santa Cruz公司;其他试剂为国产分析纯。酶标仪为 VICTOR™X3 Multilabel Plate Reader,购自 PerkinElmer公司;Mastercycler梯度PCR仪器为Eppendorf公司产品;荧光显微镜型号为Olympus MX7600。细胞培养瓶购自Corning公司;96孔酶标板购自Costar公司。
1.2 方法
1.2.1 生物素或FITC标记的DNA适配子A15的制备

图1 适配子A15的筛选过程[7] (DNA文库与对照细胞 (红色) 孵育后,将与之不结合的 DNA (灰色) 再与靶细胞 (绿色) 孵育,得到与靶细胞结合的 DNA(黄色),通过不对称PCR大量扩增获得DNA单链,即为亚库,接着进行下一轮筛选)Fig. 1 The procedure of cell-SELEX[7]. In cell-SELEX,a DNA library is first incubated with a non-target cell(shown in red) in a counter-selection step. Non-specific membrane proteins are shown in grey. Unbound aptamers are then recovered and incubated with the target cell (shown in green), which may express either a particular target (specific target membrane proteins shown in color) or target cell type. Bound aptamers(yellow) are recovered by phenol extraction and amplified via asymmetrical PCR.
通过cell-SELEX的方法 (图1) 从随机DNA文库中筛选到能与 U87-EGFRvⅢ细胞特异结合的DNA适配子A15,其中筛选过程中引入负筛细胞 U87 (EGFRvⅢ表达阴性) 细胞作为对照,以去除细胞表面相同蛋白分子的干扰。相应的DNA文库序列为5'-GCAATGGTACGGTACTTCC(30N)CAAAAGTGCACGCTACTTTGCTAA-3'[5]。得到的 A15适配子通过不对称 PCR法[6]用相应的标记引物扩增并切胶回收,通过乙醇沉淀法得到DNA单链。
1.2.2 A15特异识别U87-EGFRvⅢ细胞
HEK293、SH-SY5Y、U87和U87-EGFRvⅢ细胞在 48孔板中培养过夜,用洗涤缓冲液(0.45%葡萄糖-5 mmol/L氯化镁-PBS) 漂洗一次。将 200 pmol的 FITC标记的 A15加入到500 μL结合缓冲液 (0.01% BSA-0.45%葡萄糖-5 mmol/L氯化镁-PBS),95 ℃加热 5 min,冰上放置10 min后加入培养板中。放置在5% CO2、37 ℃孵箱中孵育1 h,每隔5 min振摇一次。孵育完毕,用洗涤缓冲液洗涤3次,4%多聚甲醛固定15 min后用PBS洗涤3次,然后用1 mg/mL DAPI (1∶1 000) 染色10 min,在荧光显微镜下观察 (40倍物镜)。整个实验过程避光操作。
1.2.3 细胞酶联反应法 (cell-ELA) 检测A15对U87-EGFRvⅢ细胞的亲和力
在 96孔板中接种密度为 1×105个/mL的U87-EGFRvⅢ细胞,在37 ℃、5% CO2孵箱培养24 h后用0.1 mol/L PBS (pH 7.2~7.4) 漂洗一次,再用4%的多聚甲醛固定15 min。用1×PBST(0.01%的Tween-20) 洗板3次,5% BSA于37 ℃封闭2 h后弃封闭液,洗板3次。用PBS (加入0.01% 鲑鱼精DNA) 对生物素标记的初始DNA文库、A15进行倍比稀释,样品终浓度分别为:2 000、1 000、500、250、125、62.5、31.25、15.625 nmol/L,各样品在95 ℃处理5 min,立刻置于冰上10 min后加入至U87-EGFRvⅢ细胞的培养孔中37 ℃孵育1 h,1×PBST洗板3次。每孔加入 50 μL辣根过氧化物酶标记的链亲和素-PBS溶液 (1∶1 000),37 ℃孵育30 min后洗涤3次,每孔加入50 μL TMB显色液,37 ℃避光显色10 min。最后每孔加50 μL 2 mol/L硫酸终止反应,在酶标仪上测定450 nm的吸光度值。用SigmaPlot 12.0软件处理数据,拟合曲线方程Y=BmaxX/Kd+X,求出Kd值。用抗EGFR的抗体代替生物素标记的A15加入至U87-EGFRvⅢ细胞中,用 5% BSA作为缓冲液倍比稀释抗体,样品终浓度为:4 ng/μL (27.59 nmol/L),2 ng/μL(13.79 nmol/L),1 ng/μL (6.90 nmol/L),0.5 ng/μL(3.45 nmol/L), 0.25 ng/μL (1.72 nmol/L),0.125 ng/μL (0.86 nmol/L) , 0.0625 ng/μL(0.43 nmol/L),0.03125 ng/μL (0.21 nmol/L),羊抗兔HRP标记二抗1∶5 000稀释,检测方法同上。实验设置空白 (不加一抗) 对照。阴性对照为生物素标记的初始DNA文库。
1.2.4 数据的统计和分析
用SigmaPlot 12.0软件中非线性回归分析作图,求出曲线方程,比较方程中参数。
2 结果
2.1 生物素或FITC标记的适配子A15的制备
以A15的菌液为模板进行不对称PCR扩增,其中上游引物带有生物素标记,扩增的两条链的比例为1∶4,扩增较多的短链即为标记上生物素或FITC的单链,大量切胶回收纯化后,用7 mol/L尿素-8%聚丙烯酰胺变性胶电泳鉴定 (图2)。结果表明通过不对称PCR法获得了生物素或FITC标记的DNA适配子。
2.2 A15特异识别U87-EGFRvⅢ细胞
通过Western blotting实验用EGFR抗体对U87和 U87-EGFRvⅢ两种细胞进行鉴定。结果显示 U87细胞表达 EGFR (170 kDa),而U87-EGFRvⅢ过表达 EGFRvⅢ,它是 EGFR缺失了胞外区2~7个外显子的的Ⅲ型突变体,条带分子量为 145 kDa (图 3)。FITC-A15分别与HEK293、SH-SY5Y、U87和U87-EGFRvⅢ细胞孵育,用荧光显微镜观察结合特异性情况,结果见图 4。未加 FITC-A15的细胞未观察到荧光,即为细胞背景,加入了 FITC-A15后,在U87-EGFRvⅢ细胞上可观察到绿色荧光,HEK293、SH-SY5Y和 U87细胞都观察不到明显的荧光,DAPI染色后,细胞核呈蓝色。结果表明,A15能特异识别U87-EGFRvⅢ细胞。
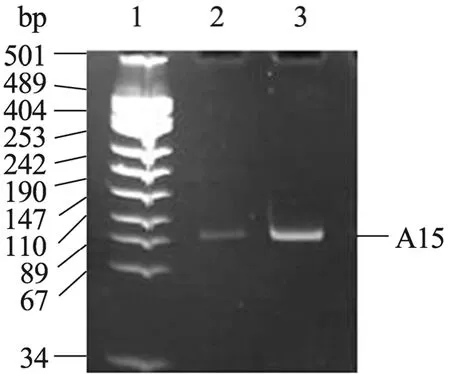
图2 不对称PCR切胶回收得到的biotin和FITC标记的适配子A15Fig. 2 Biotinylated and FITC-labeled A15 aptamers were generated by asymmetrical PCR. 1: pUC18 DNA/Msp l; 2: biotin-A15; 3: FITC-A15.

图3 EGFR抗体鉴定U87和U87-EGFRvⅢ细胞的免疫印迹实验Fig. 3 U87 and U87-EGFRvIII cells identified by Western blotting. 1: U87 cells expressing EGFR(170 kDa); 2: U87-EGFRvIII cells overexpressing EGFRvIII (145 kDa).
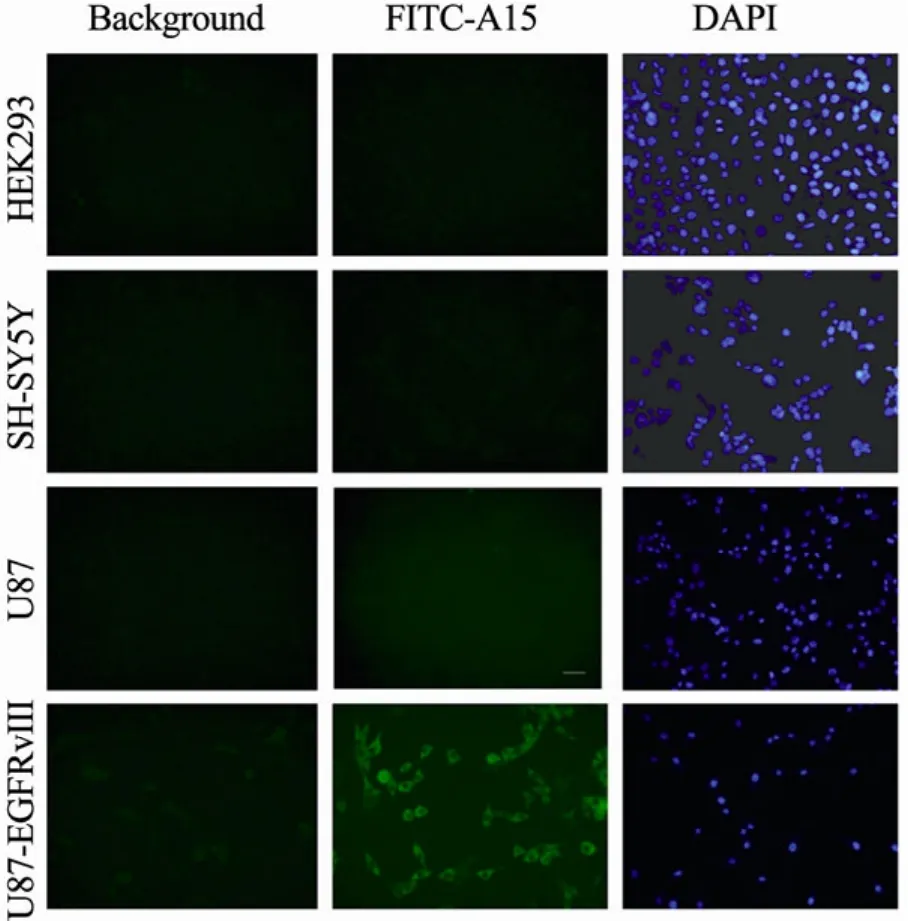
图 4 荧光显微镜检测适配子 FITC-A15对U87-EGFRvⅢ的特异性结合 (40×)Fig. 4 Fluorescent microscope imaging of U87-EGFRvIII, U87, and HEK293 cells stained with FITC-A15. U87 and HEK293 cells served as negative controls (40×).
2.3 Cell-ELA检测初始DNA文库、A15适配子和EGFR抗体三者对U87-EGFRvIII细胞的亲和力
用cell-ELA方法检测生物素标记的初始DNA文库、A15和抗 EGFR的抗体对 U87-EGFRvⅢ细胞的亲和力,测定450 nm时的吸光度值,在Sigmaplot 12.0软件中进行非线性回归分析,拟合曲线见图 5。分析结果发现 A15的 Kd值为(14.09±2.95) nmol/L (R=0.9653),小于 100 nmol/L,与以往报道过的 cell-SELEX筛选到的适配子亲和力在pmol/L~nmol/L范围内相符。适配子A15的Kd值与抗体EGFR对U87-EGFRvⅢ细胞的亲和力 (Kd=0.32±0.01 nmol/L,R=0.9989) 相比,A15的亲和力已经接近抗体的亲和力。阴性对照初始DNA文库的Kd值为 (726.28±200.75) nmol/L(R=0.9952),由此可见,相比初始DNA文库而言,A15和 EGFR抗体对U87-EGFRvⅢ细胞有较高的亲和力。

图5 Cell-ELA法测定初始DNA文库、适配子A15或抗体EGFR对U87-EGFRvⅢ细胞的亲和力Fig. 5 The binding affinity of the initial library,aptamer A15 and an anti-EGFR antibody for U87-EGFRvIII cells as measured by cell-ELA.
3 讨论
目前在 cell-SELEX技术应用中,测定适配子 Kd值的方法有流式细胞技术[8](Flow cytometry,FCM)、荧光定量分析[9](Fluorescence quantification analysis,FQA)、放射免疫法[10](Radioimmunoassay,RIA)、酶联免疫吸附试验(Enzyme-linked immunosorbent assay,ELISA)等。其中ELISA试验用于cell-SELEX中适配子与靶分子亲和力的测定具有如下优点:生物素标记的适配子比荧光标记的要稳定;整个 ELISA试验操作简便经济;所用试剂成本低且无污染;测定的结果灵敏度高。ELISA在适配子的亲和力的测定已经有了很多应用,早在 2004年已有研究小组建立了酶联寡聚核苷酸吸附试验(Enzyme-1inked oligonucleotide assay,ELONA)方法用于hTNF-α的检测[11]。CHO等[12]用ELISA方法来检测针对SARS COV (冠状病毒) N蛋白的DNA适配子是否富集及其亲和力,得到适配子 1 (Kd=4.93±0.30 nmol/L) 和适配子 11(Kd=9.02±1.89 nmol/L)。Kazem 等[13]在 4 ℃条件下用 ELISA方法测定不同筛选轮数 DNA 适配子对HER2高表达细胞及对照细胞的结合力,随着筛选轮数的增加,吸收值也增加,表明适配子在富集。Zhu等[14]筛选针对MPT64抗体的DNA适配子,并尝试用 ELISA方法来检测临床血清样本,数据证明其最低检测限为0.2 mg/L,是用于肺结核病人血清诊断的一种有效的方法。
EGFRvⅢ是 EGFR胞外区 2~7个外显子(EGFR∆2-7) 缺失导致的突变体,与肿瘤的侵袭性和致瘤性有关[15-16]。EGFRvⅢ在正常细胞中不表达[17-21],在 25%的恶性胶质瘤中过表达[22],因此筛选特异识别 EGFRvⅢ的适配子对探求诊断和治疗恶性胶质瘤的方法具有重大意义。
本研究在 Friguet法[23]基础上,通过以全细胞表面分子作为抗原,生物素标记的适配子作为抗体,利用生物素-链亲合素系统应用于 ELISA试验[24]来测定适配子 A15与 U87-EGFRvⅢ的Kd值。结果显示与抗体相比,适配子的亲和力接近抗体的亲和力。荧光显微镜观察也证实适配子对靶细胞具有特异识别能力。目前荧光定量分析方法检测适配子对靶细胞的亲和力,标记探针用的荧光染料有异硫氰酸荧光素 (FITC)、羟基荧光素 (FAM)、四氯荧光素 (TET) 等,但此类荧光素类衍生物有共同的缺点:光淬灭率高、pH敏感性强和发射波谱宽等[25-27]。Cell-ELA法克服了荧光标记方法进行定量分析的缺点,实现非荧光标记定量检测微量物质的目的,解决了筛选中适配子亲和力的定量分析问题,为建立cell-SELEX筛选技术平台提供了重要保证。但以细胞作为抗原进行 cell-ELA有诸多的缺点,比如:空白值与单纯的纯化蛋白相比要高;由于细胞的贴壁特性,不能使用双抗夹心法[11]等更加灵敏的 ELISA方法用来测定适配子与靶分子的亲和力。
Cell-ELA可以代替荧光标记法进行定量分析,进一步证明了ELISA在cell-SELEX中测定适配子亲和力的应用可行性;适配子和单克隆抗体一样,可以作为检测分子用于诊断某些疾病。
[1]Osborne SE, Matsumura I, Ellington AD, et al.Aptamers as therapeutic and diagnostic reagents:problems and prospects. Curr Opin Chem Biol,1997, 1(1): 5−9.
[2]Famulok M, Hartig JS, Mayer G, et al. Functional aptamers and aptazymes in biotechnology,diagnostics, and therapy. Chem Rev, 2007, 107(9):3715−3743.
[3]Navani NK, Li Y. Nucleic acid aptamers and enzymes as sensors. Curr Opin Chem Biol, 2006,10(3): 272−281.
[4]Jiang Y, Zhu C, Ling L, et al. Specific aptamer-protein interaction studied by atomic force microscopy. Anal Chem, 2003, 75(9):2112−2116.
[5]Li S, Xu H, Ding H, et al. Identification of an aptamer targeting hnRNP A1 by tissue slide-based SELEX. J Pathol, 2009, 218(3): 327−336.
[6]Cao XX, Li SH, Chen LC, et al. Combining use of a panel of ssDNA aptamers in the detection ofStaphylococcus aureus. Nucl Acids Res, 2009,37(14): 4621−4628.
[7]Cerchia L, de Franciscis V. Targeting cancer cells with nucleic acid aptamers. Trends Biotechnol,2010, 28(10): 517−525.
[8]Sefah K, Shangguan D, Xiong X, et al.Development of DNA aptamers using Cell-SELEX.Nat Protoc, 2010, 5(6): 1169−1185.
[9]Graham JC, Zarbl H. Use of cell-SELEX to generate DNA aptamers as molecular probes of HPV-associated cervical cancer cells. PLoS ONE,2012, 7(4): e36103.
[10]Ulrich H, Martins AH, Pesquero JB. RNA and DNA aptamers in cytomics analysis. Cytometry A,2004, 59(2): 220−231.
[11]Yan XR, Gao XW, Yao LH, et al. Novel methods to detect cytokines by enzyme-linked oligonucleotide assay. Chin J Biotech, 2004, 20(5):679−682 (in Chinese).
严馨蕊, 高绪文, 姚立红, 等. 应用核酸适配子检测细胞因子的新方法-ELONA法. 生物工程学报, 2004, 20(5): 679−682.
[12]Cho SJ, Woo HM, Kim KS, et al. Novel system for detecting SARS coronavirus nucleocapsid protein using an ssDNA aptamer. J Biosci Bioeng, 2011,112(6): 535−540.
[13]Dastjerdi K, Tabar GH, Dehghani H, et al.Generation of an enriched pool of DNA aptamers for an HER2-overexpressing cell line selected by cell SELEX. Biotechnol Appl Biochem, 2011,58(4): 226−230.
[14]Zhu C, Liu J, Ling Y, et al. Evaluation of the clinical value of ELISA based on MPT64 antibody aptamer for serological diagnosis of pulmonary tuberculosis. BMC Infect Dis, 2012, 12(4): 96−103.
[15]Gan HK, Kaye AH, Luwor RB, et al. The EGFRvIII variant in glioblastoma multiforme. J Clin Neurosci, 2009, 16(6): 748−754.
[16]Kanu OO, Mehta A, Di C, et al. Glioblastoma multiforme: a review of therapeutic targets. Expert Opin Ther Targets, 2009, 13(6): 701−718.
[17]Garcia de Palazzo IE, Adams GP, Sundareshan P,et al. Expression of mutated epidermal growth factor receptor by non-small cell lung carcinomas.Cancer Res, 1993, 53(14): 3217−3220.
[18]Gan HK, Kaye AH, Luwor RB. The EGFRvIII variant in glioblastoma multiforme. J Clin Neurosci, 2009, 16(6): 748−754.
[19]Emrich JG, Brady LW, Quang TS, et al.Radioiodinated (I-125) monoclonal antibody 425 in the treatment of high grade glioma patients:ten-year synopsis of a novel treatment. Am J Clin Oncol, 2002, 25(6): 541−546.
[20]Wikstrand CJ, Hale LP, Batra SK, et al.Monoclonal antibodies against EGFRvIII are tumor specific and react with breast and lung carcinomas and malignant gliomas. Cancer Res, 1995, 55(14):3140−3148.
[21]Moscatello DK, Holgado-Madruga M, Godwin AK,et al. Frequent expression of a mutant epidermal growth factor receptor in multiple human tumors.Cancer Res, 1995, 55(23): 5536−5539.
[22]Nikiforova MN, Hamilton RL. Molecular diagnostics of gliomas. Arch Pathol Lab Med,2011, 135(5): 558−568.
[23]Friguet B, Chaffotte AF, Djavadi-Ohaniance L, et al. Mea-surements of the true affinity constant in a solution of antigen-antibody complexes by enzyme-linked immunosor-bent assay. J Immunol Meth, 1985, 77(2): 305−319.
[24]Zhu J, Zhang JH. Biotin-avidin ELISA method to detect ANEP Ⅲ aptamers affinity quantitatively. J Cell Mol Immunol, 2007, 23(6): 580−581 (in Chinese).
祝军, 张景海. 生物素-亲合素ELISA法对ANEPⅢ寡核苷酸适配子亲和力的定量测定. 细胞与分子免疫学杂志, 2007, 23(6): 580−581.
[25]Fan W, Zhang YL. Application of fluorescent labeling in drug research. Foreign Med Sci Sect Pharm, 2007, 34(1): 52−55 (in Chinese).
范雯, 张燕玲. 荧光标记在药物研究中的应用.国际药学研究杂志, 2007, 34(1): 52−55.
[26]King MA. Detection of dead cells and measurement of cell killing by flow cytometry. J Immunol Methods, 2000, 243(1/2): 155−166.
[27]Shapiro HM. Microbial analysis at the single-cell level: tasks and techniques. J Microbiol Method,2000, 42(1): 13−16.
November 13, 2012; Accepted: February 1, 2013
Xingmei Zhang. Tel: +86-20-61648215; E-mail: zhangxm@smu.edu.cn
Cell-ELA-based determination of binding affinity of DNA aptamer against U87-EGFRvIII cell
Yan Tan1, Huiyu Liang1, Xidong Wu1, Yubo Gao2, and Xingmei Zhang1
1Department of Neurobiology,School of Basic Medical Sciences,Southern Medical University,Guangzhou510515,Guangdong,China
2First Clinical Medicine College,Southern Medical University,Guangzhou510515,Guangdong,China
A15, a DNA aptamer with binding specificity for U87 glioma cells stably overexpressing the epidermal growth factor receptor variant III (U87-EGFRvIII), was generated by cell systematic evolution of ligands by exponential enrichment (cell-SELEX) using a random nucleotide library. Subsequently, we established a cell enzyme-linked assay(cell-ELA) to detect the affinity of A15 compared to an EGFR antibody. We used A15 as a detection probe and cultured U87-EGFRvIII cells as targets. Our data indicate that the equilibrium dissociation constants (Kd) for A15 were below 100 nmol/L and had similar affinity compared to an EGFR antibody for U87-EGFRvIII. We demonstrated that the cell-ELA was a useful method to determine the equilibrium dissociation constants (Kd) of aptamers generated by cell-SELEX.
cell enzyme-linked assay (cell-ELA), cell based systematic evolution of ligands by exponential enrichment(cell-SELEX), DNA aptamer, Kd
book=670,ebook=397
Supported by: National Natural Science Foundation of China (Nos. 81272509, 30973481).
国家自然科学基金 (Nos. 81272509,30973481) 资助。
(本文责编 郝丽芳)
