应用单核苷酸多态性芯片研究单亲二体型来源及致病机制*
2013-04-13郭佩玲尹玉竹侯红瑛
邓 妮, 郭佩玲, 尹玉竹, 章 钧, 田 琪, 侯红瑛
(中山大学附属第三医院产科,广东 广州 510630)
单亲二体型(uniparental disomy,UPD)是指一对同源染色体均来自一个亲本,在新生儿中发生率约为0.029%,至今文献已报道了约1 100例整体型UPD和120例部分型UPD[1]。据Kotzor等[2]统计,文献报道的母源性16号染色体UPD(maternal UPD of chromosome 16,mUPD 16)约为50余例,其临床表型多变,从无任何异常临床表型到智力障碍、发育迟缓、结构畸形等均见报道。mUPD 16由于已知病例均属于涉及整条染色体的整体型单亲二体型,因此16号染色体上因UPD而致病的区段及基因迄今无法定位,其致病机制也难以分析。虽然仅涉及16号染色体部分片段的部分型UPD 16病例可以帮助研究人员确定导致具体临床症状的染色体区段,并进一步确定致病的关键基因、明确基因功能和致病机制,但迄今尚无报道。我院自2012年起,应用单核苷酸多态性芯片(single nucleotide polymorphism array, SNP array)技术对124例孕妇羊水中的胎儿细胞及父母亲外周血细胞进行全基因组拷贝数分析,共发现2例部分型mUPD 16。本研究对这2例部分型mUPD 16进行回顾性分析,探讨SNP array在单亲二体型来源和致病机制研究及遗传咨询中的应用价值。
材 料 和 方 法
1研究对象
我院自2012年起,对具有唐氏综合征高风险、需行羊水胎儿细胞G 显带染色体核型分析的124例孕妇,在征得孕妇及丈夫的同意后,同时应用SNP array对羊水中的胎儿细胞进行全基因组拷贝数分析,共发现2例部分型mUPD 16。2例孕妇经核实孕周后,在妊娠20周前的超声检查中发现胎儿双顶径和股骨长均小于正常孕龄的第10位百分数[3],存在均称型胎儿生长受限征象,并已排除其它可能导致均称型生长受限的高危因素的存在,如TORCH综合征、宫内感染、抗磷脂抗体综合征、存在狼疮抗凝物等。妊娠22~26周三维彩超结果未发现明显的胎儿畸形,羊水细胞G显带核型分析显示核型正常,见表1。

表1 2例病例的临床资料、核型分析和SNParray结果
G1P0:gravidity time is 1 and parity time is 0; G4P0:gravidity times are 4 and parity time is 0; BPD: biparietal diameter; FL: femur length.
2方法
2.1胎儿及父母亲样本提取与处理羊膜腔穿刺术中,在超声引导下经腹抽吸羊水20 mL,分别装在2支消毒试管内,加盖,其中1管行羊水细胞培养G显带染色体核型分析。羊水细胞培养G显带核型分析按经典方法进行,羊水细胞分离后加入羊水培养基(Gibco)培养7 d后,分离细胞经洗涤、低渗、固定、滴片、后固定、脱水、老化、染色后在显微镜下根据染色体条带进行判读、分析[1]。另外1管羊水细胞悬液经离心后获取羊水细胞,使用QIAmp DNA Mini Kit(Qiagen)提取羊水细胞基因组DNA。另外,用肝素钠抗凝管和EDTA抗凝管分别抽取孕妇及其配偶肘静脉血各2 mL,分别用于胎儿父母外周血淋巴细胞培养G显带染色体核型分析和提取DNA。
2.2母血污染鉴定分别对羊水DNA标本和母亲DNA标本进行短串联重复序列 (short tandem repeat, STR)扩增(Devyser V3)并比对[4]。按说明书要求进行结果分析,凡可能存在母源性污染的羊水沉渣样本一律舍弃,重新分离培养以后的羊水细胞并提取DNA用于下一步分析。
2.3SNP array检测和基因组拷贝数变异分析选用Illumina的HumanCytoSNP-12对羊水细胞基因组DNA和胎儿父母亲外周血淋巴细胞DNA进行拷贝数变异(copy number variation, CNV)和杂合性缺失(loss of heterozygosity,LOH)检测,芯片构成见www.illumina.com。严格按照产品操作手册进行全基因组扩增、标记、杂交、洗涤及荧光扫描,扫描结果采用Karyotyping和GenomeStudio软件进行分析。选取Illumina公司提供的人类基因组单倍型数据作为实验对照。根据原始荧光值和对照数据计算Log R Ratio值和B等位基因频率(B allele frequency,BAF) 判断等位基因拷贝数和基因型。Log R Ratio值低于1表示存在拷贝数缺失,大于1表示存在拷贝数重复。BAF值为0.5代表杂合型(AB),0和1分别代表纯合型(AA和BB)。软件根据连续位点拷贝数和基因型计算CNV的连续区段,进行t检验并给予Confidence值。分别选择Confidence>50,片段长度>100 kb作为CNV入选标准以及Confidence>50,片段长度>5 Mbp作为LOH入选标准。筛选获得的片段在UCSC数据库(www.ucsc.edu)中进行分析,并与目前国际上已有的正常人群数据库DGV数据库(http://projects.tcag.ca/variation)比对,通过查询表型数据库DECIPHER (https://decipher.sanger.ac.uk/)、PubMed数据库(www.ncbi.nlm.nih.gov/pubmed/)和OMIM数据库(www.ncbi.nlm.nih.gov/omim)分析CNV和LOH的临床意义。将CNV和LOH进行分类:(1)多态:相似的重复或缺失出现在3个或3个以上的正常人;(2)良性:与异常表型存在或增高的风险无关,遗传自正常的父母;(3)致病:与已知的异常表型相关,或与不同外显的异常表型相关或与异常表型增高的风险相关;(4)未知:不确定遗传性,或不包含已知致病基因,或不确定临床重要性[5]。
3结果验证
CNV结果采用多重连接依赖性探针扩增(multiplex ligation-dependent probe amplication,MLPA)或real-time PCR进行验证,胎儿LOH结果通过父母SNP array检测并做连锁分析进行验证[6]。
结 果
1孕妇1羊水中胎儿细胞的SNParray结果
用SNP芯片技术对羊水细胞进行遗传学分析,结果显示为部分型UPD 16,分别位于16号染色体短臂末端和长臂末端,具体位点是p12.2~13.3,长度约为21.0 Mbp;16q24.1~24.3,长度约为4.1 Mbp,见表1和图1。
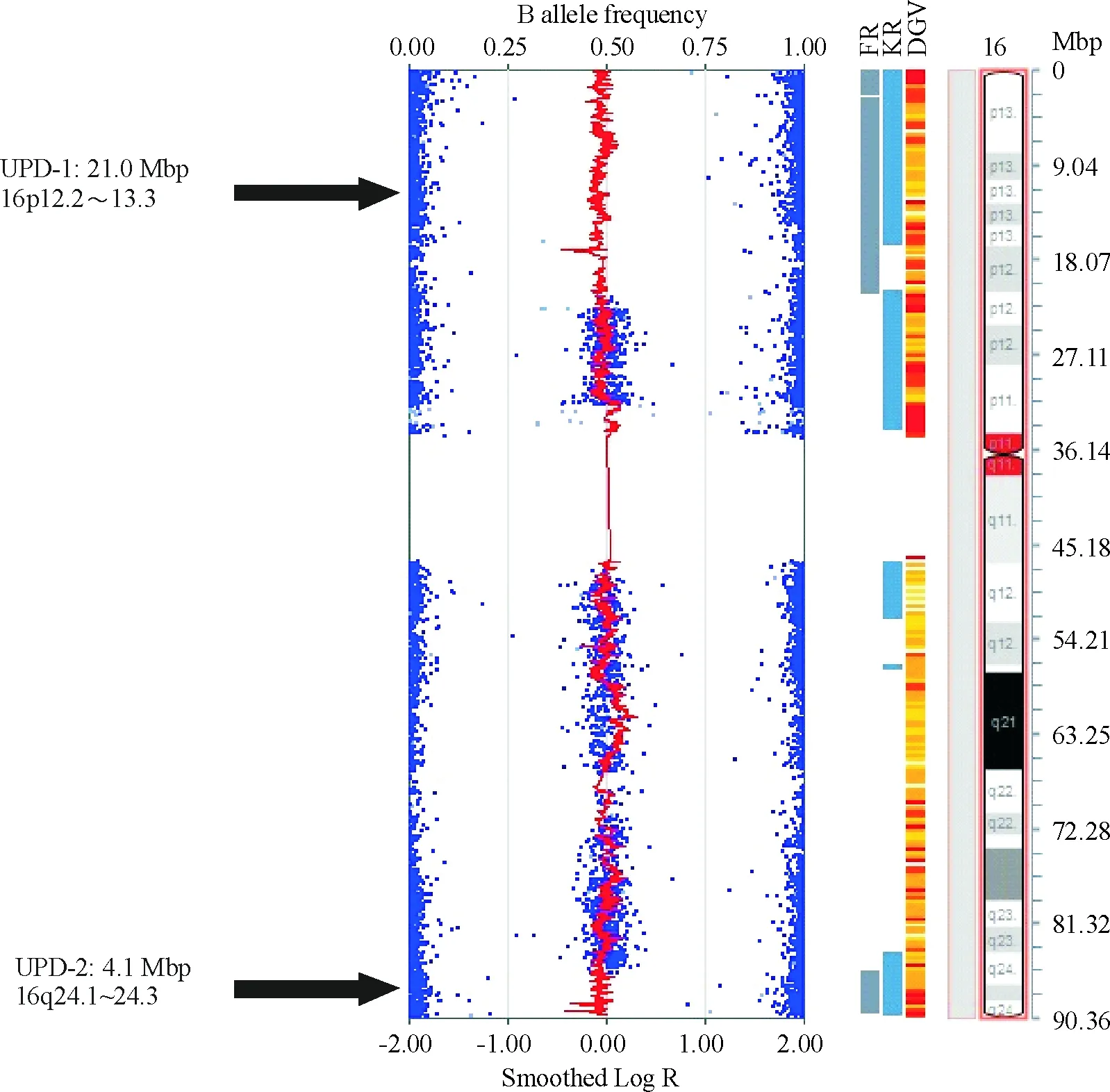
Figure 1. Human genome-wide SNP array result of Case 1. FR: found region; KR: known region; DGV: Database of Genomic Variants.
图1例1的人类全基因组SNP分型芯片检测结果
2孕妇2羊水中胎儿细胞的SNParray结果
用SNP芯片技术对羊水细胞进行遗传学分析,结果显示为部分型UPD 16,存在于16号染色体长臂末端,具体位置为16q21~24.3,大小约24.1 Mbp,见表1和图2。
正常对照样本的人类全基因组SNP分型芯片检测结果见图3。
在图1~3中,左侧方框内蓝色点为等位基因的BAF,位于0.5位置表示基因型为杂合子(AB),位于两侧表示基因型为纯合子(AA或BB);左侧方框内红色曲线为Log R值,提示等位基因的拷贝数,0表示拷贝数中性,无缺失重复,往左偏表示存在片段缺失,往右偏表示存在重复。中间柱状图分别为该样本的变异区域(found region),已知的致病的变异区域(known region)和已知的正常多态性区域(数据来自Database of Genomic Variants,DGV)。最右侧G带核型图提示变异区段对应的染色体位置。
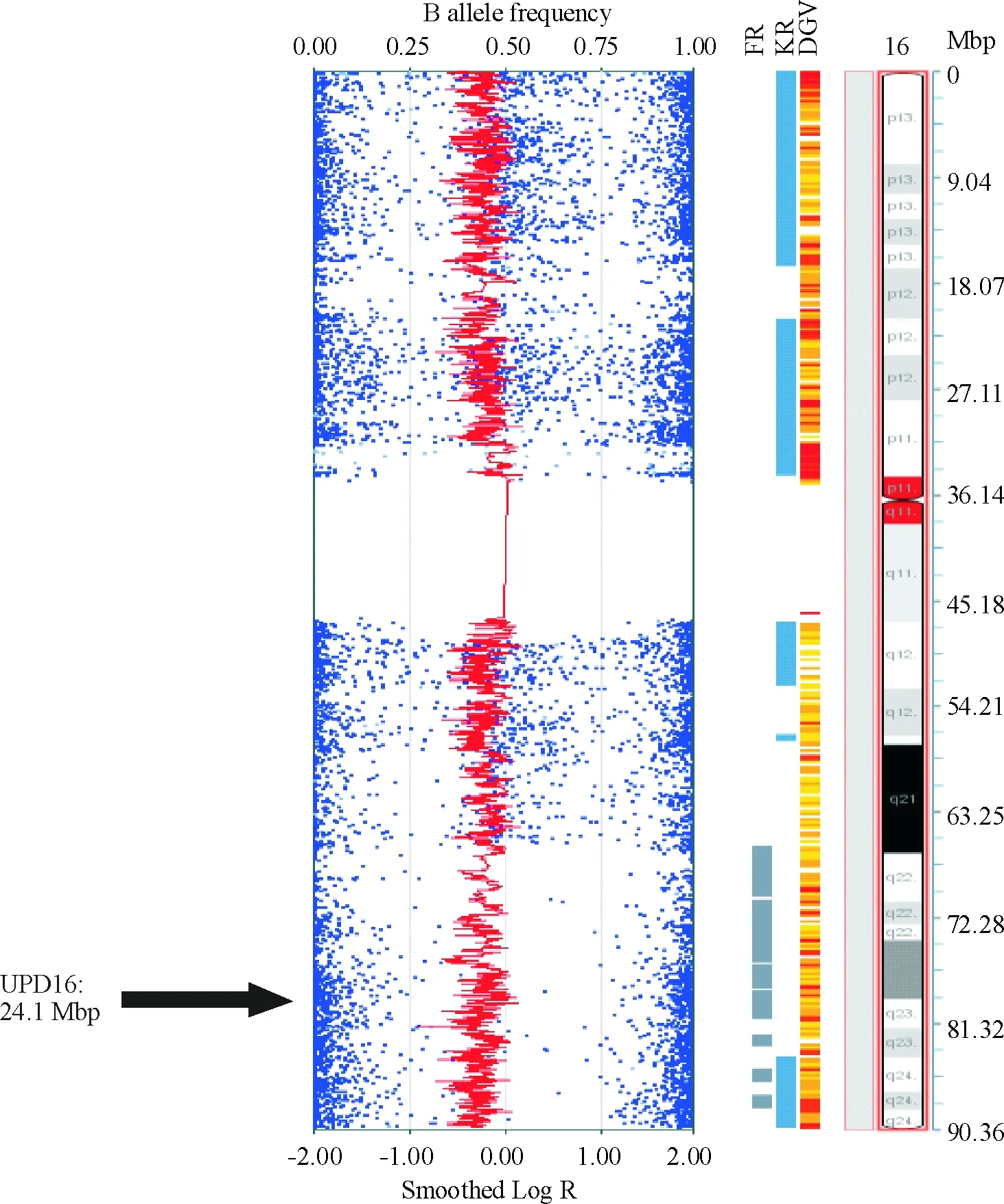
Figure 2. Human genome-wide SNP array result of Case 2. FR: found region; KR: known region; DGV: Database of Genomic Variants.
图2例2的人类全基因组SNP分型芯片检测结果
3父母外周血细胞G显带染色体核型分析和SNParray结果
2例胎儿的核型分别为46,XX和46,XY,父母核型分析亦都正常。SNP array检测并做同家系3人的基因型连锁分析发现,2例胎儿的16号染色体非单亲二体型部分与双亲的基因型相似度均高于99%,提示该部分染色体分别遗传自父母双方;而16号染色体单亲二体型部分与父亲基因型比较可以排除父源性遗传可能性,而与母亲基因型相似度均高于99%,证实该染色体区段存在父源性缺失,为母源性单亲二体型。
讨 论
SNP array技术为本研究2例胎儿存在的均称型生长受限阐释了常规G显带染色体核型分析技术无法发现的细微片段的染色体改变。针对染色体疾病的诊断,学术界近年来已逐渐形成共识,染色体微阵列技术包括SNP array和比较基因组杂交芯片技术,应该逐渐成为发育障碍或先天畸形个体的首选遗传学诊断方法[6]。本研究中,除这2例mUPD 16外, SNP array检测还发现了Williams综合征、1p36微缺失综合征、angleman综合征、Smith-Meganes综合征等多种已知遗传综合征和微缺失微重复综合征。
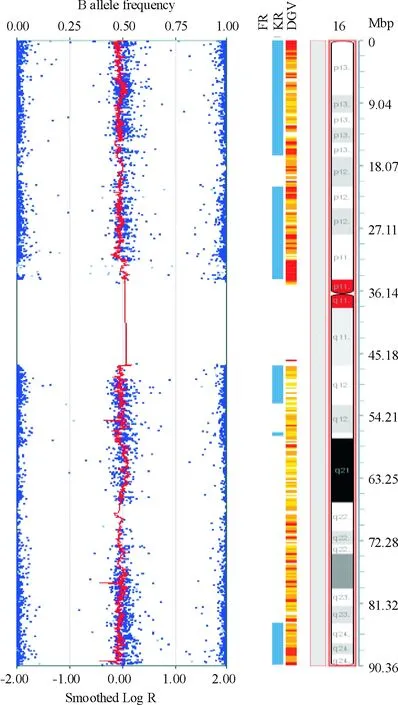
Figure 3. Human genome-wide SNP array result of normal control. FR: found region; KR: known region; DGV: Database of Genomic Variants.
图3正常对照样本的人类全基因组SNP分型芯片检测结果
不同类型的mUDP 16对胎儿发育的影响不尽相同[2]。单亲二体型的形成与染色体不分离有关, 并通过三体补救、配子互补、单体补救等方式避免非整倍体的出现,从而形成不同类型的单亲二体型存活下来[5]。SNP array除了是目前诊断单亲二体型的最佳方法外[6],可以帮助研究人员推测染色体不分离发生的不同时期并确定导致具体临床症状的染色体区段,为进一步进行相关区域的基因序列分析、确定致病的关键基因及明确基因功能打下基础。 (1)单亲异二体型是指一对同源染色体分别来自父亲或母亲的2条同源染色体,其染色体不分离发生在减数分裂Ⅰ期。(2)整体型单亲二体型的染色体不分离发生在减数分裂Ⅱ期,为姐妹染色单体的不分离。(3)部分型单亲二体型又可分为包含着丝粒在内和不包含着丝粒在内2种类型。单亲二体型区域包含着丝粒在内的部分型的形成机制与整体型相类似,只是在姐妹染色单体不分离形成的二体配子与单体配子结合成三体型时发生了部分染色体的交换。本研究发现的2例16单亲二体型均为单亲二体型区域未包含着丝粒在内的部分型。 目前,这一类型单亲二体型形成的具体机制并未被完全阐明,推测可能发生在有丝分裂期。在有丝分裂过程中,同源染色体中一条染色体发生了不包含着丝粒在内的部分缺失,其杂合性缺失由另一染色体的相对应部分进行复制来弥补[6]。这2例病例单亲二体型的位置分别位于16号染色体长、短臂末端(例1)和长臂末端(例2),两者均在妊娠20周前出现了胎儿生长受限的症状,而例1生长受限的症状更为明显。结合其它报道[2],提示16号染色体长、短臂末端可能均存在与胎儿发育或胎盘功能相关的基因。鉴于该2例病例均未出现结构畸形等症状,提示导致结构畸形的相关基因可能位于长、短臂近着丝粒的位置。在以后的研究中,应进行相关区域的基因序列分析来确定致病的关键基因及明确基因功能。
对胎儿双亲来说,显然更关注的是单亲二体型对胎儿可能造成的影响及其再次妊娠的复发几率。从形成机制来看,单亲二体型属于新发染色体变异,即变异个体的双亲任何一方体细胞中都不存在的染色体畸变,按孟德尔遗传规律来说,再次妊娠复发的几率极低。而在产前诊断中,对新发染色体变异胎儿的预后评估往往比较复杂,更多的是依靠经验统计数据。尽管目前文献累积的部分型mUPD 16病例尚不多见,但既然已有相当一部分mUPD 16患者出现胎儿生长受限和(或)胎儿畸形的报道,这2例胎儿的双亲应该被告知不能排除胎儿生长受限进一步加重以及胎儿和新生儿以后出现畸形的可能性。另外,本研究通过SNP array对胎儿双亲的外周血细胞进行遗传连锁分析后发现,这2例UPD 16均为母源性,若母亲16号染色体上相关区域存在着特定的隐性致病基因,单亲二体型则意味着该基因会成为纯合子,胎儿双亲应被告知隐性疾病致病基因的暴露可能会导致一些罕见常染色体隐性疾病的出现[7]。
在本研究中,SNP array 还发现了相当一部分临床意义不明确的拷贝数变异。SNP array的分辨率高达0.7 kb[8],而与SNP array技术的高分辨率相随而生的正是临床意义不明确的拷贝数变异的高检测率[9]。随着SNP array诊断流程逐步规范化及人们对基因组结构和功能相关性理解的逐步改善,人类全基因组SNP array技术在产前诊断中的应用会越来越广泛。
[参考文献]
[1]Liehr T.Cytogenetic contribution to uniparental disomy (UPD).Mol Cytogenet, 2010,3:8.
[2]Kotzot D, Utermann G. Uniparental disomy (UPD) other than 15: phenotypes and bibliography updated[J]. Am J Med Genet A,2005,136A(3):287-305.
[3]严英榴,杨秀雄.产前超声诊断学[M].第2版.北京:人民卫生出版社,2012:52-57.
[4]Grati FR, Malvestiti F, Grimi B, et al. QF-PCR as a substitute for karyotyping of cytotrophoblast for the analysis of chorionic villi: advantages and limitations from a cytogenetic retrospective audit of 44,727 first-trimester prenatal diagnoses[J]. Prenat Diagn,2013,33(5):502-508.
[5]Conlin LK, Thiel BD, Bonnemann CG,et al. Mechanisms of mosaicism, chimerism and uniparental disomy identified by single nucleotide polymorphism array analysis[J].Hum Mol Genet, 2010,19(7):1263-1275.
[6]Miller DT, Adam MP, Aradhya S,et al. Consensus statement: chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies[J].Am J Hum Genet, 2010,86(5):749-764.
[7]Malvagia S, Papi L, Morrone A, et al. Fatal malonyl CoA decarboxylase deficiency due to maternal uniparental isodisomy of the telomeric end of chromosome 16[J].Ann Hum Genet, 2007, 71(6):705-712.
[8]Siggberg L, Sirpa AM, Tarja L,et al. High-resolution SNP array analysis of patients with developmental disorder and normal array CGH results[J]. BMC Med Genet,2012,13:84.
[9]Ganesamoorthy D, Bruno DL, McGillivray G, et al. Meeting the challenge of interpreting high-resolution single nucleotide polymorphism array data in prenatal diagnosis: does increased diagnostic power outweigh the dilemma of rare variants?[J].BJOG, 2013,120(5):594-606.
