一株猪源豕链球菌的分离鉴定
2012-08-14王楷宬邓明俊吴振兴岳志芹郑小龙肖西志
陈 煜,吴 达,孙 敏,王楷宬,邓明俊,吴振兴,岳志芹,郑小龙,孙 涛,肖西志*
(1.福建农林大学生命科学学院,福建福州350002;2.甘肃出入境检验检疫局,甘肃兰州730020;3.山东出入境检验检疫局,山东青岛266002;4.中国动物卫生与流行病学中心,山东青岛266032)
链球菌科包括2个属,链球菌属和乳球菌属,链球菌属包括89种链球菌,豕链球菌和猪链球菌同属于链球菌属。Collins于1984年将豕链球菌归入兰氏血清群E、P、V、U以及其他3个抗原群,即C1、5916T和7155A,豕链球菌的模式株包括NTCC10999、SS-1029和 ATCC43138[1]。而猪链球菌的血清群包括C、D、E和L群,在血清学上两者可能存在交叉反应。两者在生长表现及形态学上非常相似,即在血琼脂平板上菌落都为光滑、圆润的呈现明显的β型溶血,在普通光学显微镜下,经革兰染色后都表现为革兰阳性、长短不一的球菌。
据报道,豕链球菌通常与猪的化脓性感染有关,如子宫颈淋巴管炎、脓肿、肺炎、败血症和心内膜炎[2]。有报道称该菌可引起猪流产[3-4]。据报道从健康猪也能分离到该菌[5]。从女性的生殖系统可分离到该菌[6],作为致病菌,主要感染女性的生殖系统[7],可引起死胎[8]。也有该菌引起皮肤外伤感染的报道[9]。通过对25株分离自人的豕链球菌与17株参考菌株的比较分析,结果显示人源豕链球菌与猪源有明显的差异[10]。Bekal S等[11]利用16SrRNA 测序分析表明,人类的豕链球菌在生化特点上与来源于猪的豕链球菌虽然相同,但分子生物学上存在差异,从而将人类的豕链球菌命名假豕链球菌(Streptococcus pseudoporcinus)。Kevin A S等[12]通过流行病学调查发现假豕链球菌主要存在于黑人女性中,而且与其他生殖道疾病相关。我国关于豕链球菌的报道很少,仅见有陈明辉的牛源豕链球菌的生物学特性报道[13]。
山东出入境检验检疫局检验检疫技术中心近期对送检的一例病死猪进行了针对猪链球菌病的检测,绵羊血琼脂上分离到疑似猪链球菌菌株,菌株呈明显的β型溶血。为进一步的确证该菌,我们进行了生化、血清学和分子生物学鉴定,同时我们进行了小鼠感染试验以确认其是否具有致病性。
1 材料与方法
1.1 材料
绵羊血琼脂平板为OXOID公司产品;VITEK革兰阳性鉴定卡、链球菌乳胶凝集分型诊断试剂盒为法国梅里埃公司产品;猪链球菌分型血清为中国动物卫生流行病中心猪病实验室提供;细菌鉴定卡和药敏鉴定卡为美国BD公司产品;猪链球菌通用型、1型、2型、7型和9型特异性引物由农业部动物卫生与流行病学中心提供;清洁级试验小鼠购自青岛食品药品管理局实验动物中心。
1.2 方法
1.2.1 病料的分离培养 无菌采集死猪的杆、心、脾、肺、肾、扁桃体等组织,分别研磨,然后接种于绵羊血琼脂平板,37℃,24h,挑取明显β溶血、表面光滑、透明菌落,进行革兰染色镜检。对可疑菌落用绵羊血琼脂平板进行纯培养。
1.2.2 可疑菌的生化鉴定 取可疑菌的纯培养物分别按照VITEK2(法国梅里埃微生物鉴定系统)和PHOENIX-100(美国BD公司微生物鉴定系统)配制菌悬液。
1.2.3 血清学鉴定 首先用链球菌分群血清进行分群,然后用猪链球菌的分型血清(35种)进行分型,分群血清采用的是乳胶凝集试验,分型则应用玻片凝集试验。
1.2.4 PCR鉴定 采用猪链球菌通用型引物及1型、2型、7型和9型特异性引物对纯培养物的核酸提取物进行扩增,同时设立阳性对照和阴性对照,所用引物序列见表1。
1.2.5 16SrDNA的扩增及序列测定 用猪链球菌16SrDNA的通用引物27F和1492R对分离的纯培养物核酸进行扩增,序列见表1。PCR产物经琼脂糖凝集回收试剂盒回收后送南京金斯瑞生物公司测序,测序结果在NCBI中的GenBank进行比对。
1.2.6 小鼠感染试验 将分离菌株接种于血清肉汤,37℃,12h后离心,沉淀用生理盐水稀释到1×106cfu/mL,腹腔注射小鼠4只,同时设生理盐水作为对照。
2 结果
2.1 分离株的生长表现
分离到的可疑菌株在绵羊血琼脂平板上37℃培养12h后表现为圆形、光滑、透明的小菌落,呈β溶血;在血清肉汤中为试管底部有轻微的浑浊沉淀。
2.2 生化试验鉴定
经PHOENIX-100和VITEK2微生物鉴定仪鉴定,结果均为豕链球菌。可信值分别为99%和97%,结果见表2和表3。
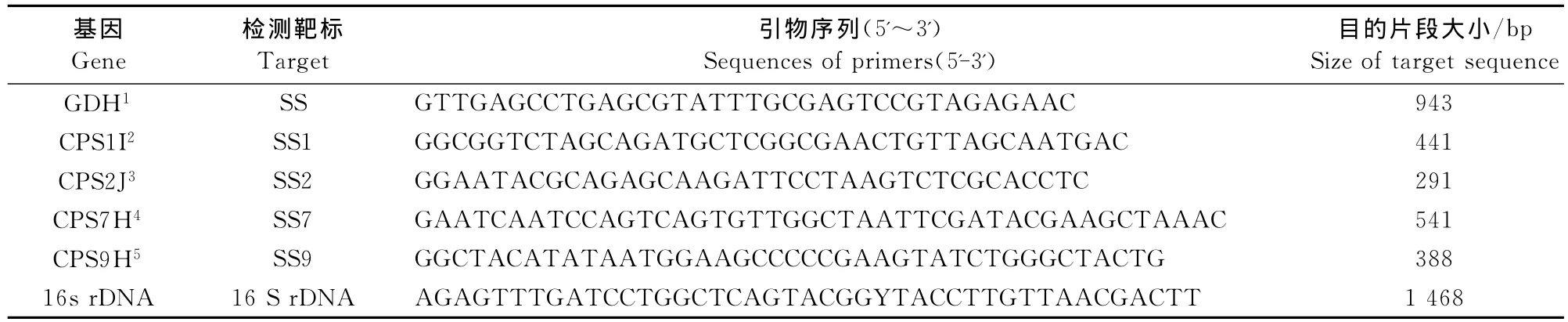
表1 所使用的引物及目的片段大小Table 1 Primers and size of target sequences

表2 可疑菌的PHOENIX-100生化试验鉴定结果Table 2 Identification result of suspect bacterium by PHOENIX-100

表3 可疑菌的VITEK2生化试验鉴定结果Table 3 Identification result of suspect bacterium by VITEK 2
2.3 血清学分型
用链球菌乳胶凝集法检测试剂盒(可检测链球菌A、B、C、D、F和G群)对分离到的可疑菌的分群结果显示,6种分群血清均不能与分离到的可疑菌株凝集。同时,32种猪链球菌分型血清也不能与可疑菌凝集。
2.4 PCR鉴定结果
用猪链球菌通用引物及猪链球菌的1、2、7、9型特异性引物对可疑菌提取的基因组DNA的扩增结果显示均没有目标条带出现,对照组试验结果成立,结果见图1和图2。
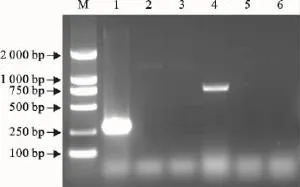
图1 可疑猪链球菌的PCR鉴定Fig.1 PCR identification of suspect S.suis
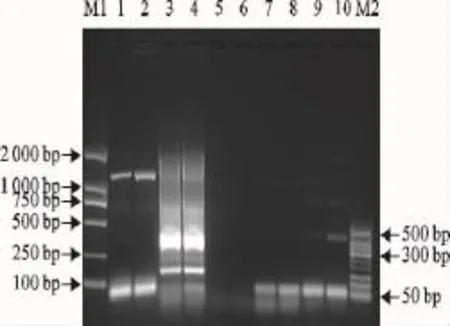
图2 分别用猪链球菌通用引物及1型、2型、7型和9型对可疑菌进行PCR鉴定Fig.2 PCR identification of suspect bacterium amplified by general primers of S.suis,1,2,7and 9respectively
2.5 16SrDNA序列比对及与猪链球菌的同源性分析
细菌16SrDNA的通用引物27F和1492R对可疑菌的核酸提取物扩增出1 500bp左右的目标条带,对扩增产物的序列表明,与发表的豕链球菌具有98%的同源性,而与猪链球菌2型的同源性为93%。
2.6 攻毒试验结果
试验组小鼠在注射可疑菌的菌悬液后没有出现明显的发病症状,和对照组小鼠在表现上无明显差异,经1周后仍正常。
3 讨论
随着分子生物学技术的发展,链球菌属的分类已经从21种[14]增加到89种[1],那些以前未被分离到或误判为某种链球菌的链球菌属新成员被重新命名并定位。这也意味着一些新的病原体被发现并逐渐受到重视。
虽然VITEK2和PHOENIX-100的鉴定结果均为Streptococcus porcinus,两者的可信度分别达到了97%和99%,但由于生化试验的结果还不足以得出鉴定结论,甚至生化试验有时会给出错误的结果[15]。因此,随后的试验有两个目的,一是排除猪链球菌;二是对该菌进行鉴定及致病性的初步分析。
豕链球菌和猪链球菌在形态学及生长表现上很相近,在血琼脂上都呈现明显的β型溶血。然而这两种菌在致病性方面具有很大差别,猪链球菌是一种重要的人畜共患细菌性传染病,不仅可给养猪业带来重大损失,而且人一旦感染也会引起发病甚至死亡。因此,当我们分离到疑似猪链球菌时,所设计的试验都是按照猪链球菌标准进行。
所分离的豕链球菌的血清学分群结果与Christian等的报道结果相同,暗示着所分离的豕链球菌并不是医学中常见的B型,有可能是E、U、V、P中的一种,还需要进一步确认。用猪链球菌分型血清进行的凝集试验和随后的针对猪链球菌的PCR鉴定结果排除了猪链球菌的可能。
16SrDNA作为细菌分类的活化石,已经广泛应用于细菌的分类和鉴定[16-23],并以此开发出许多快速的鉴定方法[24-27]。根据 Bosshard P P[28]的判断标准,16SrDNA的同源性在95%~99%之间的可认为是同属细菌,大于99%的可认为是同种细菌。因此,通过生化鉴定和分子生物学鉴定,可确定所分离到的链球菌为豕链球菌。
本研究的结果表明,虽然所分离到的豕链球菌不具备致病性或具有较弱的致病性,但解剖及试验分析表明,该菌可存在于健康猪体内,并可能会成为继发感染的病原体之一,应引起重视。
[1] George M G,Julia A B,Timothy G L.Taxonomic outline of the prokaryotes Bergey’s manual of systematic bacteriology[M].New York,Berlin,Heidelberg:Springer,2004:205.
[2] Katsumi M,Kataoka Y,Takahashi T,et al.Biochemical and serological examination ofβ-hemolytic streptococci isolated from slaughtered pigs[J].J Vet Med Sci,1998,60(1):129-131.
[3] Hommez J,Devriese L A,Castryck F,et al.Bèta-hemolytic streptococci from pigs:bacteriological diagnosis[J].Zentralbl Vet Med B,1991,38(6):441-444.
[4] Plagemann O.Streptococcus porcinus as a cause of abortion in swine[J].Zentralbl Vet Med B,1988,35(10):770-772.
[5] Katsumi M,Kataoka Y,Takahashi T,et al.Bacterial isolation from slaughtered pigs associated with endocarditis,especially the isolation of Strptococcus suis [J].J Vet Med Sci,1997,59(1):75-78.
[6] Robin R C,Carol J W,Rebecca A J,et al.Evaluation of a chromogenic agar for detection of group B Streptococcus in pregnant women[J].J Clin Microbiol,2010,48(9):3370-3371.
[7] Facklam R,Elliott J,Pigott N,et al.Identification of Streptococcus porcinus from human sources[J].J Clin Microbiol,1995,33(2):385-388.
[8] Martin C,Fermeaux V,Eyraud J L,et al.Streptococcus porcinus as a cause of spontaneous preterm human stillbirth[J].J Clin Microbiol,2004,42(9):4396-4398.
[9] Steven D M,Jill E C.Thumb infection caused by Streptococcus pseudoporcinus[J].J Clin Microbiol,2009,47(9):3041-3042.
[10] Duarte R S,Barros R R,Facklam R R,et al.Phenotypic and genotypic characteristics of Streptococcus orcinus isolated from human sources[J].J Clin Microbiol,2005,43(9):4592-4601.
[11] Bekal S,Gaudreau C,Laurence R A,et al.Streptococcus pseudoporcinus sp.nov.,a novel species isolated from the genitourinary tract of women[J].J Clin Microbiol,2006,44:2584–2586.
[12] Kevin A S,Lorna K R,Michele N A,et al.Incidence and epidemiology of Streptococcus pseudoporcinus in the genital tract[J].J Clin Microbiol,2011,49(3):883-886.
[13] 陈明辉,王开胜.新疆牛源豕链球菌的部分生物学特性[J],石河子大学学报:自然科学版,2010,28(2):180-183.
[14] 布坎南R E,吉本斯N E.伯杰细菌鉴定手册[M].北京:科学出版社,1984:677.
[15] Bascomb S,Manafi M.Use of enzyme tests in characterization and identification of aerobic and facultatively anaerobic gram-positive cocci[J].Clin Microbiol Rev,1998,11(2):318-340.
[16] Angharad P D,Michelle R,Stephen J H,et al.Identification of clinical isolates ofα-hemolytic s treptococci by 16SrRNA gene sequencing,matrix-assisted laser desorption ionization–time of flight mass spectrometry using MALDI biotyper,and conventional phenotypic methods:a comparison [J].J Clin Microbiol,2012,50(12):4087-4090.
[17] Patrick C Y W,Jade L L T,Jeff K L W,et al.Guidelines for interpretation of 16SrRNA gene sequence-based results for identification of medically important aerobic Gram-positive bacteria[J].J Med Microbiol,2009,58(8):1030-1036.
[18] Claire J,Clare L L,Holly L C,et al.Detection and identification of bacteria in clinical samples by 16SrRNA gene se-quencing:comparison of two different approaches in clinical practice[J].J Med Microbiol,2012,61(4):483-488.
[19] Patricia S C,Patrick R M,Adrian M Z.Evaluation of the integrated database network system (IDNS)smart gene software for analysis of 16SrRNA gene sequences for identification of Nocardiaspecies[J].J Clin Microbiol,2010,48(8):2995-2998.
[20] Elisabeth E A,Jan W B,Jessica R C,et al.Identification of clinical coryneform bacterial isolates:comparison of biochemical methods and sequence analysis of 16SrRNA and rpoB genes[J].J Clin Microbiol,2008,46(3):921-927.
[21] Joann L C,Dag H,Peter C I,et al.Comparison of traditional phenotypic identification methods with partial 5’16S rRNA gene sequencing for species-level identification of nonfermenting gram-negative bacilli[J].J Clin Microbiol,2010,48(4):1442-1444.
[22] Church D L,Simmon K E,Jan S,et al.Identification by 16S rRNA gene sequencing of negativicoccus succinicivorans recovered from the blood of a patient with hemochromatosis and pancreatitis[J].J Clin Microbiol,2011,49(8):3082-3084.
[23] Ulrik S J,Marianne N S,Elisa K,et al.16SrRNA gene sequencing in routine identification of anaerobic bacteria isolated from blood cultures[J].J Clin Microbiol,2010,48(3):946-948.
[24] Kirstin J E,Julie M J L,Sally L,et al.Utility of real-time amplification of selected 16SrRNA gene sequences as a tool for detection and identification of microbial signatures directly from clinical samples[J].J Med Microbiol,2012,61(5):645-652.
[25] Christian F P S,Knud P,Mogens K,et al.Novel molecular method for identification of Streptococcus pneumoniae applicable to clinical microbiology and 16SrRNA sequence-based microbiome studies[J].J Clin Microbiol,2012,50(6):1968-1973.
[26] Patrick C Y W,Jade L L T,Juilian M Y Y,et al.Automated identification of medically important bacteria by 16S rRNA gene sequencing using a novel comprehensive database,16SpathDB[J].J Clin Microbiol,2011,49(5):1799-1809.
[27] Jade L L T,Ming Y Y,Geoffrey Y,et al.In silico analysis of 16SrRNA gene sequencing based methods for identification of medically important aerobic Gram-negative bacteria[J].J Med Microbiol,2011,60(9):1281-1286.
[28] Bosshard P P,Abels S,Zbinden R,et al.Ribosomal DNA sequencing for identification of aerobic Gram-positive rods in the clinical laboratory(an 18-month evaluation)[J].J Clin Microbiol,2003,41(9):4134-4140.
