人朊蛋白Glu 200残基突变的分子动力学研究
2011-03-08刘梦源程先超王润玲
周 慧,刘梦源,杨 冰,程先超,王润玲
(1.天津医科大学药学院,天津300070;2.北京协和医学院、中国医学科学院放射医学研究所,天津分子核医学重点实验室,天津 300192)
朊蛋白(prion protein,PrP)是一种不同于细菌、病毒或类病毒的在分类上尚未定论的病原因子[1-5]。其本质为由正常宿主细胞基因编码的、构象异常的蛋白质,目前尚未检出任何核酸成分,是人和动物的传染性海绵状脑病(transmissible spongiform encephalopathies,TSEs)的病原体。能造成哺乳动物脑部病变的是PrPSC(传染型PrP),另一种是不具有侵染力的PrPC(细胞型PrP)。有观点认为是由正常形式的蛋白错误折叠成致病蛋白而形成的,两种结构异型的蛋白PrPC和PrPSC来源于同一基因,具有相同的氢基酸序列和共价修饰,但是其理化性质和结构有很大的差异。目前研究表明,朊蛋白E200K突变会使PrPC转变为PrPSC[6-9]导致人和动物的传染性海绵状脑病,但其转变机制尚不明确。本研究采用分子动力学的方法,进行长时间分子动力学模拟,研究E200K突变所导致的朊蛋白变构过程。
1 材料与方法
1.1 分子动力学模型构建 从布鲁克海文蛋白数据库(RCSB data bank,http://www.rcsb.org)下载人的野生型朊蛋白NMR结构(PDB ID:1QM0)和人的E200K突变型朊蛋白NMR结构(PDB ID:1QM2)。
利用Desmond 9.0分子动力学软件包[10-11]向上述两个人朊蛋白模型中加入溶剂分子,溶剂分子采用简单点电荷水分子(SPC水分子),周期边界条件中将生成的盒子形状定为Cubic,盒子的Buffer值为10Å。向体系中加入反离子,使模拟体系达到电中性,同时再向体系中加入0.15mol/L的氯化钠,使整个体系与人体体液环境等渗。
1.2 分子动力学模拟条件 分子动力学模拟采用等温等压系综,键长限制算法为SHAKE,温度耦合方法为Nose-Hoover,模拟温度为300K,弛豫时间为1ps。压力耦合方法为Martyna-Tobias-Klein,弛豫时间为2ps,模拟压力为1.01325bar(1bar=100000Pa),Couplingstyle为Isotropic,可压系数为4.5×10-5。短程静电相互作用阈值为9Å,长程静电作用计算采用Smooth particle mesh Ewald(PME)[12-13]。为防止朊蛋白在分子动力学模拟过程中本身部分移出盒子外,给朊蛋白中心残基的α碳原子施加2kcal/mol/ Å2的力。积分步长为2fs,首先平衡500ps,平衡采用Desmond默认的Relaxation protocol,再进行10ns的正式分子动力学模拟。在模拟过程中,每2ps记录一次轨迹文件。
2 结果
2.1 人朊蛋白的二级结构 图1为人朊蛋白二级结构模型缎带图,由图可以看出人朊蛋白主要由3个α螺旋组成,其中α1螺旋包括144~152残基,α2螺旋包括174~193残基,α3螺旋包括200~228残基。朊蛋白的N端主要包括一些loop结构,C端主要以α螺旋为主。

图1 人朊蛋白二级结构图Fig 1 The secondary structure of human prion protein
2.2 E200K突变对朊蛋白整体结构影响
2.2.1 野生型和E200K突变型朊蛋白Cα的均方根偏差(root mean square deviation,RMSD)随时间变化比较 野生型(WT)朊蛋白在10ns的分子动力学模拟过程中RMSD值变化不大,从1Å逐渐升高到2Å,而E200K突变型朊蛋白RMSD变化非常明显,在前4000ps内,RMSD值从1Å迅速升高到4.5Å,此后基本在4~5Å范围内波动,见图2。

图2 野生型和E200K突变型朊蛋白Cα的RMSD随时间变化曲线Fig 2 RMSD of α-carbon atoms of wild type and E200K mutant prion protein
从RMSD随时间变化曲线可以看出,野生型朊蛋白在10ns的分子动力学模拟过程中结构波动不大,但突变型朊蛋白RMSD波动很大,说明在分子动力学模拟过程中,结构发生了显著变化。
2.2.2 野生型和E200K突变型朊蛋白Cα的均方根波动(root mean square fluction,RMSF)随时间变化比较 野生型的朊蛋白RMSF在1~3Å之间保持稳定波动,突变型的朊蛋白RMSF在C端残基RMSF值较大,在8Å左右,见图3。

图3 野生型和E200K突变型朊蛋白Cα的RMSF变化曲线Fig 3 RMSF of α-carbon atoms of wild type and E200K mutant prion protein
从RMSF曲线图可以看出,相对于分子动力学模拟过程中的平均结构而言,突变型朊蛋白C端残基骨架波动很大,说明在突变型的朊蛋白的C端蛋白结构发生了显著变化。
2.2.3 分子动力学模拟10ns后人朊蛋白二级结构变化 图4是10ns后的人朊蛋白二级结构图,由此图可以看出,E200K会导致α2和α3螺旋发生解旋,同时,在10ns分子动力学模拟后,相比野生型朊蛋白,E200K突变型朊蛋白的β折叠会延长,因此,需进一步分析造成上述结构变化的原因。

图4 10ns后人朊蛋白二级结构图Fig 4 The secondary structure of human prion protein after 10ns MD simulations
2.2.3.1 α2螺旋骨架氢键概率统计情况:由表1可以看出,E200K的突变会使朊蛋白α2螺旋大部分骨架氢键稳定程度降低,从而导致突变体的α2螺旋在10ns的分子动力学模拟后出现解旋现象。
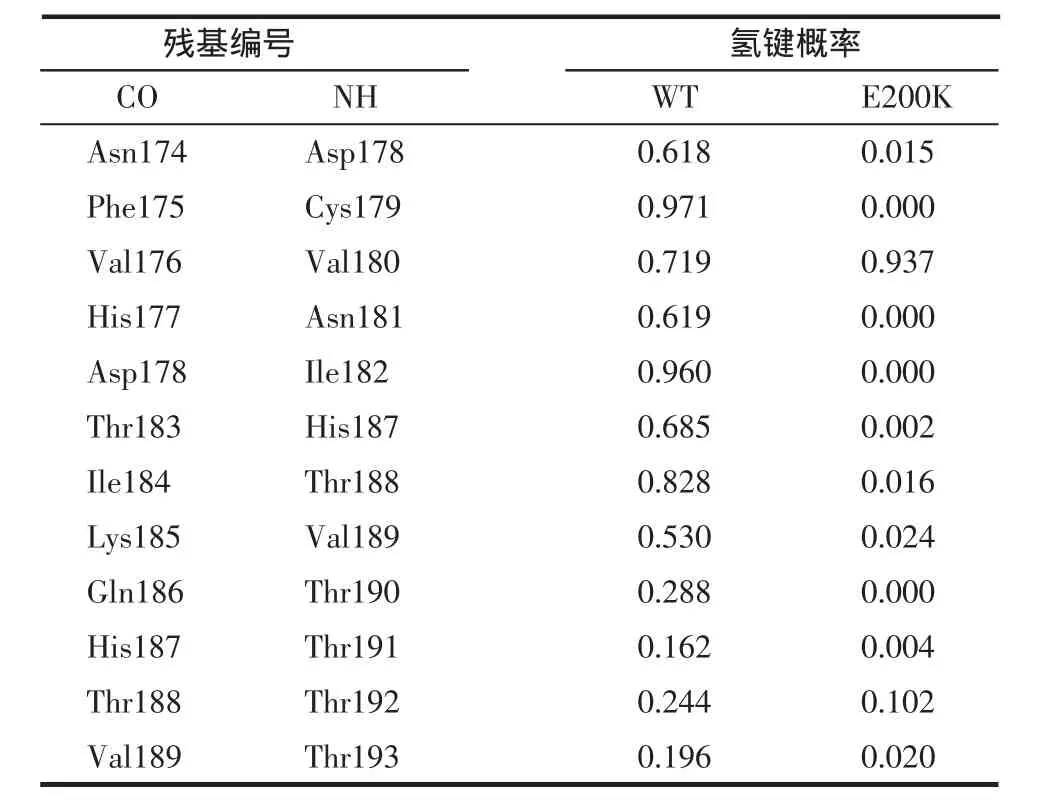
表1 α2螺旋骨架氢键概率统计情况Tab 1 Hydrogen bond occupancy of α2 helix backbone hydrogen bonds
2.2.3.2 α3螺旋骨架氢键概率统计情况:由表2可以看出,E200K的突变会使朊蛋白α3螺旋骨架中Ser222与Tyr226、Ala224与Arg228氢键稳定程度降低,所以E200K会使C端的α3螺旋骨架氢键稳定程度降低,从而导致突变体的α3螺旋在10ns的分子动力学模拟后出现解旋的现象。

表2 α3螺旋骨架氢键概率统计情况Tab 2 Hydrogen bond occupancy of α3helix backbone hydrogen bonds
2.2.3.3 β折叠氢键概率统计情况:由表3可见,野生型和E200K突变型朊蛋白β折叠区域残基骨架氢键概率影响不是很大。除Gly131骨架的羰基与Val161骨架的氨基形成氢键的概率较低外,其它残基骨架氢键概率非常高。因此可以看出,E200K突变对朊蛋白β折叠区域残基骨架氢键稳定性影响不是很大。

表3 β折叠氢键概率统计情况Tab 3 Hydrogen bond occupancy of β sheet backbone hydrogen bonds
3 讨论
3.1 PrPC和PrPSC理化性质差异(1)PrPC存在于正常组织及感染动物的组织中,是正常基因的产物,通常情况下是无害的,对蛋白酶K敏感。(2)PrPSC仅存在于感染动物的组织中,与致病和传染有关,对蛋白酶K有抗性。这两种朊蛋白的差异主要是后者β折叠所占比例较前者高。目前认为,PrPC转变为PrPSC是疾病发生的基本条件,PrPSC在中枢神经系统细胞内聚集而导致疾病的发生[14-18]。
3.2 关于分子动力学模拟条件选择 在分子动力学模拟过程中,对于原子间非共价键静电相互作用,包括短程静电相互作用和长程静电相互作用两部分。Cut off算法只计算体系的短程静电相互作用不计算长程静电相互作用,体系的波动就会比较剧烈,体系的稳定性会降低。PME是分子动力学计算长程静电作用的常用计算方法,通过计算体系在分子动力学模拟过程中的长程静电相互作用情况,可以更为精确地计算模拟过程的势能情况,也可以防止体系在模拟过程中波动过于剧烈[19]。
目前,大量研究表明,人体内正常朊蛋白中某些残基的突变会导致其二级结构的变化,从而导致传染性海绵状脑病的发生,但迄今为止,对其致病机制尚无明确结论。
本研究中采用的分子动力学模拟是近些年来新兴的分子模拟方法,是结合物理、数学和化学的综合技术。分子动力学是一套分子模拟方法,该方法主要是依靠牛顿力学来模拟分子体系的运动,以在由分子体系的不同状态构成的系综中抽取样本,从而计算体系的构型积分,并以构型积分的结果为基础进一步计算体系的热力学量和其他宏观性质。利用分子动力学方法可以从原子水平模拟出蛋白的变构过程,弥补实验研究的不足。
因此,本研究采用分子动力学的模拟方法,分别对人野生型和E200K突变型朊蛋白进行了10ns分子动力学模拟。结果表明,E200K的突变会使朊蛋白二级结构发生显著变化,使朊蛋白二级结构中的β折叠长度增加,使α2和α3螺旋解旋,其解旋的主要原因是由于突变体会导致α2和α3螺旋骨架氢键的稳定性下降,这样β折叠所占的比例就会升高,α螺旋由于解旋所占比例就会下降,而这一二级结构的变化最终可能会导致PrPC转化为PrPSC。该研究成果会为今后传染性海绵状脑病致病机制的研究提供一定的理论依据。
[1] 李俊,林东海.朊病毒蛋白的三维结构[J].生命的化学,2007,27(1):53
[2] Zou WQ,Gambetti P.Prion:the chameleon protein[J].Cell Mol Life Sci,2007,64(24):3266
[3] Mehrpour M,Codogno P.Prion protein:From physiology to cancer biology[J].Cancer Lett,2010,290(1):1
[4] Sakudo A,Xue G,Kawashita N,et al.Structure of the Prion Protein and its gene:an analysis using bioinformatics and computer simulation[J].Curr Protein Pept Sci,2010,11(2):166
[5] Martins VR,Beraldo FH,Hajj GN,et al.Prion protein:orchestrating neurotrophic activities[J].Curr Issues Mol Biol,2010,12(2):63
[6] Pezza JA,Serio TR.Prion propagation:the role of protein dynamics [J].Prion,2007,1(1):36
[7] van der Kamp MW,Daggett V.The consequences of pathogenic mutations to the human prion protein[J].Protein Eng Des Sel, 2009,22(8):461
[8] Hasegawa K,Mohri S,Yokoyama T.Fragment molecular orbital calculations reveal that the E200K mutation markedly alters local structural stability in the human prion protein[J].Prion,2010,4(1): 38
[9] Mancuso M,Siciliano G,Capellari S,et al.Creutzfeldt-Jakob disease with E200K PRNP mutation:a case report and revision of the literature[J].Neurol Sci,2009,30(5):417
[10]Shaw DE.A fast,scalable method for the parallel evaluation of distance-limited pairwise particle interactions[J].J Comput Chem, 2005,26(13):1318
[11]Klepeis JL,Lindorff-Larsen K,Dror RO,et al.Long-timescale molecular dynamics simulations of protein structure and function [J].Curr Opin Struc Biol,2009,19(2):1
[12]Darden T,York D,Pedersen L.Particle mesh ewald:an N.log(N) method for ewald sums in large systems[J].J Chem Phys,1993,98(12):10089
[13]Essmann U,Perera L,Berkowitz ML,et al.A smooth particle mesh ewald potential[J].J Chem Phys,1995,103(19):8577
[14]Nicolas O,Gavin R,del Rio JA.New insights into cellular prion protein(PrPC)functions:The“ying and yang”of a relevant protein [J].Brain Res Rev,2009,61(2):170
[15]Westergard L,Christensen HM,Harris DA.The cellular prion protein (PrPC):Its physiological function and role in disease[J]. Biochim Biophys Acta,2007,1772(6):629
[16]Sakudo A,Ikuta K.Prion protein functions and dysfunction in prion diseases[J].Curr Med Chem,2009,16(3):380
[17]Kupfer L,Hinrichs W,Groschup MH.Prion protein misfolding[J]. Curr Mol Med,2009,9(7):826
[18]Linden R,Martins VR,Prado MAM,et al.Physiology of the prion protein[J].Physiol Rev,2008,88(2):673
[19]El-Bastawissy E,Knaggs MH,Gilbert IH.Molecular dynamics simulations of wild-type and point mutation human prion protein at normal and elevated temperature[J].J Mol Graph Model, 2001,20(2):145
