高性价比寡核苷酸基因表达谱芯片的制备方法
2010-12-01任永红张科孙清岚王亚辉庄远红张亮
任永红,张科,孙清岚,王亚辉,庄远红,张亮
随着人类基因组学研究的不断进展,基因芯片正逐步成为检测基因表达最强大的分子生物学技术方法。根据芯片上点制的探针不同,基因芯片主要可分为cDNA 芯片和寡核苷酸(Oligo)芯片 2 种类型。cDNA 芯片是将一些基因片段通过 PCR 方式制备探针,在早期应用比较常见[1]。然而随着越来越多的生物物种基因组测序的完成,Oligo 探针的设计成为可能,由于Oligo 芯片的特异性较cDNA 芯片好,并且制备起来不需要经过繁琐的PCR 扩增和纯化回收过程,因此 Oligo 芯片已逐渐成为基因芯片中的应用主流。目前制备 Oligo 芯片主要有 2 种方式[2]:第一种是在线方式(on line)的原位制作技术[3-4],即在基片介质上直接合成DNA 探针,例如美国 Affymetrix公司利用光刻掩膜方式在硅基片表面原位合成 25-mer 长度的Oligo 探针、美国 Agilent公司利用喷墨(ink-jet)方式在玻片表面合成 60-mer 长度的Oligo 探针、美国 LC Science公司利用酸碱法在硅基片表面非光刻掩膜合成 Oligo 探针等;第二种是离线方式(off line)的点制芯片技术[5-6],即先利用 DNA 合成仪合成 Oligo 探针,然后用芯片点样仪将 Oligo探针精确点制到基片表面上。第一类芯片制备方式需要复杂的设备、技术以及大量资金投入,只能由专门的实验室制备;第二种芯片制备方法需要的硬件设备和操作技能都可以被一般的分子生物学实验室所接受,并且点制芯片的制备方法是一个开放的平台,研究者可以根据自己的研究目的制备多种类型的基因芯片,非常适合实验室自行建立自己的基因芯片平台。
目前国际上一些商业芯片公司在点制芯片的时候多用 60~70-mer 长度的探针,例如美国的Operon公司(拥有包括人、大鼠、小鼠等多个物种的全基因组Oligo 库,其探针长度通常为70-mer)。从 Oligo 探针的合成效率和合成难度角度考虑,探针越长,单位价格越高。现今国内大多数公司就是以 60-mer 长度为界,探针合成价格相差 3 倍,但目前还没有详细的关于60-mer 长度以上和以下探针对实验结果影响的研究报道。另外在检测基因表达时,由于原核生物的mRNA 不含PolyA,难于针对 mRNA 进行特异性标记,因此针对原核生物的基因表达检测要远比真核生物困难。鉴于此,我们选择大肠杆菌基因表达信息作为实验模型,对 59-mer和70-mer 长度 Oligo 探针的杂交信号进行分析比较,以探讨探针长度对寡核苷酸基因芯片性价比优势的影响。
1 材料与方法
1.1 材料
大肠杆菌 E.coli ATCC11775 菌株、晶芯 cRNA扩增标记试剂盒(Cat#.360060)、DNA 点样液、PersonalArrayer16TM芯片点样仪、BioMixerTMII 芯片杂交仪、SlideWasherTM8 芯片清洗仪、LuxScanTM10K-A 激光共聚焦扫描仪、LuxScan 3.0数据分析软件均为博奥生物有限公司产品,溶菌酶(lysozyme)(Cat#.RT401)为天根生化科技(北京)有限公司产品,NucleoSpin RNA II 试剂盒(Cat#.740.955.10)为德国 Machery Nagel(MN)公司产品,MessageAmp II-Bacteria 试剂盒(Cat#.1790)为美国 Ambion公司产品,ND-1000 型紫外分光光度计为美国 NanoDrop公司产品。
1.2 方法
1.2.1 菌株培养 从平板上挑取新的活化后E.coli ATCC11775 单菌落,接种于5ml LB 液体培养基中,37 ℃ 振荡培养 12 h。取 50 μl 菌液转入 5ml LB 中,37 ℃ 振荡培养 1~1.5 h 至对数生长期、吸光度(A600)值为0.5 左右。
1.2.2 菌株 RNA 提取 在1.5ml EP 管中加入1ml 菌液,14000×g 离心 2 min,吸弃上清。加入 100 µl 新鲜配制的1×TE 缓冲液(含有0.2 mg/ml 溶菌酶)重悬菌体,轻弹管壁混匀,37 ℃孵育 10 min。然后按照 NucleoSpin RNA II 试剂盒说明书提取 E.coli RNA。提取的RNA 样品用甲醛变性琼脂糖凝胶电泳进行质量检测,紫外分光光度计测量浓度,计算 RNA 总量,将 2个样品等量混合用于后续芯片实验。
1.2.3 探针设计与芯片点制 根据博奥生物有限公司已有大肠杆菌全基因组芯片数据选择覆盖高、中、低杂交信号强度的大肠杆菌基因共 20个。针对该 20个基因分别设计 59-mer和70-mer 长度的Oligo 探针(探针均由上海英骏生物技术有限公司合成),探针序列见表1。将 Oligo 探针溶于点样液中并调至终浓度为20 μmol/L,利用芯片点样仪点制在经过氨基修饰的基片上,每个探针重复2个点,点间距为250 μm,每个点阵共包含 10 行、10 列,探针分布示意图见图 1。点制于芯片上的样品还包括用于监控实验过程的外标探针 Y1-Y8、用于监控核酸固定的Hex 探针 PC,以及阴性对照点样液 NC。同时制备 4 张完全相同的芯片,点制好的芯片干燥后室温保存备用。

表1 20个大肠杆菌基因的59-mer和70-mer 长度寡核苷酸探针序列Table1 The 59-mer and 70-mer oligo probes sequences of 20 E.coli genes

图1 59-mer和70-mer 长度寡核苷酸探针的芯片点阵分布Figure1 The probe layout of the 59-mer and 70-mer oligo probes on the array
1.2.4 芯片检测
1.2.4.1 RNA 样品标记 首先采用 MessageAmp II-Bacteria 试剂盒对 RNA 进行 Ploy A 加尾,利用 T7 Oligo(dT)引物进行反转录合成一链 cDNA,DNA 聚合酶合成 cDNA 第二链,以双链 DNA 为模板经体外转录系统合成 cRNA。参照文献[7-11]改良方法,利用 cRNA 扩增标记试剂盒对 cRNA进行反转录得到 DNA,最后在Klenow 酶的作用下利用随机引物进行扩增,延伸过程中加入荧光素,具体标记步骤见图 2。
1.2.4.2 芯片预处理 将芯片点制有探针的一面在65 ℃ 水浴锅上水合处理 2 次,每次 10 s。250 mJ 紫外强度下交联,42 ℃ 预热 0.5% SDS 清洗 10 min,42 ℃ 预热蒸馏水清洗 2 min,离心甩干。
1.2.4.3 芯片杂交 将标记后样品加入含有 3×SSC、0.2% SDS、5×Dehart’t、25% 甲酰胺的杂交缓冲液中,42 ℃ 杂交 12~16 h,在芯片清洗仪中用 42 ℃ 预热的洗液 I(2×SSC、0.2% SDS)和42 ℃ 预热的洗液 II(0.2% SSC)分别清洗 4 min,离心甩干。重复杂交 4 张芯片。
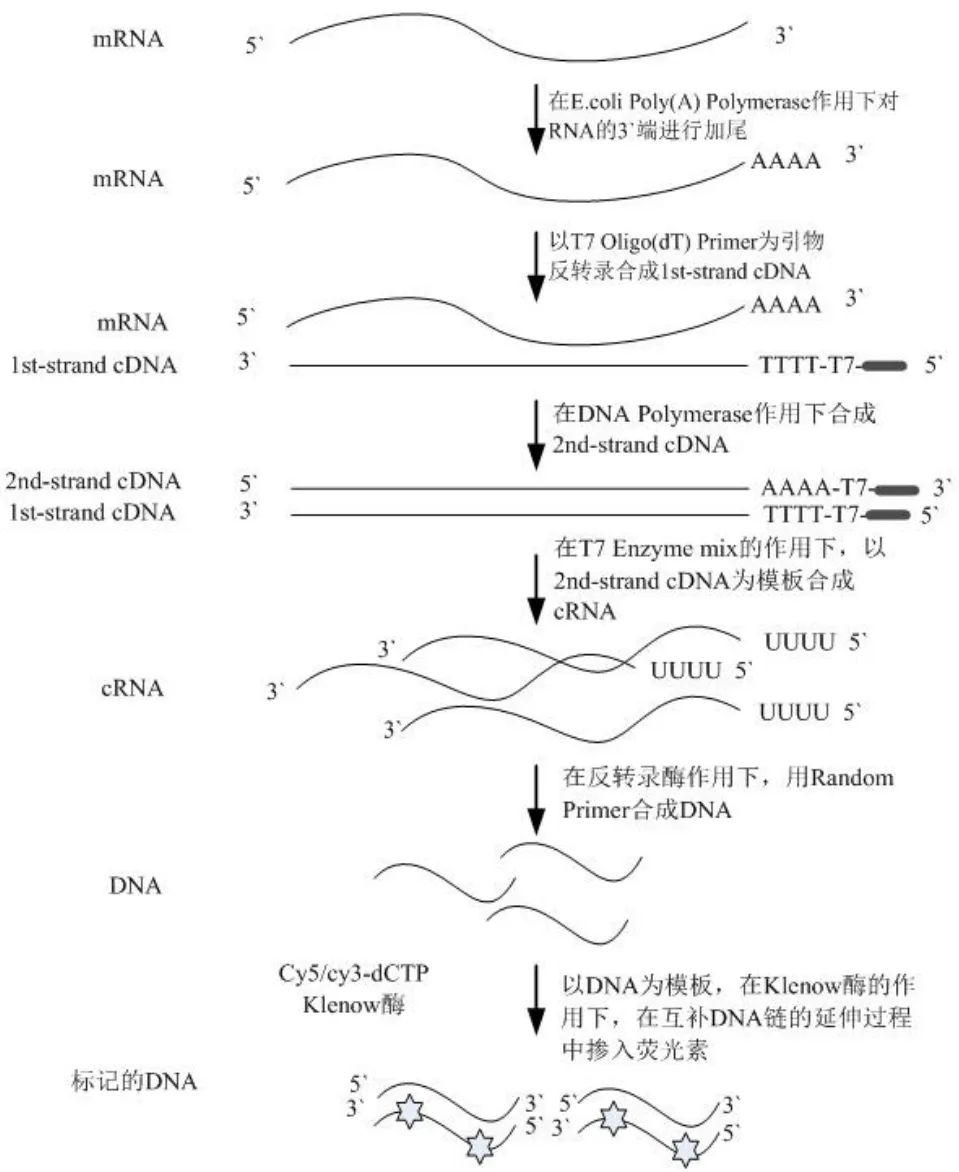
图2 大肠杆菌 RNA 样品扩增标记过程示意图Figure2 Flow diagram for labeling E.coli RNA samples
1.2.4.4 芯片扫描 采用 LuxScanTM10K-A 激光共聚焦扫描仪扫描芯片获取图像,利用 LuxScan 3.0数据分析软件将图形文件转换为数字文件,提取获得 59-mer和70-mer 长度探针的杂交信号值。
1.3 统计学处理
应用 SPSS 11.0 统计学软件进行数据处理,采用 Paired t test 进行显著性分析,利用 Excel 计算各探针的杂交信号值(),以 P<0.05 为差异有统计学意义。
2 结果
2.1 探针设计
根据博奥生物有限公司已有大肠杆菌全基因组芯片数据选择覆盖高、中、低杂交信号强度的20个大肠杆菌基因分别设计 59-mer和70-mer长度 Oligo 探针,使得探针更具有生物学代表性;同时将 59-mer 长度探针设计位于70-mer 长度探针的内部,以防止芯片杂交信号的差异与碱基组成有关。
2.2 大肠杆菌 RNA 质检
甲醛变性琼脂糖凝胶电泳检测结果显示大肠杆菌 28S rRNA和18S rRNA 条带清晰,无降解带出现,质量合格(图 3)。
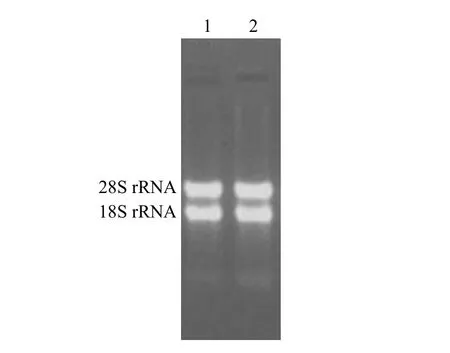
图3 大肠杆菌 RNA的甲醛变性琼脂糖凝胶电泳检测Figure3 Formaldehyde denatured agarose gel electrophoresis of E.coli total RNA
2.3 芯片检测
我们将 59-mer和70-mer 长度探针点制在同一个芯片点阵中,一次杂交过程即可并行比较 2 种长度探针的杂交信号,以防止 2 种探针由于分布在不同芯片中所导致的杂交信号差异,共进行了4 张芯片的重复实验。芯片杂交结果显示同一个芯片中的59-mer和70-mer 长度探针的杂交信号没有差异(图 4),阳性对照探针 Y1-Y8、PC 出现阳性杂交信号,阴性对照点样液 NC 未检测到杂交信号,符合质控要求,因此判断芯片杂交实验成功。
2.4 杂交信号强度分析

图4 59-mer和70-mer 长度寡核苷酸探针芯片杂交图Figure4 Hybridization figures of 59-mer and 70-mer oligo probes
为了直观地比较 2 种探针的杂交信号,利用Excel 计算各探针的杂交信号值,并采用 Paired T test 进行显著性分析,结果表明 59-mer和70-mer长度探针的杂交效率和杂交信号差异无统计学意义(P=0.9810)(图 5)。
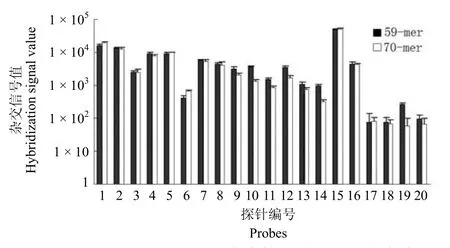
图5 59-mer和70-mer 长度寡核苷酸探针的杂交信号强度Figure5 The hybridization signal intensities of 59-mer and 70-mer oligo probes
3 讨论
以不同长度 Oligo 探针基因芯片进行基因表达检测最主要的影响因素是探针的杂交信号,若杂交信号弱,会导致检测信息丢失,出现假阴性。已有研究表明,短探针的杂交信号会较长探针弱,并且稳定性差[12-13],例如美国 Affymetrix公司制备的基因芯片探针长度只有 25-mer,得到的杂交信号较弱,为了弥补该缺陷,减少由于探针杂交信号弱导致假阴性的出现,他们针对一个基因设计了多条探针,并且分为完全匹配和错配的探针,利用多条探针的杂交信号值检测丰度很低的基因。Affymetrix公司设计完全匹配和错配的多条探针检测基因表达的技术在一般实验室难以进行,而其他的芯片公司,例如美国 Agilent和Roche NimbleGen公司都是利用 60-mer 以上长度的探针检测包括原核和真核生物细胞的基因表达信息,这样设计的芯片不用考虑设计完全匹配和错配等多条探针,通过设计一条可靠的探针就能检测到一个基因的表达信息,这种Oligo 探针基因芯片设计方法在一般实验室都可以实现。但在国外已有的研究中都没有考虑到探针合成的成本问题;而在国内,60-mer 长度就是探针合成长度的一个分界线,价格差异很大,长度在60-mer 以上探针的合成费用是 60-mer 以下探针的3 倍以上,例如目前国内 59-mer 长度探针的合成价格约为每个碱基 1.4 元,而 70-mer 长度探针的合成价格约为每个碱基 4.0 元。因此,结合探针长度对于杂交信号的影响以及探针的合成成本,我们对 59-mer和70-mer 长度探针对杂交信号强度的影响进行了分析比较。
在本实验中,我们选择了基因表达检测难度较大的原核生物大肠杆菌的基因表达为模型,筛选出覆盖高、中、低杂交信号强度的20个大肠杆菌基因,比较了 59-mer和70-mer 长度探针的杂交信号,结果表明 59-mer和70-mer 长度探针的杂交信号没有显著差异。而对于真核生物,已有研究表明 50~70-mer 长度的Oligo 探针均具有良好的特异性和灵敏度[14-17],我们也成功应用 59-mer 长度探针检测了定制的60个大鼠基因的表达信息[18],因此 59-mer 长度探针也可以用于检测真核生物的基因表达信息。综上,59-mer 长度探针合成费用不仅远远低于70-mer 长度探针,而且杂交信号强度也没有明显降低,可用于制备具有高性价比优势的寡核苷酸基因芯片,从而推动基因芯片技术更为广泛的应用。
[1]Schena M, Shalon D, Davis RW, et al.Quantitative monitoring of gene expression patterns with a complementary DNA microarray.Science, 1995, 270(5235):467-470.
[2]Stears RL, Martinsky T, Schena M.Trends in microarray analysis.Nat Med, 2003, 9(1):140-145.
[3]Fodor SP, Read JL, Pirrung MC, et al.Light-directed, spatially addressable parallel chemical synthesis.Science, 1991, 251(4995):767-773.
[4]Singh-Gasson S, Green RD, Yue Y, et al.Maskless fabrication of light-directed oligonucleotide microarrays using a digital micromirror array.Nat Biotechnol, 1999, 17(10):974-978.
[5]Schena M, Shalon D, Heller R, et al.Parallel human genome analysis:microarray-based expression monitoring of 1000 genes.Proc Natl Acad Sci U S A, 1996, 93(20):10614-10619.
[6]Schena M.Genome analysis with gene expression microarrays.Bioessays, 1996, 18(5):427-431.
[7]Patterson TA, Lobenhofer E, Fulmer-Smentek SB, et al.Performance comparison of one-color and two-color platforms within the MicroArray Quality Control (MAQC) project.Nat Biotechnol, 2006,24(9):1140-1150.
[8]MAQC Consortium, Shi L, Reid LH, et al.The MicroArray Quality Control (MAQC) project shows inter- and intraplatform reproducibility of gene expression measurements.Nat Biotechnol, 2006, 24(9):1151-1161.
[9]Guo Y, Guo H, Zhang L, et al.Genomic analysis of anti-hepatitis B virus (HBV) activity by small interfering RNA and lamivudine in stable HBV-producing cells.J Virol, 2005, 79(22):14392-14403.
[10]Shi YH, Zhu SW, Mao XZ, et al.Transcriptome profiling, molecular biological, and physiological studies reveal a major role for ethylene in cotton fiber cell elongation.Plant Cell, 2006, 18(13):651-664.
[11]Xiang G, Pan L, Xing W, et al.Identification of activity-dependent gene expression profiles reveals specific subsets of genes induced by different routes of Ca(2+) entry in cultured rat cortical neurons.J Cell Physiol, 2007, 212(1):126-136.
[12]Bates SR, Baldwin DA, Channing A, et al.Cooperativity of paired oligonucleotide probes for microarray hybridization assays.Anal Biochem, 2005, 342(1):59-68.
[13]Chou CC, Chen CH, Lee TT, et al.Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression.Nucleic Acids Res, 2004, 32(12):e99.
[14]Liebich J, Schadt CW, Chong SC, et al.Improvement of oligonucleotide probe design criteria for functional gene microarrays in environmental applications.Appl Environ Microbiol, 2006, 72(2):1688-1691.
[15]He Z, Wu L, Fields MW, et al.Use of microarrays with different probe sizes for monitoring gene expression.Appl Environ Microbiol, 2005,71(9):5154-5162.
[16]He Z, Wu L, Li X, et al.Empirical establishment of oligonucleotide probe design criteria.Appl Environ Microbiol, 2005, 71(7):3753-3760.
[17]Kane MD, Jatkoe TA, Stumpf CR, et al.Assessment of the sensitivity and specificity of oligonucleotide (50mer) microarrays.Nucleic Acids Res, 2000, 28(22):4552-4557.
[18]Wang XY, Li XL, Hong SQ, et al.Electroacupuncture induced spinal plasticity is linked to multiple gene expressions in dorsal root deafferented rats.J Mol Neurosci, 2009, 37(2):97-110.
