儿童过敏性紫癜口腔菌群及其代谢产物特征
2024-05-18王晴雯张淑雅熊维霖胡晓磊李紫薇郭庆寅
王晴雯 张淑雅 熊维霖 胡晓磊 李紫薇 郭庆寅
1河南中医药大学 (郑州 450003);2河南中医药大学第一附属医院 (郑州 450003)
过敏性紫癜(HSP)是儿童最常见的系统性小血管炎,30% ~ 50%的HSP 会进展为过紫癜性肾炎(HSPN)[1],少数进展为终末期肾病[2]。HSP 的主要触发因素是黏膜感染[3],尤其是呼吸道黏膜[4]。黏膜菌群组成和代谢产物与感染密切相关[5-6]。之前的研究中没有结合代谢产物,且未将这二者关联起来。因此,本研究以唾液为研究样本通过16S rRNA 测序与代谢组学相结合的方法,整合探究HSP 的发病机制,为临床研究提供新的诊断与治疗方向。
1 资料与方法
1.1 研究对象 收集河南中医药大学第一附属医院2022 年1 - 7 月儿科收治的初发HSP 患儿20 例、初发HSPN 患儿20 例,同时征集20 例健康正常儿童作为对照组。纳入标准:各组年龄为5 ~ 12 岁;病程< 4周者。HSP患儿符合《儿童过敏性紫癜循证诊治建议》[7]诊断标准。HSPN 患儿符合《紫癜性肾炎诊治循证指南》[8]诊断标准。对照组为同期在我院体检的健康儿童。排除标准:2 周前使用激素、免疫抑制剂、微生态制剂治疗者;心脑血管、肺脏等脏器严重病变及精神疾病者;过敏性鼻炎、鼻窦炎、龋齿和其他口腔疾病患者。所有参与儿童的监护人均签署知情同意书;该研究已得到河南中医药大学第一附属医院伦理委员会的批准(编号:2020HL-101-01)。因16S rRNA测序中HSPN组1例样品质量不合格而排除。各组临床特征差异无统计学意义(P> 0.05),见表1。
表1 各组人口学基线特征比较Tab.1 Comparison of demographic baseline characteristics among groups ±s

表1 各组人口学基线特征比较Tab.1 Comparison of demographic baseline characteristics among groups ±s
项目性别(例)男女年龄(岁)身高(cm)体质量(kg)对照组(n = 20)14 6 8.10 ± 1.89 132.25 ± 7.23 30.75 ± 4.89 HSP组(n = 20)13 7 7.35 ± 2.16 127.15 ± 10.70 28.65 ± 4.83 HSPN组(n = 19)11 8 8.74 ± 2.28 130.37 ± 8.25 31.79 ± 8.54统计值0.210 1.931 1.446 1.364 P值0.895 0.132 0.236 0.260
1.2 标本采集 采集唾液前2 h 内禁食,收集唾液3 ~ 5 mL。离心后置于-80 ℃超低温冰箱。
1.3 16S rRNA 测序 采用特异性引物扩增16S rRNA/18S rRNA/ITS 基因。用Qiagen Gel Extraction Kit(Qiagen)纯化PCR 产物,应用TruSeq®DNA PCR-Free Sample Preparation Kit(Illumina)构建文库,于Illumina NovaSeq6000 平台系统上机测序,最后对OTU 序列进行物种注释,丰富度(Chao1、Observed_species)及多样性(Simpson 指数),PCoA 降维分析,LDA 效应量及ANOSIM 和MRPP 分析。
1.4 代谢组学多元数据分析 经过代谢物提取,采用超高效液相色谱仪及液相色谱柱进行色谱分离,最后采用软件Simca(V16.0.2)对数据处理和分析。采用正交偏最小二乘判别分析来分析结果。差异代谢物的筛选卡值标准是studentt检验。采用京都基因与基因组百科全书(KEGG)的通路数据库(http://www.kegg.jp/kegg/pathway.html)筛选通路,找到与HSP 中各组之间差异性代谢物相关的关键通路。
1.5 相关性分析 对从唾液16S rRNA 测序中筛选出的菌属和从唾液代谢组学中筛选出的差异性代谢产物进行Spearman 相关分析。
1.6 统计学方法 采用SPSS 26.0软件。符合正态分布且方差齐的计量资料,采用t检验分析组间差异;不完全符合正态分布或方差不齐的计量资料,采用两独立样本Wilcoxon 秩和检验。以P< 0.05为差异有统计学意义。
未加权和加权的UniFrac 距离,使用PCoA 评估了3 组之间β 多样性,并区分菌群结构组间差异。采用基于秩检验的LEfSe分析(LDA评分> 4)及t检验区分各组的口腔菌群。t检验区分各组的口腔差异代谢产物,气泡图显示各组口腔差异代谢通路。Spearman 相关分析菌群和代谢产物间的相关性。以P< 0.05为差异有统计学意义。
2 结果
2.1 口腔菌群的丰富性和多样性 HSPN组丰富度高于HSP组,而多样性显著低于HSP组(P< 0.05);HSPN 组丰富度高于对照组,而多样性显著低于对照组(P< 0.05);HSP 组丰富度和多样性低于对照组,但差异无统计学意义(P> 0.05)。见表2。
表2 各组丰富度和多样性比较Tab.2 Comparison of richness and diversity among groups ±s

表2 各组丰富度和多样性比较Tab.2 Comparison of richness and diversity among groups ±s
组别HSPN组HSP组健康对照组Chao1 880.85 ± 420.40 478.94 ± 253.91 508.97 ± 256.70 Observed_species 686.68 ± 328.06 387.40 ± 153.15 424.55 ± 195.32 Simpson 0.897 ± 0.059 0.930 ± 0.018 0.937 ± 0.022
2.2 口腔菌群的特征 比较3 组之间门到属水平的口腔菌群相对丰度在前10 的菌群。门水平的菌群组成表明,Firmicutes(厚壁菌门)、Proteobacteria(变形菌门)、Bacteroidetes(拟杆菌门)和Fusobacteriota(梭杆菌门)在各组中均为绝对优势菌门,占总序列所有菌群的90%以上。属水平上,Streptococcus(链球菌属)、Prevotella(普雷沃氏菌属)和Neisseria(奈瑟氏菌属)在四组中均为优势菌属,三者总和占所有菌群的40%以上,链球菌属最为丰富,见图1。

图1 各组菌群相对丰度及OTUs 的比较Fig. 1 Comparison of relative abundance of flora and OTUs in each group
2.3 口腔菌群的结构 PCoA 结果表明,各组菌群都有较强的聚集性,见图2。并分析口腔菌群结构组间差异,见表3。

图2 各组之间菌群的PCoA 分析Fig. 2 PCoA analysis of flora between groups

表3 口腔菌群结构组间差异分析Tab. 3 Analysis of differences between oral flora structure groups
2.4 口腔菌群差异分类丰度 LEfSe 分析结果表明在属水平上,HSPN 组中假单胞菌属显著高于HSP 组和对照组(P< 0.05),见图3。HSPN 组中假单胞菌属和Parabacteroides(副拟杆菌属)显著高于HSP 组和对照组(P< 0.05),见图4。
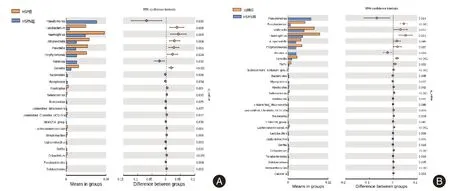
图4 t 检验确定HSPN 组与HSP 组及对照组间的属水平差异物种Fig. 4 Species with genus level difference between HSPN group, HSP group and control group determined by t-test
2.5 口腔代谢产物分析 HSPN 组与HSP 组对比,见表4。HSPN 组与对照组对比,见表5。HSP组与对照组对比,未筛选出主要的差异性代谢产物,无代谢通路。

表4 HSPN 组与HSP 组差异性代谢途径的筛选结果Tab.4 Screening results of differential metabolic pathways between HSPN group and HSP group
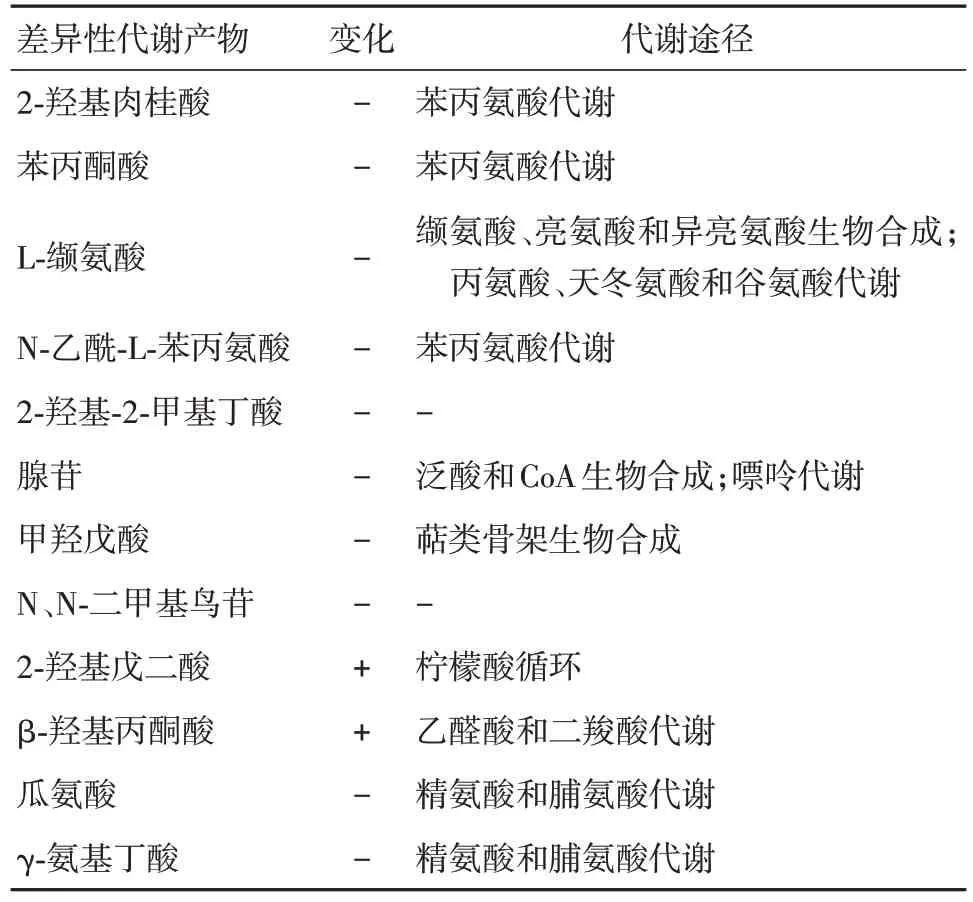
表5 HSPN 组与对照组差异性代谢途径的筛选结果Tab.5 Screening results of different metabolic pathways between HSPN group and control group
2.6 口腔代谢通路分析 HSPN 组与HSP 组对比,苯丙氨酸代谢通路尤为显著;HSPN 组与对照组对比,苯丙氨酸代谢通路尤为显著,见图5。HSP 组与对照组对比,无代谢通路。
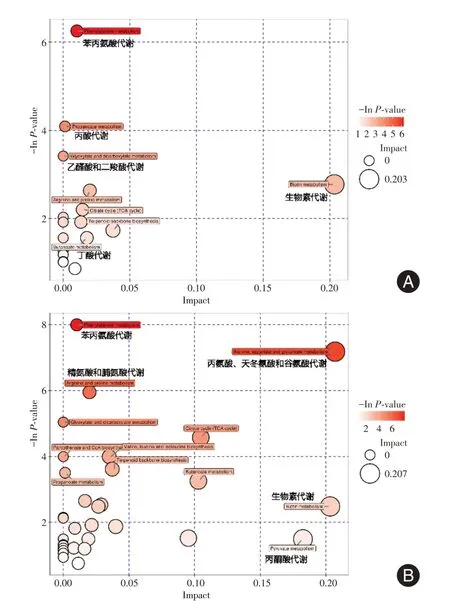
图5 组间代谢通路分析Fig. 5 Analysis of metabolic pathways between groups
2.7 口腔菌群与代谢产物的相关性分析 HSPN
组与HSP 组中假单胞菌属、副拟杆菌属与苯丙氨酸代谢途径产物呈负相关;HSPN 组与对照组中假单胞菌属、副拟杆菌属与苯丙氨酸代谢途径产物呈负相关,见表6、7。其中苯丙氨酸代谢途径产物包括2-羟基肉桂酸、苯丙酮酸、N-乙酰基-L-苯丙氨酸。
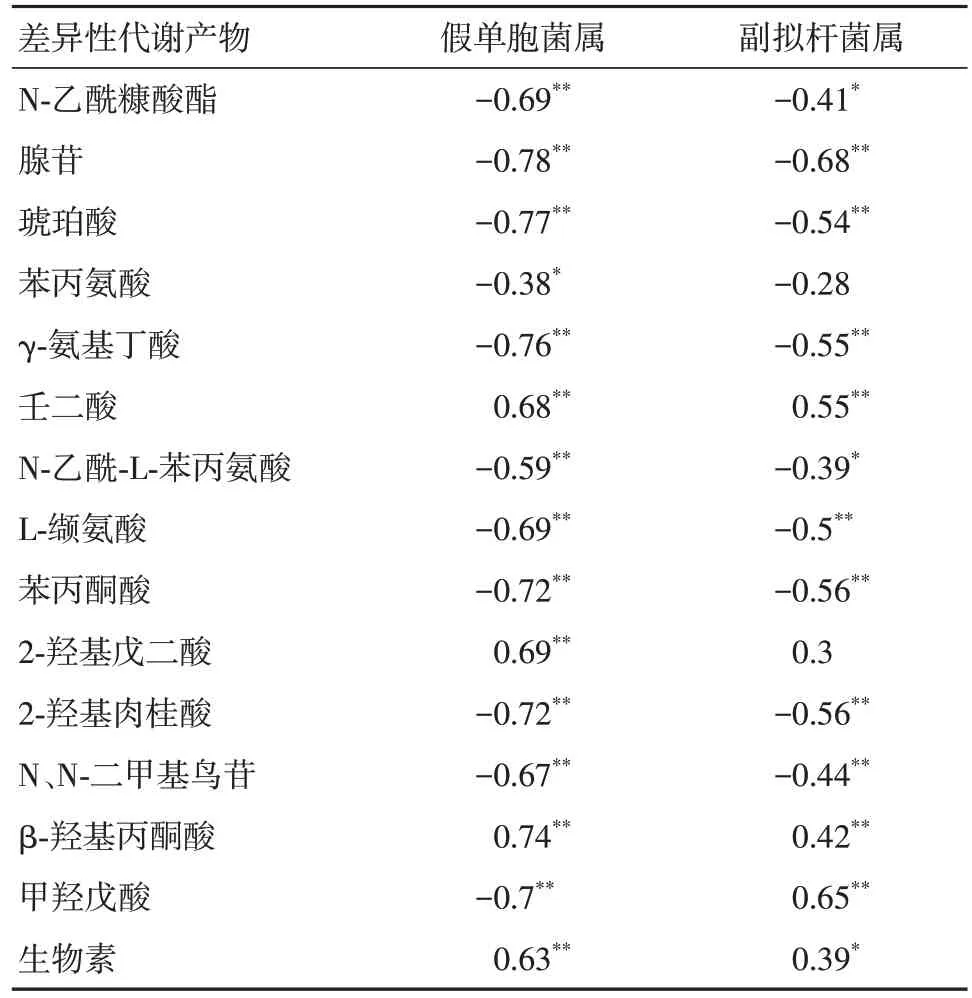
表6 HSPN 组与HSP 组间口腔菌群与代谢产物的相关性Tab.6 Correlation between oral flora and metabolites between HSPN group and HSP group

表7 HSPN 组与健康对照组组间口腔菌群与代谢产物的相关性Tab. 7 Correlation between oral flora and metabolites between HSPN group and healthy control group
3 讨论
已有研究表明,HSP 与口腔中的菌群的丰富度和多样性有关。PANG 等[6]发现,HSPN 患儿舌苔上的菌群丰富度和多样性明显低于健康对照组,此结果与本研究结果一致。口腔菌群随着唾液吞咽定植在消化道,口腔和肠道中的菌群与疾病状态有关系[5,9-10]。而且研究[11-12]表明,肠道和口腔菌群之间出现一致性,表明不同身体部位物种的丰度和功能存在重叠。与对照组相比,HSPN组儿童在口腔菌群中表现出更低的多样性这与既往在肠道菌群中的发现部分一致,更趋近肠道菌群的失衡;与对照组相比,HSP 组儿童在口腔菌群结构组成差异不明显和多样性差异不大,这可能是无肾脏损伤的HSP 菌群失调不明显。这与既往研究在肠道菌群中的发现不同,后者表明患有HSP 的儿童中α 多样性减少,结构组成差异明显[11-12]。且ZHOU等[14]发现,HSP或HSPN儿童表现出肠道微生物群多样性差异显著。
在本研究中,α 多样性分析表明了HSP 发病与口腔中的菌群有关。且HSPN 比无肾脏损伤的HSP 菌群失调更加明显。相比于健康儿童,HSP初发期患儿口腔菌群的整体变化小,这可能是患儿发病后短时间内口腔菌群因时间较短还未出现明显改变。口腔微生物区系属水平上组成差异,与健康儿童和HSP 相比,HSPN 患儿中链球菌属升高但无统计学意义,假单胞菌属和副拟杆菌属差异有统计学意义。链球菌是口腔群落的优势属之一,也是存在的口腔潜在病原体菌群。链球菌水平越高的HSPN 患者更容易出现血尿和低蛋白血症,且HSPN 患者肠道微生物群失调明显,肠黏膜链球菌感染明显,与严重程度密切相关[15]。已知嗜血杆菌属与链球菌属是竞争共存关系,两者只有一种细菌占优势[16]。假单胞菌属在肠道和口腔多样性降低与多种自身免疫性疾病相关[17]。链球菌属和假单胞菌属的细菌通过分泌胞外多糖等物质形成的生物被膜附着在黏膜表面,引起黏膜感染[17]。副拟杆菌属是传染病中的机会致病菌属,能够产生抗菌药物耐药性。已有研究[13]表明,Parabacteroidesspp.丰度与肠道通透性相关的主要紧密连接蛋白(occludin 和ZO-1)的结肠mRNA 水平呈负相关,HSPN 患者的肠道通透性增加。链球菌属、假单胞菌属、副拟杆菌属这3 种菌属的相对丰度增加可能与HSPN 发病相关。与HSP 组相比,HSPN 组菌群结构组成变化明显且多样性降低,这表明HSPN 致病菌群增多且增殖更快,菌群出现明显的紊乱,HSPN 进展较迅速且严重。
通过代谢组学和代谢通路研究分析表明,HSPN 组与HSP 组及对照组间的口腔代谢产物有明显的差异,主要集中于苯丙氨酸代谢相关的代谢产物,如2-羟基肉桂酸、苯丙酮酸、N-乙酰基-L-苯丙氨酸,这与本研究中的差异性菌群有相关性。本研究结果表明,假单胞菌属、副拟杆菌属与苯丙氨酸代谢途径产物呈负相关。其中N-乙酰-L-苯丙氨酸可由细菌中生物酶催化L-苯丙氨酸和乙酰辅酶A 合成,在微生物中对防御、运动和代谢很重要,可在细菌争夺有限资源的斗争中作为抗菌药物[18]。2-羟基肉桂酸是一种邻香豆酸,具有抗氧化和抗菌等生物活性。它是由细菌通过代谢苯丙氨酸解氨去氧产生的[19]。苯丙酮酸能显著降低葡萄糖-6-磷酸脱氢酶活性,引起NADPH 产生的损害并最终改变细胞氧化还原状态[19]。这3 种物质产生减少,口腔的抗氧化和抗菌环境减少的变化表明优势菌群相对减少,潜在致病菌相对增加。由此推测,苯丙氨酸代谢的产物2-羟基肉桂酸、苯丙酮酸、N-乙酰基-L-苯丙氨酸可作为HSPN 发病过程中的代谢产物。
在本研究中,通过使用16S rRNA 测序和代谢组学方法分析不同类型的HSP 患儿唾液样本中的细菌物种和代谢产物。这一研究表明HSP 的发病可能与口腔微生态的失调相关。唾液样本中属水平上链球菌属、假单胞菌属、副拟杆菌属优势可能作为HSPN 发病的触发因素之一,苯丙氨酸代谢可能是HSPN 发病过程中的通路之一。这为临床诊断HSP 疾病提供了指导,并且为临床治疗HSP 疾病提供新思路和新切入点。
【Author contributions】WANG Qingwen performed the experiments and wrote the article. GUO Qingyin designed the study and revised the article. ZHANG Shuya and LI Ziwei performed part of the experiments. XIONG Weilin, HU Xiaolei revised the article. All authors read and approved the final manuscript as submitted.
【Conflict of interest】The authors declare no conflict of interest.
