m1A/m5C/m6A/m7G调控基因预测胃癌预后及免疫关联性
2024-05-18陈小梅王安奇杨积祯于淼
陈小梅 王安奇 杨积祯 于淼
甘肃省人民医院1国家胃肠肿瘤诊治重点实验室,2临床研究与转化医学研究所,3超声医学科(兰州 730000)
目前,胃癌患者的预后主要通过检查病理分型和临床分期来评估。早期发现、及时适当的干预、合理的预后评价是影响胃癌预后的关键因素。因此,探索与胃癌显著相关的独特生物分子对于提高患者的长期生存至关重要。研究[1]表明,表观遗传调控在间充质肿瘤的发生中起重要作用。由于其对基因表达的显著影响,RNA 修饰近年来受到越来越多的关注。甲基化是已知RNA 修饰中最常见的,包括n1-甲基腺苷(m1A)、5-甲基胞嘧啶(m5C)、n6-甲基腺苷(m6A)和7-甲基鸟苷(m7G)[2]。作为RNA 修饰研究的热点,m1A、m5C、m6A、m7G甲基化修饰在多种生物过程中发挥重要作用[3]。异常甲基化与人类癌症进展有关,特别是在胃癌中[4]。
胃癌在分子和表型上都是一种高度异质性的恶性肿瘤。肿瘤微环境除肿瘤细胞外,还含有细胞外基质和基质细胞、免疫细胞等多种细胞,共同构成肿瘤免疫微环境(tumor immune microenvironment, TIME)。癌症中有许多重要的过程导致了TIME 的复杂性,包括不受控制的癌细胞增殖、代谢和血管发育缺陷[5-6]。TIME类似于肿瘤免疫促进和抑制的战场,极大地影响免疫治疗的反应性,基于甲基化调控基因的基因表达特征可能是预测癌症患者免疫检查点阻断(immune checkpoint blockade, ICB)治疗反应的有力方法[7]。为了充分阐明m1A/m5C/m6A/m7G 调控基因在胃癌进展中的作用及其对TIME 的影响,这项研究分析了来自TCGA和GEO 数据集的707 个胃癌样本的基因组变化,建立了基于m1A/m5C/m6A/m7G 调控基因的新型预后RS 模型,检验其对胃癌患者预后的预测能力,并进一步分析该模型与TIME 的关联性。
1 资料与方法
1.1 数据收集和预处理 TCGA-胃癌数据集(n=407)来自UCSC-Xena 数据库,包括具有临床信息的32 个正常对照样本和350 个胃癌样本,作为训练数据集。GSE62254 数据集(n= 300)可从Gene Expression Omnibus (GEO)数据库获得[8],此数据集被指定为验证数据集。
1.2 鉴定m1A/m5C/m6A/m7G 调控基因在胃癌中的差异表达 从文献中获得m1A/m5C/m6A/m7G 调控基因[9-10]。对于训练数据集,使用R4.2.1中的“limma”软件包 (v3.54.1)来识别正常和胃癌样本之间的差异表达基因(differentially expressed genes, DEGs),选择FDR < 0.05 和|Log2 FC| > 0.5作为筛选阈值;采用“cor”函数计算DEGs 之间的相关性。
1.3 RS 模型的建立与验证 使用R4.2.1 中的survival包(v3.5-0),进行单因素Cox回归分析,选取P< 0.05 作为筛选显著相关的阈值,筛选与疾病预后显著相关的基因。利用lars 包(v1.3)中的LASSO算法对目标基因集进行回归分析,筛选最佳基因组合。最后,根据各基因的LASSO 预后系数和表达水平,构建RS模型:Risk Score(RS) = ∑Coefgenes×Expgenes。Coefgenes为靶基因的LASSO 预后系数,靶基因的表达量用Expgenes表示。分别计算TCGA-胃癌和GSE62254 数据集的RS 值,并以RS 中值为界将样本分为低、高风险两个风险组。在R4.2.1中使用survival 包(v3.5-0)的Kaplan-Meier 曲线统计方法对模型可靠性进行验证。
1.4 建立具有独立生存预后因素的nomogram 生存模型 在R4.2.1 中使用survival 包(v3.5-0)对样本的临床因素进行单因素和多因素Cox 回归分析,检测显著相关性的阈值均设置为P< 0.05。将上述步骤得到的独立预后因素与预后RS 模型判别出的风险信息结合使用R4.2.1 中rms 包(v6.2-0)构建nomogram 生存模型。使用R4.2.1 中的survcomp 包(v1.48.0)计算nomogram预后模型的C-index系数。
1.5 胃癌细胞与正常胃细胞的培养 人胃癌细胞系AGS、HGC27、MKN45 和SNU1 来源于武汉普诺赛生命科学技术有限公司,人胃黏膜上皮细胞系(GES-1)由上海赛百慷生物科技有限公司提供。培养条件均为含10%胎牛血清的RPMI-1640 培养基,37 ℃, 5% CO2。
1.6 RT-qPCR 验证 使用Trizol 试剂(批号:15596018,Ambion, Life Technologies,USA)从细胞系中提取总RNA。使用FastKing gDNA RT 试剂盒(批号:KR118,天根生物科技有限公司,中国)进行逆转录合成cDNA。使用TB Green Premix DimerEraser (批号:RR037A,TaKaRa,日本)和Quant-Studio DX PCR 仪(Applied Biosystems, Inc.,美国)进行qPCR 分析。引物由上海生工提供。
1.7 免疫基因景观分析 采用CIBERSORT 算法计算TCGA-胃癌数据集中22 个免疫细胞的比例,并评估两组之间免疫细胞亚群分布的差异。在R4.2.1 中使用estimate 包来计算比较两组中各免疫评分分布的差异,使用ggpubr 包分析两组中免疫检查点基因的表达差异。
1.8 统计学方法 所有分析均使用R语言(v4.2.1)进行。采用学生t检验和χ2检验进行配对比较。对于两组以上的方差,采用单因素方差分析或Kruskal-Wallis 检验。为了评估变量之间的关系,采用Spearman 相关检验。P< 0.05 为差异有统计学意义。
2 结果
2.1 差异基因识别 在训练集中鉴定出29个差异表达的m1A/m5C/m6A/m7G 调控基因(P< 0.005)(图1)。
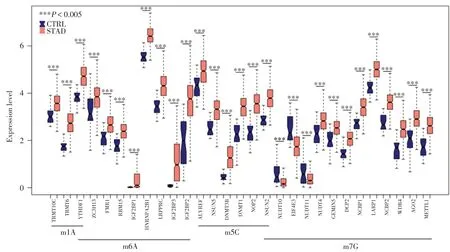
图1 胃癌患者m1A/m5C/m6A/m7G 调控基因DEGs 的筛选Fig.1 Screening for DEGs of m1A/m5C/m6A/m7G regulated genes in GC patients
2.2 建立m1A/m5C/m6A/m7G 调控基因相关RS模型 单因素Cox回归分析产生11个与生存有显著预后相关性的基因(图2A),之后利用LASSO算法获得8 个最佳组合基因(图2B)。利用TCGA-胃癌数据集中8 个基因及其表达量的LASSO 回归系数,构建RS 公式:Risk score = (-0.003627555) × ExpTRMT10C+ (-0.030350865) × ExpTRMT6+ (-0.100825842) ×ExpRBM15+ (-0.025329733) × ExpWDR4+ (-0.015785251)× ExpHNRNPA2B1+ (0.057529045) × ExpNUDT10+(0.076060498) × ExpNUDT11+ (0.146531852) × ExpDCP2。

图2 森林图描绘了与预后有显著关系的11 个基因Fig.2 A forest plot depicting the 11 genes that have a significant relationship with prognosis
2.3 RS模型的效能评价 Kaplan-Meier曲线算法分析表明,在验证集(HR= 1.805,P= 0.000 53;图3A)和训练集(HR= 1.779,P= 0.000 44;图4A) 中,患者OS 与分组状态之间显著相关。图3B 和图4B 分别显示了两个数据集中两个风险组的RS 值和生存时间分布。图3C 和4C 显示了基于RS 模型的受试者工作特征(ROC)曲线的结果,TCGA-胃癌数据集的AUC 为 0.863(95%CI:0.724 ~ 0.858,P<0.001);GSE62254 数据集的AUC 为 0.810(95%CI:0.621 ~ 0.847,P< 0.001)。
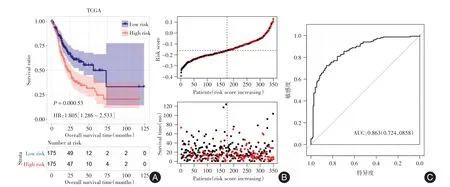
图3 TCGA 训练数据集Fig.3 TCGA training dataset.

图4 GSE62254 验证数据集Fig.4 GSE62254 validation dataset.
2.4 Nomogram 生存模型的建立 单因素和多因素Cox 分析表明,RS 模型、放疗、肿瘤复发、病理分期和年龄是与OS 相关的独立预后因素(表1)。构建的nomogram 生存模型可以很好的预测胃癌患者的生存期(图5A),校准曲线(图5B)显示通过nomogram 生存模型预测的1 年(C-index=0.703)、3 年(C-index=0.729)和5 年(C-index=0.734)生存率与实际生存率的一致性较好(P< 0.001)。

表1 临床预后因素与预后的相关性分析Tab.1 Analysis of the correlation between clinical prognostic factors and prognosis

图5 构建生存预测nomogram 模型并评估其预测能力Fig.5 Creating a survival prediction nomogram and assessing its predictive power
2.5 验证RS模型基因在胃癌细胞系中的表达 与胃黏膜细胞相比,胃癌细胞中DCP2、HNRNPA2B1、NUDT10、NUDT11、RBM15、TRMT10C、TRMT6 和WDR4 的mRNA 表达水平显著升高(P< 0.05)(图6)。

图6 利用RT-qPCR 方法验证8 个优化基因在胃癌细胞系中的mRNA 表达水平Fig.6 Verifying mRNA expression levels of the 8 optimized genes in GC cell lines using RT-qPCR method
2.6 免疫浸润模式的鉴定 免疫细胞浸润分析(图7A)共筛选到8 种免疫细胞(记忆B 细胞、滤泡辅助T 细胞、调节性T 细胞、活化肥大细胞、静息肥大细胞、巨噬细胞M0、巨噬细胞M1、巨噬细胞M2)在各组间的分布具有显著差异(P< 0.05)。各组之间的ESTIMATE 评分亦具有显著差异(P<0.001)(图7B)。图7D显示了8个RS模型基因与免疫细胞和ESTIMATE评分之间的相关性。免疫检查点基因差异分析结果显示,高危组有14 个免疫检查点基因(BTLA、CD200、CD200R1、CD226、CD28、CD40LG、CD44、CD48、CD86、CD96、HAVCR2、IDO2、LAIR1、NRP1、PDCD1LG2、TIGIT、TNFSF14、TNFSF4)上调,而低危组只有3 个免疫检查点基因(LGALS9、PVR、TNFRSF12A)上调(图7C)。

图7 风险分组与免疫浸润模式的相关性分析Fig.7 Analysis of correlations between risk grouping and immune infiltration patterns.
3 讨论
最近的证据表明,活跃的RNA 甲基化在癌症进展中起重要作用,并可能成为癌症患者的新药靶点[11]。然而,对胃癌中RNA 甲基化的研究主要集中在少数RNA 甲基化调控基因上,特别是m6A相关的调控基因,其他类型的RNA 修饰及其综合作用尚不清楚。尽管许多研究使用生物标志物预测胃癌预后,但没有研究将m1A/m5C/m6A/m7G 调节基因作为预后生物标志物。因此,建立基于m1A/m5C/m6A/m7G 调控基因的胃癌预后RS 模型至关重要,且有助于阐明胃癌TIME 的特征和开发新的胃癌治疗策略。
本研究利用TCGA-胃癌数据集构建并验证了m1A/m5C/m6A/m7G 调控基因相关RS 模型。与正常样本相比,胃癌样本中DCP2、WDR4、HNRNPA2 B1、NUDT10、NUDT11、RBM15、TRMT10C和TRMT6基因表达显著上调,这8 个基因的mRNA 表达水平亦在胃癌细胞株中得到了验证。HNRNPA2B1作为m6A 结合蛋白之一,通过抑制细胞凋亡、促进细胞增殖和增加细胞迁移侵袭,参与维持胃癌的恶性表型[12]。HNRNPA2B1 高表达的胃癌患者预后不良[12]。RBM15 是m6A 甲基转移酶之一,在喉癌和肝细胞癌中被发现显著升高[13]。作为m1A甲基化的编辑器,TRMT6 和TRMT10C 参与妇科癌症的恶性行为,并与肝细胞癌患者的不良预后相关[10,14]。DCP2 是第一个也是研究最广泛的真核解旋酶,通过影响细胞迁移和凋亡等过程参与癌症的发病机制[15]。作为Nudix 蛋白家族(NUDT)的成员,NUDT10 与胃癌的TNM 分期、淋巴结转移和局部浸润深度显著相关[17];NUDT11 可促进结肠癌、前列腺癌、乳腺癌和肺癌的细胞增殖[18]。WDR4 是一种m7G 甲基转移酶,目前已知WDR4在多种恶性肿瘤中作为肿瘤启动子[19]。因此,这八个RS 模型基因与肿瘤密切相关,它们在胃癌中的作用机制值得深入研究。这项研究中,低危组和高危组的临床结局存在显著差异,表明该RS 模型能够有效预测胃癌预后。此外,K-M 验证结果表明,该RS 模型在训练集和验证集中的5 年AUC分别为0.863 和0.810,表明其在长期随访中具有更好的预测价值,高于近期一些研究[20-21]。
近年来,免疫浸润评估在肿瘤治疗和预后预测中越来越重要。免疫疗法在很大程度上依赖于TIME。对接受ICB 治疗的患者的回顾性分析显示,对ICB 有反应的肿瘤与其TIME 模式相关[22]。有研究发现M2 巨噬细胞与肿瘤进展密切相关,M2 巨噬细胞有助于胃癌腹膜播散[23]。肥大细胞激活特征与胃癌患者肿瘤进展加快和患者生存率降低相关[24]。在小鼠胃癌模型中观察到使用肥大细胞稳定剂色甘酸钠可以防止肥大细胞活化,从而有效降低肿瘤生长[25]。这些证据表明丰富的M2 巨噬细胞和活化的肥大细胞浸润对胃癌的临床结局和治疗反应效果有负面影响。相反,一些免疫细胞的丰度与良好的肿瘤预后密切相关,如滤泡辅助性T 细胞和M1 巨噬细胞。M1 巨噬细胞具有促炎特性(如分泌IL-12 和活性氧),从而促进抗肿瘤活性[26]。肿瘤中M1 巨噬细胞的高密度预示着更长的总生存期[25]。滤泡辅助性T 细胞作为主要的IL-21 产生细胞,在实体瘤的免疫应答中发挥积极作用;滤泡辅助性T 细胞通过分泌IL-21 间接增强CD8+T 细胞介导的抗肿瘤免疫[27]。同样,我们的研究结果提示,预后较好的患者有较高水平的滤泡辅助T 细胞浸润和M1 巨噬细胞浸润。这些发现很好地解释了我们研究结果中不同风险组的预后差异,这可能在很大程度上是由于免疫浸润细胞分布的差异。我们还发现在高危组患者中,高ESTIMATE 评分伴随着低水平的肿瘤纯度,提示免疫评分低的胃癌患者比免疫评分高的胃癌患者生存期更长,而肿瘤纯度低的胃癌患者生存期更短。我们的观察结果与GONG 等人和ZENG等人的一致[28-29]。此外,这项研究显示大多数免疫检查点基因在高危组中过度表达。免疫检查点基因的高表达可形成免疫抑制TIME,促进肿瘤免疫逃逸。因此,不同的免疫细胞浸润模式是影响胃癌预后的重要因素。总体来说,m1A/m5C/m6A/m7G调控基因参与了TIME 的重编程。
综上所述,我们基于m1A/m5C/m6A/m7G 调控基因建立的胃癌预后相关的RS 模型展现出优秀的预测能力,同时在免疫治疗和化疗用药评估方面也显示出潜力,为胃癌的预后和治疗提供了新的视角。
【Author contributions】The study protocol was co-created by CHEN Xiaomei and YU Miao. The manuscript was written by CHEN Xiaomei.CHEN Xiaomei and YU Miao worked together to analyze data from public databases. The qRT-PCR experiments were carried out by WANG Anqi and YANG Jizhen. All authors read and approved the final manuscript as submitted.
【Conflict of interest】The authors declare no conflict of interest.
