TMT 定量蛋白质组学解析Rummeliibacillus suwonensis 3B-1 生长及己酸代谢机制
2024-04-17陈晓松刘超杰郑佳乔宗伟罗惠波邹伟
陈晓松 刘超杰 郑佳 乔宗伟 罗惠波 邹伟,2
(1.四川轻化工大学生物工程学院,宜宾 644005;2.中国轻工业浓香型白酒固态发酵重点实验室,宜宾644007;3.宜宾五粮液股份有限公司,宜宾 644007)
白酒是一种传统的蒸馏酒[1],根据风格的不同,可分为三大香型(浓香型、酱香型、清香型)和九种小香型(兼香型、凤香型、米香型、药香型、芝麻香型、豉香型、特香型、老白干香型、馥郁香型[2-3])。浓香型白酒中的风味物质高达1 200 余种[4],己酸乙酯为浓香型白酒的主体香味物质,其含量高低决定了白酒的品质和风格的典型性。己酸菌作为浓香型白酒发酵过程中最为重要的功能微生物之一,其主要代谢产物为己酸,而己酸是合成己酸乙酯的前体物质,因此己酸菌对白酒风味的形成和白酒品质的提高有着重要影响[5]。
己酸的合成依赖于乙醇的逆β-氧化(reverse β-oxidation, RBO)循环羧酸链延长。在该过程中,乙醇或乳酸作为电子供体被氧化为乙酰辅酶A[6-8],然后与乙酸缩合生成丁酸,丁酸再与另一个乙酰辅酶A 缩合生成己酸[9-11]。水源拉梅尔芽孢杆菌首次发现于2013 年[12],是拉梅尔杆菌属物种,前期研究发现,水源拉梅尔芽孢杆菌在厌氧和好氧条件下均能正常生长,且主要代谢产物为己酸,然而该菌在厌氧和好氧条件下的生长与代谢差异及其机制尚未明确。
近年来,蛋白质组学研究方法发展迅速,基于TMT 的定量蛋白质组学技术是目前差异蛋白质定量分析方法中通量最高、系统误差最小、功能最强大的分析方法之一[13],具有高灵敏度、高分离能力、高通量等特点。本研究以R.suwonensis 3B‑1 为研究对象,利用TMT 定量蛋白质组学技术分析该菌株在厌氧和好氧条件下的蛋白质差异,筛选与菌株生长及己酸代谢相关的关键蛋白,在蛋白水平阐明氧气对该菌生长的影响及其己酸代谢的分子机制,以期为水源拉梅尔芽孢杆菌的基因工程改造提供一定技术基础,对提高水源拉梅尔芽孢杆菌的发酵能力具有重要意义。
1 材料与方法
1.1 材料
1.1.1 菌株 R.suwonensis 3B‑1 分离自四川某浓香型酒厂窖泥。
1.1.2 培养基 菌株R.suwonensis 3B‑1 的培养和发酵分别采用乙醇乙酸钠培养基(ES 培养基)和乙醇乙酸钠发酵培养基[14]。
1.2 方法
1.2.1 菌株R.suwonensis 3B‑1 生长曲线的测定 将R.suwonensis 3B‑1 活化好的种子液以5%的接种量接种至ES 培养基中,分别置于35℃厌氧工作站和35℃摇床中(200 r/min)培养,初期每隔12 h 测一次OD600nm值;在生长对数期,每隔6 h 测一次;平稳期每隔2 h 测一次,直至绘制出完整的生长曲线。
1.2.2 菌株R.suwonensis 3B‑1 发酵液的测定 使用气相质谱(GC‑MS)分别测定好氧与厌氧条件下发酵液中丁酸和己酸的含量。将R.suwonensis 3B‑1 种子液接种至ES 液体培养基中,分别置于35℃厌氧工作站和35℃摇床中(200 r/min)培养10 d,完成发酵。取发酵液通过0.22 μm 有机相滤膜收集至EP管内,以5 000 r/min 离心10 min,吸取1 mL 离心后的发酵液于进样瓶进行样品含量的测定。
1.2.3 菌株R.suwonensis 3B‑1 样品的制备 具体的制备步骤如下:(1)吸取厌氧条件下培养且处于对数生长期的9 mL 菌液至10 mL 已灭菌离心管中,转速5 000 r/m,离心5 min 后收集菌体。(2)菌体沉淀后,加入1 mL xPBS 缓冲液(购自Thermo 公司),混合均匀,于7 000 r/min 下离心5 min,收集菌体,重复2-3 次,直至离心后的上清液呈现澄清透明的状态。(3)舍弃上清液,保留沉淀,记录每个样品的名称与编号,将样品放于装满干冰的泡沫箱内,密封后送至上海派森诺公司进行后续实验。
1.2.4 蛋白样品的提取和肽段酶解 (1)采用SDT 裂解法提取样品;(2)采用二喹啉甲酸(bicin‑choninic acid,BCA)法定量蛋白;(3)采用FASP(filter‑aided sample preparation)法进行胰蛋白酶酶解;(4)用C18Cartridge 对肽段进行脱盐;(5)肽段定量(OD280nm)[15]。
1.2.5 TMT 标记 各样品分别取100 μg 肽段,按照Thermo 公司TMT 标记试剂盒说明书进行标记。
1.2.6 基于LC‑MS/MS 的数据采集 采用纳升流速的 HPLC 液相系统 Easy nLC 对每份样品进行分离。缓冲液 A 液为 0.1%甲酸水溶液, B 液为 0.1%甲酸乙腈水溶液(乙腈为 84%)。色谱柱以 95%的 A 液平衡,样品由自动进样器上样到上样柱(Thermo Sci‑entific Acclaim PepMap100, 100 μm×2 cm, nanoVi‑per C18),经 过 分 析 柱(Thermo scientific EASY col‑umn, 10 cm, ID7 5 μm, 3 μm, C18‑A2)分 离,流 速 为300 nL/min。
样品经色谱分离后用 Q‑Exactive 质谱仪进行质谱分析。检测方式为正离子,母离子扫描范围 300-1 800 m/z,一级质谱分辨率为 70 000(200 m/z),AGC(automatic gain control)target 为 1×106,Maxi‑mum IT 为 50 ms,动态排除时间为 60.0 s。多肽和多肽碎片的质量电荷比按照下列方法采集:每次全扫描后采集 20 个碎片图谱(MS2 scan),MS2 activation type 为HCD,isolation window 为2 m/z,二级质谱分辨率17 500(200 m/z),normalized collision energy 为30 eV,underfill 为 0.1%[16]。
1.2.7 蛋白质鉴定与定量分析 采用Proteome Discoverer 1.4 和Mascot 2.2 软件对原始数据文件进行质谱分析,便于查库鉴定及定量分析[17]。
1.2.8 生物信息学分析 将鉴定到的蛋白质序列分别与亚细胞定位、基因本体论(gene ontology,GO)、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)、STRING 或 IntAct 等数据库进行比对,获得蛋白在各数据库的注释信息,分析DEPs 互相作用关系。
2 结果
2.1 菌株生长及代谢情况
如图1‑A 所示,就菌体生长而言,好氧条件下,菌株R.suwonensis 3B‑1 的生长速度明显快于厌氧条件,最大生物量约为厌氧条件下的2 倍,但二者达到最大生长量的时间基本相同;菌株R.suwonensis 3B‑1 的代谢产物产量如图1‑B 所示,在厌氧条件下,该菌株的丁酸、己酸产量都高于好氧条件下的产量,其中,丁酸产量提高了1.14 倍,己酸产量提高了1.97 倍。

图1 3B-1 好氧和厌氧条件生长及代谢情况Fig.1 Growth and metabolism of 3B-1 under aerobic and anaerobic conditions
2.2 蛋白样本质控
经BCA 法测定比较组蛋白浓度,结果如表1 所示,蛋白定量结果显示,好氧条件下(RH1‑RH3)的菌株蛋白总量普遍高于厌氧条件(RY1‑RY3)。取一定量蛋白样本进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis, SDS‑PAGE),经考马斯亮蓝染色,结果如图2‑A 所示,所有样品蛋白完整性较好,条带组间不平行,组内相对平行,条带清晰,蛋白量满足后续实验要求。肽段离子质量偏差分布、肽段离子得分分布分别如图2‑B、2‑C 所示,所有鉴定肽段的质量偏差主要分布在10 ppm 以内,结合严格的分析工具MASCOT 对MS 图谱数据进行分析,图2‑C显示,超过69.37%的肽段得分大于20 分,肽段得分中位数为30 分。
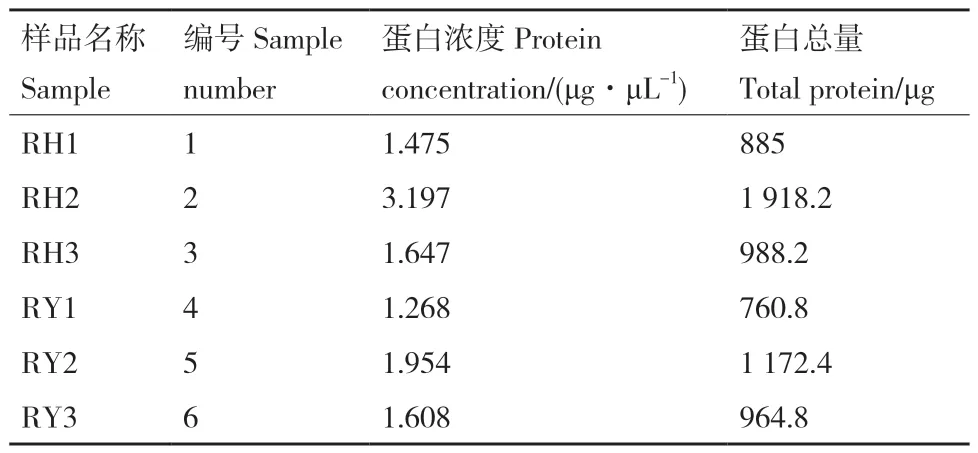
表1 BCA 蛋白定量结果Table 1 BCA protein quantification results

图2 蛋白样品质量控制Fig.2 Quality control of protein sample
2.3 蛋白鉴定及定量分析
由图3‑A 可知,经Mascot 2.2 鉴定和Proteome Discoverer 1.4 定量分析共获得623 624 张总谱图(total spectra)。其中蛋白谱图(spectra)26 634 张;肽段(peptide)数量为7 675 个,其中特有肽段(unique peptide)数量为5 573 个;蛋白(protein)数量为1 804 个,定量到的蛋白数量为1 803 个。以表达倍数(fold change, FC)大于1.2 倍或小于0.83(上调大于1.2 倍或下调小于0.83 倍)且P value<0.05为标准进行比较组间DEPs 筛选,结果见图3‑B,筛选获得总差异表达蛋白810 个,其中显著上调蛋白423 个,显著下调蛋白387。DEPs 火山图如图3‑C所示,其中显著下调的蛋白质标注为蓝色,显著上调标注为红色,无差异则为灰色;采用层次聚类算法(hierarchical cluster)对比较组DEPs 进行分层聚类分析,结果如3‑D 所示,好氧组内(RH‑1、RH‑2和RH‑3)差异蛋白表达量的上调和下调趋势较一致,RH‑1 和RH‑3 先聚为一类,再与RH‑2 聚为一类;厌氧组内(RY‑1、RY‑2 和RY‑3)差异蛋白表达量的上调和下调趋势较一致,与好氧组组间的表达趋势相反,RY‑1 和RY‑2 先聚为一类,再和RY‑3 聚为一类,最终形成与好氧组明显不同的簇。
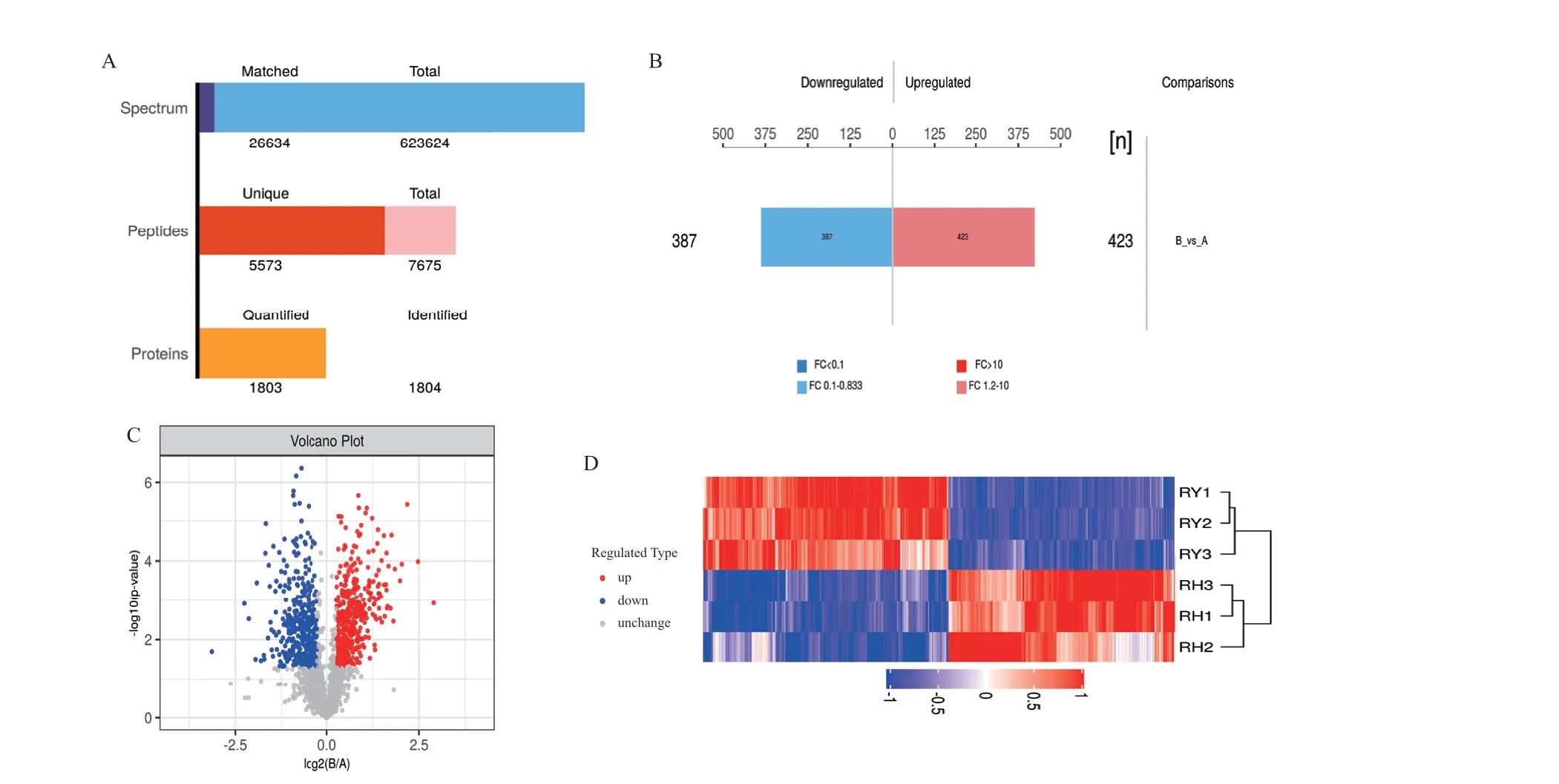
图3 蛋白鉴定和差异表达结果Fig.3 Results of protein identification and differential expression
2.4 差异表达蛋白功能注释
利用亚细胞结构预测软件CELLO 对DEPs 进行亚细胞定位分析,725 个DEPs 定位到6 个条目上,分别是细胞质(cytoplasmic)蛋白567 个,细胞膜(cytoplasmic membrane)蛋白138 个,细胞壁(cellwall)蛋白9 个以及其他蛋白11 个[胞外(extracellular)蛋白6 个,孢子(spore)蛋白3 个和鞭毛(flagellar)蛋白2 个];对DEPs 进行GO 功能注释,结果如图4‑A 所示,生物过程(biological process)方面主要涉及细胞过程(cellular process)蛋白394 个,代谢过程蛋白(metabolic process)373 个,生物调节蛋白(biological regulation)65 个等;分子功能(molecular function)方面主要涉及催化活性(catalytic activity)蛋白478 个,结合功能(binding)蛋白413 个和结构分子活性(structural molecule activity)蛋白55 个等;细胞组分(cellular component)方面主要涉及细胞(cell)蛋白280 个,细胞部分(cell part)蛋白276 个和细胞膜(membrane)蛋白55 个等。对DEPs 进行GO 功能富集分析,GO 三大分类下的GO 条目富集情况结果如图4‑B-D 所示,在该比较组中细胞酰胺代谢过程(cellular amide metabolic process)、肽的生物合成过程(peptide biosynthetic process)、翻译(translation)和肽代谢过程(peptide metabolic process)等生物学过程;核糖体的结构组成(structural constituent of ribosome)、结构分子活性(structural molecule activity)、rRNA、RNA 结 合(rRNA binding、RNA binding)和嘌呤核苷结合(purine nucleoside binding)等分子功能;核糖体(ribosome)、核糖核蛋白复合物(ribonucleoprotein complex)、细胞质部分(cytoplasmic part)和细胞内非膜结合细胞器(intracellular non‑membrane‑bounded organelle)等细胞组分发生了显著变化。

图4 比较组DEPs 的GO 注释图Fig.4 GO annotation map of DEPs in comparative group
2.5 差异表达蛋白KEGG注释
将所有DEPs 进行KEGG 代谢通路注释,结果如图5 所示,其中DEPs 主要参与的通路为核糖体(56 个,且全部上调),辅因子生物合成(44 个,上调25 个,下调19 个),双组分系统(30 个,上调8个,下调22 个)和ABC 转运系统(24 个,上调13个,下调11 个)等途径。在碳水化合物代谢中,厌氧条件显著提高了磷酸戊糖代谢通路中蛋白的表达,其中有10 个DEPs 的表达量上调,6 个下调;淀粉和蔗糖代谢(HAD 家族磷酸酶),糖酵解/糖异生代谢通路[果糖二磷酸醛缩酶、磷酸甘油酸激酶、烯醇化酶、磷酸烯醇式丙酮酸羧激酶(ATP)]中的DEPs 表达量上调。在氨基酸代谢中,厌氧条件降低了氨基酸降解酶的表达,如缬氨酸、亮氨酸和异亮氨酸等。在能量代谢中,涉及氧化磷酸化(NADH:醌还原酶、无机焦磷酸酶、F 型H+转运ATP 酶亚基a 等)和硫代谢(半胱氨酸合成酶、硫转移酶等)途径中的DEPs 表达量上调。

图5 KEGG 富集代谢通路Fig.5 KEGG enriched metabolic pathways
2.6 蛋白质互作网络
利用CytoScape 软件构建比较组DEPs 相互作用网络结构图,如图6‑A 所示,关联度排名前20位的蛋白如图6‑B、表2 所示,其中关联度最高的是苯丙氨酸‑tRNA 连接酶β 亚基(A0A3N6AUL8),关联度为149;其次是核黄素生物合成蛋白RibD(A0A143HEW7),关联度为147,两者在厌氧条件下均呈下调特征。呈上调特征的DEPs 主要集中在5-13 位,关联度为105 左右。

表2 比较组蛋白互作网络中关联度最高的20 个蛋白质Table 2 Top 20 proteins with the highest correlation in the interaction network of DEPs

图6 比较组DEPs 互作网络分析结果Fig.6 Results of the interaction network analysis of DEP in the comparative groups
3 讨论
3.1 菌株R.suwonensis 3B‑1的生长情况
菌株R.suwonensis 3B‑1 在好氧和厌氧条件下,表现出明显的生长和代谢差异,本研究通过对DEPs 的挖掘发现,DEPs 主要与代谢过程相关,包括能量代谢、碳水化合物代谢、氨基酸代谢和核苷酸代谢等,其中糖酵解/糖异生、三羧酸循环和丙酮酸代谢是DEPs 最集中的代谢通路。厌氧条件下,影响糖酵解/糖异生通路的主要上调蛋白为磷酸甘油酸激酶(A0A511C542)和磷酸丙酮酸水合酶(A0A3N6AA05)。作为糖酵解/糖异生通路的关键酶,磷酸甘油酸激酶在催化三磷酸腺苷(ATP)的形成中有着至关重要的影响[18],为菌株的后续生长代谢提供能量,其表达量的上调使菌株的能量供应得到保障。磷酸丙酮酸水合酶是一种多功能酶,可通过催化2‑磷酸‑D‑甘油酸脱水生成磷酸烯醇丙酮酸[19],该酶在厌氧条件下的表达上调,使得菌株3B‑1 内的磷酸烯醇丙酮酸产量高于好氧条件。主要下调蛋白为丙酮酸脱氢酶(A0A3N5ZV13)和丙酮酸激酶(A0A3N5ZTV7),丙酮酸脱氢酶可减弱丙酮酸对2‑羟基-乙基‑Thpp 的抑制作用[20],由于其在厌氧条件下的表达量下调,使得菌株体内的丙酮酸含量较低。丙酮酸激酶在调节丙酮酸的合成与代谢中起着重要作用,可催化磷酸烯醇丙酮酸和ADP转化为丙酮酸和ATP[21],该酶表达量的下调,导致菌株体内丙酮酸和能量供应不足。此外,丙酮酸激酶还是糖酵解途径中的限速酶,其表达量的下调导致菌株在厌氧条件下的生长发育缓慢。
双组分系统富集到较多的DEPs,其中的转录调节蛋白ResD 通过接收环境信号并调节下游基因的转录活性,参与菌体的适应性反应和生理调节,有研究发现,ResD 在调控有氧和无氧呼吸过程中起着重要作用[22-23],该蛋白在厌氧条件下呈下调趋势,符合菌株3B‑1 在厌氧条件下的生长特征。在氨基酸和辅因子合成途径中,厌氧条件导致菌株在亮氨酸、精氨酸、甲硫氨酸等氨基酸代谢和硫胺素代谢结合蛋白(A0A3N5ZSI0)、生物素蛋白连接酶(A0A3N5ZTP5)的表达呈现下调特征,这可能表明菌株3B‑1 更适合在好氧环境中生长。在蛋白质互作网络中发现的两个高关联度的蛋白:苯丙氨酸‑tRNA连接酶β 亚基和核黄素生物合成蛋白RibD,两者在厌氧条件下均呈下调特征。苯丙氨酸‑tRNA 连接酶β 亚基主要参与在核糖体中的mRNA 分子蛋白质的生物合成,其通过与ATP、镁离子、锌离子或tRNA结合发挥生物学功能[24],该蛋白在厌氧条件下呈现下调趋势,导致该菌株在厌氧条件下对于镁离子、锌离子的结合能力较弱,从而影响相应的生物学功能。核黄素生物合成蛋白RibD 主要参与核黄素的生物合成[25-26],该蛋白表达量的下调导致菌株在核黄素的生物合成方面能力较弱。
厌氧条件下,糖酵解阶段丙酮酸脱氢酶和丙酮酸激酶表达量的下调,导致菌株R.suwonensis 3B‑1的能量供应量及丙酮酸合成量较低,加之相关氨基酸代谢和辅因子生物合成相关蛋白表达量的下调,进而影响菌株R.suwonensis 3B‑1 的生长发育,图1‑A也表明菌株R.suwonensis 3B‑1 的最大生物量和生长速度低于好氧条件。综上所述,菌株R.suwonensis 3B‑1 更适合在好氧条件下生长。
3.2 菌株R.suwonensis 3B‑1的代谢差异
菌株R.suwonensis 3B‑1 在好氧和厌氧条件下的生长与其代谢产物产量有着明显差异:相较于好氧条件,厌氧条件下的菌体生物量和生长速度有着明显下降,但是己酸、丁酸的产量却表现为上升。为从蛋白水平阐述此差异的分子机制,本研究着重关注菌株3B‑1 己酸代谢相关蛋白的表达情况,并构建了己酸代谢通路图(图7)。乙酰辅酶A 是连接糖酵解/糖异生、三羧酸循环和己酸代谢途径的关键代谢物,其积累量在一定程度上影响菌体的生长发育和己酸代谢。在糖酵解/糖异生途径中,由于丙酮酸激酶表达量的下调,导致由磷酸烯醇丙酮酸生成丙酮酸的速率较低,因此乙酰辅酶A 的积累量较少,但磷酸丙酮酸水合酶的上调,可催化2‑磷酸‑D‑甘油酸脱水生成磷酸烯醇丙酮酸,使得磷酸烯醇丙酮酸在菌株体内大量积累。同时,三羧酸循环途径中各催化酶的表达量均呈现上调,可由此积累大量的草酰乙酸,由三羧酸循环生成的草酰乙酸可由磷酸烯醇丙酮酸羧化激酶催化生成磷酸烯醇丙酮酸[27],磷酸烯醇丙酮酸羧化激酶在厌氧条件下呈上调特征,使得磷酸烯醇丙酮酸在菌株体内进一步积累,并将三羧酸循环和糖酵解/糖异生途径偶联,为后续乙酰辅酶A 的生成提供丰富的前体物。此外,厌氧条件显著提升了磷酸戊糖途径相关蛋白的表达,由磷酸戊糖途径会生成大量的NADPH 和磷酸核糖[28],而己酸的合成依赖于乙醇的逆β-氧化循环羧酸链延长,此过程需要丰富的还原当量和乙酰辅酶A[29-30],磷酸戊糖途径相关蛋白的表达上调为己酸合成提供大量的NADPH。可以说,糖酵解/糖异生、三羧酸循环和磷酸戊糖代谢途径为后续己酸代谢提供了必要的物质基础和能量供应。在己酸代谢通路中,乙酰辅酶A 乙酰转移酶(A0A3N6AQ32)的蛋白表达量有所降低,可能由于菌株在厌氧条件生长初期,乙酰辅酶A 的产量和积累量相对较少,菌株首先用其满足生长发育需求。与之相似的是3‑羟基丁酰辅酶A 脱氢酶(A0A3N5ZT88),该酶的表达量也呈现下调,因为在厌氧条件初期,基础的生长代谢仍然不充分,没有足够的物质准备,无法进行后续的己酸代谢。酰基辅酶A 硫酯酶(A0A3N6BPG2)可催化己酰辅酶A 生成己酸[31],该酶在厌氧条件下呈现上调,在菌株完成相应的物质准备后,由此催化己酰辅酶A 生成己酸,从而提高己酸的产量。

图7 DEPs 在中心代谢和己酸代谢途径中的分布Fig.7 Distribution of DEPs in the central metabolic and caproic acid metabolic pathways
4 结论
在厌氧与好氧条件下,在R.suwonensis 3B‑1 中共鉴定到810 个DEPs,其中上调蛋白423 个,下调蛋白387 个。在厌氧条件下,亮氨酸、精氨酸、甲硫氨酸等氨基酸代谢呈现下调特征,硫胺素结合蛋白、生物素蛋白连接酶等辅因子表达下调,同时,苯丙氨酸‑tRNA 连接酶和核黄素生物合成蛋白的下调进一步表明该菌更适合在好氧环境中生长。在己酸合成方面,酰基辅酶A 硫酯酶的表达量显著上调,此外,糖酵解/糖异生途径、三羧酸循环和磷酸戊糖途径为己酸合成提供了充足的前体物质和还原当量,共同促进了己酸合成。
