济麦44/ 济麦229 重组自交系群体籽粒蛋白质含量QTL 分析
2024-04-01单宝雪刘秀坤肖延军展晓孟黄金鑫刘百川张玉梅李豪圣刘建军高欣曹新有赵振东
单宝雪 刘秀坤 肖延军 展晓孟 黄金鑫 刘百川 张玉梅 李 豪圣 刘建军 高欣 曹新有 赵振东

摘要:小麦籽粒蛋白质含量与面团流变学特性及加工特性的关系密不可分。 本研究在2020—2021 年济南试验基地(E1)及2021—2022 年济南试验基地(E2)和济阳试验基地(E3)三个环境下,以“济麦44×济麦229”构建的包含285 个家系的重组自交系群体(F2∶6 RILs)为材料,利用小麦55K SNP 芯片构建高密度遗传连锁图谱,对籽粒蛋白质含量进行QTL 分析。 结果共筛选到2 344 个SNP 标记用于构建遗传连锁图谱,图谱总长度3 349.95 cM,平均标记密度为1.43 cM/ 标记。 通过对籽粒蛋白质含量的QTL 分析,共检测到18 个籽粒蛋白质含量相关QTL,分布在1A、1B、2B、3D、4B、4D、5A、5B、5D、7A、7B 共11 条染色体上。 Qpc.saas.4B-1 在E1、E2 和E3 三个环境和BLUE(最佳线性无偏估计)值中均被稳定检测到,可以解释3.26% ~23.79%的表型变异。 Qpc.saas.4D 和Qpc.saas.5A 在兩个环境和BLUE 值中被检测到,分别解释2.42% ~ 11.18%和2.48% ~5.47%的表型变异,且Qpc.saas.4D 与小麦矮秆基因Rht-D1b 物理位置重合。 本研究中检测到的QTL 新位点Qpc.saas.4B-1、Qpc.saas.4D 和Qpc.saas.5A 是控制籽粒蛋白质含量的主效基因,具有高表型变异解释率。 本研究结果可为小麦品质育种提供分子标记及理论参考。
关键词:小麦;籽粒蛋白质含量;QTL 分析;重组自交系;55K SNP 芯片
中图分类号:S512.1+ 10.3文献标识号:A文章编号:1001-4942(2024)01-0001-09
小麦是世界上种植最广泛的粮食作物之一,也是人类碳水化合物和蛋白质的重要来源[1] 。小麦籽粒中蛋白质约占8%~20%,小麦面粉及面筋中蛋白质含量与面团的流变学特性和面包加工品质关系密切[2-3] 。 面包、馒头和面条等最终产品的质量,在很大程度上受到由蛋白质和淀粉决定的小麦品质性状的影响[4] 。
小麦多数性状为数量性状,同时受基因型与环境的影响。 小麦品质相关性状,如籽粒蛋白质含量、籽粒淀粉含量、面团稳定时间、SDS-沉降值等,受遗传因素影响较大,因此利用基因型改良小麦品质是可行的[5] 。 小麦基因组巨大,且大多为重复序列,在不同染色体组之间具有部分同源性。单核苷酸多态性(SNP)广泛存在于生物基因组内,对构建高密度遗传连锁图谱具有积极作用,现已成为研究小麦数量性状控制基因的主要方法[6-8] 。 已有研究针对不同群体利用基因芯片[9-10] 、SRAP 标记[11] 和SSR 标记[12] 等方式检测控制小麦品质性状的QTL 位点,检测到大量微效QTL 位点和QTL 富集区,分布于小麦基因组的21条染色体上。
常用的遗传作图群体包括永久性作图群体和暂时性作图群体。 永久性作图群体家系内基因型纯合且相同,家系间基因型差异较大,包括双单倍体(DH)群体、重组自交系(RIL)群体和近等基因系(NIL)群体等。 Zhao 等[13] 、Ilaria 等[14] 、孔忠新等[15] 分别以DH、RIL、NIL 群体为作图群体,对小麦籽粒蛋白质含量和粒重等数量性状相关QTL进行定位,检测到多个环境稳定的QTL。 永久性作图群体能进行多年试验,受环境影响较小,适合高精度作图,但难以分析显性效应。 暂时性作图群体内个体基因型杂合,后代会发生基因分离,包括F2群体和回交(BC)群体等,构建群体耗时短,难以进行多年研究。 王彦青等[16] 、侯北伟等[17]分别利用F2 和BC 群体对小麦数量性状进行定位,确定了与性状紧密连锁的标记。 RIL 群体可以在多年多点进行重复实验,且连续多代自交使染色体重组的机会增加,可以更精确地定位紧密连锁的位点,是研究基因型与环境互作的理想材料[18]。
小麦籽粒蛋白质含量是多基因控制的数量性状,在多个遗传背景中检测到的位点较少,缺乏稳定有效的分子标记用于该性状的育种改良。 已有研究确定了几个控制小麦籽粒蛋白质含量的基因,包括位于6BS 染色体上的高蛋白质含量基因Gpc-B1[19] 和与水稻OsAAP6 同源的TaAAP6-3B-Ⅰ优异等位基因[20] 。 济麦44 与济麦229 是近年育成的小麦新品种,其中济麦44 含有较高的籽粒蛋白质含量,该性状在RIL 群体中有明显的超亲分离现象[21] 。 本研究利用“济麦44×济麦229”构建的RIL 群体为材料,对三个环境下的小麦籽粒蛋白质含量进行测定,并利用小麦55K SNP 芯片构建高密度连锁图谱,以期挖掘小麦籽粒蛋白质含量相关QTL,为用分子标记辅助育种改良小麦品质提供理论依据。
1 材料与方法
1.1 试验材料
供试亲本材料为济麦44(JM44,母本,超强筋小麦品种)和济麦229(JM229,父本,强筋小麦品种),JM44 与JM229 高分子量麦谷蛋白的亚基组成均为1,7+8,5+10。 供试RIL 群体由两亲本杂交后经单粒传法得到,包含285 个家系。
1.2 田间试验设计
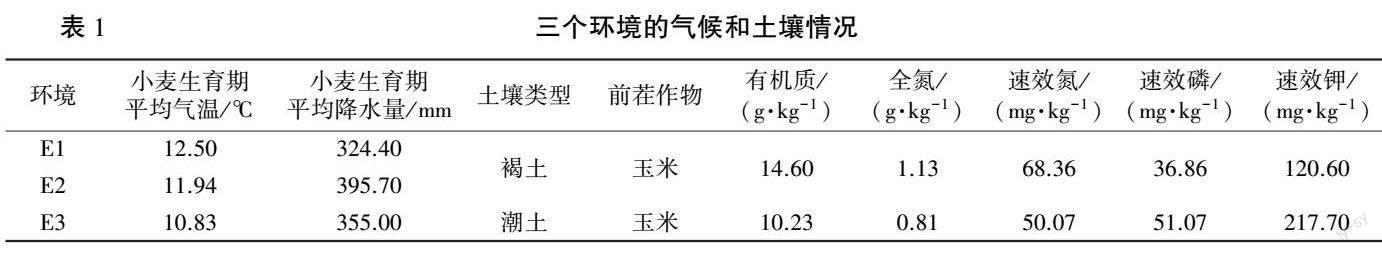
试验在三个环境(E1—E3)中进行,即将试验材料于2020—2021 年种植在济南试验基地(E1)、2021—2022 年分别种植在济南(E2)和济阳(E3)试验基地。 三个环境的气候和土壤条件见表1。
采用随机区组设计,小区面积9 m2,每个小区种植6 行,行长6 m,行距25 cm,机播180 g 种子。 按常规方法进行大田试验管理。
1.3 籽粒蛋白质含量测定与分析方法
小麦自然成熟后机器收获,风干入库,储存3个月后利用Infratec 1241 谷物分析仪(丹麦福斯)对籽粒蛋白质含量进行测定。
利用Microsoft Excel 2016 进行数据整理和分析,使用SAS 9.4 软件(http:/ / www.sas.com)进行基本统计量分析( UNIVARIATE) 和方差分析(ANOVA),并根据方差分析结果评估该性状在群体中的广义遗传率,利用CORR 程序计算Pearson相关性。 广义遗传率计算公式[22] 如下:
1.4 遗传图谱构建与QTL 定位分析
利用中玉金标记(北京)生物技术股份有限公司开发的小麦55K SNP 芯片对RIL 群体及亲本材料进行基因型分析,删除杂合率≥10%和缺失率≥10%的标记,用TASSEL V5.0 软件过滤掉偏分离标记(Min ∶ Max = 0.3 ∶0.7)。 在QTL Ici ̄Mapping V4.2 软件(https:/ / isbreeding.caas.cn/ rj/qtllcmapping/294445.htm)中假设基因型的固定效应,使用AOV(ANOVA of multi ̄environmental tri ̄als)工具跨环境计算最佳线性无偏估计(BLUE)值。 在QTL IciMapping V4.2 中整理剩余的标记并进行QTL 分析,选择复合完备区间作图(ICIMADD)法,以LOD = 2.5 为阈值判定QTL 存在与否。 利用Map Chart 绘制遗传图谱,利用AdobeIllustrator 对染色体遗传图谱进行修饰。在两个及两个以上环境中均发现的QTL 认为是稳定的QTL。 检测到的QTL 位点以“Q+性状名称英文缩写+研究机构名称缩写+染色体编号+序号”的方式进行命名[23] 。
2 结果与分析
2.1 RIL 群体籽粒蛋白质含量的统计分析
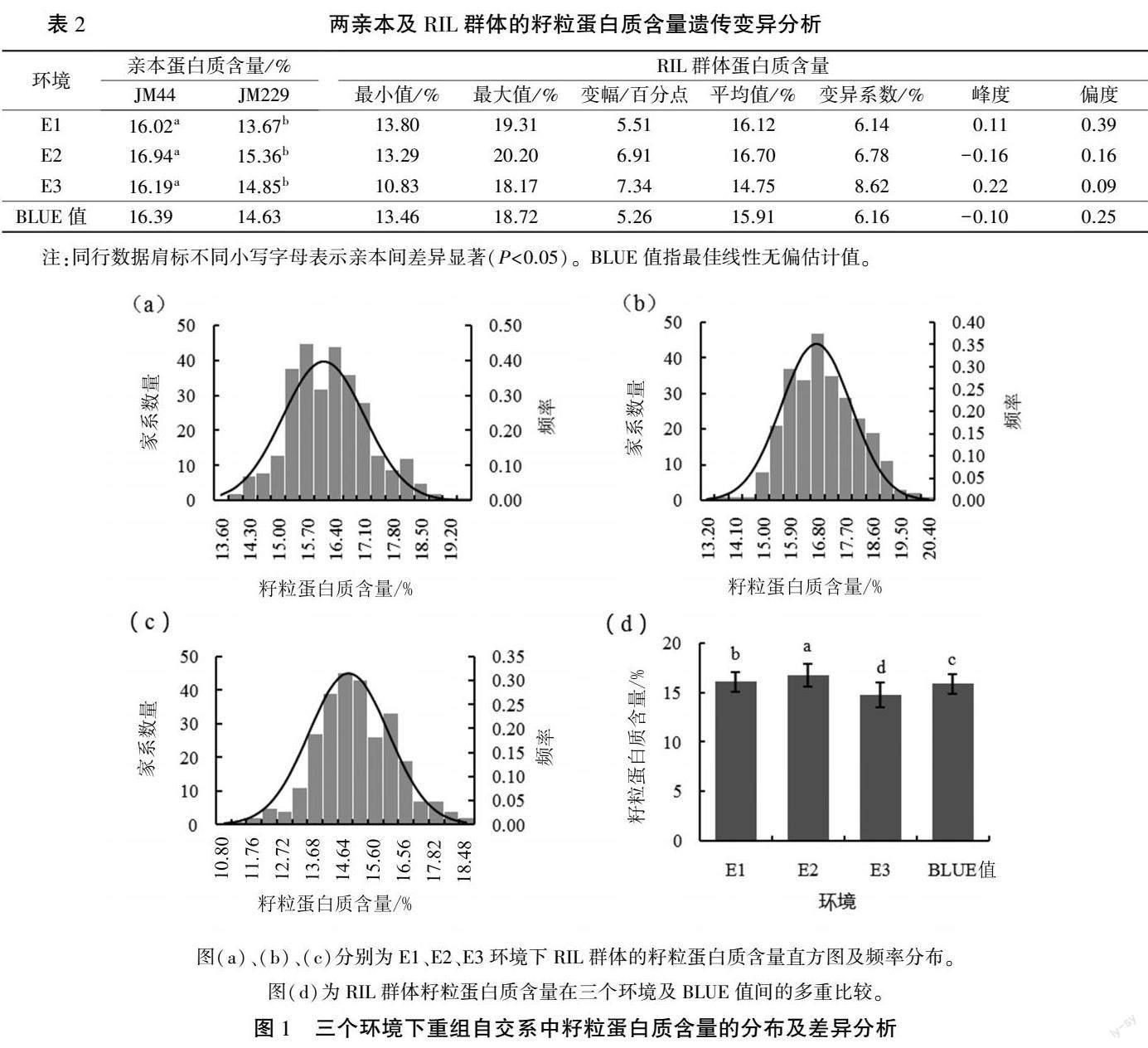
在三个环境中,JM44、JM229、RIL 群体的籽粒蛋白质含量分别为16.02%~16.94%、13.67%~15.36%、10.83%~20.20%,JM44 的籽粒蛋白质含量显著高于JM229,RIL 群体的籽粒蛋白质含量存在超亲分离现象(表2)。
Pearson 相关性分析结果表明,RIL 群体在三个环境中籽粒蛋白质含量的相关性均超过0.965 8;RIL 群体籽粒蛋白质含量在E1、E2、E3中的广义遗传率分别为0.996 9、0.997 8、0.998 1。表明小麦籽粒蛋白质含量主要受遗传因素的影响。 RIL 群体的籽粒蛋白质含量分布见图1,可以看出,其存在一定的环境差异,整体表现为E2>E1>E3,这是因为籽粒蛋白质含量是多基因控制的数量性状,也易受环境因素的影响。
RIL 群体籽粒蛋白质含量的变异系数在三个环境中均大于6,偏度与峰度绝对值均小于1,均表现为连续的正态分布(图1),可以对其进行QTL 定位分析。
2.2 遗传连锁图谱的构建
经过小麦55K SNP 芯片分析,两亲本间具有多态性的SNP 标记共53 064 个,其中2 344 个可用于遗传连锁图谱的构建。 这些SNP 多态性标记覆盖了小麦基因组的全部21 条染色体(图2),
共29 个连锁群,其中1A、2B、2A、4B、5D、6B、7A、7B 染色体上各有两个连锁群。 构建的连锁图谱全长3 349.95 cM,平均为1.43 cM/ 标记。 各染色体组间分子标记的数目差异较大,表现为B 组>A组>D 组,其中,B 组的SNP 标记最多(936 个)且长度最短,为1 091.06 cM;A 组、D 组分别含有815、593 个标记,覆盖染色体长度分别为1 123.57、1 135.32 cM。
2.3 RIL 群体籽粒蛋白质含量QTL 定位
从RIL 群体中共检测到18 个控制籽粒蛋白质含量的QTL,分布在1A、1B、2B、3D、4B、4D、5A、5B、5D、7A、7B 共11 条染色体上,单个QTL可以解释0.96%~23.79%的表型变异(表3)。 其中位于4B 染色体上的Qpc. saas.4B - 1 在三个环境和BLUE 值中均被稳定地检测到,加性效应平均达到-0.41,可以解释3.26%~23.79%的表型变异,等位基因来源于JM229。 Qpc. saas.4D 在E2、E3 两个环境和BLUE 值中被检测到,可以解释2.42%~11. 18% 的表型变异,等位基因来源于JM44。 Qpc. saas. 5A 可以在E1、E2 两个环境和BLUE 值中被检测到,可以解释2.48% ~5.47%的表型变异,等位基因来源于JM229。 Qpc. saas.3D和Qpc.saas.5B-1 均在一个环境和BLUE 值中被检测到,分别可以解释1.60% ~2.77%和1.58% ~3.40%的表型变异,等位基因均来源于JM44。 还有13 个QTL 仅在一个环境下被检测到或仅被BLUE 估计到, 是环境特异的QTL, 可以解释0.96%~12.26%的表型变异。 可在多个环境下被检测到的QTL 在染色体上的分布见图3。
3 讨论与结论
小麦籽粒蛋白质含量是多基因控制的数量性状,受遗传因素和非遗传因素的影响。 本研究以“济麦44×济麦229”构建的包含285 个家系的重组自交系群体(F2∶6 RILs) 为材料,在E1、E2、E3三个环境下对其籽粒蛋白质含量进行遗传变异分析,并利用小麦55K SNP 芯片构建高密度遗传连锁图谱,对控制籽粒蛋白质含量的QTL 进行定位分析。 结果表明,RIL 群体的籽粒蛋白质含量为10.83% ~ 20.20%,存在超亲分离现象,且三个环境间的相关性均超过0.965 8,广义遗传率均高于0.996,表明该性状主要受遗传因素的影响。
蛋白质是小麦籽粒中主要的含氮成分,因而影响小麦氮素吸收与积累的土壤、气候等因素也是影响小麦籽粒蛋白质含量的主要环境因素。 研究表明,全生育期增温能促进小麦籽粒中高分子量麦谷蛋白亚基的积累[24] ;适度提高施氮量可促进小麦植株花后氮素的吸收同化能力,提高籽粒蛋白质含量[25] 。 本研究中,E1、E2 環境小麦生育期的平均温度以及土壤中全氮、速效氮含量均高于E3 环境,RIL 群体的籽粒蛋白质含量也表现为E2>E1>E3,进一步证实了温度和氮素对小麦籽粒蛋白质的影响。 韩占江等[26] 研究发现,随着拔节期和开花期灌水量的增加,小麦氮素利用效率呈先增高后降低的趋势。 本研究中RIL 群体两年中在同一个试验地(E1、E2)种植,小麦生育期的降雨量在两个环境间具有明显差异(E2>E1),且在小麦拔节期间E2 降雨量(40.2 mm) 高于E1(27.8 mm),这有利于提高小麦的水分利用效率[27] ,促进植株生长,RIL 群体的籽粒蛋白质含量也表现为E2>E1。
本研究利用小麦55K SNP 芯片构建高密度连锁图谱,有利于将QTL 定位在较小的区间内。染色体水平上整体表现为标记数量不平衡,其中,B 染色体组标记数量最多(936 个),D 染色体组标记数量最少(593 个)。 平均标记间的遗传距离为1.43 cM。
本研究在三个环境中共检测到18 个控制小麦籽粒蛋白质含量的QTL,分布在1A、1B、2B、3D、4B、4D、5A、5B、5D、7A、7B 共11 条染色体上,单个QTL 可以解释0.96%~23.79%的表型变异。
在三个环境和BLUE 值中可以被稳定检测到的主效QTL Qpc. saas. 4B - 1 侧翼标记为AX -109998354—AX - 109866370,定位在4B 染色体423.78—431.27 Mb 处,可以解释3.26% ~23.79%的表型变异,加性效应为负值,位点增效等位基因来源于父本JM229,与控制籽粒蛋白质含量的QGpc. hwwgr - 4BS (位于4B 染色体上82. 66—110.70 Mb 和396.69—425.08 Mb 区间)[28] 可能是同一个QTL。 4B 染色体上有两个控制面团流变特性参数的QTL 簇,但均与Qpc.saas.4B-1 距离较远[29] 。
Qpc.saas.4D 和Qpc.saas.5A 可以在两个环境及BLUE 值中被检测到。 其中,Qpc.saas.4D 位于4D 染色体上16. 15—37. 49 Mb 处, 可以解释2.42%~11.18% 的表型变异,侧翼标记为AX -111616151—AX-111322614,加性效应为正值,位点增效等位基因来源于母本JM44。 Qpc. saas.4D与小麦矮秆基因Rht-D1b(4D,33.61 Mb)物理位置完全重合,前期也已经验证RIL 群体中含有矮秆基因Rht - D1b,来源于亲本JM44,因此推测Qpc.saas.4D 可能与Rht -D1b 为同一个QTL。 另有研究表明,控制籽粒蛋白质含量的QTL 位点QGpc. caas - 4D. 2, 侧翼标记为gwp93025—wmc331,在4D 染色体12.50—43.00 Mb 之间[30] ,与Qpc.saas.4D 的物理位置存在部分重合;有研究将部分QTL 簇定位到矮秆基因Rht-D1b 附近,与籽粒硬度[30] 、蛋白质含量[31] 等相关;Tian 等[32]
鉴定到15 个控制SDS-沉降值的QTL 位点,其中QSsv.cau-4B、QSsv.cau-4D 与矮秆基因Rht-B1b、Rht-D1b 物理位置邻近。 因此Qpc. saas.4D 也可能与Rht-D1b 邻近。 Qpc.saas.5A 位于5A 染色体691. 66—702.07 Mb 处, 侧翼标记为AX -111018517—AX - 109370078,可以解释2.48% ~5.47%的表型变异,加性效应为负值,位点增效等位基因来源于父本JM229。 有研究在5A 染色体34、96、102 Mb 处定位到多个控制籽粒蛋白含量的QTL[33] ,但可能与本研究定位到的Qpc.saas.5A是不同的QTL。
Qpc.saas.3D 和Qpc.saas.5B-1 可以在一个环境和BLUE 值中被检测到。 其中,Qpc.saas.3D 位于3D 染色体444.00—453.81 Mb 之间,侧翼标记为AX - 108813316—AX - 111612043, 可以解释1.60%~2.77%的表型变异,加性效应为正值,位点增效等位基因来源于母本JM44。 胡文静等[33]在3D 染色体wsnp _Ex _rep _c78526 _74641162—BS00058654_51 标记之间检测到一个环境特异性QTL,与Qpc.saas.3D 距离较近。 Qpc.saas.5B-1 位于5B 染色体7.33—10.55 Mb 之间,侧翼标记为AX - 110714323—AX - 109650482, 可以解释1.58%~3.40%的表型变异,加性效应为正值,位点增效等位基因来源于母本JM44。 Mann 等[34]在单个环境中鉴定到5B 染色体5—8 Mb 处有控制籽粒蛋白质含量的QTL,可以解释7%的表型变异,与本研究定位到的Qpc.saas.5B-1 可能是同一个QTL。
Qgpc2B 是在2B 染色体上定位到的控制籽粒蛋白質含量的稳定QTL 位点, 位于基因组165.29—180. 82 Mb 处[35] ,与本研究定位到的Qpc.saas.2B-1 可能是同一个QTL。 但Qpc. saas.2B-1 只在BLUE 值中被检测到,可能是环境特异的QTL。 本研究在1A(480.36—577.08 Mb)、4B(423.78—667.89 Mb)、5B(283.76—438.16 Mb)、5D(408.44—440.81 Mb)同一染色体区段上定位到多个环境特异的QTL 位点,可能是控制籽粒蛋白质含量的微效QTL 簇,受环境因素的影响。目前用于籽粒蛋白质含量的测定方法较多,
本研究利用近红外光谱技术测定籽粒蛋白质含量,测定方法简单便捷且不破坏种子结构,然后利用小麦55K SNP 芯片数据,鉴定到Qpc.saas.4B-1、Qpc.saas.4D、Qpc.saas.5A 三个稳定的控制籽粒蛋白质含量的QTL 位点。 今后可在此基础上继续进行基因的精细定位,为选育高品质小麦新品种提供理论支撑。
参 考 文 献:
[1] Smidansky E D,Meyer F D,Blakeslee B,et al. Expression of amodified ADP ̄glucose pyrophosphorylase large subunit in wheatseeds stimulates photosynthesis and carbon metabolism [ J].Planta,2007,225(4):965-976.
[2] Uthayakumaran S,Grasp W,Stoddard F L,et al. Effect of var ̄ying protein content and glutenin to gliadin ratio on the func ̄tional properties of wheat dough[J]. Cereal Chemistry,1999,76:389-394.
[3] 何中虎,夏先春,陈新民,等. 中国小麦育种进展与展望[J].作物学报,2011,37(2):202-215.
[4]Sun H Y, Lü J H, Fan Y D, et al. Quantitative trait loci(QTLs) for quality traits related to protein and starch in wheat[J]. Progress in Natural Science,2008,18:825-831.
[5] 伍玲,董亚超,戴常军,等. 长江上游小麦新品种(系)品质分析[J]. 麦类作物学报,2020,40(4):1-11.
[6] 朱秀志,彭正松,向成华. SNPs 分析技术及其在小麦遗传育种中的应用研究[J]. 天津农业科学,2005,11(1):12-15.
[7] Gao L F,Zhao G Y,Huang D W,et al. Candidate loci involvedin domestication and improvement detected by a published 90Kwheat SNP array[J]. Scientific Reports,2017,7(1):44530.
[8] Sun C W,Dong Z D,Zhao L,et al. The wheat 660K SNP arraydemonstrates great potential for marker ̄assisted selection in pol ̄yploid wheat[J]. Plant Biotechnology Journal,2020,18:1354-1360.
[9] 严勇亮,时晓磊,张金波,等. 春小麦籽粒主要品质性状的全基因组关联分析[J]. 中国农业科学,2021,54(19):4033-4047.
[10] 陈巧灵. 基于全基因组关联分析对中国地方小麦蛋白质含量性状的解析[D]. 雅安:四川农业大学,2020.[11] 郭利建,王竹林,汪世娟,等. 基于SRAP 和SSR 标记的小麦品质相关性状的QTL 定位[J]. 麦类作物学报,2016,36(10):8.
[12] 王掌军,姚明明,余慧霞,等. 宁春4 号×河东乌麦F2:5 家系遗传图谱构建与籽粒蛋白质性状QTL 分析[J]. 浙江农业学报,2021,33(8):1367-1378.
[13] Zhao L,Zhang P K,Liu B,et al. A comparison of grain proteincontent QTLs and flour protein content QTLs across environ ̄ments in cultivated wheat[J]. Euphytica,2010,174(3):325-335.
[14] Marcotuli I,Gadaleta A,Mangini G,et al. Development of ahigh ̄density SNP ̄based linkage map and detection of QTL forβ ̄glucans, protein content, grain yield per spike and headingtime in durum wheat [ J]. International Journal of MolecularSciences,2017,18(6):1329.
[15] 孔忠新,程瑞如,张利伟,等. 小麦粒重主效QTL 近等基因系的构建和效应评价[J]. 麦类作物学报,2017,37(3):312-318.
[16] 王彥青,许娜丽,余慧霞,等. 普通小麦及其杂交F2 代的粒重、粗蛋白与湿面筋含量及白粉病抗性分析[J]. 山东农业科学,2022,54(7):32-38.
[17] 侯北伟,窦秉德,李生强,等. 利用回交群体对小麦雌性不育基因的SSR 标记[J]. 淮阴师范学院学报(自然科学版),2005,4(4):318-322,326.
[18] 林谦,毛孝强,杨德,等. QTL 作图主要统计方法及主要作图群体[J]. 云南农业大学学报,2004,19(2):121-127.
[19] 胡云,徐如宏. 小麦高蛋白质含量基因GPC-B1 的分子标记研究[J]. 天津农业科学,2014,20(9):6-10.
[20] Jin X F,Feng B,Xu Z B,et al. TaAAP6 - 3B, a regulator ofgrain protein content selected during wheat improvement[J].BMC Plant Biology,2018,18:71.
[21] ValeriuR,Georgeta D,Mihail M,等. 基于玉米单倍体性状表现的轮回选择试验研究[J]. 玉米科学,2015,23(6):39-44.
[22] 王姗,胡润雨,于士男,等. 小麦淀粉RVA 特性的QTL 定位及效应分析[J]. 核农学报,2023,37(10):1957-1967.
[23] 胡文静,朱冬梅,别同德,等. 扬麦16 籽粒灌浆速率相关性状的QTL 定位(小麦15K SNP 芯片法) [J]. 麦类作物学报,2020,40(8):915-920.
[24] 曹敏旭. 全生育期增温下施氮量对小麦产量和品质形成的影响及其生理机制[D]. 南京:南京农业大学,2012.
[25] 王月福,于振文,李尚霞,等. 氮素營养水平对冬小麦氮代谢关键酶活性变化和籽粒蛋白质含量的影响[J]. 作物学报,2002,28(6):743-748.
[26] 韩占江,于振文,王东,等. 测墒补灌对冬小麦氮素积累与转运及籽粒产量的影响[J]. 生态学报,2011,31(6):1631-1640.
[27] 苏盛楠. 不同生育期灌水对小麦产量形成和水分利用效率的影响[D]. 扬州: 扬州大学,2018.
[28] 孙宇慧. 小麦籽粒蛋白质含量主效QTL———QGpc. hwwgr ̄4BS 的精细定位及分子标记开发[D]. 杨凌:西北农林科技大学,2020.
[29] Jin H,Wen W E,Liu J D,et al. Genome ̄wide QTL mapping forwheat processing quality parameters in a Gaocheng 8901/Zhoumai 16 recombinant inbred line population[J]. Frontiersin Plant Science,2016,7:1032.
[30] Li Y Y,Song Y X,Zhou R,et al. Detection of QTLs for bread ̄making quality in wheat using a recombinant inbred line popu ̄lation[J]. Plant Breeding,2009,128(3):235-243.
[31] Liu T T,Liu K,Wang F F,et al. Conditional and unconditionalQTLs mapping of gluten strength in common wheat (Triticumae ̄stivumL.) [J]. Journal of Integrative Agriculture,2017,16:2145-2155.
[32] Tian S,Zhang M H,Li J H,et al. Identification and validationof stable quantitative trait loci for SDS ̄sedimentation volume incommon wheat (Triticum aestivum L.)[J]. Frontiers in PlantScience,2021,12:747775.
[33] 胡文静,裔新,李东升,等. 扬麦13/ C615 重组自交系籽粒蛋白质含量和硬度性状QTL 分析[J]. 麦类作物学报,2021,41(8):930-936.
[34] Mann G,Diffey S,Cullis B,et al. Genetic control of wheat qual ̄ity: interactions between chromosomal regions determining pro ̄tein content and composition, dough rheology, and sponge anddough baking properties[J]. Theoretical and Applied Genetics,2009,118(8):1519-1537.
[35] 吴胜男. 小麦几个农艺和加工品质相关性状的QTL 定位[D]. 杨凌:西北农林科技大学,2020.
