基于GEO数据库探讨UBE2T在肺腺癌中的作用
2024-03-31李晶汪巍彭敦钰
李晶 汪巍 彭敦钰
[摘要] 目的 旨在通過整合单细胞测序数据与TCGA数据库,探究在肺腺癌中泛素连接酶E2T(ubiquitin binding enzyme EZT, UBE2T)的作用,为揭示UBE2T在肺腺癌发生机制中的具体作用提供帮助。方法 本研究从GEO数据库(GSE117570)下载了单细胞测序数据,并使用R语言进行数据质控和分析。同时,利用在线工具分析了TCGA数据库中与肺腺癌相关的数据。通过整合TCGA数据库与单细胞测序数据,分析UBE2T的潜在作用靶点。结果 差异分析结果表明,UBE2T可作为肺腺癌独立的预后因子,并与患者预后不良相关。单细胞测序数据的整合分析显示,与UBE2T相关的10个基因主要分布在单核细胞和T细胞簇中。通过CancerSEA数据库的分析,发现UBE2T可能与细胞周期调控密切相关。结论 UBE2T可能通过调控细胞周期来促进肺腺癌的发生和发展。
[关键词] 肺腺癌;单细胞分析;TCGA数据库
[中图分类号] R734 [文献标识码] A [DOI] 10.3969/j.issn.1673-9701.2024.08.005
Investigating the role of UBE2T in lung adenocarcinoma based on GEO database
LI Jing1, WANG Wei2, PENG Dunyu1
1.Department of Anesthesia, Peoples Hospital of Wuhan University, Wuhan 430060, Hubei, China; 2. Department of Thoracic Surgery, Peoples Hospital of Wuhan University, Wuhan 430060, Hubei, China
[Abstract] Objective To investigate the role of ubiquitin binding enzyme EZT (UBE2T) in lung adenocarcinoma by integrating single-cell sequencing data with the TCGA database, in order to provide insights into the specific molecular mechanisms of UBE2T in lung adenocarcinoma. Methods Single-cell sequencing data was downloaded from the GEO database (GSE117570), and R language was used for quality control and analysis of the data. Additionally, online tools were employed to analyze lung adenocarcinoma-related data from the TCGA database. The potential target genes associated with UBE2T were identified by integrating TCGA and single-cell sequencing data. Results Differential analysis using the TCGA database successfully demonstrated that UBE2T could serve as an independent prognostic factor in lung adenocarcinoma, and it was correlated with poor patient outcomes. Integration of single-cell sequencing data revealed that UBE2T-associated genes were mainly distributed in mononuclear cells and T cells. Furthermore, analysis using the CancerSEA database suggested a close association between UBE2T and cell cycle regulation. Conclusion UBE2T may play a role in promoting the occurrence and development of lung adenocarcinoma through the regulation of the cell cycle.
[Key words] Lung adenocarcinoma; Single-cell analysis; TCGA database
肺癌是全球主要致死性癌症之一,其发病率不断上升且年轻化趋势明显,对人类健康构成严重威胁[1]。多靶点治疗已被广泛研究证明在癌症治疗中优于单一药物治疗,因此寻找肺腺癌的治疗靶点并揭示其发生机制显得尤为重要。肿瘤组织由多种不同的肿瘤细胞构成,其特征与对治疗的敏感性密切相关。随着生物信息学技术的发展,从传统的总体测序到单细胞测序的转变为进一步揭示肺腺癌发病机制提供了帮助[2]。
泛素连接酶E2T(ubiquitin binding enzyme EZT, UBE2T)属于UBE2超家族,在泛素化的第二步中起基础作用。以往研究表明UBE2T参与范可尼贫血(Fanconi anemia, FA)DNA修复的调控,最近的研究发现UBE2T还参与人类癌症患者的肿瘤发生、转移过程并影响生存和预后[3-4]。癌症组织样本和细胞系中常见到异常的UBE2T表达[5]。相关实验研究证实,UBE2T过表达可促进细胞增殖,而UBE2T敲低则可抑制细胞增殖[6]。然而,目前尚不清楚UBE2T是否为典型的致癌基因,也不清楚UBE2T除致癌作用外是否参与其他功能。此外,除了泛素化外,UBE2T是否还具有其他作用机制,以及其在肺腺癌中的表现如何仍不清楚。为了回答这些问题,需要对UBE2T进行进一步深入的研究。
本研究利用GEO数据库公开的单细胞数据,选择本研究课题组长期研究的基因UBE2T,并结合TCGA数据库,分析UBE2T在肺腺癌发病机制中的可能角色,以期为研究UBE2T的具体分子信号通路提供帮助。
1 资料与方法
1.1 數据收集与处理
本研究从GEO数据库下载了GSE117570的单细胞测序数据,并对数据进行了质量控制。质量控制的条件:①每个细胞至少表达3个基因;②每个基因至少在50个细胞中表达;③剔除线粒体基因占比超过5%的细胞;④每个细胞至少检测到50个RNA分子。
1.2 单细胞Seurat包的使用
本研究使用R语言中的Seurat包进行单细胞数据分析,并设置相关参数条件:|LogFC|>1,P<0.05[7]。在进行数据质量控制后,本研究使用主成分分析(principal component analysis, PCA)进行降维分析。使用FindCluster函数对细胞群进行识别,并使用tSNE函数进行可视化降维。然后使用Scanpy函数进行细胞轨迹分析,并使用clusterProfiler包进行GO富集分析,同时对相关的6个通路进行可视化。
1.3 TCGA数据库
本研究使用在线网站GEPIA(http://gepia.cancer- pku.cn/)对TCGA数据库进行在线分析,分析UBE2T在正常组和肺腺癌组中的差异表达情况,并对生存率和分期进行差异统计,结果显示正常组与肿瘤组的生存率差异有统计学意义[8]。
1.4 CancerSEA数据库
本研究使用CancerSEA数据库(http:// biocc.hrb mu.edu.cn/CancerSEA/)分析UBE2T调控的与癌症相关的细胞过程,包括细胞周期、细胞代谢、炎症等7个癌症相关的细胞过程。通过下载相关数据并进行进一步分析,使用气泡图进行可视化[9]。
1.5 HPA数据库
本研究从HPA数据库(http://www.proteinatlas.org/)中下载了免疫荧光图像,初步确定UBE2T在细胞中的定位。HPA数据库中的所有图像都可以获得授权下载[10]。
1.6 统计学方法
本研究使用R软件(R*64 4.0.2 版本)对数据进行处理分析(使用相关的R包:pheatmap、ggplot2)。本研究使用Log-rank检验确定显著性,并使用Spearman系数表示变量之间的关联。P<0.05为差异有统计学意义。
2 结果
2.1 UBE2T生存分析与细胞定位
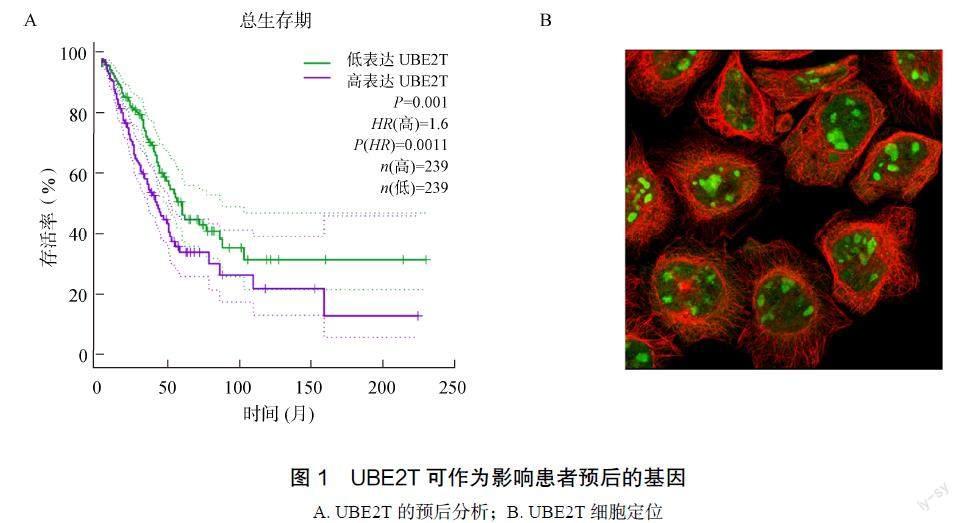
通过利用TCGA数据库中肺腺癌的数据,对正常组与肿瘤组中UBE2T的表达量进行统计分析,结果提示在肿瘤组织中UBE2T表达水平显著下降,进一步根据UBE2T表达的中位值将样本分为高低表达两组,发现在低表达组中患者生存期明显缩短(P<0.001),这与肿瘤组中UBE2T表达水平下降的结果相符。同时,UBE2T对患者分期也具有显著影响。综合以上分析结果可以得出,UBE2T可能是一个能够影响患者预后的靶基因。为进一步探明UBE2T的分子功能,本研究从HPA数据库中下载了UBE2T表达的荧光图,绿色为靶蛋白,发现UBE2T大部分表达于细胞内,这提示UBE2T可能与细胞内部的信号通路调控相关(图1)。
2.2 UBE2T相关基因单细胞分析
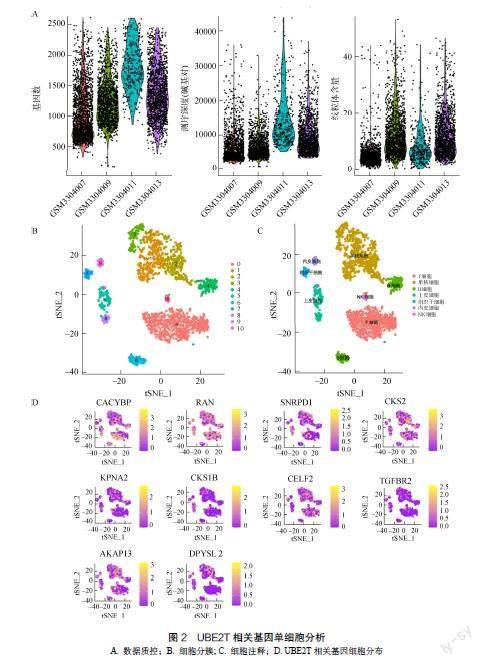
为进一步揭示UBE2T的分子功能,本研究从TCGA中下载的肺腺癌数据中,筛选出与UBE2T最显著相关的10个基因进行单细胞分析(CACYBP、RAN、SNRPD1、CKS2、KPNA2、CKS1B、CELF2、TGFBR2、AKAP13、DPYSL2)。图2A展示了单细胞数据质控结果,分别代表基因数,测序深度和线粒体含量,图2B和图2C展示了将细胞分为10个簇,每个簇代表的细胞种类,图2D展示了与UBE2T最相关的10个基因的细胞分布。通过以上分析,本研究发现,与UBE2T最相关的基因主要分布于单核细胞与T细胞中。
2.3 细胞轨迹分析
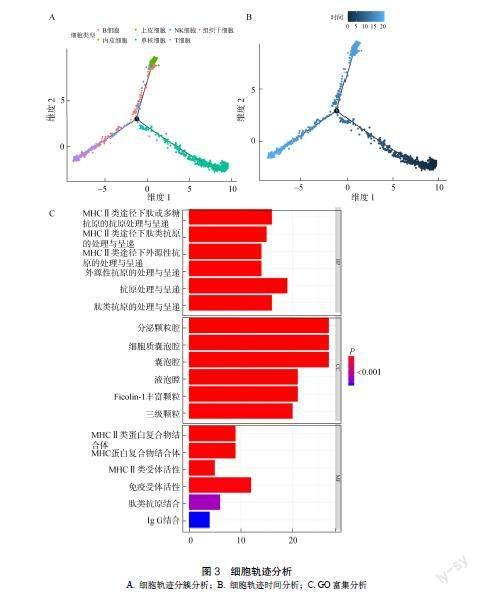
与UBE2T相关的基因主要集中在T细胞和单核细胞中,为了进一步明确结果,本研究做了细胞轨迹分析(图3A),通过细胞轨迹分析,本研究得出,随着时间的增加,细胞可大致有3个走向(图3B),其中与UBE2T相关的细胞簇大致位于第2支,因此,本研究对UBE2T相关细胞簇所在的一支做了GO富集分析(图3C)。
2.4 UBE2T与细胞周期密切调控相关
单细胞分析结果表明,与UBE2T最为密切的10个基因主要分布在单核细胞和T细胞组中,然而,由于测序深度和数据的限制,本研究无法在单细胞图中展现UBE2T的具体分布,因此,本研究利用在线网站(CancerSEA)分析了UBE2T可能的细胞学功能(图4)。结果显示,UBE2T与细胞周期调控关系最为密切,为进一步量化这个结果,本研究从CancerSEA數据库中下载了相关数据,并计算了相关系数(R=0.71),且P<0.001。
3 讨论
UBE2T是一种编码蛋白的基因,它能够编码催化泛素与蛋白质共价结合的蛋白质,从而影响该蛋白的表达[11]。该基因存在缺陷会导致T细胞功能异常[12]。目前的研究证实,UBE2T通过影响多种信号通路(包括p53/AMPK/mTOR信号通路)来影响肺腺癌的发生和发展[13]。同时,UBE2T与患者的预后不良相关,并与患者的分期有显著关系[14]。本研究系统探讨了UBE2T的具体功能,并进行了验证。结果显示,在多数肿瘤的正常组和肿瘤组中,UBE2T差异有统计学意义,并且与肺腺癌患者的预后不良相关。本研究发现,与UBE2T相关的基因主要分布在单核细胞和T细胞中,并可能调控T细胞抗原提呈过程,从而影响肺腺癌的发生、发展。
研究结果表明,UBE2T与Th2细胞浸润相关,且在乳腺癌中被证明可通过免疫调节来影响肿瘤细胞的浸润[15-16]。本研究的实验结果与这些研究结果相符合,进一步验证了结果的准确性。最后,本研究进行了泛癌分析,发现UBE2T与细胞周期调控密切相关,因此,笔者猜想,UBE2T是否能够通过影响T细胞分化周期进而影响其表达,这需要更多的相关研究来证明。
总之,本研究通过GEO单细胞测序数据和TCGA数据库联合分析,探讨了UBE2T在肺腺癌中的作用。结果表明,该基因能够通过调控细胞周期来影响肺腺癌的发生与发展,并与患者预后有关。然而,本研究仍存在一些不足之处,例如对数据的分析并不具有代表性。因此,在后续的研究中,需要对更多的患者进行研究验证,以进一步完善本研究对UBE2T在肺腺癌发生发展中作用的认识。
利益冲突:所有作者均声明不存在利益冲突。
[参考文献]
[1] NASIM F, SABATH B F, EAPEN G A. Lung cancer[J]. Med Clin North Am, 2019, 103: 463–473.
[2] KATZENELENBOGEN Y, SHEBAN F, YALIN A, et al. Coupled scRNA-Seq and intracellular protein activity reveal an immunosuppressive role of TREM2 in cancer[J]. Cell, 2020, 182(4): 872–885.
[3] SATIJA R, FARRELL J A, GENNERT D, et al. Spatial reconstruction of single-cell gene expression data[J]. Nat Biotechnol, 2015, 33: 495–502.
[4] TANG Z, LI C, KANG B, et al. GEPIA: A web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Res, 2017, 45(W1): W98–W102.
[5] YUAN H, YAN M, ZHANG G, et al. CancerSEA: A cancer single-cell state atlas[J]. Nucleic Acids Res, 2019, 47: D900–D908.
[6] LEI Y, YU T, LI C, et al. Expression of CAMK1 and its association with clinicopathologic characteristics in pancreatic cancer[J]. J Cell Mol Med, 2021, 25: 1198–1206.
[7] MACHIDA Y J, MACHIDA Y, CHEN Y, et al. UBE2T is the E2 in the Fanconi anemia pathway and undergoes negative autoregulation[J]. Mol Cell, 2006, 23: 589–596.
[8] MA N, LI Z, YAN J, et al. Diverse roles of UBE2T in cancer (Review)[J]. Oncol Rep, 2023, 49(4): 69.
[9] ZHU J, AO H, LIU M, et al. UBE2T promotes autophagy via the p53/AMPK/mTOR signaling pathway in lung adenocarcinoma[J]. J Transl Med, 2021, 19: 374.
[10] DENG Y, CHEN X, HUANG C, et al. Screening and validation of significant genes with poor prognosis in pathologic stage-I lung adenocarcinoma[J]. J Oncol, 2022, 2022: 3794021.
[11] XIAO Y, DENG Z, LI Y, et al. ANLN and UBE2T are prognostic biomarkers associated with immune regulation in breast cancer: a bioinformatics analysis[J]. Cancer Cell Int, 2022, 22: 193.
[12] WANG Z, CHEN N, LIU C, et al. UBE2T is a prognostic biomarker and correlated with Th2 cell infiltrates in retinoblastoma[J]. Biochem Biophys Res Commun, 2022, 614: 138–144.
(收稿日期:2023–05–15)
(修回日期:2023–11–25)
