细菌程序性细胞死亡及其在抗菌治疗中的应用前景
2023-12-26刘伟佳方芳朱靓薛云新王岱
刘伟佳?方芳?朱靓?薛云新?王岱

摘要:细菌程序性细胞死亡(programmed cell death, PCD)是细菌在压力环境下自发牺牲部分亚群以提高细菌群体生存而产生的编程性死亡,该利他现象受到越来越多的关注。耐药菌的出现为病原菌杀伤带来挑战,鉴于细菌PCD在耐药菌产生与治疗方面的重要作用,本文从PCD角度详细分析细菌应对抗生素压力的方式,阐明PCD为抗菌治疗提供的新策略,为合理使用这把“双刃剑”提供理论支持。
关键词:细菌;程序性细胞死亡;抗生素耐药;抗菌治疗;利他主义
中图分类号:Q932, TQ465文献标志码:A
Bacterial programmed cell death and its application prospect in antimicrobial therapy
Liu Wei-jia, Fang Fang, Zhu Liang, Xue Yun-xin, and Wang Dai
(Department of Experimental Medicine, State Key Laboratory of Molecular Vaccinology and Molecular Diagnostics, Xiamen 361102)
Abstract Bacterial programmed cell death (PCD) is a programming death in stressful conditions, at the cost of spontaneously sacrificing sub-population as to improve survival of bacterial population. The altruistic behavior is getting an increase attention. The emergence of drug-resistant bacteria has always posed a challenge to the killing of pathogens. Since bacterial programmed cell death plays an important role in both the generation and treatment of drug-resistant bacteria, we analyzed the response modes of bacteria to antibiotic pressure from the perspective of PCD, and illustrated new strategies of PCD for antimicrobial treatment, so as to provide theoretical support for the rational use of this double-edged sword.
Key words Bacteria; Programmed cell death; Antibiotic resistance; Antimicrobial therapy; Altruism
程序性细胞死亡(programmed cell death, PCD) 在多細胞真核生物中普遍存在,前期研究通常以真核细胞表型定义PCD,但近期研究表明PCD于细菌群体也至关重要[1-5]。面对不同压力刺激时,野油菜黄单胞菌(Xanthomonas campestris)[6]、金黄色葡萄球菌 (Staphylococcus aureus)[7]、铜绿假单胞菌(Pseudomonas aeruginosa)[8]、大肠埃希菌(Escherichia coli)[9]和结核分枝杆菌(Mycobacterium tuberculosis)[10]等均易产生PCD。对于单个细菌细胞,PCD会导致菌体死亡,但当视为群体的一部分时,部分细菌主动死亡可为压力环境下的群体提供养分与生存空间,有利于其余菌群存活[1,3,11-12]。该“利他主义”行为展现出菌群协同进化过程中的精妙合作。
2021年,世界卫生组织宣布抗微生物药物耐药 (antimicrobial resistance, AMR)是人类面临的全球十大公共卫生威胁之一[13]。据Lancet杂志报道,在2019年全球约有近500万例与细菌性AMR相关的感染死亡[14]。同年美国疾病控制与预防中心统计发现,美国每年有280多万起AMR病例感染,超过3.5万人因此死亡[15]。2016年英国政府有关审查报告指出:若缺乏有效途径加以抑制,预计2050年,全世界每年约有1000万人死于AMR感染[16]。可见抗微生物药物耐药给人类带来极大危害,增强现有抗菌药物杀伤效果和研发新型抗菌药物至关重要。
细菌PCD是耐药菌最初产生的主要原因,合理利用细菌PCD又能杀伤病原菌。鉴于细菌PCD在耐药菌产生以及治疗方面的重要作用,本文对细菌PCD及其诱发因素进行综述,详细介绍PCD用于抵抗病原菌的新策略,阐明PCD与耐药菌的“双刃剑”关系,为PCD在原核细菌群体中抗菌治疗方向提供应用思路。
1 细菌的程序性细胞死亡
1995年,Science杂志首次报道关于限制修饰系统 (restriction-modification system, RM系统) 介导大肠埃希菌PCD的研究成果,引起广泛关注[17-18]。自此,“细菌PCD”理论逐步成为原核生物研究领域的热点方向之一。Kenneth[19]提出细菌PCD是由遗传编码决定的高度协调过程,当修复损伤细菌细胞成本高于构建新细胞成本时,细菌才自主启动PCD响应损伤。“亲缘选择理论”是解释细菌群体PCD 的假设之一:若受益个体是遗传亲支并且“成本/收益”比值小于“亲缘关系度”,则代价高昂的个体行为可以进化[20-22]。
过去10年,细胞死亡命名委员会(Nomenclature Committee on Cell Death, NCCD)就形态学、诱发机制与功能作用角度对多种死亡类型做出定义[23]。目前对真核生物PCD过程研究较为透彻,主要分为细胞凋亡和自噬[24]。作为一个非炎症过程,细胞凋亡形态特征表现为细胞质、细胞核收缩,DNA损伤及凋亡体形成[25]。目前已知的细胞凋亡主要途径有3种,包括内源的线粒体介导细胞凋亡(又名BCL-2调节途径)、外源的死亡受体介导细胞凋亡和内质网途径的细胞凋亡[24]。细胞凋亡早期的重要特征是半胱氨酸依赖的磷脂酰丝氨酸(phosphatidylserine, PS)外翻引发“胞葬作用”(efferocytosis),吞噬细胞和溶酶体进一步清除凋亡细胞。与之相反,核心的自噬机制与吞噬细胞几乎无关,由30多个自噬相关基因编码,特征是垂死细胞本身存在自噬液泡,细胞质封存在自噬体内部,以便溶酶体进行本体降解[26]。
已有报道提出细菌PCD过程涉及的内在途径与真核细胞PCD相比高度保守,如细菌中用来调节死亡与裂解的holin样蛋白具有类似于促凋亡BCL-2蛋白的死亡效應[19]。除去真核生物特有的细胞结构,细菌的凋亡样程序性死亡 (apoptosis-like death, ADL) 与真核细胞凋亡在形态学上无明显差异,相似度极高(表1)[10,27]。
2 影响细菌PCD发生因素
2.1 外在不利压力引发PCD
研究表明多种压力应激源可诱导细菌产生PCD:热休克、极端pH、营养饥饿、抗生素杀伤、噬菌体攻击、暴露于异种生物和遗传毒性分子等外环境因素不适宜细菌生存,可诱发细菌“凋亡样”死亡过程,导致细胞分裂、染色体缩合和破碎、核糖体RNA降解、膜去极化和重塑,直至死亡[12,28-29]。其中,营养饥饿引发的PCD受鸟苷四磷酸(ppGpp)调控[11]。面对饥饿环境,细胞内快速积累ppGpp,严谨反应下停止大部分代谢活动,抑制核糖体和tRNA,整体转录水平下降,因而减弱mazEF基因转录[12,30]。MazEF属于II型毒素-抗毒素系统(toxin-antitoxin system, TA),由毒素MazF与抗毒素MazE组成,该系统与ppGpp介导的大肠埃希菌PCD有关。营养饥饿时MazEF转录与翻译位点在ppGpp作用下被抑制,不稳定蛋白质MazE被ClpAP丝氨酸蛋白酶降解,MazF以不依赖核糖体的方式切割RNA,进一步引发大肠埃希菌PCD[12]。
2.2 内源性ROS引发PCD
另一方面,细胞内在应激也会诱发细菌PCD,包括体内活性氧(reactive oxygen species, ROS)积累、DNA损伤等[30-31]。ROS通常包括超氧化物、过氧化氢和羟基自由基,过量ROS导致DNA断裂、膜去极化、蛋白质羰基化及细胞壁损伤,直接诱导细胞内在凋亡,且其作用通常是累计级联反应,即ROS介导的损伤可进一步引发ROS生成,加剧细胞破坏[32-33]。研究表明假微型海链藻(Thalassiosira pseudonana)中ROS含量与PCD标志物水平呈显著相关。在用ROS清除剂预处理后,所有PCD标志物百分比降低,死细胞比率也降低,表明随着细胞ROS清除,假微型海链藻群体中PCD发生率随之降低[31]。
2.3 群体感应系统引发PCD
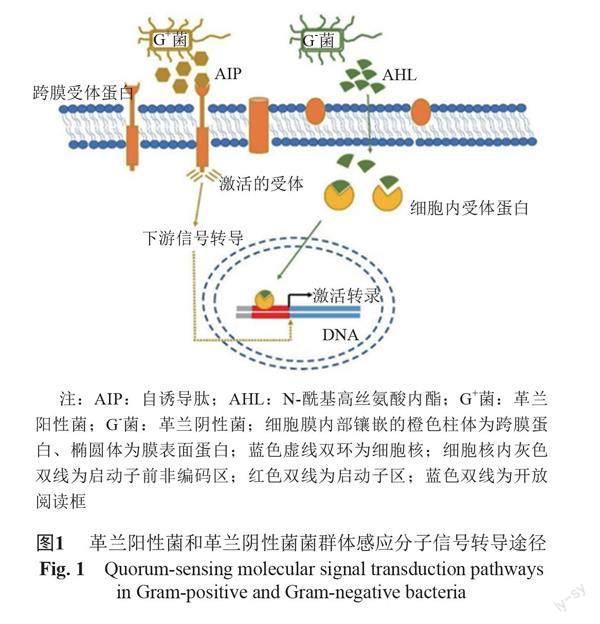
当细菌生长密度达到阈值时,微生物可分泌群体感应分子进行细胞间通讯,控制群体生命活动与行为[34]。群体感应(quorum sensing, QS)系统根据分泌的自诱导剂性质及其信号转导模式主要分为革兰阳性(Gram-positive)细菌和革兰阴性(Gram-negative)细菌两类(图1)[35]。上述信号分子不仅有利于种群内细菌之间通讯,也为种间交流奠定生物学基础。
高密度细菌群体营养物质有限,不利于细菌生长与代谢,因此控制细胞密度以优化代谢产生至关重要。QS系统在控制种群密度上发挥关键作用,通过引发部分细菌死亡为群体提供高质量代谢环境,与“利他主义”原理相吻合[35-36]。
如革兰阳性菌金黄色葡萄球菌通过Agr QS系统进行调节,由反应调节子AgrA和组氨酸激酶感应器AgrC组成,AgrC可通过表达RNA III调节子来响应自诱导多肽。RNA III和agrA的缺失株比野生型菌株存活率更高,表明野生型中存在由RNA III本身而非其编码的δ毒素介导的Agr系统发挥作用,导致部分细胞失活[37]。G-菌铜绿假单胞菌的QS依赖于3个相互联通的回路,las、rhl和pqs系统。作为其中最主要的QS系统,las可通过激活转录因子LasR来调节rhI和pqs。研究发现lasR突变体在培养36~54 h后细胞密度几乎维持恒定,而野生型生长密度已显著下降,证明LasR可介导细胞溶解与死亡[38]。在与宿主相互作用时,铜绿假单胞菌的QS系统还能调控宿主细胞PCD。QS分子N-(3-氧代十二酰基)-高丝氨酸内酯通过掺入哺乳动物的质膜诱导真核细胞脂质结构域溶解,引发caspase 3-caspase 8介导的细胞凋亡,抑制宿主天然免疫反应,增强自身在宿主内生存定植,可见其促凋亡作用是调节免疫活性的主力军[39-40]。
3 细菌利用PCD应对抗生素压力
抗生素杀伤细菌主要通过靶向30S或50S核糖体亚基抑制蛋白质合成,或抑制细胞壁、核酸、ATP合成等,进一步扰动细胞代谢水平造成细菌损伤,细胞代谢状态改变又会反向影响其对抗生素敏感性[41-42]。面对抗生素压力时细菌会利用PCD“利他性”实现群体利益更大化:一方面PCD指向降低环境中抗生素浓度途径,如死细菌透过膜吸收抗生素或释放酶降解抗生素以抵抗外界压力[43-44];另一方面,PCD指向促进抗菌物质释放途径,如细菌种间与细菌种内竞争时产生抗菌物质实现竞争优势[1,45]。可见抗生素压力下,PCD与抗菌物质的产生与减少都密不可分。
3.1 PCD促进抗菌物质释放
3.1.1 细菌种间竞争
在压力应激时,细菌能够自发产生广谱抗菌物质抵抗其他与之竞争的细菌群体,如被人熟知的抗菌肽。虽然细菌细胞不能自发分泌抗菌化合物,但可通过裂解释放抗菌物质。如在不同细菌间相互竞争时,大肠埃希菌产生的抗菌肽被释放后,可以杀死竞争细菌,有利于剩余大肠埃希菌群生存[45]。
3.1.2 细菌种内竞争
在细菌发育进程中不仅存在种间竞争,还存在种群内杀伤:在抗生素作用下某一亚群分泌抗菌物质以杀死另一亚群,以此获取胞外同源DNA增强遗传多样性,最终使种群存活率升高,该现象被认为是表型异质性的极端形式之一[1]。如在枯草芽胞杆菌中,同类相食是一种分化策略,可增强生物膜形成,在饥饿条件下延缓甚至阻止内孢子形成,同时可保护生物膜内细胞免受竞争物种侵害[1]。
综上可见,细菌群体可通过分泌抗菌物质实现对竞争群体和同类细菌群的部分杀伤,不仅利于同种亲缘菌群,也能抑制其他物种生长[21]。
3.2 PCD减少环境中抗生素浓度
3.2.1 死细胞降低抗生素浓度
研究发现抗生素压力下的死亡细菌能降低环境中抗生素浓度以保护其他活菌群体。如人源性LL37抗菌肽能透化大肠埃希菌细胞膜以杀死部分亚群,但随之形成的死细胞会借助破膜渗透的方式从环境中吸收剩余LL37来降低抗菌肽疗效,从而保护更多活菌,形成集体耐受[43]。基于数学建模分析,结合群体与单细胞实验,发现“细菌吸收LL37的时间与其细胞质膜的渗透时间一致”,该现象证实了死细菌能够吸收抗菌肽[43]。
3.2.2 细菌释放酶降解抗生素
在一定程度上可用利他性死亡来解释高浓度抗生素杀菌的“eagle effect”现象,即病原菌整体密度表现出与抗生素作用浓度非单调的对应关系,高抗生素浓度时杀菌效果反而降低。具体表现在抗生素压力下,部分细菌裂解并释放β-内酰胺酶,快速降解环境中抗生素,以更快地恢复种群密度减弱抗生素杀伤[44]。
4 基于细菌PCD行为的抗菌新策略
抗生素的泛用、滥用使得越来越多的耐药菌涌现。耐药菌的产生是细菌群体在最初面临抗生素压力时,少量抗性突变体以自我牺牲为代价,通过产生如吲哚类的信号分子打开药物外排泵将抗生素输送至细胞外、产生氧化应激保护机制,为相对药物敏感的细胞提供保护,从而提高整个种群生存能力[46-47],这也是PCD为最初产生耐药菌所做的贡献。提升现有抗菌药物杀伤病原菌能力以及研发新型抗菌药物是目前抗菌药物研发两大主要方向,PCD在这两方面均能提供新策略。
4.1 诱发PCD促进细菌细胞死亡
就提升目前抗菌药物的杀伤力方面,激活PCD可以增强细菌死亡程度,实现有效杀伤。例如原核生物中保守性较高的TA系统已被发现可以作为抗生素靶标开发的重要靶点之一[48]。早在2004年已证明抗生素如氯霉素、大观霉素与利福平可通过抑制转录和翻译、激活MazEF途径来诱发细菌PCD[3,49]。一方面可追求抗毒素快速降解,使对应毒素更易释放。另一方面,设计抗毒素结构类似物与底物竞争性结合也是手段之一。但这些终归是间接的,开发新型化合物直接靶向这些死亡途径而非间接激活死亡途径是新型抗生素研发的重要途径。
4.2 干扰PCD抑制细菌抵抗能力
短期看PCD激活能杀伤部分细菌群,为病原菌治疗提供思路。但根据在群体生成PCD的生物学原理,从长远看会产生更多“公共利益”,而这些公共代谢物反向缓解压力,使更多病原菌恢复菌量成为可能[50]。可见PCD效应与“公共利益”是非线性关系,把握二者平衡至关重要。从PCD基于群体合作的角度看,降低PCD以减少产生“公共利益”更有利于细菌杀伤。
4.2.1 干扰反馈回路以加强细菌杀伤
抗菌肽作用下已经被杀死的细菌能反向吸收环境中抗菌肽,降低浓度以减少杀伤[43]。由此提出的治疗策略是干扰死菌反向吸收作用,该反馈回路强调抗菌肽治疗期间死亡细菌对群体生存的保护。如使用肽佐剂阻碍死细菌的反向吸收,降低病原菌群体中死细胞对其余活细胞的保护能力,以提升抗菌药物疗效[43]。
4.2.2 群体感应抑制剂相关新型抗菌剂
病原菌中群体感应诱发PCD使得更多细菌群体得以生长、进一步产生感染,因此QS抑制剂这类新型抗菌剂研发成为阻断PCD产生的重要手段之一。QS抑制剂的研发起源于不同微生物争夺相同生态位,已知革兰阴性菌的N-酰基高丝氨酸内酯(AHL) 可与革兰阴性菌金黄色葡萄球菌的细胞膜相互作用,降低外毒素产生,抑制其Agr系统表达[51]。近年来,临床上研发QS抑制剂是防治铜绿假单胞菌定植和致病的主要方向之一,如设计LasR受体拮抗剂中断级联反应、降低病原体毒力[52]。Kalia等[53]从数据库中筛选得到8个铅类化合物,通过分子对接软件模拟,发现这些化合物利用不同氨基酸残基与烯酰还原酶FabI结合形成氢键以抑制AHL酰基链生成,从酶类药物靶标角度为铜绿假单胞菌QS抑制劑的开发提供了可能。
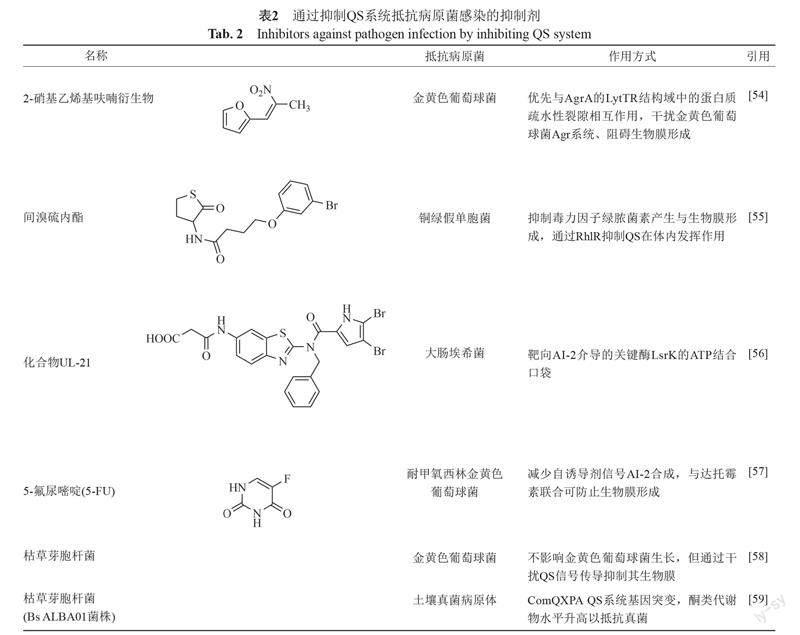
QS小分子抑制剂可作为预防细菌致病性的潜在治疗药物,具有广阔发展前景。表2罗列了通过抑制QS系统抵抗病原菌感染的物质名称与作用方式等。
可见,QS抑制剂作为干扰PCD反馈回路的重要分支,主要通过影响生物膜形成来抵抗致病菌,在抗感染治疗方面有卓越潜力。目前通过技术手段对QS分子抑制剂进行筛选成为主要趋势,如成立数据储存库、开发新型灵敏发光测定法等,可快速高通量地对潜在抑制剂进行虚拟筛选,进而通过分析检测数据评估其与拟定靶点的吻合程度[53,56,60]。
5 总结与展望
综合上述内容表明,细菌PCD对于抗菌治疗是一把“双刃剑”,应合理把握平衡。在抗生素用药的基础上人为诱发细菌PCD或拮抗细菌QS通讯以抑制群体内部利他性质的PCD,使细菌群体更容易被杀伤;而值得注意的是PCD也能被病原菌加以利用作为其适应宿主环境生存进化的有力武器。
因而,需要推出新抗菌感染策略以應对耐药性增强的病原菌和新病原菌。人工合成的化合物或从生物中分离得到的小分子抗菌蛋白具有良好治疗潜力,它们稳定性高,耐药风险低,对哺乳动物细胞无毒性,具有促凋亡活性,有望在不久的将来成为治疗病原菌的候选药物。目前,迫切需要深入研究来阐明这类抗菌药物诱导细菌PCD的靶点。从机制而言,细菌PCD的产生路径远不如真核细胞PCD通路研究成熟。现在尚不清楚是否有任何基因或蛋白质与抗菌治疗期间细菌的异质性行为有关。鉴于目前全基因组测序已成为广泛使用的研究工具,获取耐药菌株基因组序列有助于寻找新的抗菌蛋白,也有利于筛选靶向致病菌PCD的新治疗靶点。
致谢:感谢厦门大学公共卫生学院研究生王炜捷、朱伟伟对文章撰写所提出的宝贵意见及建议。
参 考 文 献
Popp P F, Mascher T. Coordinated cell death in isogenic bacterial populations: Sacrificing some for the benefit of many?[J]. J Mol Biol, 2019, 431(23): 4656-4669.
Smith R P, Barraza I, Quinn R J, et al. The mechanisms and cell signaling pathways of programmed cell death in the bacterial world[J]. Int Rev Cell Mol Biol, 2020, 352: 1-53.
Peeters S H, de Jonge M I. For the greater good: Programmed cell death in bacterial communities[J]. Microbiol Res, 2018, 207: 161-169.
Zemke A C, Bomberger J M. Microbiology: Social suicide for a good cause[J]. Curr Biol, 2016, 26(2): R80-R82.
Allocati N, Masulli M, Di Ilio C, et al. Die for the community: An overview of programmed cell death in bacteria [J]. Cell Death Dis, 2015, 6(1): e1609.
Wadhawan S, Gautam S, Sharma A. Involvement of proline oxidase (PutA) in programmed cell death of Xanthomonas[J]. PLoS One, 2014, 9(5): e96423.
Rice K C, Mann E E, Endres J L, et al. The cidA murein hydrolase regulator contributes to DNA release and biofilm development in Staphylococcus aureus[J]. Proc Natl Acad Sci U S A, 2007, 104(19): 8113-8118.
McFarland K A, Dolben E L, LeRoux M, et al. A self-lysis pathway that enhances the virulence of a pathogenic bacterium[J]. Proc Natl Acad Sci USA, 2015, 112(27): 8433-8438.
Wadhawan S, Gautam S. Rescue of Escherichia coli cells from UV-induced death and filamentation by caspase-3 inhibitor[J]. Int Microbiol, 2019, 22(3): 369-376.
Burgess Tornaletti L, Manina G. Delving into the functional meaning of phenotypic variation in mycobacterial persistence: Who benefits the most from programmed death of individual cells?[J]. Microbiol Insights, 2020, 13: 1178636120945304.
Aizenman E, Engelberg-Kulka H, Glaser G. An Escherichia coli chromosomal "addiction module" regulated by guanosine [corrected] 3', 5'-bispyrophosphate: A model for programmed bacterial cell death[J]. Proc Natl Acad Sci U S A, 1996, 93(12): 6059-6063.
Ramisetty B C, Natarajan B, Santhosh R S. mazEF-mediated programmed cell death in bacteria: “what is this?”[J]. Crit Rev Microbiol, 2015, 41(1): 89-100.
World Health Organization. Antimicrobial resistance[EB/OL]. (2021-11-17)[2022-10-13] https: //www.who.int/en/news-room/fact-sheets/detail/antimicrobial-resistance.
Antimicrobial Resistance Collaborators. Global burden of bacterial antimicrobial resistance in 2019: A systematic analysis[J]. Lancet, 2022, 399(10325): 629-655.
U.S. Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019 [EB/OL]. (2019-12)[2022-10-13]. https: //www.cdc.gov/drugresistance/biggest-threats.html.
Oneill J. The review on antimicrobial resistance[EB/OL]. (2016-5)[2022-10-13]. https: //amr-review.org/.
Naito T, Kusano K, Kobayashi I. Selfish behavior of restriction-modification systems[J]. Science, 1995, 267(5199): 897-899.
Yarmolinsky M B. Programmed cell death in bacterial populations[J]. Science, 1995, 267(5199): 836-837.
Bayles K W. Bacterial programmed cell death: Making sense of a paradox[J]. Nat Rev Microbiol, 2014, 12(1): 63-69.
Michod R E. The theory of kin selection[J]. Annu Rev Ecol Syst, 1982, 13(1): 23-55.
Durand P M, Choudhury R, Rashidi A, et al. Programmed death in a unicellular organism has species-specific fitness effects[J]. Biol Lett, 2014, 10(2): 20131088.
Durand P M, Sym S, Michod R E. Programmed cell death and complexity in microbial systems[J]. Curr Biol, 2016, 26(13): R587-R593.
Galluzzi L, Vitale I, Aaronson S A, et al. Molecular mechanisms of cell death: Recommendations of the Nomenclature Committee on Cell Death 2018[J]. Cell Death Differ, 2018, 25(3): 486-541.
DArcy M S. Cell death: A review of the major forms of apoptosis, necrosis and autophagy[J]. Cell Biol Int, 2019, 43(6): 582-592.
Zhang Y, Chen X, Gueydan C, et al. Plasma membrane changes during programmed cell deaths[J]. Cell Res, 2018, 28(1): 9-21.
Bedoui S, Herold M J, Strasser A. Emerging connectivity of programmed cell death pathways and its physiological implications[J]. Nat Rev Mol Cell Biol, 2020, 21(11): 678-695.
Kroemer G, El-Deiry W S, Golstein P, et al. Classification of cell death: Recommendations of the nomenclature committee on cell death[J]. Cell Death Differ, 2005, 12 (Suppl 2): 1463-1467.
Nam Y, Goo E, Kang Y, et al. Membrane depolarization and apoptosis-like cell death in an alkaline environment in the rice pathogen Burkholderia glumae[J]. Front Microbiol, 2021, 12: 755596.
Dwyer D J, Camacho D M, Kohanski M A, et al. Antibiotic-induced bacterial cell death exhibits physiological and biochemical hallmarks of apoptosis[J]. Mol Cell, 2012, 46(5): 561-572.
Lee H, Lee D G. Programmed cell death in bacterial community: Mechanisms of action, causes and consequences[J]. J Microbiol Biotechnol, 2019, 29(7): 1014-1021.
Zhao Y, Tang X, Qu F, et al. ROS-mediated programmed cell death (PCD) of Thalassiosira pseudonana under the stress of BDE-47[J]. Environ Pollut, 2020, 262: 114342.
李娇. 超声场胁迫下大肠杆菌O157: H7的致死效应及分子机制研究[D]. 杭州: 浙江大学, 2019.
Hong Y, Zeng J, Wang X, et al. Post-stress bacterial cell death mediated by reactive oxygen species[J]. Proc Natl Acad Sci U S A, 2019, 116(20): 10064-10071.
Abisado R G, Benomar S, Klaus J R, et al. Bacterial quorum sensing and microbial community interactions[J]. mBio, 2018, 9(3): e02331-17.
Wu S, Liu J, Liu C, et al. Quorum sensing for population-level control of bacteria and potential therapeutic applications[J]. Cell Mol Life Sci, 2020, 77(7): 1319-1343.
Diggle S P, Griffin A S, Campbell G S, et al. Cooperation and conflict in quorum-sensing bacterial populations[J]. Nature, 2007, 450(7168): 411-414.
Paulander W, Varming A N, Bojer M S, et al. The agr quorum sensing system in Staphylococcus aureus cells mediates death of sub-population[J]. BMC Res Notes, 2018, 11(1): 503.
Heurlier K, Dénervaud V, Haenni M, et al. Quorum-sensing-negative (lasR) mutants of Pseudomonas aeruginosa avoid cell lysis and death[J]. J Bacteriol, 2005, 187(14): 4875-4883.
Song D, Meng J, Cheng J, et al. Pseudomonas aeruginosa quorum-sensing metabolite induces host immune cell death through cell surface lipid domain dissolution[J]. Nat Microbiol, 2019, 4(1): 97-111.
祝司霞. 铜绿假单胞菌群体感应代谢产物通过细胞表面脂质结构域溶解诱导宿主淋巴細胞死亡的机制[J]. 中国免疫学杂志, 2021, 37(8): 922-926.
Stokes J M, Lopatkin A J, Lobritz M A, et al. Bacterial metabolism and antibiotic efficacy[J]. Cell Metab, 2019, 30(2): 251-259.
Hutchings M I, Truman A W, Wilkinson B. Antibiotics: Past, present and future[J]. Curr Opin Microbiol, 2019, 51: 72-80.
Wu F, Tan C. Dead bacterial absorption of antimicrobial peptides underlies collective tolerance[J]. J R Soc Interface, 2019, 16(151): 20180701.
Tanouchi Y, Pai A, Buchler N E, et al. Programming stress-induced altruistic death in engineered bacteria[J]. Mol Syst Biol, 2012, 8: 626.
Mavridou D A I, Gonzalez D, Kim W, et al. Bacteria use collective behavior to generate diverse combat strategies[J]. Curr Biol, 2018, 28(3): 345-355.e344.
Lee H H, Molla M N, Cantor C R, et al. Bacterial charity work leads to population-wide resistance[J]. Nature, 2010, 467(7311): 82-85.
李昕, 曾洁, 王岱, 等. 细菌耐药耐受性机制的最新研究进展[J]. 中国抗生素杂志, 2020, 45(2): 113-121.
海洋, 王小雨, 谢建平. 细菌毒素-抗毒素系统在噬菌体流产感染中的作用[J]. 生物工程学报, 2022, 38(9): 3291-3300.
Engelberg-Kulka H, Sat B, Reches M, et al. Bacterial programmed cell death systems as targets for antibiotics[J]. Trends Microbiol, 2004, 12(2): 66-71.
Tanouchi Y, Lee A J, Meredith H, et al. Programmed cell death in bacteria and implications for antibiotic therapy[J]. Trends Microbiol, 2013, 21(6): 265-270.
Qazi S, Middleton B, Muharram S H, et al. N-acylhomoserine lactones antagonize virulence gene expression and quorum sensing in Staphylococcus aureus[J]. Infect Immun, 2006, 74(2): 910-919.
Duplantier M, Lohou E, Sonnet P. Quorum sensing inhibitors to quench P. aeruginosa pathogenicity[J]. Pharmaceuticals (Basel), 2021, 14(12): 1262.
Kalia M, Yadav V K, Singh P K, et al. Designing quorum sensing inhibitors of Pseudomonas aeruginosa utilizing FabI: An enzymic drug target from fatty acid synthesis pathway[J]. 3 Biotech, 2019, 9(2): 40.
Oliveira D, Borges A, Ruiz R M, et al. 2-(2-Methyl-2-nitrovinyl) furan but not furvina interfere with Staphylococcus aureus Agr quorum-sensing system and potentiate the action of fusidic acid against biofilms[J]. Int J Mol Sci, 2021, 22(2): 613.
O'Loughlin C T, Miller L C, Siryaporn A, et al. A quorum-sensing inhibitor blocks Pseudomonas aeruginosa virulence and biofilm formation[J]. Proc Natl Acad Sci U S A, 2013, 110(44): 17981-17986.
Gatta V, Toma?i? T, Ila? J, et al. A new cell-based AI-2-mediated quorum sensing interference assay in screening of Lsrk-targeted inhibitors[J]. Chembiochem, 2020, 21(13): 1918-1922.
Sedlmayer F, Woischnig A K, Unterreiner V, et al. 5-fluorouracil blocks quorum-sensing of biofilm-embedded methicillin-resistant Staphylococcus aureus in mice[J]. Nucleic Acids Res, 2021, 49(13): e73.
Qiu M, Feng L, Zhao C, et al. Commensal Bacillus subtilis from cow milk inhibits Staphylococcus aureus biofilm formation and mastitis in mice[J]. FEMS Microbiol Ecol, 2022, 98(7): fiac065.
Albarracín Orio A G, Petras D, Tobares R A, et al. Fungal-bacterial interaction selects for quorum sensing mutants with increased production of natural antifungal compounds[J]. Commun Biol, 2020, 3(1): 670.
Wu S, Liu C, Feng J, et al. QSIdb: quorum sensing interference molecules[J]. Brief Bioinform, 2021, 22(4): bbaa218.
收稿日期:2022-11-08
基金項目:国家自然科学基金(No. 81971905;No. 82272349)
作者简介:刘伟佳,女,生于1998年,在读硕士研究生,主要研究方向为肠出血性大肠埃希菌中T3SS的作用,E-mail: 1319945429@qq.com.
*通信作者,E-mail: daiwang@xmu.edu.cn
