山东某地区产气荚膜梭菌的分子型别与耐药特征
2023-12-26钟佳鑫王媛媛白璐璐郑浩然周海健吴媛卢金星
钟佳鑫?王媛媛?白璐璐?郑浩然?周海健?吴媛?卢金星
摘要:目的 分析山东省某地区临床及动物中分离的产气荚膜梭菌的分子型别及耐药特征。方法 对山东省56株产气荚膜梭菌进行PFGE、MLST分子分型、药敏试验和耐药基因检测,比较分析同一来源菌株内部和不同来源菌株间的型别和耐药特征。结果 56株产气荚膜梭菌分为53个PFGE型,有50株菌表现出独特的型别。MLST分型将其分为46个ST型,其中40种ST型仅包含一株菌。本研究中菌株均对万古霉素,美罗培南和头孢西丁表现为敏感;对甲硝唑、青霉素、莫西沙星和氯霉素的耐药率相对比较低,分别为3.57%、7.14%、8.93%和1.79%;然而,对克林霉素的耐药率高达53.57%,人类来源菌株与动物来源菌株的耐药谱呈现出较为明显的差异。基于全基因组的耐药基因分析显示,所有56株产气荚膜梭菌均携带mprF基因,其次是tetA(P) (98.21%),tetB(P) (87.5%)以及ermQ (51.79%)。结论 我国山东某地区临床来源与动物来源产气荚膜梭菌耐药谱存在显著差异,同一地区的菌株分子型别具有很强的多样性。需要开展更全面的的系统监测为制定控制措施提供流行病学依据。
關键词:产气荚膜梭菌;脉冲场凝胶电泳分型;多位点序列分型;抗菌药物敏感性;耐药基因;分子流行病学
中图分类号:R978.1 文献标志码:A
Molecular typing and drug resistance characteristics of Clostridium perfringens in Shandong Area
Zhong Jiaxin, Wang Yuanyuan, Bai Lulu, Zheng Haoran , Zhou Haijian , Wu Yuan, and Lu Jinxing
(State Key Laboratory of Infectious Disease Prevention and Control, Institute of Infectious Disease Prevention and Control, Chinese Center for Disease Control and Prevention, Beijing 102206)
Abstract Objective To analyze the molecular type and drug resistance characteristics of Clostridium perfringens isolated from clinics and animals in a region of Shandong Province. Methods The molecular typing of PFGE and MLST, drug sensitivity tests, and drug resistance gene detection were performed on 56 strains of Clostridium perfringens in Shandong Province, and the phenotypic and drug resistance characteristics within and between strains of the same origin were compared and analyzed. Results The 56 Clostridium perfringens strains were classified into 53 PFGE types, with 50 strains exhibiting unique phenotypes. The MLST typing classified them into 46 ST types, with 40 ST types containing only one strain. All strains in this study showed sensitivity to vancomycin, meropenem, and cefoxitin. Resistance rates to metronidazole, penicillin, moxifloxacin and chloramphenicol were relatively low. The resistance rates were 3.57%, 7.14%, 8.93%, and 1.79% respectively. However, resistance rates to clindamycin were as high as 53.57%, showing a more pronounced difference in the resistance profiles of strains of human versus animal origin. Genome-wide resistance gene analysis showed that all 56 Clostridium perfringens strains carried the mprF gene, followed by tetA(P) (98.21%), tetB(P) (87.5%), and ermQ (51.79%). Conclusion There are significant differences in the drug resistance profiles of Clostridium perfringens from clinical and animal sources in one region of Shandong, China. Molecular strains showed high diversity in the same region. More comprehensive and systematic surveillance is needed to provide an epidemiological basis for control measures.
Key words Clostridium perfringens; Pulsed-field gel electrophoresis typing; Multilocus sequence typing; Antimicrobial susceptibility; Drug resistance genes; Molecular epidemiology
产气荚膜梭菌是一种在自然界中广泛存在的革兰阳性厌氧菌。该细菌在动物和人类中都能引起气性坏疽、肠道疾病和食物中毒,是全球公认的最重要的胃肠道病原体之一[1]。其重要的致病机制是产生多种毒素和酶[2]。抗生素相关性腹泻(antibiotic-associated diarrhea,AAD)在住院患者和社区中仍然是一个主要的医疗问题,住院患者中产气荚膜梭菌作为AAD相关细菌的患病率为14.9%,位于第二[3-4]。近年来,产气荚膜梭菌的耐药性有所增加[5],一些调查表明,大多数动物来源的产气荚膜梭菌为多重耐药菌株(MDR)[6]。人类来源的产气荚膜梭菌多重耐药菌株比例很低[7]。然而,对中国产气荚膜梭菌耐药性的研究尚且不足。
脉冲场凝胶电泳(PFGE)是最重要的检测细菌基因型多样性分子分型方法之一,由于其结果能够检测90%以上的细菌基因组,并且适用于广泛的病原体,因此依旧被公认为是食源性病原菌分型的金标准[8]。本研究收集了2013—2020年山东地区一家三级综合医院从临床标本中分离的菌株及山东地区3家养殖场分离的56株产气荚膜梭菌,以脉冲场凝胶电泳(PFGE)和多位点序列分型(MLST)为分子工具并结合抗菌药物敏感性检测,旨在了解山东地区人与动物源产气荚膜梭菌分离株的异同。
1 材料和方法
1.1 材料
1.1.1 菌株来源及基本情况
56株产气荚膜梭菌来源于2017—2020年山东省某医院抗生素相关腹泻患者和山东某三家养殖场中的猪、牛和羊,人来源样本按照AAD病例定义由临床实验室收集;动物来源样本采集自新鲜的动物粪便;样本均带回本课题组实验室检测,经16S rRNA測序对分离菌株进行鉴定。PFGE标准Marker菌株H9812由国家致病菌识别网PulseNet China提供。
1.1.2 主要试剂与仪器
药敏试验采用E-test条法,E-test条购自公司利飞驰,质控菌株产气荚膜梭菌ATCC13124购自美国模式菌种收集中心,细菌基因组DNA的提取使用天根生化科技有限公司的试剂盒,DNA Marker、Premix Taq购于北京奥科生物工程有限公司,PCR仪(德国Senso Quest Labcycler公司),PFGE图像形成使用Bio-Rad公司的脉冲场凝胶电泳仪和Gel Doc XR+ 凝胶成像系统。
1.2 方法
1.2.1 细菌培养、鉴定及细菌基因组DNA提取
在血琼脂平板上按照三区划线原则进行接种,置于恒温培养箱(37℃±1℃)在厌氧环境下培养18~24 h,采用16S rRNA测序进行产气荚膜梭菌的鉴定。采用DNA提取试剂盒(天根生化科技有限公司)提取菌株DNA。
1.2.2 PFGE实验和分析
参照国家致病菌识别网实验室使用的革兰阳性菌标准化PFGE分子分型方法,对分离纯化的产气荚膜梭菌进行DNA胶块的制备,经限制性内切酶酶切后进行脉冲场凝胶电泳分型。电泳条件:电压6V/cm,脉冲参数0.5~40 s,电泳22 h。利用BioNumerics软件读取并聚类分析PFGE电泳图谱。
1.2.3 多位点序列分型
8个管家基因引物序列colA、groEL、sodA、plc、gyrB、sigK、pgk和nadA参照PubMLST数据库(https://pubmlst.org/organisms/clostridium-perfringens)。引物序列由北京奥科生物工程有限公司合成。
PCR反应总体积为25 μL,Premix Taq 12.5 μL,前后引物各0.5 μL,模板1.5 μL,无菌水10 μL。PCR反应条件:94℃预变性2 min,一个循环;94℃变性30 s,55℃退火60 s,72℃延伸60 s,共35个循环;72℃延伸8 min,1个循环[9]。取4 μL PCR产物进行琼脂糖凝胶电泳,PCR产物阳性的,送至北京奥科生物工程有限公司进行双向测序,将序列提交到PubMLST官网数据库(https://pubmlst.org/organisms/clostridium-perfringens)进行比对,确定每株菌株的等位基因号与序列型(ST)并提交新的等位基因与ST型。
1.2.4 药敏实验
采用E-test条法,以美国临床和实验室标准化协会(CLSI)M100-S30, 2020文件和欧洲抗菌药物敏感性测试委员会(EUCAST)v.10.0, 2020文件作为折点判读标准,对克林霉素、氯霉素、万古霉素、头孢西丁、美罗培南、甲硝唑、青霉素和莫西沙星8种抗菌药物的最小抑菌浓度(minimal inhibitory concentration,MIC)进行检测(万古霉素的MIC在CLSI中未标明,以EUCAST中的折点作为判读标准。其余7种抗菌药物的MIC均以CLSI为为折点判读标准。克林霉素(≥8 ?g/mL)、氯霉素(≥32 ?g/mL)、利福平(≥64 ?g/mL)、美罗培南(≥16 ?g/mL)、甲硝唑(≥32 ?g/mL)、青霉素(≥2 ?g/mL)、莫西沙星(≥2 ?g/mL)、万古霉素(>2 ?g/mL)。质控菌株为产气荚膜梭菌ATCC13124,购自美国典型培养物保藏中心。
挑取单克隆菌株接种于血琼脂平板,37℃厌氧状态下培养18~24 h,于布氏肉汤中将菌液浓度调整为3×108 CFU/mL。用无菌棉签蘸取菌液均匀涂布在添加5%羊血、5 mg/L氯化血红素和1 mg/L维生素K1的布氏平板上,待平板表面水分吸收后,贴E-test试条。置于37℃厌氧状态下培养18~24 h后取出,进行结果判读,培养基表面水滴状抑菌环指向的浓度值为检测的MIC。
1.2.5 基于全基因组的耐药基因分析
在注释时,使用CARD数据库中的工具软件rgi将基因的蛋白序列分别数据库进行比对,找出比对上的数据库中对应序列,并得到对应的耐药基因和耐药基因相关信息。并设置参数“ ≥80% identity and ≥50% coverage”,比对上的基因描述于文中。
1.2.6 统计分析
耐药谱构成比差异使用Pearson χ2检验或Fisher确切概率法进行检验,检验水准α为0.05。P<0.05为统计学差异显著。最终使用Fisher确切概率法检验结果。
2 结果
2.1 PFGE聚类分析
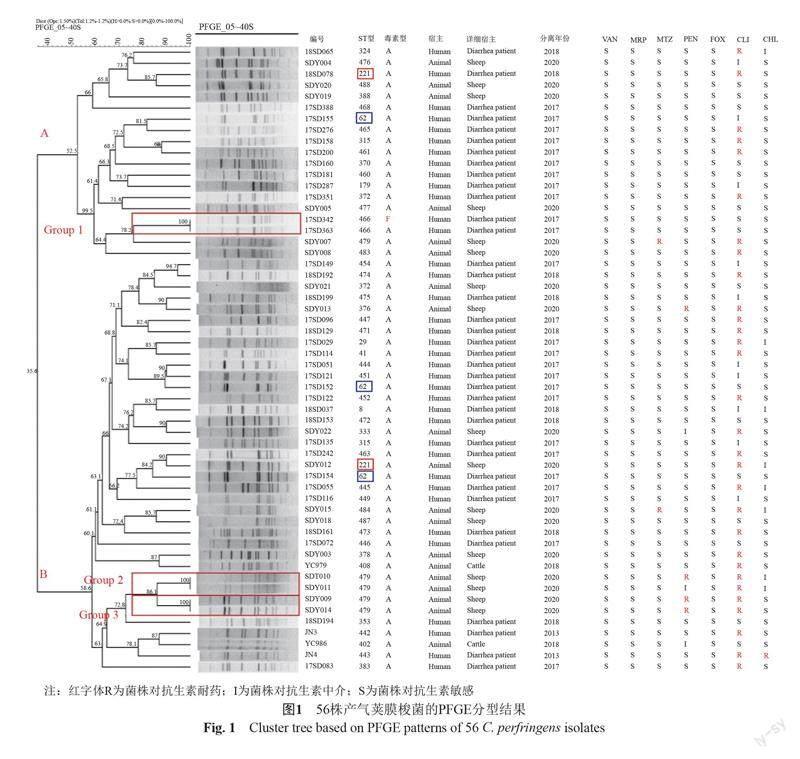
56株山东地区产气荚膜梭菌共分为53个型别,其中3个型别包含2株菌,50个型别仅包含1株菌,即有50株菌表现出独特的型别(图1)。56株菌的PFGE图谱相似性指数为35.6%~100%。通过PFGE分析,将56株菌分为2个主要类群即:“A”(19株)和“B”(37株),见图1。这些菌株并没有呈现出耐药菌株聚为某一特定克隆群或抗生素相关性腹泻菌株聚为某一克隆群,整体聚类特征表现出较强的多样性。
这56株菌中37株来源于抗生素相关性腹泻患者,共有36种带型,Group 1包含两个重复出现的带型,在菌株17SD342和菌株17SD363中共有;19株来源于养殖场中的猪、牛和羊,Group 2(菌株SDY010与菌株SDY011)和Group 3(菌株SDY009与菌株SDY014)分别包含两个重复出现的带型。抗生素相关性腹泻患者来源菌株和动物来源菌株并没有存在相同的带型。
通过菌株的背景信息和基因指纹图谱结合分析,分离自两位抗生素相关性腹泻患者的菌株(17SD342、17SD363)具有100%的遗传相似性,代表同一种PFGE型别,且前一组同属ST466。这一组条带完全一致的菌株分离自山东一家医院,且两位患者的就医时间接近,怀疑在就医过程中产气荚膜梭菌存在潜在的传播。分离自山东绵羊的菌株(SDY010和SDY011)与(SDY009和SDY014)同属于ST479,分别具有100%的遗传相似性,这两组条带完全一致的菌株来自同一家绵羊养殖场,提示在养殖场内可能存在产气荚膜梭菌的传播。
然而,整体PFGE结果提示不同宿主来源,差异较大,具有较强的遗传多样性。即使是分离自同一所医院患者的菌株或分离自同一家养殖场的菌株也具有很强的遗传多样性。
本研究结果表明,PFGE和MLST在产气荚膜梭菌分型方面均具有较高的分辨力。PFGE 类型与ST型别之间的一对一相关性表明,属于同一ST型的一些分离株具有不同的PFGE型别,例如SDY009、SDY010、SDY011和SDY014同属于ST479但被分为两种不同的PFGE型别(图1);18SD078与SDY012同属ST221但被分为两种不同的PFGE型别;17SD155,17SD152,17SD154同属ST62,但被分为3种不同的PFGE型别。而研究中相同PFGE分子型别的3组菌株(Group 1、Group 2、Group 3)分别属于相同的ST型别(ST466、ST479、ST479)。因此,对于产气荚膜梭菌来说,PFGE分型可能具有更高的分辨力,更适合用于产气荚膜梭菌的溯源研究。
2.2 MLST分型
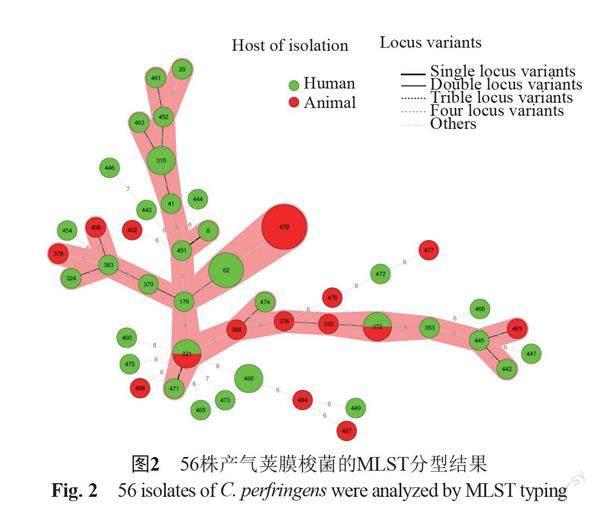
56株产气荚膜梭菌MLST分型共发现46个ST型,具有很强的遗传多样性,其中33株分离株出现新序列型,共27个新序列型,占分离株的58.93%。优势型别为ST479(5株),ST62(3株),ST221(2株),ST315(2株),ST372(2株),ST466(2株),其余40种ST型各1株。动物来源菌株和人来源菌株的分布并没有呈现出明显的差异或聚为不同的克隆群。其中动物来源的分离株优势型别为ST479(5株),人类来源的分离株优势型别为ST62(3株)。
使用BioNumerics软件分析,得到最小生成树(MST)(图2)。根据参数(相邻等位基因差异个数≤5,所包含的ST型≥3)[10],本研究中属于不同STs的山东来源产气荚膜梭菌大部分(36株)聚在一起,表明按照此参数大部分山东地区的菌株相对亲缘关系较近。其余菌株(20株)围绕该克隆群呈散在分布,彼此之间存在较远的遗传距离(管家基因差异≥6)。但如果缩小相邻等位基因差异个数,例如将参数调为相邻等位基因差异个数≤3,所包含的ST型≥3,则呈现出多个克隆群,呈现出较强的多样性。
2.3 产气荚膜梭菌的药敏情况和耐药基因检测
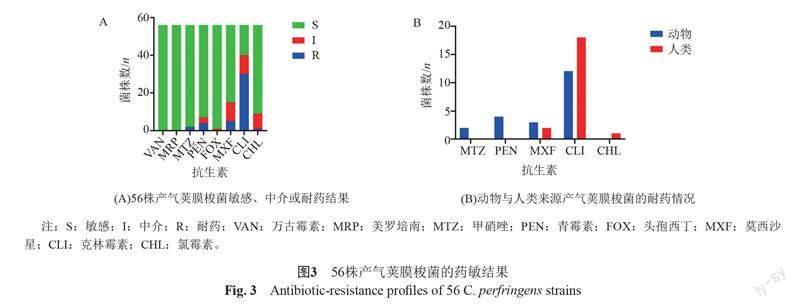
所有试验菌株均对万古霉素,美罗培南和头孢西丁表现为完全敏感;对甲硝唑、青霉素、莫西沙星和氯霉素的耐药率相对比较低,分别为3.57%、7.14%、8.93%和1.79%;然而,对克林霉素的耐药率高达53.57%(图3A)。同时本研究发现,山东地区人类来源菌株与动物来源菌株的耐药谱存在显著的统计学差异(P=0.006)(图3B)。人类来源的部分菌株对莫西沙星、克林霉素、氯霉素耐药,而动物来源的部分菌株对甲硝唑、青霉素、莫西沙星、克林霉素耐药。按动物与人类来源分组的产气荚膜梭菌耐药谱见图3B,无论物种,大多数菌株(63.16%与48.65%)都对克林霉素耐药。对于人类来源的分离株,均对万古霉素、美罗培南、甲硝唑、青霉素和头孢西丁敏感,莫西沙星(5.41%)和氯霉素(2.70%)的耐药率较低,而克林霉素的耐药率(48.65%)则处于相对较高的水平。对于动物来源的分离株,均对万古霉素、美罗培南、头孢西丁和氯霉素敏感,对测试抗生素耐药率由高到低依次为克林霉素(63.16%)、青霉素(21.05%)、莫西沙星(15.79%)、甲硝唑(10.53%)。本研究中菌株的多重耐药率较低,多重耐药菌株(对大于等于3类抗菌药物耐药[11])的比例为3.57%(2/56),分别从绵羊(SDY007)和腹泻患者(JN4)中分离得到,属于ST479与ST443,SDY007对甲硝唑、莫西沙星、克林霉素;JN4对莫西沙星、克林霉素、氯霉素3种抗生素耐藥(图1)。没有分离株对4种及以上的抗生素有耐药性(图1)。
耐药基因检测结果显示,56株产气荚膜梭菌中肽类抗生素中mprF基因携带率最高,为100%(56/56),其他基因未携带;氨基糖苷类抗生素中AAC(6')-Ie-APH(2")-Ia基因携带率最高,为33.93%(19/56);ANT(6)-Ib基因携带率为21.43%(12/56),其他基因未携带;大环内酯类/林可酰胺类/链阳霉素类(MLS)抗生素中ermQ基因携带率最高,为51.79%(29/56),ermA、ermB、ermT基因携带率分别为1.79%(1/56),8.93%(5/56),1.79%(1/56),其他基因未携带;林可酰胺抗生素中lnuP基因携带率为26.78%(15/56),其他基因未携带;酚类抗生素中fexA基因携带率为1.79%(1/56),其他基因未携带;恶唑烷酮类抗生素、苯酚类抗生素中optrA基因携带率1.79%(1/56),其他基因未携带;四环素类抗生素中tetA(P)、tetB(P)和tet44基因的携带率为98.21%(55/56)、87.5%(49/56)、21.43%(12/56),均存在较高的携带率。
3 讨论
产气荚膜梭菌是一种重要的食源性传播病原体,是食物中毒最常见原因之一[4]。在美国产气荚膜梭菌每年导致近100万例食物中毒。同时研究表明,产气荚膜梭菌存在于健康的养殖经济动物中,如鸡、牛、猪和羊等,并且可在零售市场的肉制品中分离出来[12-13]。因此,产气荚膜梭菌是1种全球公认的食源性传播的人畜共患病原体,食源性传播和动物源性传播是人类感染的主要来源。然而在中国对于不同宿主中的产气荚膜梭菌分子特征研究依旧相对缺乏,需要利用PFGE和MLST分析其遗传多样性和种群结构,以提高对该病原体的全面认识,为进一步的监测和调查研究提供前提。
在本研究的PFGE分析中,确定了两个水平的多样性:一方面,56株来自临床患者与经济养殖动物的样本共分为53个型别,这表明产气荚膜梭菌在不同的宿主中具有极强的遗传多样性;另一方面,17株健康绵羊中分离的产气荚膜梭菌共分为15种不同的PFGE型别(图1),37株临床抗生素相关性腹泻患者中分离的产气荚膜梭菌共分为36种不同的PFGE型别,这表明产气荚膜梭菌在同一宿主内也具有很强的基因组多样性。
从传播链的角度来看,PFGE结果显示,个别动物来源与人类来源菌株的亲缘关系范围较近,相似度高达90%,如18SD099和SDY013,18SD153和SDY022,17SD242和SDY012,推测这种病原体可能存在养殖动物和人类之间的传播。同一家医院中分离的菌株和同一家绵羊养殖场中分离的菌株分别出现了相同的PFGE分子型别,并且对应相同的MLST分子型别,推断在小范围区域内(AAD人群中和养殖绵羊中)可能存在菌株的克隆传播。PFGE结果并不支持不同宿主(人与动物来源)之间链条传播,但是鉴于时间间隔性和地域间隔性,日后可再进行一次大规模采集反映更加真实的情况。
产气荚膜梭菌是一种多重耐药病原体。近年来的国内外的研究表明在经济养殖动物中,多重耐药菌比例都极高[6,14]。青霉素已被农业农村部批准作为食品性动物生产的生长促进剂,并已用于控制革兰阳性菌,包括产气荚膜梭菌;甲硝唑虽不允许用于动物生产,但已明确收入国标31650中允许动物治疗使用,这些饲养过程抗生素的添加可能是菌株耐药率升高的原因。然而,在本研究中仅发现1株动物来源多重耐药菌,这可能与本研究所选抗生素多偏向临床用药相关,也可能是由于不同地区养殖场抗生素使用情况不同。对于临床分离株而言,抗生素的使用情况不同是不同地区产气荚膜杆菌耐药率具有较大差异的主要驱动因素。国外的耐药情况较为乐观。例如,在匈牙利、斯洛文尼亚和台湾北部的一些研究表明,头孢西丁、美罗培南和甲硝唑成功应用于产气荚膜菌感染治疗,只有少数菌株对青霉素(2.6%)或克林霉素(3.8%)耐药。然而,在本研究中,发现在山东地区的人源性菌株对克林霉素有很高的耐药性(48.65%)。此外,最近来自意大利的数据显示,克林霉素的耐药性在梭菌属中超过20%[6-14]。事实上,克林霉素一直被认为是治疗产气荚膜梭菌感染的一线药物。因此,产气荚膜梭菌高克林霉素耐药研究结果表明克林霉素作为经验治疗应引起重视。
本研究中,四环素耐药基因(tet)和抗防御素基因(mprF)在产气荚膜梭菌中检出率极高,这与近年来的研究结果相一致[15]。四环素耐药基因是该病原体中唯一广泛传播的AMR特异性基因,自20世纪80年代以来,编码四环素外排蛋白的tetA(P)和tetB(P)是产气荚膜梭菌中最常携带的抗性基因[15]。mprF基因(参与抗人类防御素)在所有产气荚膜梭菌基因组中都是保守的,这与其在宿主肠道定植的能力有关。其同源物可以抑制氨基糖苷类临床抗生素,如万古霉素和庆大霉素,然而本研究中菌株对于万古霉素完全敏感,这些菌株携带耐药基因,但无耐药表型,可作为耐药基因的“储存库”。另外值得关注的是,产气荚膜梭菌可从其所在的环境(如肠道)获得新的AMR基因,前抗生素时代的菌株基因组并没有编码tet[16],这揭示了产气荚膜梭菌在过去80年的进化史上的抗生素选择压力。
然而,本研究依旧存在一些不足之处,还需不断加大监测力度,补充菌株以扩大数据库,将PFGE分子型别、MLST分子型别和产气荚膜梭菌的耐药特征进行关联性分析,以期发现耐药菌株是否存在优势PFGE分子型别与MLST分子型别,进而预测菌株的耐药流行趋势并控制耐药菌的传播。
长期以来,由于产气荚膜梭菌属于条件致病菌,并未受到临床重视,然而近年来该菌耐药性逐步升高,严重时甚至导致院内的暴发感染,导致严峻的的公共卫生问题[17-18],医院应对产气荚膜梭菌的耐药菌监测和流行病学调查给予重视,本研究通过对山东地区的产气荚膜梭菌菌株进行PFGE与MLST分型并结合耐药表型和耐药基因分析以期對临床和动物养殖中在预防和治疗该类细菌感染时有所帮助。
參 考 文 献
Kiu R, Hall L J. An update on the human and animal enteric pathogen Clostridium perfringens[J]. Emerg Microb Infect, 2018, 7(1): 1-15.
Rood J I, Adams V, Lacey J, et al. Expansion of the Clostridium perfringens toxin-based typing scheme[J]. Anaerobe, 2018, 53: 5-10.
Carman R J. Clostridium perfringens in spontaneous and antibiotic-associated diarrhoea of man and other animals[J]. Rev Med Microbiol, 1997, 8: S46.
Motamedi H, Fathollahi M, Abiri R, et al. A worldwide systematic review and meta-analysis of bacteria related to antibiotic-associated diarrhea in hospitalized patients[J]. PLoS One, 2021, 16(12): e260667.
Azimirad M, Gholami F, Yadegar A, et al. Prevalence and characterization of Clostridium perfringens toxinotypes among patients with antibiotic-associated diarrhea in Iran[J]. Scient Rep, 2019. doi: 10.1038/s41598-019-44281-5.
Bendary M M, Abd El-Hamid M I, El-Tarabili R M, et al. Clostridium perfringens associated with foodborne infections of animal origins: Insights into prevalence, antimicrobial resistance, toxin genes profiles, and toxinotypes[J]. Biology, 2022, 11(4): 551.
Lopez-Canovas L, Martinez Benitez M B, Herrera Isidron J A, et al. Pulsed field gel electrophoresis: Past, present, and future[J]. Analyt Biochem, 2019, 573: 17-29.
Deguchi A, Miyamoto K, Kuwahara T, et al. Genetic characterization of type A enterotoxigenic Clostridium perfringens strains[J]. PLoS One, 2009, 4(5): e5598.
Xiao Y, Wagendorp A, Moezelaar R, et al. A wide variety of Clostridium perfringens type A food-borne isolates that carry a chromosomal cpe gene belong to one multilocus sequence typing cluster[J]. Appl Environm Microbiol, 2012, 78(19): 7060-7068.
M100-S11, Performance standards for antimicrobial susceptibility testing[J]. Clin Microbiol Newsl, 2001, 23(6): 49.
Verma A K, Abdel-Glil M Y, Madesh A, et al. Multilocus sequence typing of Clostridium perfringens strains from neonatal calves, dairy workers and associated environment in India[J]. Anaerobe, 2020, 63: 102212.
Lin Y T, Labbe R. Enterotoxigenicity and genetic relatedness of Clostridium perfringens isolates from retail foods in the United States[J]. Appl Environ Microbiol, 2003, 69(3): 1642.
Xiu L, Liu Y, Wu W, et al. Prevalence and multilocus sequence typing of Clostridium perfringens isolated from 4 duck farms in Shandong province, China[J]. Poultry Sci, 2020, 99(10): 5105-5117.
Sarvari K P, Schoblocher D. The antibiotic susceptibility pattern of gas gangrene-forming Clostridium spp. clinical isolates from south-eastern hungary[J]. Infect Dis (Lond), 2020, 52(3): 196-201.
Kiu R, Caim S, Alexander S, et al. Probing genomic aspects of the multi-host pathogen Clostridium perfringens reveals significant pangenome diversity, and a diverse array of virulence factors[J]. Front Microbiol, 2017, 8: 2485.
Lyras D, Rood J I. Genetic organization and distribution of tetracycline resistance determinants in Clostridium perfringens[J]. Antimicrob Agents Chemother, 1996, 40(11): 2500-2504.
Maslanka S E, Kerr J G, Williams G, et al. Molecular subtyping of Clostridium perfringens by pulsed-field gel electrophoresis to facilitate food-borne-disease outbreak investigations[J]. J Clin Microbiol, 1999, 37(7): 2209-2214.
Brzychczy-W?och M, Bulanda M. Analysis of genetic similarities between Clostridium perfringens isolates isolated from patients with gas gangrene and from hospital environment conducted with the use of the PFGE method. [J]. Polski Przegl Chirurg, 2014, 86(3): 141-146.
收稿日期:2022-11-16
基金項目:传染病预防控制国家重点实验室自主研究课题(No. 2021SKLID207)
作者简介:钟佳鑫,女,生于1997年,在读硕士研究生,主要研究方向为产气荚膜梭菌分子流行病学,疾病预防与控制,E-mail:
zhongjiaxin97@163.com
*通信作者,E-mail: lujinxing@icdc.cn
