番茄短节间基因BRACHYTIC(BR)的精细定位及其候选基因特异性分子标记的开发
2023-11-28杨沛宁宇魏凯刘晓林杨孟霞李珊珊王孝宣国艳梅刘磊李鑫杜永臣李君明黄泽军
杨沛 宁宇 魏凯 刘晓林 杨孟霞 李珊珊 王孝宣 国艳梅 刘磊 李鑫 杜永臣 李君明 黄泽军
(中国农业科学院蔬菜花卉研究所,蔬菜生物育种全国重点实验室,北京 100081)
株高是理想株型的重要指标之一。20 世纪60 年代的“绿色革命”正是源于水稻和小麦矮秆及半矮秆品种的育成和大面积推广(Sakamoto &Matsuoka,2004;Salas et al.,2009)。番茄(Solanum lycopersicum)是中国乃至世界上最重要的蔬菜作物之一,按照用途可以分为加工番茄和鲜食番茄两大类。加工番茄目前已经基本实现机械化栽培和采收。随着用工费用增加、劳动力日渐匮乏,种植者也越来越倾向于选择能够实现轻简化栽培的鲜食番茄品种(杨斌,2022)。因此,培育株型紧凑、适合轻简化栽培的番茄新品种已成为趋势。
番茄株高主要决定于主茎的节间数量和节间长度。现已发现了多个与番茄株高相关的突变体,如:影响节间数量的突变体有self-pruning(sp)(MacArthur,1932)、semideterminate(sdt)(Elkind et al.,1991)和suppressorofsp(ssp)(Park et al.,2014)。其中SP基因是CETS 基因家族成员之一(McGarry & Ayre,2012),编码1 个开花抑制因子。sp突变体中的SP-interactingG-BOX(SPGB)基因突变会造成ssp表型。影响节间长度的突变体有brachytic(br)(MacArthur,1931)、dwarf(d)(Bishop et al.,1996)、ElongatedInternode(EI)(Sun et al.,2019)、gibberellindeficient-1(gib-1)、gib-2、gib-3(Koornneef et al.,1990)、procera(pro)(Jupe et al.,1988)、shortinternode(si)(Kwon et al.,2020)和tomatointernodeelongated-1(tie-1)(Schrager-Lavelle et al.,2019)等。其中D基因编码1 个参与油菜素内酯合成的P450 蛋白(Bishop et al.,1996;Bishop et al.,1999;Marti et al.,2006)。短节间ShortInternode(SI)基因与拟南芥ERECTA(ER)基因同源,编码一个受体激酶(Kwon et al.,2020)。而EI、tie-1和pro等突变体的节间伸长、植株变高,与正常植株喷施外源赤霉素后的表型类似。EI与tie-1这两个突变体是由于SlGA2ox7基因的功能缺失导致活性赤霉素转化为非活性赤霉素的过程受阻、活性赤霉素积累的结果(Schrager-Lavelle et al.,2019;Sun et al.,2019)。而PRO基因则编码1 个赤霉素信号转导负调控因子SlDELLA 蛋白(Bassel et al.,2008)。还有多位研究者挖掘到了多个控制番茄株高的遗传位点并克隆了几个主效基因(Zhou et al.,2016;Sun et al.,2019;Liu et al.,2020)。
番茄br突变体已发现多年,将br位点转育到株高正常的番茄中可以降低株高(MacArthur,1931;Barton et al.,1955;Balint-Kurti et al.,1995)。经典遗传学研究表明番茄短节间br位点位于1 号染色体(MacArthur,1931;Balint-Kurti et al.,1995)。Lee 和Hutton(2018)将br位点定位到分子标记brM4 和brM7 之间约763 kb 的区段内。根据番茄基因组基因注释ITAG3.2 版本,该区段编码55 个基因。本试验将br位点定位到14.9 kb的区间内,该区间编码2 个基因,其中1 个基因的大部分序列缺失,根据该序列差异开发了1 个共显性PCR 分子标记BRD,试验结果将有助于番茄株型分子改良。
1 材料与方法
1.1 试验材料及表型调查
番茄参考基因组测序材料加工番茄Heinz1706和樱桃番茄VFNT Cherry(syn.LA1221,含br突变位点)的种子来自加利福尼亚州戴维斯番茄遗传资源中心(Tomato Genetics Resource Center,Davis,CA,USA)。由Heinz1706 和VFNT Cherry 构建的F2群体(152 株)于2017 年春季定植于中国农业科学院北京市顺义区杨镇试验基地大棚内,后代鉴定群体也定植于该基地大棚内。
植株采用单干整枝方式,每株保留5~6 穗果。当番茄第4 穗果实成熟后,使用卷尺测量地面至第4 穗果的高度作为株高;测量第1 穗果下面第1 片叶着生节位到第4 穗果下面第1 片叶着生节位之间的长度,除以之间的节位数量,即为节间长度。
1.2 序列差异分析与分子标记开发
VFNT Cherry 的重测序深度为30×(魏凯 等,2020)。利用BWA 软件(Li,2013)将测序得到的Clean Data 与参考基因组Heinz1706 进行比对,使用Samtools(Li et al.,2009)进行排序和索引,使用GATK(McKenna et al.,2010)对基因组进行变异分析。根据两亲本的序列差异,选取适当的InDel(Insertion/Deletion)和SNP,利用LaserGene(https://www.dnastar.com)设计引物,开发了InDel 和CAPS 标记。利用上述标记进行基因型分析。主要分子标记信息见表1。分子标记PCR 扩增、酶切和电泳检测等方法参照魏凯等(2020)。分子标记的扩增体系为10 μL:DNA 模板1 μL(100 ng · μL-1)、正向及反向引物各0.2 μL(10 μmol ·L-1)、2×Taqmaster Mix 5 μL(MF002-01,北京聚合美生物科技有限公司)、双蒸水3.6 μL。PCR 扩增程序设置为:94 ℃预变性4 min;94 ℃变性30 s,52 ℃退火 30 s,72 ℃延伸30 s,35 个循环;72 ℃终延伸5 min。InDel 标记使用3%的琼脂糖凝胶电泳检测PCR 产物,CAPS 标记 PCR 产物先酶切然后再使用3%的琼脂糖凝胶电泳进行检测。15 μL酶切体系:PCR 产物10 μL、内切酶0.2 μL(10 000 U · mL-1,New England Biolabs,MA,USA)、10×缓冲液1.5 μL、双蒸水3.3 μL,在最适反应温度下酶切2 h。

表1 试验所用分子标记的信息
1.3 BR 基因的精细定位
挑选F2群体中94 株无限生长型的植株进行表型调查和基因型分析,确定与株高连锁的分子标记并筛选交换单株。交换单株后代鉴定按下列方法进行:每株交换单株的自交种子播种于3 个32 孔穴盘中,常规日照及水肥管理。在幼苗期,取植株幼嫩叶片于96孔取样盒中,用CTAB法提取DNA后,用位于杂合区段的1~3 个分子标记进行基因型分析。分别选择基因型为1(与Heinz1706 基因型相同)的植株12 株,基因型为3(与VFNT Cherry基因型相同)的植株12 株,定植于中国农业科学院北京市顺义区杨镇试验基地大棚内。同时,从这些幼苗中挑选交换单株和杂合植株(如果没筛选到交换植株则使用杂合植株代替)定植,自交扩繁种子,用于后续的交换单株筛选或后代鉴定。第4 穗果实成熟后,调查后代鉴定群体植株的株高和节间长度。采用Student’st-test 进行数据统计。
1.4 FPF1 蛋白多重序列比对与进化树分析
拟 南 芥AtFPF1(Y11988) 和AtFLP1(AL353995)、 白芥SaFPF1(Y11987)、 烟草NtFPF1(AY496934)、玉米ZmFPF1(ACG44143)、水 稻OsRAA1(AY659938) 和OsFPFL4(AK120187)、棉花GhFPF1(AHI62966)等FPF1蛋白序列从NCBI(National Center for Biotechnology Information)数据库下载。以拟南芥AtFPF1 蛋白序列为查询序列,采用BLASTP(e-value < 1 e-10)检索番茄全基因组蛋白序列(ITAG 4.0 版本),获得番茄FPF1 蛋白同源序列。利用ClustalX2 进行蛋白多序列比对,利用MEGA6 软件采用邻接法(NJ,Neighbor-joining)构建FPF1 蛋白的进化树。
2 结果与分析
2.1 表型分析
以VFNT Cherry(含br突变位点,无限生长型)为轮回亲本,Heinz1706(有限生长型)为供体,利用BR基因定位区间内的分子标记辅助选择,回交5 代后自交2 代,构建了BR基因近等基因系NIL-WT 和NIL-br,两者的株高(4 穗果高度)分别为58.6 cm 和45.9 cm,节间长度分别为4.3 cm和3.7 cm,差异显著(图1)。

图1 近等基因系NIL-WT 和NIL-br 植株
2.2 BR 基因的精细定位
利用番茄1 号染色体上的分子标记对F2群体中的94 株无限生长型植株进行基因分析,结合其表型数据,经过单因素方差分析,发现分子标记HP1797 与控制株高性状br位点连锁较为紧密。挑选11 株交换位点位于分子标记HP1797 两侧的交换单株,收集自交种子。2018 年春季,对上述11株交换单株进行后代鉴定,将br位点定位到分子标记HP1737 和HP1797 之间。随后经过2018 年秋季、2019 年春季、2019 年秋季和2020 年春季4 轮的交换单株后代鉴定,最终将br位点定位到分子标记HP5589 和ZP273 之间、物理距离约为14.9 kb的区段内(图2)。

图2 br 的精细定位
2.3 候选基因分析
根据番茄参考基因组基因注释ITAG 4.0版本,br位点精细定位区间编码2 个基因:Solyc01g066970和Solyc01g066980(图2)。功能注释显示这两个基因均为开花促进因子(Flowering PromotingFactor1,FPF1)的同源基因(表2)。多重序列比对和进化树分析表明,Solyc01g066970和Solyc01g066980与已经报道的开花促进因子FPF1及其同源基因,拟南芥AtFPF1和AtFLP1、白芥SaFPF1、烟草NtFPF1、棉花GhFPF1、水稻OsRAA1和OsFPFL4、玉米ZmFPF1为同源基因(图3)。结合第2 代重测序分析,发现VFNT Cherry 的Solyc01g066980基因大部分序列缺失(图4)。PCR产物测序证明VFNT Cherry 中缺失了1 个607 bp的片段,具体物理位置为SL4.0ch01:68064387-68064993(图5)。故推测Solyc01g066980基因为BR的候选基因。
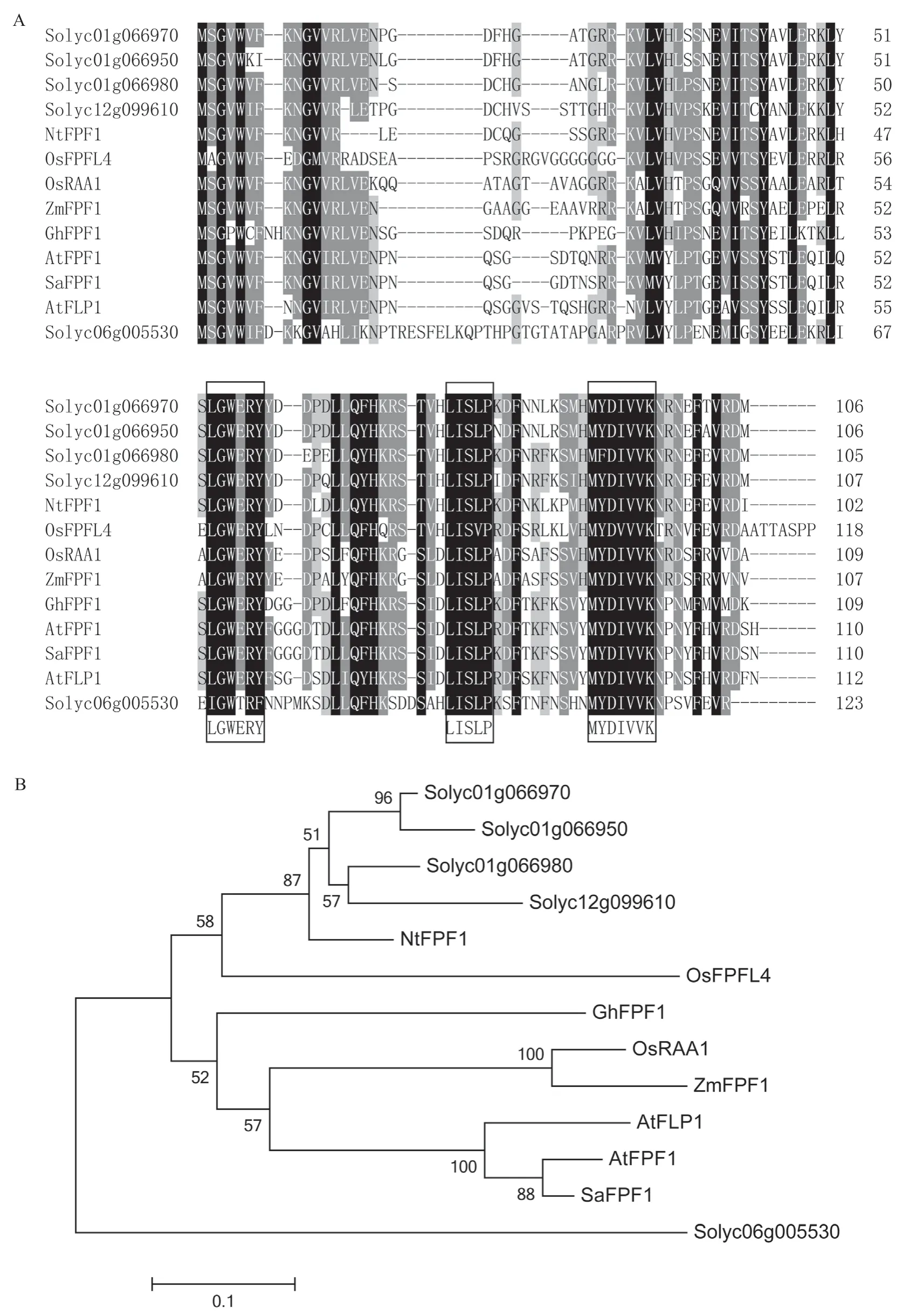
图3 FPF1 蛋白多重序列比对(A)及进化树分析(B)
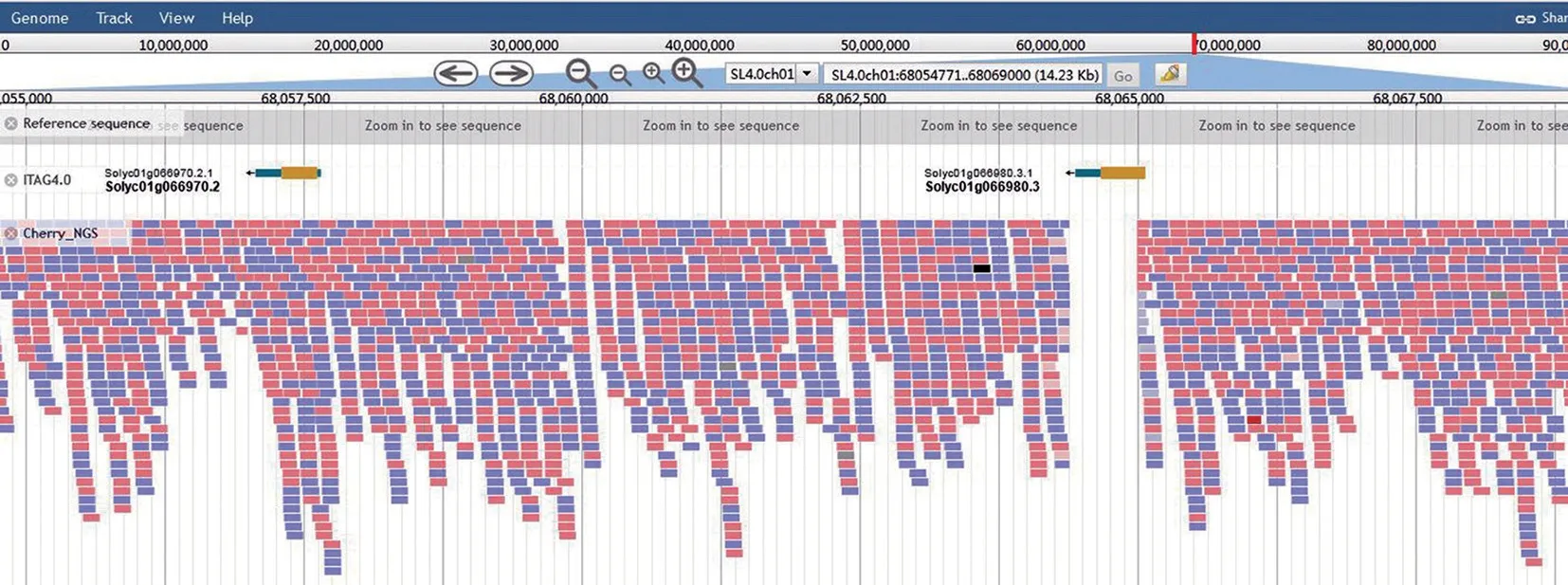
图4 br 精细定位区间的序列多态性分析

图5 BR 候选基因Solyc01g066980 的序列变异(A)及分子标记BRD PCR 产物的电泳检测结果(B)

表2 br 候选基因及其注释信息
2.4 候选基因特异性分子标记开发及应用
基于VFNT Cherry 中Solyc01g066980基因的缺失序列(图5),设计了3 条引物(表1,图5),开发了1 个共显性PCR 分子标记BRD。PCR 产物的琼脂糖凝胶电泳检测显示,含有短节间(br)纯合突变位点的VFNT Cherry 的电泳条带长度为416 bp,br位点为正常纯合(野生型)的Heinz1706 的电泳条带长度为296 bp。VFNT Cherry和Heinz1706 组配的分离群体中,呈现出3 种基因型,其中杂合单株的PCR 产物同时含有上述2 条条带(图5)。番茄遗传育种课题组以VFNT Cherry为供体、大果型番茄高代育种材料ZS768 为轮回亲本,利用BRD 进行分子标记辅助回交6 次、自交2 次后,得到转育了br位点的高代育种材料ZS768br。与ZS768 相比,ZS768br 株高(4 穗果高度)约降低13%。
3 讨论
株高是一个重要的农艺性状,对种植密度、水肥管理、抗倒伏能力、机械化采收等都有影响,因此理想株高成为了重要的育种目标(宋蒙飞 等,2022;马彩娥 等,2023;郭江怡 等,2023)。番茄株高主要由节间数量和节间长度决定。突变体sp、sdt和ssp主要影响节间数量(MacArthur,1932;Elkind et al.,1991;Park et al.,2014),突变体br、d、EI、gib-1、gib-2、gib-3、pro、si和tie-1则影响节间长度(MacArthur,1931;Jupe et al.,1988;Koornneef et al.,1990;Bishop et al.,1996;Sun et al.,2019;Schrager-Lavelle et al.,2019;Kwon et al.,2020)。 且qtph1.1、EI、h4t2a、h4t3a和h4t7a等控制株高的QTL 位点也逐渐被发现(周慧等,2015;Sun et al.,2019;Liu et al.,2020)。然而,可用于番茄株高改良的分子标记报道较少。
番茄br突变体已发现多年,其可以降低株高,且对单果质量没有影响,已经用于番茄育种(MacArthur,1931;Barton et al.,1955;Balint-Kurti et al.,1995)。然而,br是隐性突变体,通过表型进行选择,育种效率较低。本试验将br位点定位到14.9 kb 的区间内,根据番茄参考基因组基因注释ITAG 4.0 版本,该区间编码2 个基因:Solyc01g066970和Solyc01g066980。其中含br突变位点的VFNT Cherry 中Solyc01g066980基因大部分序列缺失。Lee 等(2022)对Solyc01g066980进行基因编辑后,可以降低番茄株高。因此,可以确定Solyc01g066980为BR的候选基因。
根据ITAG 4.0 的注释信息,Solyc01g066980为开花促进因子(FloweringPromotingFactor1,FPF1)的同源基因。FPF1基因家族成员参与开花调控和株高调控(韩琳和王佳伟,2018)。多重序列比对和进化树分析表明,Solyc01g066980与已经报道的开花促进因子FPF1及其同源基因,拟南芥AtFPF1和AtFLP1(Kania et al.,1997)、 白芥SaFPF1(Kania et al.,1997)、烟草NtFPF1(Smykal et al.,2004)、棉花GhFPF1(Wang et al.,2014)、水稻OsRAA1和OsFPFL4(Ge et al.,2004;Guo et al.,2020)、 玉 米ZmFPF1(Su et al.,2018)为同源基因。FPF1基因家族成员SaFPF1、AtFPF1、GhFPF1和OsRAA1在拟南芥中过量表达,均呈现出下胚轴伸长和早开花等表型。扫描电镜分析表明,AtFPF1在拟南芥中过量表达可使下胚轴细胞伸长(Kania et al.,1997)。进一步的分析表明,AtFPF1可能通过提高植株对赤霉素的敏感性而促进开花(Kania et al.,1997)。与此相似,NtFPF1在烟草中过量表达也可以促进开花并使植株节间伸长。经外源赤霉素处理,NtFPF1过量表达植株的早开花和节间伸长表型更加明显(Smykal et al.,2004)。因此,在拟南芥和烟草中,FPF1基因可能参与赤霉素信号途径,从而调节株高。Lee等(2022)分析br突变体在外源GA 处理下的转录组,推测番茄FPF1可能在抑制根部GA 的生物合成中起作用,但是具体分子机理有待进一步研究。
4 结论
番茄株型矮化紧凑有利于轻简化栽培。本试验将已经用于番茄矮化育种的短节间br位点定位到14.9 kb 的区间内,该区间编码2 个基因:Solyc01g066970和Solyc01g066980。其中含br突变位点的VFNT Cherry 中Solyc01g066980基因大部分序列缺失。推测Solyc01g066980为BR的候选基因。同时根据br位点的Solyc01g066980基因序列变异开发了1 个共显性PCR 分子标记BRD,该标记将有助于番茄株高的分子改良。
