不同稻蟹共作模式下寒区稻田土壤微生物的高通量测序研究
2023-11-16刘金雨张瑞罗亮王世会郭坤白庆利徐伟赵志刚
刘金雨,张瑞,罗亮,王世会,郭坤,白庆利,徐伟,赵志刚
(1.中国水产科学研究院黑龙江水产研究所,黑龙江省冷水性鱼类种质资源及增养殖重点开放实验室,黑龙江 哈尔滨 150070;2.上海海洋大学水产与生命学院,上海 201306)
稻渔综合种养是将水稻种植和水产养殖相结合的复合农业生产方式[1],利用水稻与水生生物之间的互利共生关系,提升种养系统物质循坏和能量流动的效率[2]。目前,稻渔综合种养已形成稻-鱼、稻-虾、稻-蟹、稻-鳖、稻-鳅等5 大类19 种典型模式[3],具有稳粮、促渔、提质、增效和可持续发展的特点[4,5]。全国稻蟹综合种养面积占稻渔综合种养总面积的4.97%,主要分布在北方地区[6]。黑龙江省作为我国水稻种植面积最大的省区之一,种植面积达400 多万公顷,发展稻渔综合种养潜力巨大,稻蟹综合种养也是目前黑龙江省发展稻渔综合种养的主要模式之一。
关于寒区稻蟹综合种养目前多集中于通过机体生长、水质理化因子、浮游生物和底栖生物等参数来评价种植、施肥和投喂等技术层面的研究[7-11],缺乏种养系统土壤微生物评价等的基础研究[12,13],尤其对寒区不同稻蟹共作模式下土壤微生物群落结构和多样性研究尚未见报道。稻田土壤系统中的微生物是土壤物质循环的重要组成部分,在稻田系统中发挥着至关重要的作用[14,15]。水产动物的摄食、排泄等活动通过食物网影响稻渔综合种养系统的物质循环和能量流动[16]。研究稻田土壤微生物群落组成和多样性有助于明晰稻渔综合种养的共作机制。细菌是微生物的主要组成部分,研究土壤细菌的群落结构和多样性可以在一定程度上掌握土壤微生物的结构特征。土壤中大部分细菌因较难分离培养,传统方法无法满足土壤微生物群落结构的研究需要[17,18]。Illumina MiSeq 测序平台高效准确的特点更适用于土壤微生物群落结构和多样性的研究[19,20]。本研究采用高通量测序技术比较分析了寒区稻-蟹共作(RC)、稻-蟹-鳅共作(RCL)和水稻单作(CK)三种模式下表层土壤细菌(16S rDNA)的多样性,探讨稻-蟹共作和稻-蟹-鳅共作模式对水稻表层土壤微生物群落结构和多样性的影响,为寒区稻蟹综合种养模式的建立提供理论依据。
1 材料与方法
1.1 实验设计
实验在黑龙江省绥化市国家级稻渔综合种养示范区进行。实验田采用:稻田单作模式(CK)、稻蟹共作模式(RC)和稻-蟹-鳅共作模式(RCL),分别设3 个重复,共9 个单元,每个单元面积为300 m2,栽培管理方式一致。各单元内,在距离田埂0.6 m 处挖口宽0.5 m,底宽0.3 m,深0.4 m 的环沟,沟田比为10%。在各单元的外围利用塑料布设高于地面0.4 m 的防逃墙。共作模式的水稻均采用大垄双行,边行加密的栽培模式,水稻单作组仍采用等间距的常规稻田种植方式,水稻品种为“绥粳4 号”。稻蟹模式放养河蟹(Eriocheir sinensis)体质量为(21.45±1.45)g,放养密度为500 ind./667 m2;稻-蟹-鳅模式,河蟹体质量为(21.45±1.45)g/ind.,放养密度为350 ind./667 m2,体质量为(0.47±0.07)g 泥鳅(Misgurnus anguillicaudatus)的放养密度为2 223 inds/667 m2。河蟹和泥鳅的放养时间分别为2020 年6 月25日和7 月5 日。养殖期间投喂配合饲料,每天投喂一次,投喂量为河蟹体体质量的2%~3%。河蟹和泥鳅于2020 年9 月10 日收获,水稻于9 月20 日收割。施有机肥为基肥,化肥为提苗肥,实验期间均不使用农药。
1.2 土壤样品采集和处理
2020 年9 月20 日分别在各单元的稻田中随机选5 个点采集表层土壤样品,混合为1 个样品。样品编号分别为CK_1、CK_2、CK_3、RC_1、RC_2、RC_3、RCL_1、RCL_2 和RCL_3。鲜土去除杂质混匀装入50 mL 离心管后于-80 ℃冷冻保存,用于微生物多样性分析。
1.3 土壤微生物DNA 提取与检测
用试剂盒提取微生物基因组DNA,NanoDrop 2000 紫外-可见分光光度计检测DNA 纯度和浓度。基因组DNA 抽提后,用1%琼脂糖凝胶电泳检测DNA 完整性,PCR 预扩增检测样品。
1.4 PCR 扩增及高通量测序
采用通用引物338F(5'ACTCCTACGGGAGGGA GCA-3')[21]和806R(5'-GGACTCHV GGGTWTCTAAT-3')[22]扩增细菌16S rRNA 基因V3-V4 区,PCR 采 用TransGen AP221-02:TransStart Fastpfu DNA Pliymerase,PCR 仪为ABI GeneAmp9700 型。PCR 反应使用以下程序进行:95 ℃预变性120 s,95℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,持续25个循环周期,最后72 ℃延伸5 min,每个样品3 个重复。使用AxyPrepDNA 凝胶回收试剂盒切胶回收PCR 产物,Tris_HCL 洗脱;利用2%琼脂糖凝胶电泳检测PCR 扩增效果,借助QuantiFluorTM-ST 蓝色荧光定量系统对PCR 产物进行检测定量。高通量测序采用Illumina 公司的Miseq 平台,土壤微生物总DNA 提取和测序服务委托上海美吉生物医药科技公司完成。
1.5 生物信息分析
利用QIIME1.9.1 对原始DNA 序列进行过滤处理,去除嵌合体,嵌合体由uchime 确定,得到优化序列[23];利用UPARSE 7.1 软件按照97%相似度将非重复序列进行聚类,得到OTU(Operational Taxonomic Unit)的代表序列,在此基础上进行分类学分析,统计各个分类水平上样品群落组成。
1.6 数据分析
实验结果采用平均值±标准误差(Mean±SE)表示,数据进行单因素方差(ANOVA)分析,并进行Duncan 氏多重比较,以P<0.05 作为差异显著性标志。用Venn 图统计多个样本中所共有和独有的物种数目。Alpha 多样性分析包括Chao 指数、ACE 指数、Shannon 指数和Coverage 指数分析。Beta 多样性分析通过未加权(Unweighted UniFrac)计算距离矩阵,根据得出的距离矩阵进行主坐标分析(PCoA)。通过土壤门和属水平的细菌菌群结构,分析各模式的微生物组成。
2 结果与分析
2.1 DNA 测序数据和OTUs
由表1 可知,RC、RCL 和CK 组的9 个样品中土壤细菌V3-V4 区测序获得的平均序列数分别为69 561.33、65 007.00 和65 872.00;平均碱基数分别为28 977 495.33、27 084 291.33 和27 493 837.00;序列平均长度分别为416.57 bp、416.61 bp 和417.35 bp。以97%相似度划分,共得到6 404 个OTUs,平均OTU 数分别为5 026、5 305 和4 969。用Coverage 反映测序深度,各组覆盖率超过97.0%(表1)。用稀释曲线来反映测序深度,稀释曲线达到饱和平台表明测序数据量合理,图1 表示测序深度足以描述微生物多样性。
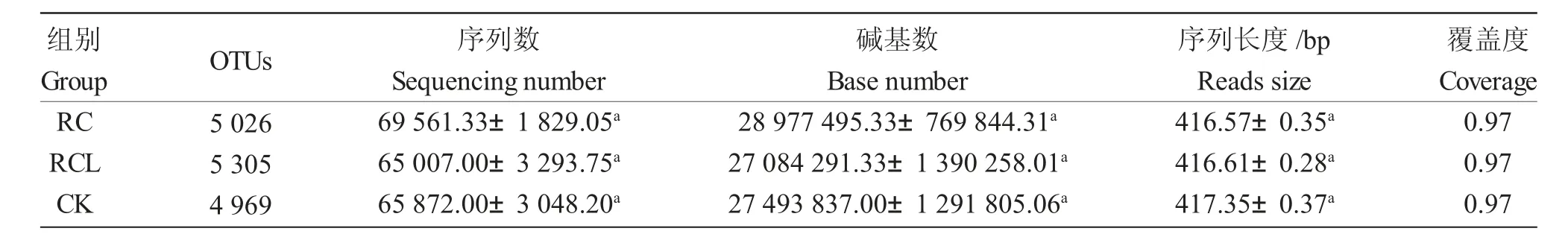
表1 不同模式下细菌高通量测序结果统计Tab.1 Illumina MiSeq sequencing results of the bacterial community in different modes
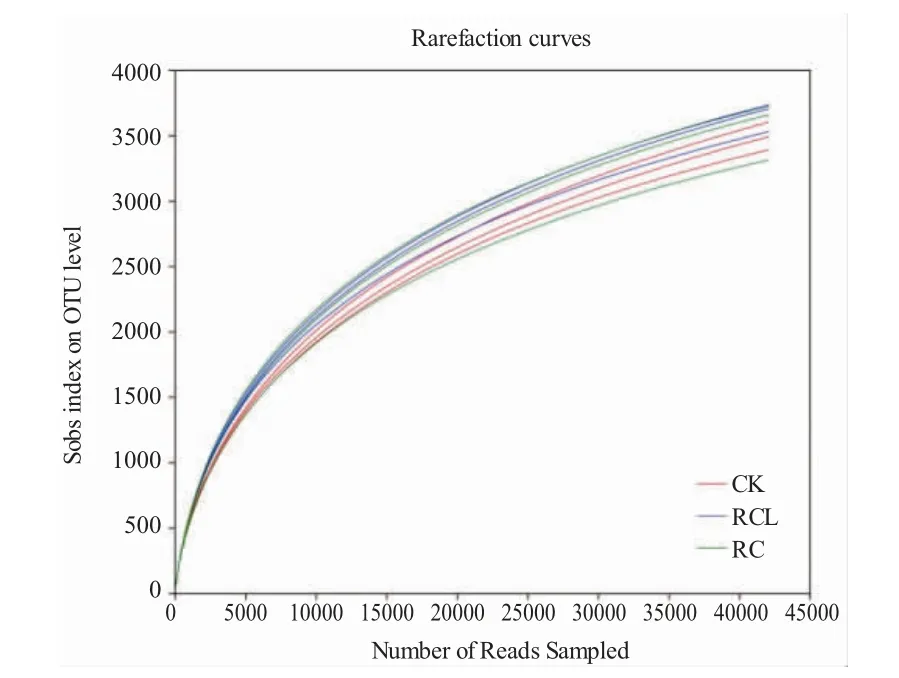
图1 三种模式稻田土壤微生物的稀释曲线Fig.1 Rarefaction curve analysis of microbial diversity in the paddy soils in three modes
Venn 图评估不同组OTU 的相似性和重叠情况见图2。图2 中数字表示所在区域的OTU 数目。结果显示:三种模式共有3 679 个OTUs,占总OTU 的57.45%,说明三种模式中的微生物种类较为相似。两两共享的OTU 中,RCL 和RC 共享数量最多,共有623 个,其次为RCL 和CK,共有550 个OTUs,最低的为CK 和RC,共有365 个OTUs。不同的OTU代表了不同的物种,特有的OTU 中,RCL 最高,为453 个,表示RCL 的特有物种最多;其次为CK,375个OTUs;RC 特有物种最少,为359 个OTUs。
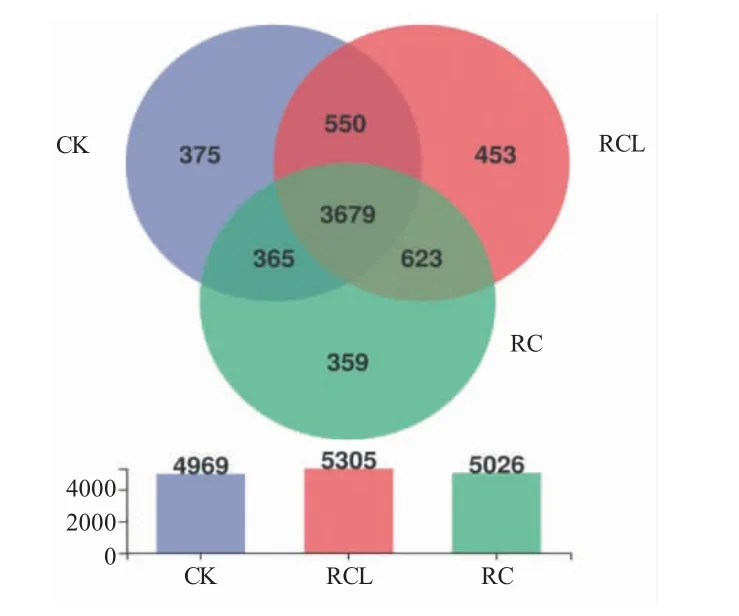
图2 三种模式稻田土壤微生物的韦恩图Fig.2 Venn diagram of microbial diversity in the paddy soils in three modes
2.2 多样性指数
α 多样性是指群落内部种的多样性,常用指标包 括ACE 指数、Chao 指数、Shannon 指数以及Simpson 指数等。ACE 指数用来估算群落物种丰富度,Chao 指数用来估计群落中含OTU 数目的指数,ACE 指数和Chao 指数越大说明细菌群落丰度越高。Shannon 指数和Simpson 指数用来反映群落物种多样性,Shannon 指数值越大或Simpson 指数越小,说明细菌群落多样性越高。与CK 相比,RC 和RCL 模式土壤细菌的ACE 指数增加了0.13%和1.58%,Chao 指数增加了0.88%和2.79%,Shannon指数增加了1.54%和2.71%(表2)。方差分析结果表明,2 种共作模式的稻田土壤细菌群落丰度和多样性均高于水稻单作,但差异并不显著(P>0.05)。
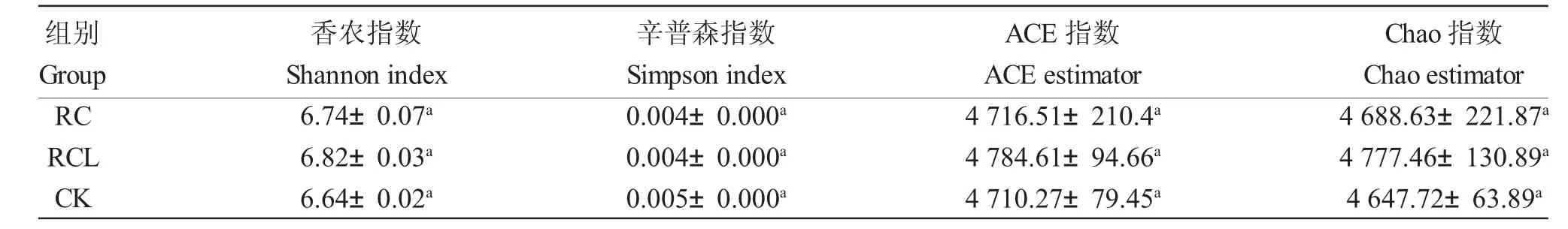
表2 不同模式下细菌群落多样性指数Tab.2 Bacterial diversity index of microbial diversity in the paddy soils in three modes
β 多样性表示的是微生物群落之间的差异。对三种模式下稻田土壤的OTU 进行PCoA 分析,第一主轴和第二主轴对细菌结构变异的贡献率分别为56.97%和12.60%(图3)。图中每个点表示一个样品,点之间的距离表示样本间微生物群落结构的相似程度,距离越近越相似。PCoA 图显示RC 和RCL距离较近,同时二者与CK 的距离较远,且出现显著性差异(R=0.4239,P=0.045)。
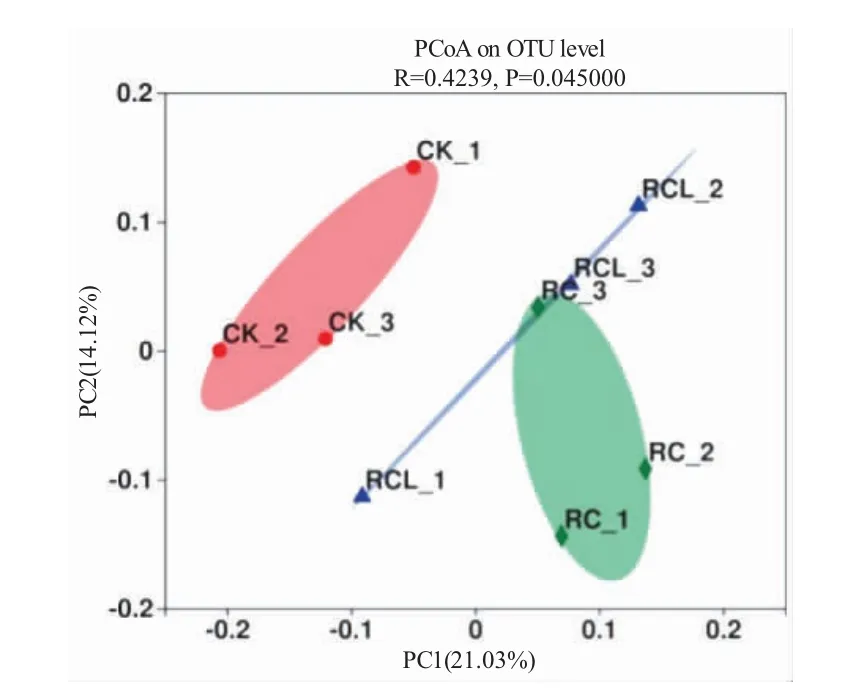
图3 不同模式微生物群落的主坐标分析(PCoA)Fig.3 Principal coordinate analysis(PCoA)of microbial diversity in the paddy soils in three modes
2.3 细菌门水平分类
本次测序中,得到的所有序列均为细菌序列。9个稻田土壤样品的6 404 个OTUs 分别属于59 个门、181 个纲、397 个目、1 093 个属、2 245 个种。图4为门水平上的细菌分类,平均丰度低于1%的物种被合并为图中的others。相对丰度大于5%的优势菌门均为相同的7 个菌门,分别为放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidota)、酸杆菌门(Acidobacteria)、厚壁菌门(Firmicutes)以及脱硫杆菌门(Desulfobacterota)。上述菌门在三种模式中丰度不同,但均未出现显著性差异(P>0.05)。CK 中相对丰度从高到低的菌门依次为:放线菌门(18.68%)、变形菌门(15.39%)、绿弯菌门(14.20%)、拟杆菌门(13.34%)、厚壁菌门(10.81%)、酸杆菌门(8.05%)以及脱硫杆菌门(7.68%)。RCL 相对丰度从高到低的菌门依次为:绿弯菌门(19.08%)、放线菌门(16.07%)、变形菌门(12.44%)、酸杆菌门(11.46%)、拟杆菌门(10.82%)、厚壁菌门(9.89%)和脱硫杆菌门(6.36%)。RC 相对丰度从高到低的菌门依次为:放线菌门(20.31%)、绿弯菌门(17.76%)、酸杆菌门(12.54%)、变形菌门(11.80%)、拟杆菌门(10.42%)、厚壁菌门(8.92%)和脱硫杆菌门(5.34%)。
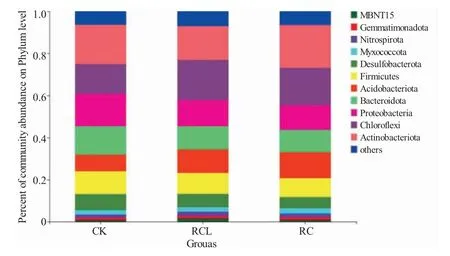
图4 三种模式稻田土壤微生物在门水平的细菌物种组成Fig.4 Bacterial community composition in the paddy soils in three modes at the phylum level
2.4 细菌属水平分类
如图5 所示,热图显示了三种模式在属水平上微生物群落的相对丰度的差异。平均丰度低于1%的物种被合并为others,其在CK、RC 和RCL 中分别占59.17%、55.74%和57.62%。相对丰度>1%的优势菌属有22 个,其中一些属没有科学名称,以norank 作为标记。CK 中相对丰度前五的优势菌属为稻田土壤菌属(Oryzihumus,4.32%)、拟杆菌属(norank_f_Bacteroidetes_vadinHA17,3.71%)、假节杆属(Pseudarthrobacter,3.85%)、(norank_f_norank_o_SJA-15,3.57 %)和(norank_f_Lentimicrobiaceae,2.84%)。RC 中相对丰度前五的菌属为稻田土壤菌属(4.86%)、假节杆属(4.08%)、(norank_f_ norank_o_norank_c_KD4-96,4.01%)、拟杆菌属(3.86%)以及(norank_f_norank_o_RBG-13-54-9,2.44%)。RCL中相对丰度前五的菌属分别为拟杆菌属(4.02%)、(norank_f_norank_o_norank_c_KD4-96,3.97%)、(no rank_f_norank_o_RBG-13-54-9,2.96%)、稻田土壤菌属(2.87%)以及假节杆属(2.81%)。
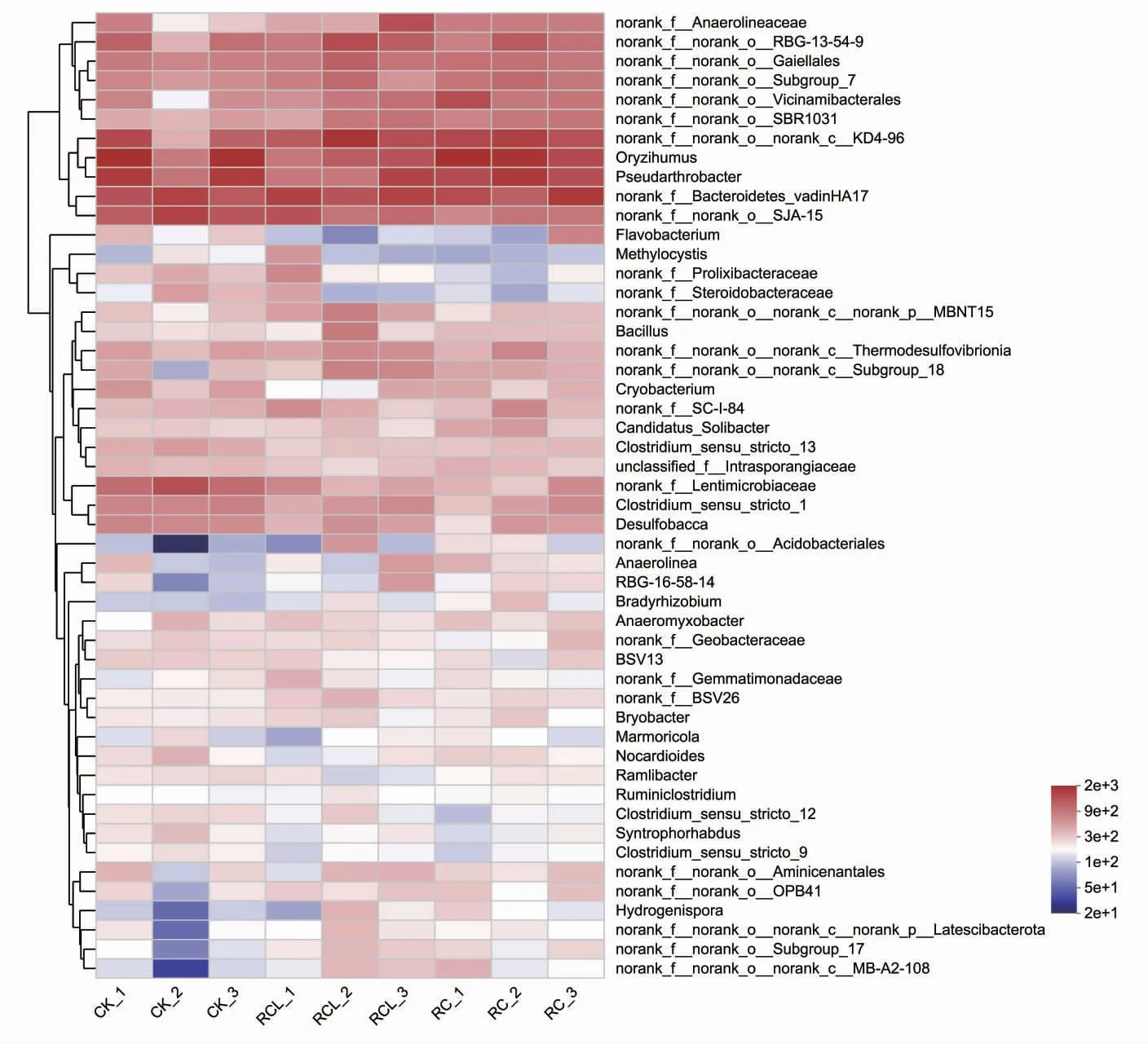
图5 三种模式稻田土壤微生物在属水平的丰度热图Fig.5 Heatmap of microbial diversity in the paddy soils in three modes at the phylum level
3 讨论
3.1 不同稻蟹共作模式土壤的细菌多样性
微生物多样性指数是评价微生物群落丰富度和多样性的综合指标,较高的生物多样性更能保证生态系统的稳定[24]。微生物的多样性变化可直接或间接影响土壤肥力[25]。在稻渔综合种养系统中,灌溉、施肥、栽培和共作方式均会影响土壤微生物[26,27],稻渔共作过程中养殖动物和饲料投喂水平也会造成土壤微生物多样性的变化[28,29]。研究表明,水产动物与水稻共作可提升稻田土壤微生物的群落结构和多样性[30-33]。本研究结果显示,在寒区进行RC 和RCL 后的稻田土壤微生物多样性指数均高于水稻单作,与上述结果一致,表明这两种寒区稻蟹共作模式均提升了稻田土壤细菌群落的多样性。同时,RCL 的Shannon 指数、ACE 指数和Chao 指数均高于RC,表明稻-蟹-鳅共作模式较稻-蟹模式更能提升稻田土壤微生物多样性,在一定程度上揭示了稻-蟹-鳅共作模式下,土壤生态系统相对更为稳定。宋宇等[33]研究结果中的PCoA 图显示,稻虾共作和稻蟹共作均与水稻单作的距离较远,而两种共作模式之间的距离较近,这与本实验PCoA 分析图中RC 和RCL 与CK 在不同区域聚类,RC 和RCL距离较近的结果相似。本实验土壤微生物不同区域聚类表明稻蟹共作模式与水稻单作模式的稻田土壤微生物群落结构差异较大,而两种稻蟹共作模式稻田土壤微生物的群落结构较为相近。稻蟹共作模式在种养过程中,其残饵、粪便可为土壤微生物提供营养;河蟹和泥鳅的活动也对土壤产生扰动,增加土壤的通透性,这些因素均可能显著影响稻蟹共作系统中稻田土壤微生物的组成和多样性。
3.2 不同稻蟹共作模式土壤的细菌群落结构组成
高通量测序技术可检测到更多丰度较低的微生物类群[34]。本研究中,3 种模式在97%的相似性下共获得6 404 个OTUs,检测到59 个门1 093 个属的微生物种群。在门的分类水平上,三种模式的优势菌门均为放线菌门、绿弯菌门、变形菌门、拟杆菌门、酸杆菌门、厚壁菌门及脱硫杆菌门。放线菌可加速土壤中动植物残体的腐烂分解[35]。宋宇等[36]研究认为,稻蟹共作土壤放线菌的丰度较水稻单作增加了1.36%。本研究中,稻蟹模式土壤放线菌的丰度增加了1.63%,土壤放线菌丰富度的增加有助于加速稻田土壤系统的物质循环进程。变形菌门大多为革兰氏阴性菌,其中大部分细菌均具有固氮能力[37]。成永旭等[38]研究表明,稻蟹共作模式土壤细菌相对丰度最高的菌门为变形菌门。罗衡等[39]对稻鳖共作模式土壤微生物的研究也认为,土壤微生物以变形菌门为主导。本研究在寒区稻蟹共作模式中也得到了相似的结果。稻鱼共作土壤系统中绿弯菌门丰度大幅上升,成为优势菌门[30]。本实验稻蟹共作模式也有效提升了土壤绿弯菌门的相对丰度,可能因共作模式稻田水淹程度较大,土壤中形成的微厌氧环境适宜该菌的生长。拟杆菌门、酸杆菌门、厚壁菌门及脱硫杆菌门均为稻田常见的细菌类群[40]。本研究在寒区两种稻蟹共作模式和水稻单作模式中也分别监测到了这些菌群。稻蟹共作模式中,宋宇等[33]检测的土壤细菌优势菌门与本实验相似,而在属分类水平上差异较大,前者优势属菌为蓝藻属(norank_c_Cyarbacteria)、硫曲菌属(Sulfuricurvum)和厌氧绳菌属(norank_f_Anaerolineareae),而本研究中的优势菌属分别为稻田土壤菌属、假节杆属、norank_f_norank_o_norank_c_KD4-96、拟杆菌属和norank_f_norank_o_RBG-13-54-9,主要原因可能是不同实验地点的气候环境差异所致。本研究在土壤主要优势菌属中,对比水稻单作,稻-蟹共作和稻-蟹-鳅共作土壤中的norank_f_norank_o_no-rank_c_KD4-96、拟杆菌属和norank_f_norank_o_RBG-13-54-9 的相对丰度均有所增加,可能因两种稻蟹共作模式中残饵、粪便提升了稻田土壤系统的肥力,在一定程度上为上述细菌类群提供了更为适宜的生长环境。
3.3 结论
本研究中,寒区“稻-蟹”和“稻-蟹-鳅”两种共作模式下稻田土壤细菌群落结构和多样性结果表明,与水稻单作相比,稻蟹共作模式和稻-蟹-鳅共作模式均能改变稻田土壤的细菌群落结构,增加稻田土壤细菌群落的丰富度和多样性。
